基于生物信息学分析肝癌发病的关键基因及通路
金 敏 王思月 王 淞 张雪梅 张 志
华北理工大学附属唐山工人医院肿瘤科 河北唐山 063000;①华北理工大学公共卫生学院;②华北理工大学附属唐山工人医院外科;③华北理工大学生命科学学院
肝细胞癌HCC(Hepatocellular Carcinoma)是世界范围内常见的恶性肿瘤,发病率呈上升趋势[1],因其高死亡率成为癌症相关死亡原因的第4位[2]。肝癌患者早期大多无明显不适,由于症状出现较晚,只有不到20%的患者可接受根治性切除或移植[3]。肝癌患者就诊时多为中晚期,治疗方法有限且复发率高[4],这些因素都导致肝癌患者生存期短,生活质量下降。肝癌是一种复杂的、异质性很强的恶性肿瘤,其发生发展机制尚未完全清楚,临床上仍然缺乏与患者预后相关的生物标志物[5-6]。因此,探寻肝细胞肝癌特异性的生物标记物和治疗靶点是急需解决的难题之一。
近年来, 高通量基因芯片和测序技术作为基因表达分析的工具, 已被广泛用于识别肿瘤发生过程中遗传信息的改变, GEO (Gene Expression Omnibus)数据库中有着丰富的肿瘤相关基因组和基因表达谱, 为研究肝细胞癌基因表达情况及发现关键基因的变化规律提供了基础[7]。本研究基于GEO数据库获取肝癌基因芯片的数据,对肝癌生存预后进行生物信息学分析,初步探索肝癌潜在的治疗靶点及信号通路,或能为肝癌的早期筛查、诊断和治疗提供新的思路。
1 材料与方法
1.1基因检索 在美国国立生物技术信息中心NCBI(https://www.ncbi.nlm.nih.gov/)的GEO (https://www.ncbi.nlm.nih.gov/geo/) 数据库中检索肝癌组织芯片数据,最终选用GSE12520与肝癌相关的mRNA芯片数据,该数据平台分别是GPL571 (Affymetrix Human Genome U133A 2.0 Array)和GPL3921(Affymetrix HT Human Genome U133A Array),包括205例肝细胞癌组织和283例正常肝组织的芯片数据。
1.2肝癌差异表达基因的筛选 用R语言软件读取下载矩阵文件,使用R语言“Affy”程序包对上述芯片的原始数据进行过滤和标准化处理,使用“limma”包[8]对两个平台数据中正常癌旁组织和肝癌组织进行分析,分别得到差异表达基因(differentially expressed genes,DEGs)。DEGs筛选标准:(|log2FC|>1,P<0.05)。并且将2个数据集中表达上调或下调的基因分别取交集,筛选出共同表达上调或下调的基因作为进一步分析的对象;通过Venn作图工具(http://bioinformatics.psb.ugent.be/webtools/venn/)挑选出两组表达谱数据中共有的差异表达基因。
1.3DEGs的功能和途径富集分析 通过DAVID(the Database for Annotation, Visualization and Integrated Discovery, https://david.ncifcrf.gov/) 数据库[9]对DEGs行基因本体论(Gene Ontology,GO) 和KEGG (Kyoto Encyclopedia of Genes and Genomes) 信号通路分析,得到DEGs的生物学过程分析结果和KEGG信号通路分析结果,P<0.05为差异有统计学意义。
1.4PPI网络中核心模块的选择和关键基因的识别 我们使用STRING(Search Tool for the Retrieval of Interacting Genes, http://www.string-db.org/)数据库评估蛋白-蛋白相互作用PPI(Protein Proteininteraction),以得分大于0.4作为标准。将进一步筛选得到的基因输入Cytoscape 3.7.2开源平台对PPI网络进行可视化分析。使用Cytoscape中的分子复合物检测算法MCODE(Molecular Complex Detection)在PPI网络中找到核心基因簇,标准为(degree cutoff=2,node score cutoff=0.2,k-core=2, max depth=100)[10]。我们使用cytoHubba插件(http://hub.iis.sinica.edu.tw/cytohubba/)采用度的算法鉴定关键基因,将度≥35的基因在网络模块中鉴定为关键基因。
1.5关键基因的验证 GEPIA(Gene Expression Profiling Interactive Analysis, http://gepia.cancer-pku.cn/)是一个交互式在线网站,可以分析来自TCGA和GTEx的9736个肿瘤和8587个正常样本的RNA测序表达数据。使用GEPIA在线工具分析HCC和正常肝组织中这些hub基因的mRNA表达水平。
KM-plotter(Kaplan Meier plotter, http://kmplot.com/analysis/)是一个在线工具,可以评估5万个基因对21种癌症类型生存率的影响。我们使用Kaplan-Meier mRNA肝癌数据库来评估HCC患者hub基因的预后分析。对于每个基因,根据mRNA表达高低的中位数将患者分为两组。采用对数秩检验来确定两组间差异的显著性,计算危险比(HR)来评估基因表达与生存之间的关系。
UALCAN(http://ualcan.path.uab.edu/analysis.html)提供了基于组学数据(TCGA和MET500)的分析。在本研究中,我们通过UALCAN的“表达分析”模块获得了hub基因的肿瘤分级和个体癌症分期的分析数据。
人类蛋白质图谱HAP(https://www.prote inatlas.org)是一个开放数据库,供工业和学术界的科学家自由获取人类蛋白质组研究的数据。使用HAP在线数据库分析具有明显预后价值的关键基因的免疫组化结果。
使用Metascape(http://metascape.org/)进行功能富集分析。P值是基于累积超几何分布计算的,而q值是使用Banjamini-Hochberg程序计算的。在对富集项进行层次聚类时,使用Kappa分数作为相似性度量,将相似度>0.3的子树视为一个聚类。选择最具有统计学意义的一个聚类项来表示该聚类。对于6个关键基因,使用以下数据库进行蛋白-蛋白相互作用富集分析:BioGrid[11]、InWeb_IM[12]、OmniPath[13]。采用分子复杂检测(MCODE)算法对密集连接的网络构件进行识别。对核心模块进行途径聚类富集分析,通过P值保留三个最好的评分项,筛选关键通路。
2 结果
2.1芯片数据 使用GEO数据库获取HCC相关表达数据集GSE14520,其中包括205例肝癌组织和283例正常肝组织数据。以(|log2FC|≥1,P<0.05)为筛选阈值,使用R语言进行分析。所得的火山结构如图1A和图1B所示。两组平台数据集GPL3921、GPL571差异分析后,共同上调的差异基因有96个,共同下调的差异基因有178个,见图2A、图2B。两组数据集取交集的差异基因总数为274个,见图2C。

图1 GSE14520 GPL571和GSE14520 GPL3921中DEGs的火山图
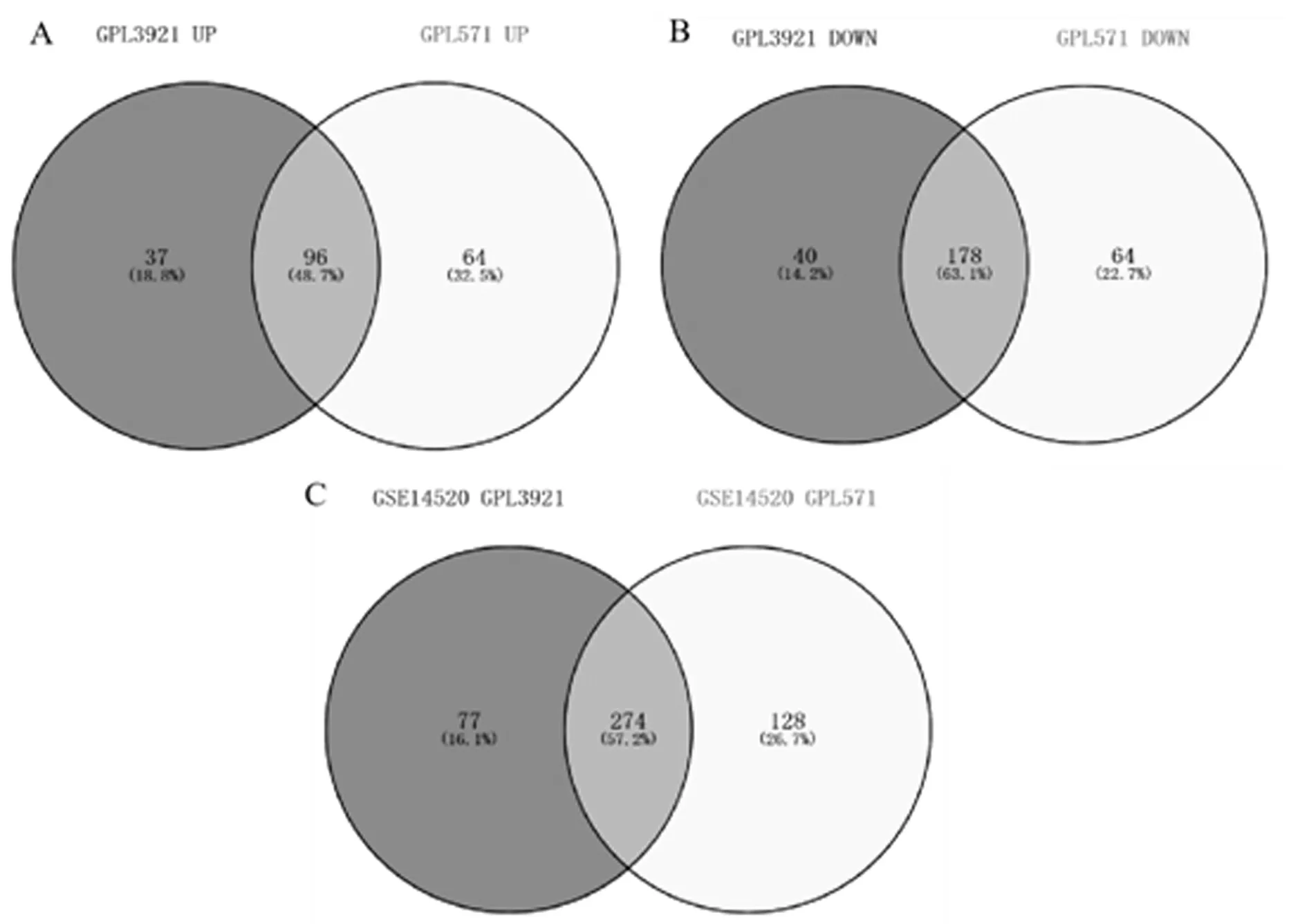
图2 GSE14520 GPL571和GSE14520 GPL3921中DEGs的维恩图
2.2DEGs的功能和途径 运用DAVID数据库分别对178个下调基因及96个上调基因进行GO分析和KEGG分析,见表1、表2。GO分析中,上调的DEGs主要参与DNA复制起始和有丝分裂细胞周期相变等生物学过程(biological process,BP)。细胞组成(cellular component,CC)表明这些基因大部分参与细胞质和核质。分子功能(Molecular Function,MF)主要集中于poly(A) RNA结合和ATP结合。下调的DEGs在生物过程BP中主要在氧化还原过程和甾体代谢过程中富集。细胞学成分CC分析显示大多在细胞外泌体和线粒体中富集。其分子功能(MF)主要包括氧化还原酶活性和蛋白质同聚活性。通过对DAVID数据库中下调基因的分析,发现KEGG通路主要富集于代谢通路和化学致癌通路。DAVID工具未能富集上调基因的相关信号通路。

表1 肝细胞癌差异表达基因的富集分析

表2 肝细胞癌差异表达基因的KEGG富集分析
2.3PPI网络构建和hub基因选择 通过Cytoscape软件构建这些基因的PPI网络,使用MCODE插件进行分析,根据评分水平高低总共得到6个具有代表性的模块。第一个核心模块得分27.286,包括29个节点和382个边,主要富集于细胞周期、卵母细胞减数分裂、DNA复制等信号通路,见图3A;核心模块2得分12.286,包括15个节点和86个边,主要富集于化学致癌途径、视黄醇代谢等途径,见图3B。使用CytoHubba插件进行分析,得到十个中心基因PPI网络图,见图3C。这10个关键基因分别是:CDK1、PCNA、CAT、CCNB2、 CDC20、AURKA、 BUB1B、RFC4、TOP2A 和NDC80。
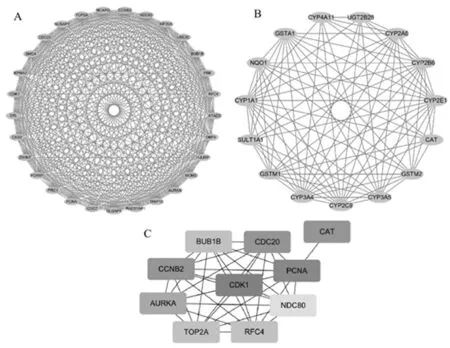
图3 利用Cytoscape软件的MCODE插件对DEGs中的核心功能模块和关键基因进行鉴定
2.4预后明显相关的六个枢纽的验证 使用Kaplan Meier-Plotter数据库来评估两个关键模块中10个hub基因的预后价值。结果显示,CDK1、CCNB2、CDC20、NDC80、BUB1B、TOP2A基因与预后有明显的相关性(P<0.01),见图4,且在肝癌组织中呈现显著高表达的状态,见图5。同时,利用Ualcan在线工具分析了这6个hub基因mRNA表达与肿瘤分级的关系,可见肿瘤的分级程度越大,基因的mRNA表达水平越高,见图6。还分析了关键基因mRNA表达和肝癌患者个体分期的关系,肿瘤患者分期越高,基因的mRNA表达水平越高,见图7。此外,使用HPA数据库来展示CDK1、CCNB2、CDC20、TOP2A蛋白水平的差异,免疫组化染色,见图8(数据库中缺少NDC80和BUB1B数据)。结果显示,CDK1、CCNB2、CDC20、TOP2A的免疫组化染色在正常组织中呈阴性,在HCC组织中呈阳性,证明了这些基因在HCC组织中明显高于正常肝组织。
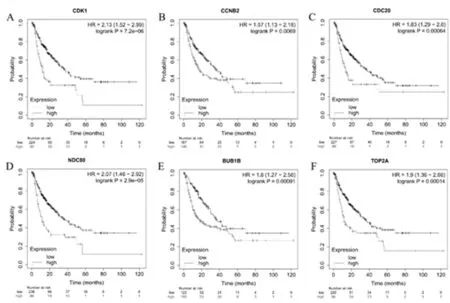
图4 基于Kaplan-Meier绘图仪数据库的6个hub基因生存分析
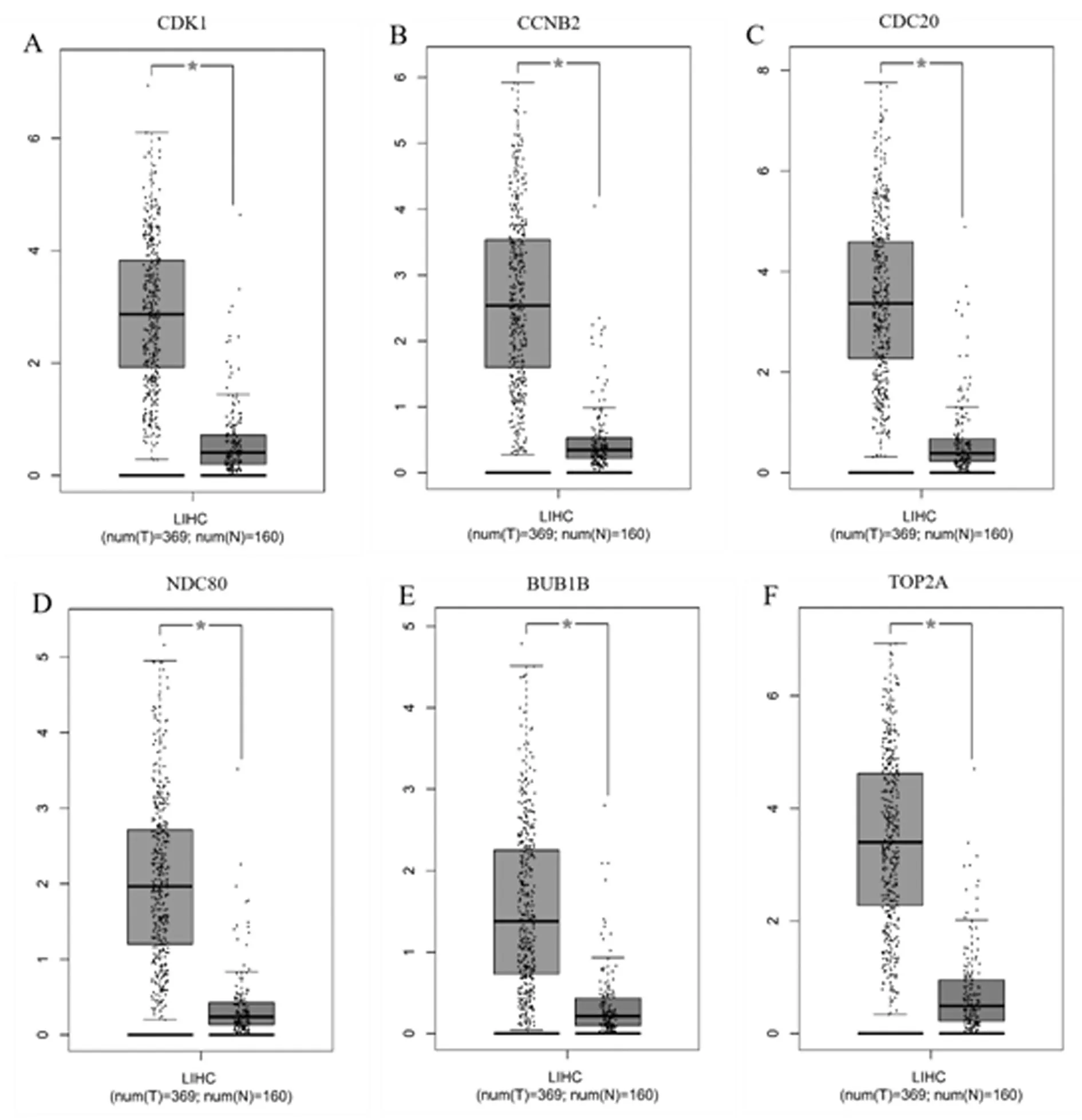
图5 与正常组织相比,6个关键基因在HCC组织中高表达且预后不良

图6 TCGA数据库中6个关键基因mRNA表达和HCC肿瘤分级的关系
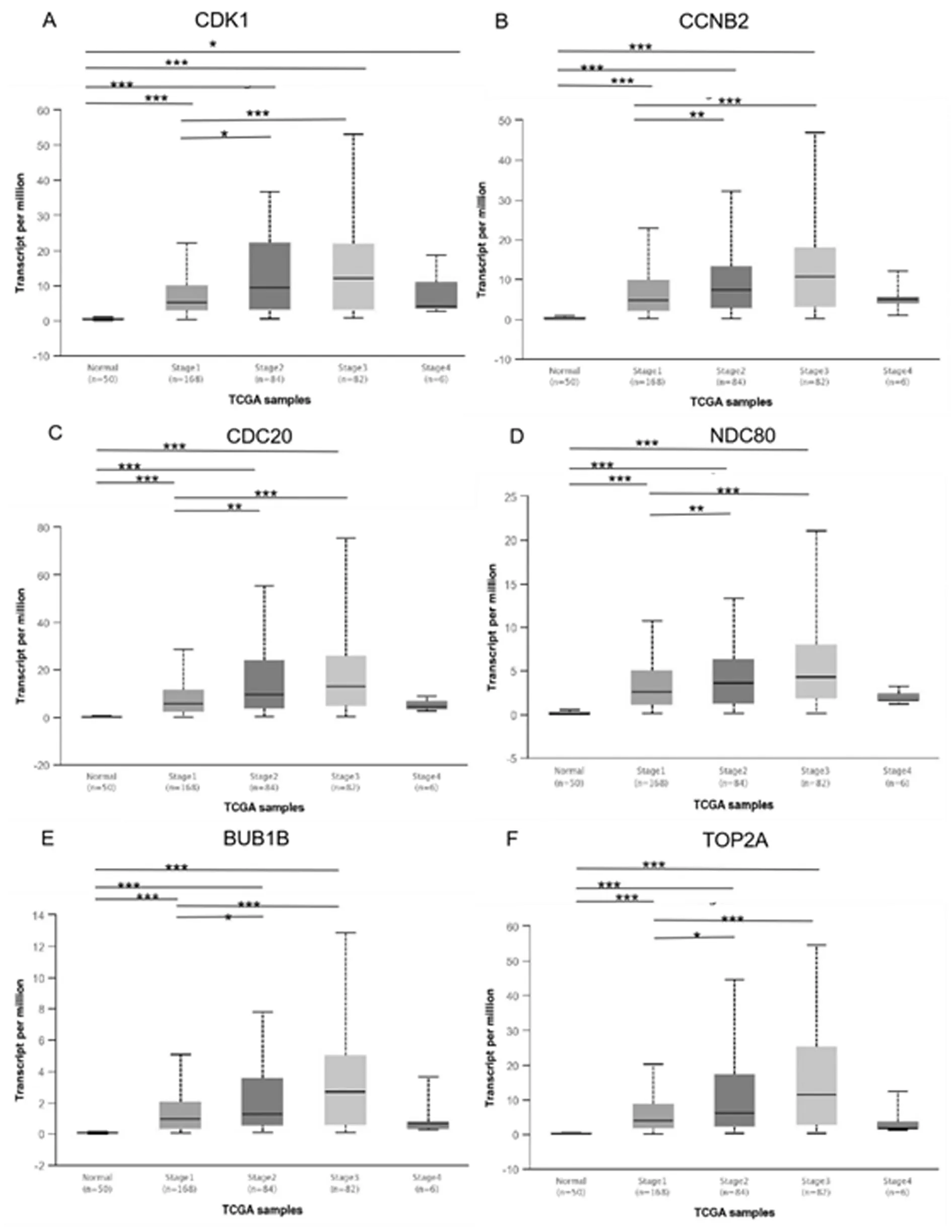
图7 TCGA数据库中6个关键基因mRNA表达和HCC分期的关系
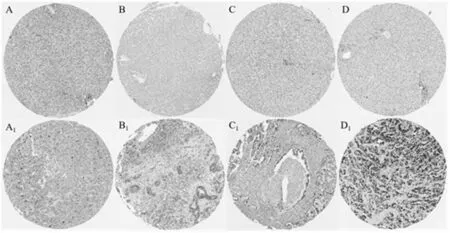
图8 基于Human Protein Altas数据库在翻译水平验证了关键基因的表达
使用GEPIA数据库分析了6个hub基因的相关性。hub基因之间的Spearman相关性分析,见图9。结果显示CDK1与其他基因呈正相关。CDK1: CDC20 (r=0.93,P=9.4e-226);CDK1: CCNB2 (r=0.96,P=1.8e-284);CDK1: NDC80 (r=0.96,P=5.2e-300);CDK1: BUB1B (r=0.94,P=1.4e-244);CDK1: TOP2A (r=0.95,P=2.8e-262)。CDK1、CCNB2、CDC20、NDC80、BUB1B、TOP2A各基因与其他基因呈正相关。
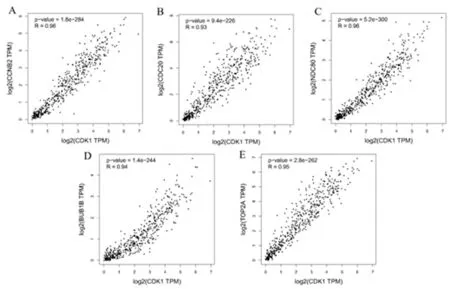
图9 GEPIA中6个关键基因的相关分析散点图
2.56个核心基因的功能和途径富集分析 使用Metascape对6个hub基因进行功能和途径富集分析。这些Metascape结果显示,与姐妹染色单体凝聚力、有丝分裂细胞周期检查点、有丝分裂细胞周期、细胞相关组件分解,见表3。该网络使用Cytoscape进行可视化,其中每个节点代表一个富集的项目,并通过其P值着色,见图10。

表3 具有代表性的前5个富集项

图10 关键基因的Metascape功能富集分析
2.6核心基因的蛋白质-蛋白质相互作用分析 Metascape使用以下数据库对核心基因进行蛋白-蛋白相互作用分析:BioGrid[11]、InWeb_IM[12]、OmniPath[13]。采用MCODE算法对密集连接的网络部件进行识别,见图11。保留P-value的3个最佳评分项作为对应组成部分的功能描述。结果显示,PID PLK1通路是关键信号通路,见表4。
图11 CDK1、CCNB2、CDC20、

表4 各MCODE组分独立富集的途径和过程分析
3 讨论
在近几年,很多肝癌的发生机制逐渐被揭示[14],大量的生物标记物已被用于肝癌的早期筛查和诊断[15]。像大多数肿瘤一样,肝癌的发生是一个复杂的生物学过程,是多个基因共同作用的结果,以往的研究多局限在单基因水平,多基因水平方面研究较少,而多基因水平研究有助于探索肝癌的发病机制。在本研究中,通过生物信息学方法分析,终于发现了高度相关的CDK1、CCNB2、CDC20、NDC80和BUB1B基因在肝癌组织的高表达与患者生存期差明显相关,可能通过PID PKL1通路参与肝癌的发生发展。存在相似的研究结果[16-17],差异在于这两项研究只阐述了差异的基因与肝癌的相关性,没有分析可能的分子通路机制。本研究利用生物信息学的方法旨在探索肝癌潜在的治疗靶点和可能的分子机制。
细胞周期蛋白依赖性激酶1(cyclin-dependent kinase 1,CDK1)是调控细胞周期有丝分裂过程的重要一员[18]。研究表明CDK1可作为非小细胞肺癌[19]、结肠癌[20]、乳腺癌[21]和卵巢癌[22]的临床预后生物标志物。Li 等[23]研究结果与本研究结果相似,CDK1基因敲除抑制PLK1基因等的表达,提示CDK1-PLK1/SGOL2/ANLN途径可能是肝癌发生发展的一个重要过程。细胞周期蛋白B2(CCNB2)与细胞的增殖密切相关,有研究表明CCNB2 mRNA在胃癌、非小细胞肺癌、肝细胞癌、前列腺癌、乳腺癌、上皮性卵巢癌和弥漫大B细胞淋巴瘤组织中表达均有上调,且与不良的预后相关[24-30]。细胞分裂周期20(CDC20)的癌组织中高表达与前列腺癌[31]、乳腺癌[32]和结肠癌[33]的不良预后有关。但是肝癌组织的表达情况与预后缺乏足够的实验数据。核分裂周期蛋白80基因(nuclear division cycle 80,NDC80)维持着染色体的稳定性,NDC80高表达的细胞可能出现纺锤体检查点功能障碍、染色体异常分离或细胞周期紊乱,这些都是肿瘤发生的原因之一[34]。研究表明,NDC80在肝癌、骨肉瘤等诸多肿瘤组织中存在过表达,可能参与肿瘤发生发展的过程[34-37]。有丝分裂检查点蛋白BUB1B(Budding Uninhibited By Benzimidazoles 1 Homolog Beta),是有丝分裂纺锤体检查点复合物的关键成分[38]。在癌症中BUB1B可能会增加胃癌[39]、前列腺癌[40]、乳腺癌[41]、肝癌[17]发生的风险。
本研究中,肝癌肿瘤组织中CDK1、CCNB2、CDC20、NDC80、BUB1B基因表达水平均显著高于癌旁非肿瘤组织,并且与肿瘤分级、分期及患者预后明显相关,提示细胞周期异常分裂可能是HCC的一个重要过程。总之,这5个核心基因与肝癌的恶性生物学行为明显相关,可能在促进肿瘤增殖恶化中通过PID PLK1通路发挥协同作用,仍需进一步研究证实。

