基于高光谱的水稻叶片氮含量估计的深度森林模型研究
李金敏 陈秀青,2 杨 琦 史良胜,*
基于高光谱的水稻叶片氮含量估计的深度森林模型研究
李金敏1陈秀青1,2杨 琦1史良胜1,*
1武汉大学水资源与水电工程科学国家重点实验室, 湖北武汉 430072;2长江勘测规划设计研究有限责任公司, 湖北武汉 430010
高光谱遥感已经成为快速诊断作物水氮状态的一种有效手段。然而, 传统的回归方法和机器学习往往难以挖掘高光谱的全部信息, 深度神经网络又通常需要大量的训练数据, 因此本研究试图探索在少量数据条件下构建深度学习模型并实现叶片氮含量的精准估计。通过在湖北省监利县开展了连续2年不同氮素胁迫水平的水稻试验, 测量了作物全生育期内的216组冠层光谱和叶片氮含量。基于一阶导数光谱, 本文构建了一种新的深度学习模型(深度森林DF)来进行叶片氮含量的反演, 并与2种经典机器学习模型(随机森林RF和支持向量机SVM)和一种深度神经网络模型(多层感知器MLP)进行比较。结果表明, 在基于少量高光谱数据的情况下, DF对水稻叶片氮含量的估算精度要高于MLP, 其中预测精度最高的模型为全波段光谱反演的DF模型(2=0.919, RMSE=0.327)。在2种经典机器学习模型中, RF的估计效果优于SVM, 但2种模型结果都不够稳定。研究表明, 深度森林可以提升高光谱反演叶片氮含量的精度和稳定性, 并且可以通过多粒度扫描相对减轻过拟合程度。该研究结果可为少量数据条件下快速监测作物叶片氮含量提供参考。
叶片氮含量; 深度学习; 机器学习; 高光谱遥感; 水稻
氮是作物生长过程中必不可少的元素, 它在作物蛋白质与核酸的合成中起到非常关键的作用。合理的氮肥供给可以促进作物生长, 但是过量的氮肥施用会带来水体污染和温室气体过量排放等问题[1-2]。快速无损的叶片氮含量估计对于实施农田精准管理是至关重要的。与传统的破坏性取样方法相比, 遥感技术已经被认为是一种快速、动态、无损监测农田环境和作物生长状况的有效手段[3-5]。自从Curran等[6]揭示了光谱信号与叶片氮含量之间的物理机制后, 研究者们开发出越来越多的算法用于解译高光谱信息, 试图更加精准地估计叶片氮含量。
线性方法, 如植被指数法、多元线性回归和偏最小二乘回归等, 由于原理简单、操作方便, 已经被广泛应用于光谱反演作物水氮及叶绿素含量的研究中[7-12]。然而, 高光谱数据通常有着很高的维度, 与叶片氮含量之间存在着高度的非线性关系, 导致这些方法很难挖掘出高光谱数据中隐藏的全部信息[7]。经典的机器学习算法如随机森林(random forest, RF)和支持向量机(support vector machine, SVM)等, 也被认为是准确高效的叶片氮含量估计方法[13-16]。但是这些机器学习算法的估计精度差异很大, 很少有一种稳健的模型可以在各种数据集上表现出稳定的预测效果。
深度学习(deep learning, DL)善于发现高维数据的信息[17], 目前在农业领域已经有了一些成功的应用, 比如作物分类、病虫害监测和产量预测等[18-20]。深度神经网络(deep neural network, DNN)是深度学习领域中最为热门的算法, 但是深度神经网络通常需要大量的数据来满足训练要求。在农田管理过程中获取叶片氮含量标签的成本很高, 导致其数据量很小(通常为几百个数据), 因此深度学习在高光谱反演叶片氮含量的研究中进展缓慢[21]。Zhou等[22]在2019年提出了深度森林算法(deep forest, DF), 可以不基于神经网络, 通过多粒度扫描和级联森林实现DNN中特征转化、层间处理和足够复杂度的3个主要功能。其中, 级联森林可以保证模型训练的深度, 而多粒度扫描可以降低数据处理的维度, 因此可以适用于少量数据的训练。目前深度森林及其改进模型已经成功地应用在高光谱图像处理[23]、雷达目标分类[24]和水稻霜害种子识别[25]等研究领域。
鉴于深度学习在高光谱估计叶片氮含量方面的研究仍不充分[26], 本文探索在数据量较少的情况下(如几百个数据)构建基于高光谱的水稻叶片氮含量估计的深度森林模型。通过设置不同水稻氮素处理水平, 获取2年水稻全生育期的216组冠层高光谱和叶片氮含量数据, 进行一阶导数预处理, 并首次将深度森林算法应用于叶片氮含量估计, 通过与传统机器学习模型(随机森林RF和支持向量机SVM)和深度神经网络模型多层感知器(multi-layer perceptron, MLP)进行对比, 尝试理解不同算法在少量数据条件下的性能差别。
1 材料与方法
1.1 试验概况
于2018年和2019年, 在湖北省监利县(29°49′21″N, 112°50′54″E)开展了连续2年不同氮素水平胁迫的水稻试验。试验小区分布、面积大小和处理水平如图1和表1所示, 其中2018年氮肥处理水平为N0 (0 kg hm–2)、N1 (43 kg hm–2)、N2 (86 kg hm–2)、N3 (130 kg hm–2), 2019年氮肥处理水平为N0 (0 kg hm–2)、N1′ (50 kg hm–2)、N2′ (100 kg hm–2)、N3′ (150 kg hm–2), 每个氮素水平设置3个重复, 在空间上随机分布, 一共12块试验田。2018年水稻试验完成10次观测, 光谱采集日期为2018年7月14日、7月22日、7月28日、8月5日、8月15日、8月25日、9月5日、9月15日、9月25日、10月12日。2019年完成8次观测, 光谱采集日期为2019年5月11日、5月21日、6月02日、6月15日、6月30日、7月17日、7月28日及8月9日。每次观测12块试验田的高光谱和叶片氮含量, 最终得到216个数据样本。
1.2 光谱采集与预处理
采用地物光谱仪(型号: FieldSpec F, R4ASD公司)获取作物的冠层光谱反射率数据(350~2500 nm), 光纤探头视场角为25°。光谱的测定选择在晴朗无云的天气下, 测量时间为10:00—14:00。每次测量将探头垂直向下高于冠层顶部1 m左右, 测量前后用白板进行校正。每个试验小区内随机选取8个点进行观测, 每个点测量10次, 最终各小区的光谱反射率为8个点10次测量的平均值。采用三阶多项式和25个数据点移动窗口的S-G滤波平滑, 并对光谱数据进行一阶导数处理。此外, 所有的光谱反射率数据都剔除了1350~1450 nm、1800~2000 nm、2400~2500 nm的水分吸收带。
1.3 叶片氮含量测定
在每次光谱测量之后, 从每个试验小区随机选取24片叶子, 随机分为3组作为重复, 用来测量作物的叶片氮含量。在105℃下杀青30 min, 使作物体内的酶失活, 然后在70℃下烘至恒重。将干燥的叶片磨碎后通过0.5 mm的筛管, 对每个样品的粉末进行半微量凯氏定氮法测定叶片总氮浓度, 叶片氮含量以百分比的形式表示, 统计结果如表2。
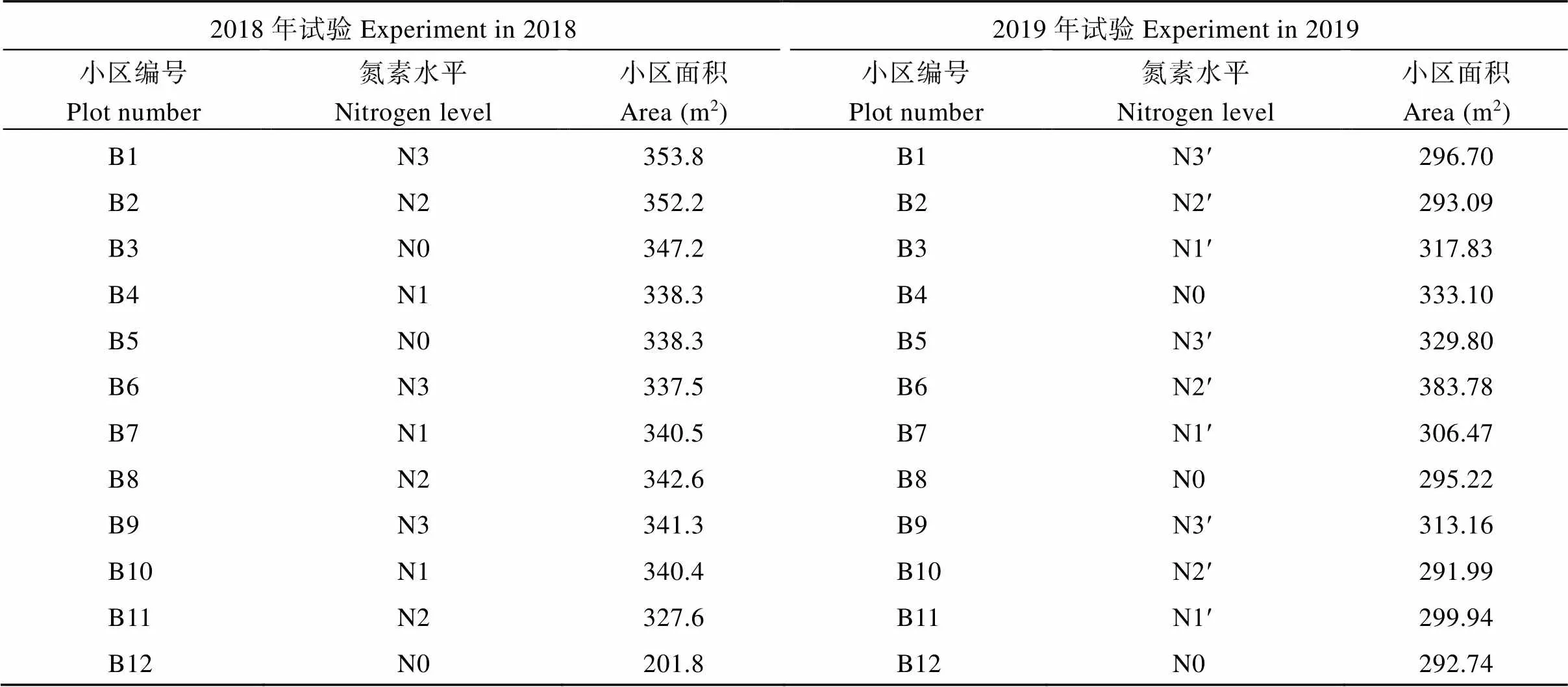
表1 2年水稻试验小区的面积和处理水平
2018年氮肥处理水平为N0 (0 kg hm–2)、N1 (43 kg hm–2)、N2 (86 kg hm–2)、N3 (130 kg hm–2), 2019年氮肥处理水平为N0 (0 kg hm–2)、N1′ (50 kg hm–2)、N2′ (100 kg hm–2)、N3′ (150 kg hm–2)。
Nitrogen level of 2018: N0 (0 kg hm–2), N1 (43 kg hm–2), N2 (86 kg hm–2), N3 (130 kg hm–2). Nitrogen level of 2019: N0 (0 kg hm–2), N1′ (50 kg hm-2), N2′ (100 kg hm–2), N3′ (150 kg hm–2).

表2 2年水稻试验叶片氮含量的统计特征
1.4 模型构建
1.4.1 机器学习模型 随机森林(RF)由Breiman[27]在2001年提出, 是一种集成机器学习学习算法, 采用自助采样法抽取样本, 利用不同的样本构建独立的决策树, 通过平均这些决策树的输出可以得到最终的模拟结果。支持向量机(SVM)通过非线性函数将原始数据映射到高维特征空间, 从而可以在更高维的空间上解决原始数据不能线性分割的难题。表3中列出了随机森林和支持向量机的主要超参数调优结果。
1.4.2 多层感知器 多层感知器(MLP)是一种典型的深度神经网络, 通过输入层神经元接收外界输入, 由多个隐藏层与输出层神经元对信号进行加工, 引入激活函数处理神经元的输出并增强非线性, 采用误差后向传播算法调整网络中的权重实现模型优化。表3列出了多层感知器的主要超参数调优结果。
1.4.3 深度森林 深度森林(DF)是一种新的深度学习算法[22-23], 在多粒度扫描阶段, 设置窗口在原始特征向量上滑动, 并利用基学习器(随机森林)对扫描出的特征进行学习, 提取出数据的原始信息。如图2所示, 假定原始特征维度为400, 通过一个维度为100的窗口滑动, 默认滑动步长为1, 从而可以扫描出301个100维的特征向量。然后将这些100维的向量进行随机森林回归训练, 可以得到301个特征值, 这些特征值被依次拼接成一个301维的特征向量作为级联森林的输入。在本研究中采用了3种不同的滑动窗口, 分别为原始特征长度的1/4、1/8和1/16, 通过不同维度的窗口滑动, 更加全面地挖掘数据信息。
在级联森林阶段, 通过基学习器(随机森林)对上一层传递的信息进行训练, 产生新的增强特征, 并将增强特征与多粒度扫描转换后的特征进行拼接,拼接后的特征向量则传递到下一个级联进行训练。除了第1个级联是直接采用多粒度扫描阶段的特征向量, 随后的每一层都是采用拼接向量作为输入。在每一层产生新的特征向量后, 都会在测试集上进行验证, 如果在测试集上的精度有所提升, 则将特征信息传递给下一个级联继续运算; 如果测试集精度没有提升, 则模型终止训练, 输出最终预测结果。深度森林模型的主要超参数设置见表3。
1.4.5 参数设置和模型评价 本研究中的模型构建与验证分析都是基于Python语言编程完成的, 5种模型调参之后的主要超参数设置见表3, 其余未列出的超参数均为Python函数库默认值。模型评价结果采用决定系数2和均方根误差RMSE作为评价指标,

表3 4种模型关键超参数设置
2 结果与分析
2.1 不同氮素水平下水稻冠层光谱特征分析
图3为2019年不同施氮处理的水稻拔节期冠层高光谱反射率(剔除水分吸收带), 可以发现在不同施氮水平下, 水稻冠层光谱反射率的变化趋势基本相同。在350~680 nm处, 光谱反射率很低, 在550 nm处出现绿峰; 从680 nm到760 nm处存在一个很明显的“红边”效应, 光谱反射率显著提升。在可见光波段处, N3′处理下的光谱反射率最小, 而N0处理下的光谱反射率最大, 而在760 nm以后, 施氮量大的N3′反射率明显高于其他, 且反射率随着施氮量的减少而降低, 这与前人研究一致[7,11]。
氮素水平: N0 (0 kg hm–2), N1′ (50 kg hm–2), N2′ (100 kg hm–2), N3′ (150 kg hm–2)。
Nitrogen level: N0 (0 kg hm–2), N1′ (50 kg hm–2), N2′ (100 kg hm–2), N3′ (150 kg hm–2).
2.2 经典机器学习模型
基于一阶导数光谱构建了随机森林(RF)和支持向量机(SVM)模型, 得到水稻叶片氮含量估计结果。从表4和图4中可以得到, RF预测集最大的2p为0.891, 相应RMSEp最小值为0.378; SVM最大2p为0.755, 相应RMSEp最小值为0.568。其中RF在可见光波段估计精度较差,2p只有0.804; 而SVM在3种波段上的2p都低于0.8。综合比较2种经典机器学习算法, 随机森林模型对于水稻叶片氮含量的估计效果要优于支持向量机。
研究表明可见光波段和近红外波段对于叶片氮含量都有吸收特征, 可见光波段主要与叶绿素有关(430 nm、460 nm、640 nm、660 nm等), 近红外波段以蛋白质的响应为主(1020 nm、1510 nm、1980 nm、2060 nm、2180 nm等)[6-7,28]。但是支持向量机模型不能很好地完成水稻叶片氮含量的估计, 随机森林模型在可见光波段估计精度也相对较差, 因此可能需要更加深度的模型来解译高光谱信息。
2.3 深度学习模型
基于少量的水稻冠层高光谱数据, 本研究构建了一种新的深度学习模型(深度森林DF)用来估计叶片氮含量, 并与传统的多层感知器MLP模型进行比较, 估计结果汇总于表5, 图5显示了2种方法估计精度最高的结果。从表中可知, MLP预测集决定系数2p最大值为0.872, 相应均方根误差RMSEp最小值为0.392; DF最大2p为0.919, 相应RMSEp最小值为0.327。综合比较2种深度学习算法可以发现, 在小数据集情况下, DF对水稻叶片氮含量的估计精度要高于MLP。与Yu等[26]的研究结果相比, 考虑到其训练数据是在实验室内对单片叶子进行光谱测量得到的, 本研究利用的是水稻田中原位观测的冠层高光谱数据, 土壤和水汽的干扰更强, 而DF在预测集上最大的2p为0.919, 仍比Yu等[26]的研究中采用SAE-FNN算法得到的0.903要大, 因此本文所构建的深度森林模型可以适用于少量数据条件下的训练, 并且预测精度比一些神经网络结构的深度学习模型还要高。
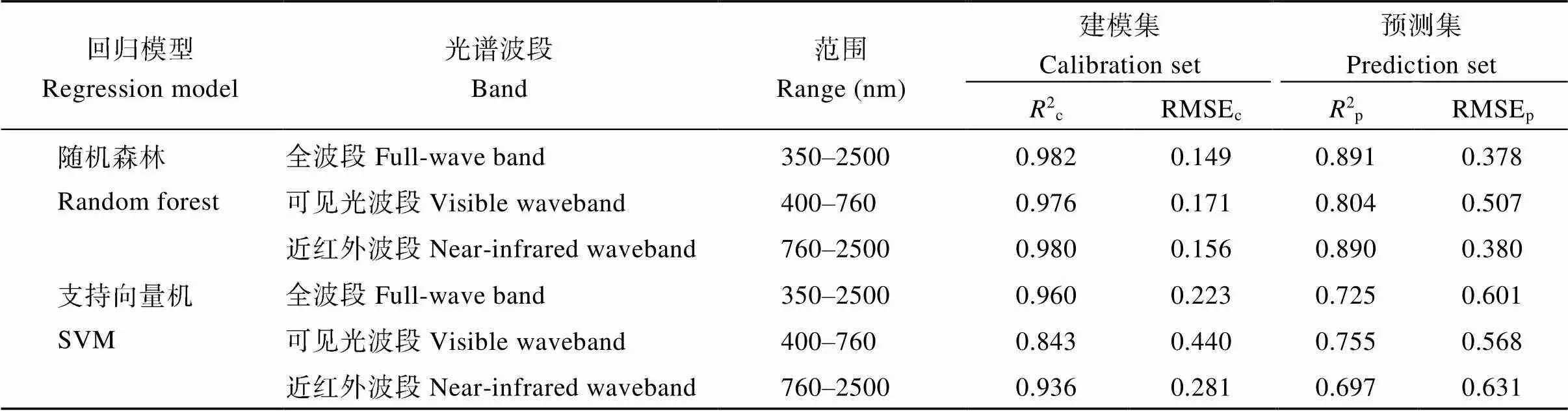
表4 随机森林和支持向量机对水稻叶片含氮量估算效果分析

表5 2种深度学习模型对水稻叶片含氮量估算效果分析
通过表4和表5的对比表明, MLP的预测精度在全波段和近红外波段上低于RF; 而DF可以显著提高经典机器学习在叶片氮含量上的估计精度。在全波段光谱反演中, DF的2p比RF提升了0.03; 在可见光波段反演中, DF的2p提升了0.10; 在近红外波段反演中, DF的2p提升了0.03。相比于在小数据集上表现不稳定的传统机器学习算法(RF和SVM), 通过多粒度扫描和级联结构构建的深度森林模型不仅大大改善了叶片氮含量的估计效果, 同时还提升了模型的稳健性。
3 讨论
高光谱遥感已经成为作物叶片氮含量快速无损监测的有效手段, 但是在高光谱反演模型中, 传统的回归方法和机器学习往往不够稳定, 深度神经网络又通常需要大量的训练数据。例如, Hu等[29]在利用深度学习进行人脸识别任务中, 认为500,000张图像样本是相对较大的样本, 而10,000张图像样本是小数据。Koppe等[30]指出相对于数据获取较易(如在线社交媒体平台或基于智能手机和移动传感器的数据)的研究领域, 在人类精神病学研究中可用的数据属于小样本, 其数量通常小于10,000个。然而, 在利用高光谱反演叶片氮含量的众多研究中, 数据量通常都不超过1000, 如Yi等[31]利用195个水稻冠层高光谱数据构建人工神经网络, 进而监测其冠层氮素水平; 田永超等[8]对360个水稻光谱数据构建植被指数模型, 实现叶片氮含量的精准估计; Tan等[32]利用192个水稻高光谱反射率数据构建BPSO-SVR模型, 定量地解译出寒地水稻的冠层全氮含量。正是由于冠层高光谱反射率和氮素标签数据获取的成本较高, 数据量较小(通常为几百), 导致其深度学习研究进展缓慢。Fu等[33]的综述研究表明, 目前只有Yu等[26]的研究中构建了针对甜菜叶片高光谱图像数据集的深度学习模型, 但是如何利用深度学习来解译野外复杂环境下的冠层高光谱信息并实现氮素水平的精准估计, 仍然缺乏相应研究。本研究以水稻为研究对象, 通过在全生育期内获取水稻的冠层光谱和叶片氮含量数据, 尝试在小数据集(216个样本)条件下构建新的深度学习模型(深度森林DF), 并与经典机器学习模型(随机森林RF和支持向量机SVM)和深度神经网络模型(多层感知器MLP)进行比较。结果表明, DF的估计精度高于MLP, 也高于同类研究中的SAE-FNN算法[26], 证明在小数据条件下深度森林模型可以实现精准的氮素估计。
此外, 还可以发现, 由于光谱数据的维度都是成百上千, 而田间试验获取的样本数量通常只有几百, 因此本研究中几乎所有的模型都存在一定的过拟合现象, 这在其他文献中也有反映。例如, Yao等[34]的高光谱估计冬小麦叶片氮含量研究中, 通过一阶导数处理的支持向量机模型建模集2为0.96, 预测集R为0.78; Yu等[26]的高光谱估计油菜叶片氮含量研究中, 深度学习模型SAE-FNN10建模集2为0.952, 预测集2为0.903。尽管过拟合现象不可避免, 但是对比随机森林RF和深度森林DF, 在全波段上建模集和预测集2之差从0.09缩减到0.06, 在可见光波段上从0.17缩减到0.08, 在近红外波段上从0.09缩减到0.06。这也许可以解释为DF中的多粒度扫描阶段可以利用窗口滑动减小数据处理的维度, 从而减轻相对过拟合的程度。
根据以往文献报道, 由于作物叶片中蛋白质和叶绿素对不同光谱的响应程度不同, 因此有必要同时利用可见光和近红外区域的吸收特征来进行叶片氮含量的估计[35]。然而, 不同方法和不同光谱数据提取出的关键波长有着很大的差别。例如, Feng等[7]的研究指出由叶绿素导致的光谱强吸收主要在可见光波段(如红、蓝波段); Wang等[36]研究表明通过对叶片氮估计构建PLSR、SPA-MLR、RFR等模型, 提取出的重要波长主要在可见光波段(400~760 nm); 而Yao等[34]研究中利用包络线去除法和植被指数法选择的关键波长主要位于红边和近红外区域。与传统的研究方法不同, 本文中采用的深度森林模型结果表明, 无论是可见光波段、近红外波段光谱, 还是全波段光谱, 深度森林都能实现精准的叶片氮含量估计(2>0.9), 这表明深度森林通过多粒度扫描和级联森林2个模块, 实现了特征转化、层间处理和足够复杂度3个功能, 从而可以充分挖掘高光谱数据中的隐藏信息。
4 结论
在基于少量高光谱数据构建的深度学习模型中,深度森林对水稻叶片氮含量的预测精度高于多层感知器, 其中预测精度最高的模型为全波段光谱反演的深度森林模型(2=0.919, RMSE=0.327)。在本文采用的2种经典机器学习模型中, 随机森林的表现优于支持向量机, 但2种模型的表现都不够稳定。深度森林可以提升经典机器学习模型对叶片氮含量的估计精度和稳定性, 并且可以通过多粒度扫描减轻模型相对过拟合程度。
[1] Zhang X, Davidson E A, Mauzerall D L, Searchinger T D, Dumas P, Shen Y. Managing nitrogen for sustainable development., 2015, 528: 51–59.
[2] Yu C, Huang X, Chen H, Godfray H C J, Wright J S, Hall J W, Gong P, Ni S, Qiao S, Huang G. Managing nitrogen to restore water quality in China., 2019, 567: 516–520.
[3] Vigneau N, Ecarnot M, Rabatel G, Roumet P. Potential of field hyperspectral imaging as a non destructive method to assess leaf nitrogen content in wheat., 2011, 122: 25–31.
[4] 高林, 杨贵军, 李长春, 冯海宽, 徐波, 王磊, 董锦绘, 付奎. 基于光谱特征与PLSR结合的叶面积指数拟合方法的无人机画幅高光谱遥感应用. 作物学报, 2017, 43: 549–557. Gao L, Yang G J, Li C C, Feng H K, Xu B, Wang L, Dong J H, Fu K. Application of an improved method in retrieving leaf area index combined spectral index with PLSR in hyperspectral data generated by unmanned aerial vehicle snapshot camera., 2017, 43: 549–557 (in Chinese with English abstract).
[5] 吾木提·艾山江, 买买提·沙吾提, 陈水森, 李丹. 基于GF-1/2卫星数据的冬小麦叶面积指数反演. 作物学报, 2020, 46: 787–797. Umut H, Mamat S, Chen S S, Li D. Inversion of leaf area index of winter wheat based on GF-1/2 image., 2020, 46: 787–797 (in Chinese with English abstract).
[6] Curran P J. Remote sensing of foliar chemistry., 1989, 30: 271–278.
[7] Feng W, Yao X, Zhu Y, Tian Y C, Cao W X. Monitoring leaf nitrogen status with hyperspectral reflectance in wheat., 2008, 28: 394–404.
[8] 田永超, 杨杰, 姚霞, 曹卫星, 朱艳. 利用叶片高光谱指数预测水稻群体叶层全氮含量. 作物学报, 2010, 36: 1529–1537. Tian Y C, Yang J, Yao X, Cao W X, Zhu Y. Monitoring canopy leaf nitrogen concentration based on leaf hyperspectral indices in rice., 2010, 36: 1529–1537 (in Chinese with English abstract).
[9] 陈兵, 韩焕勇, 王方永, 刘政, 邓福军, 林海, 余渝, 李少昆, 王克如, 肖春华. 利用光谱红边参数监测黄萎病棉叶叶绿素和氮素含量. 作物学报, 2013, 39: 319–329. Chen B, Han H Y, Wang F Y, Liu Z, Deng F J, Lin H, Yu Y, Li S K, Wang K R, Xiao C H. Monitoring chlorophyll and nitrogen contents in cotton leaf infected by Verticillium wilt with spectra red edge parameters., 2013, 39: 319–329 (in Chinese with English abstract).
[10] 吴亚鹏, 贺利, 王洋洋, 刘北城, 王永华, 郭天财, 冯伟. 冬小麦生物量及氮积累量的植被指数动态模型研究. 作物学报, 2019, 45: 1238–1249. Wu Y P, He L, Wang Y Y, Liu B C, Wang Y H, Guo T C, Feng W. Dynamic model of vegetation indices for biomass and nitrogen accumulation in winter wheat., 2019, 45: 1238–1249 (in Chinese with English abstract).
[11] 李宗飞, 苏继霞, 费聪, 李阳阳, 刘宁宁, 戴宇祥, 张开祥, 王开勇, 樊华, 陈兵. 基于高光谱数据的滴灌甜菜叶片全氮含量估算. 作物学报, 2020, 46: 557–570. Li Z F, Su J X, Fei C, Li Y Y, Liu N N, Dai Y X, Zhang K X, Wang K Y, Fan H, Chen B. Estimation of total nitrogen content in sugarbeet leaves under drip irrigation based on hyperspectral characteristic parameters and vegetation index., 2020, 46: 557–570 (in Chinese with English abstract).
[12] 陈秀青, 杨琦, 韩景晔, 林琳, 史良胜. 基于叶冠尺度高光谱的冬小麦叶片含水量估算. 光谱学与光谱分析, 2020, 40: 891–897. Chen X Q, Yang Q, Han J Y, Lin L, Shi L S. Estimation of winter wheat leaf water content based on leaf and canopy hyperspectral data., 2020, 40: 891–897 (in Chinese with English abstract).
[13] 张筱蕾, 刘飞, 聂鹏程, 何勇, 鲍一丹. 高光谱成像技术的油菜叶片氮含量及分布快速检测. 光谱学与光谱分析, 2014, 34: 2513–2518. Zhang X L, Liu F, Nie P C, He Y, Bao Y D. Rapid detection of nitrogen content and distribution in oilseed rape leaves based on hyperspectral imaging., 2014, 34: 2513–2518 (in Chinese with English abstract).
[14] 李旭青, 刘湘南, 刘美玲, 吴伶. 水稻冠层氮素含量光谱反演的随机森林算法及区域应用. 遥感学报, 2014, 18: 923–945. Li X Q, Liu X N, Liu M L, Wu L. Random forest algorithm and regional applications of spectral inversion model for estimating canopy nitrogen concentration in rice., 2014, 18: 923–945 (in Chinese with English abstract).
[15] Liang L, Di L, Huang T, Wang J, Lin L, Wang L, Yang M. Estimation of leaf nitrogen content in wheat using new hyperspectral indices and a random forest regression algorithm., 2018, 10: 1940.
[16] 依尔夏提·阿不来提, 买买提·沙吾提, 白灯莎·买买提艾力, 安申群, 马春玥. 基于随机森林法的棉花叶片叶绿素含量估算. 作物学报, 2019, 45: 81–90. Ershat A, Mamat S, Baidengsha M, An S Q, Ma C Y. Estimation of leaf chlorophyll content in cotton based on the random forest approach., 2019, 45: 81–90 (in Chinese with English abstract).
[17] LeCun Y, Bengio Y, Hinton G. Deep learning., 2015, 521: 436–444.
[18] Mohanty S P, Hughes D P, Salathé M. Using deep learning for image-based plant disease detection., 2016, 7: 1419.
[19] Yang Q, Shi L, Han J, Zha Y, Zhu P. Deep convolutional neural networks for rice grain yield estimation at the ripening stage using UAV-based remotely sensed images., 2019, 235: 142–153.
[20] Zhong L, Hu L, Zhou H. Deep learning based multi-temporal crop classification., 2019, 221: 430–443.
[21] Sun J, Yang J, Shi S, Chen B, Du L, Gong W, Song S. Estimating rice leaf nitrogen concentration: influence of regression algorithms based on passive and active leaf reflectance., 2017, 9: 951.
[22] Zhou Z, Feng J. Deep forest., 2019, 6: 74–86.
[23] Liu X, Wang R, Cai Z, Cai Y, Yin X. Deep multigrained cascade forest for hyperspectral image classification., 2019, 57: 8169–8183.
[24] Zhang J, Song H, Zhou B. SAR target classification based on deep forest model., 2020, 12: 128.
[25] Zhang L, Sun H, Rao Z, Ji H. Hyperspectral imaging technology combined with deep forest model to identify frost-damaged rice seeds., 2020, 229: 117973.
[26] Yu X, Lu H, Liu Q. Deep-learning-based regression model and hyperspectral imaging for rapid detection of nitrogen concentration in oilseed rape (L.) leaf., 2018, 172: 188–193.
[27] Breiman L. Random forests., 2001, 45: 5–32.
[28] Berger K, Verrelst J, Féret J, Wang Z, Wocher M, Strathmann M, Danner M, Mauser W, Hank T. Crop nitrogen monitoring: recent progress and principal developments in the context of imaging spectroscopy missions., 2020, 242: 111758.
[29] Hu G, Peng X, Yang Y, Hospedales T M, Verbeek J. Frankenstein: learning deep face representations using small data., 2017, 27: 293–303.
[30] Koppe G, Meyer-Lindenberg A, Durstewitz D. Deep learning for small and big data in psychiatry., 2020, 46: 176–190.
[31] Yi Q, Huang J, Wang F, Wang X, Liu Z. Monitoring rice nitrogen status using hyperspectral reflectance and artificial neural network., 2007, 41: 6770–6775.
[32] Tan K, Wang S, Song Y, Liu Y, Gong Z. Estimating nitrogen status of rice canopy using hyperspectral reflectance combined with BPSO-SVR in cold region., 2018, 172: 68–79.
[33] Fu Y, Yang G, Li Z, Li H, Li Z, Xu X, Song X, Zhang Y, Duan D, Zhao C. Progress of hyperspectral data processing and modelling for cereal crop nitrogen monitoring., 2020, 172: 105321.
[34] Yao X, Huang Y, Shang G, Zhou C, Cheng T, Tian Y, Cao W, Zhu Y. Evaluation of six algorithms to monitor wheat leaf nitrogen concentration., 2015, 7: 14939–14966.
[35] Kokaly R F. Investigating a physical basis for spectroscopic estimates of leaf nitrogen concentration., 2001, 75: 153–161.
[36] Wang J, Chen Y, Chen F, Shi T, Wu G. Wavelet-based coupling of leaf and canopy reflectance spectra to improve the estimation accuracy of foliar nitrogen concentration.l, 2018, 248: 306–315.
Deep learning models for estimation of paddy rice leaf nitrogen concentration based on canopy hyperspectral data
LI Jin-Min1, CHEN Xiu-Qing1,2, YANG Qi1, and SHI Liang-Sheng1,*
National Key Laboratory of Water Resources and Hydropower Engineering Science, Wuhan University, Wuhan 430072, Hubei, China;2Changjiang Survey Planning, Design and Research Co., Ltd., Wuhan 430010, Hubei, China
Rapid and nondestructive detection of crop nitrogen status is crucial to precision agriculture management. Hyperspectral remote sensing has been proposed to be a powerful tool for expediently monitoring crop nitrogen status. However, conventional regression methods and machine learning (ML) are difficult to utilize the full information of hyperspectral data, and deep neural networks (DNN) usually require a huge number of training data. Therefore, we attempt to construct deep learning models with a small amount of data and achieve accurate estimation of leaf nitrogen concentration (LNC). A two-year field experiment of paddy rice with four nitrogen levels were conducted at Jianli, Hubei province, China. A total of 216 samples containing canopy hyperspectral data and rice LNC were measured during two growing seasons. Based on the first derivative of hyperspectral data, a new deep learning model (deep forest, DF) was constructed for LNC estimation and compared with two traditional machine learning models (random forest, RF and support vector machine, SVM) and one deep neural network model (multi-layer perceptron, MLP). The results showed that, based on a small number of hyperspectral data, deep forest acquired higher accuracy than MLP. And the optimal estimation (2= 0.919, RMSE = 0.327) was obtained by the deep forest model based on full-wave band spectrum (350–2500 nm). Between two classical machine learning models, random forest achieved better results than SVM, but both methods were unstable. In conclusion, deep forest improved the prediction accuracy and model robustness for all band situations, and alleviated the degree of overfitting by multi-grained scanning. These results can provide a deep insight to detect crop nitrogen status rapidly when confronted with limited data.
leaf nitrogen concentration; deep learning; machine learning; hyperspectral remote sensing; paddy rice
10.3724/SP.J.1006.2021.02060
本研究由国家自然科学基金项目(51861125202)资助。
This study was supported by the National Natural Science Foundation of China (51861125202).
史良胜, E-mail: liangshs@whu.edu.cn
E-mail: jinmlee@whu.edu.cn
2020-08-24;
2020-12-01;
2021-01-04.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20210104.1140.002.html

