Bispora sp. MEY-1来源的嗜酸海藻糖酶TreA酶学性质研究
蒋肖, 涂涛, 王坤, 彤丽格, 罗会颖
(中国农业科学院饲料研究所, 农业农村部饲料生物技术重点实验室, 北京 100081)
海藻糖是一类非还原型二糖,由两个α-D-葡萄糖通过α-1,1-糖苷键连接而成,通常以二水化合物的形式存在,是所有糖中化学性最不活跃的糖[1]。它广泛存在于细菌、真菌、昆虫、植物及藻类等生物体内,可以起到为生物体提供碳源,以及提高细胞耐受性的作用[2]。
海藻糖酶(trehalase,EC3.2.1.28)可以将海藻糖水解产生两分子的葡萄糖。海藻糖酶广泛存在于动物、植物和微生物体内,目前已经从多种生物体内分离出来了海藻糖酶基因。根据氨基酸序列相似性、结构相似性等特点,海藻糖酶在碳水化合物活性酶(carbohydrate-active enzymes, CAZy)数据库中,被归类到糖苷水解酶(glucoside hydrolase, GH)第15、37和65家族[3-4]。除了毕赤酵母通过海藻糖磷酸化酶分解海藻糖以外,其他真菌系统中都是通过海藻糖酶分解海藻糖,并且对海藻糖具有严格的底物专一性。基于海藻糖酶的催化特点,海藻糖酶可以广泛应用于淀粉多糖制备、食品、临床医疗和害虫防治等生物工业中[2,5]。丁春磊[6]研究表明,在燃料乙醇生产过程中,添加海藻糖酶可有效提高乙醇的产率,改善酵母的健康状态,降低生产成本。刘迎春等[7]对金龟子绿僵菌中的海藻糖酶ReATM1p进行酶学性质鉴定和基因敲除等研究,进一步证明了海藻糖酶在害虫生物防治中的应用价值。韩隽等[8]研究表明,谷氨酸发酵生产的后期加入海藻糖酶可以提高糖酸转化率的效果,进而提高谷氨酸的生产量。
根据海藻糖酶酶促反应的最适pH不同,可以将真菌海藻糖酶分为酸性海藻糖酶和中性海藻糖酶[9]。真菌中性海藻糖酶的最适pH为7.0,通常位于细胞胞质中,其酶活性受转录调控机制的影响;酸性海藻糖酶最适pH为3.5~6.0,是一种外分泌性糖蛋白,主要位于液泡、细胞壁或者分泌到胞外[9]。除此之外,还有一种源于嗜热真菌的海藻糖酶,既具有酸性海藻糖酶最适pH反应条件的特点,又具备中性海藻糖酶可被Ca2+或Mn2+激活和被ATP抑制的特点[10-12]。目前,多种性质优良的糖苷水解酶基因资源被成功挖掘并报道,如最适pH为1.5的甘露聚糖酶[14]、最适pH为4.5的木聚糖酶[15]和最适pH为5.0的葡聚糖酶[16]等,人们研究最为清晰的是酿酒酵母中的酸性海藻糖酶。
Bisporasp. MEY-1菌株为本实验室从酸矿中分离的一株嗜酸真菌,在pH 2.5~3.0的环境中生长情况最好[13]。本研究从嗜酸真菌Bisporasp. MEY-1中克隆了一个海藻糖酶基因TreA并在毕赤酵母GS115中成功实现了异源表达,通过对其酶学性质进行鉴定分析,以期获得可以应用到工业生产的海藻糖酶,进而丰富海藻糖酶资源。
1 材料与方法
1.1 菌株、载体及试剂
Bisporasp. MEY-1菌株由本实验室分离,保存在中国普通微生物菌种保藏管理中心(China General Microbiological Culture Collection, CGMCC),登记号为CGMCC250027。Bisporasp. MEY-1的活化和培养条件参照李烨青等[17]方法。基因克隆过程中间载体为pEASY-T3,克隆宿主为大肠杆菌感受态EscherichiacoliTrans-T1,均购自北京全式金生物技术有限公司。海藻糖酶基因TreA外源表达用的载体为pPIC9,表达宿主为毕赤酵母GS115,均为本实验室保存。试验中所用的总RNA提取试剂盒购自天根生化科技有限公司;反转录试剂盒购自东洋纺生物科技有限公司;细菌质粒提取试剂盒和DNA胶回收试剂盒购自天根生化科技有限公司;限制性核酸内切酶EcoRⅠ,NotⅠ购自TaKaRa有限公司;高保真DNA聚合酶Fast pfu DNA聚合酶购自北京全式金生物技术有限公司;其他试剂,如甲醇、乙醇和乙酸等试剂均购于国药集团化学试剂有限公司。
1.2 培养基
试验中所用到的培养基有马铃薯葡萄糖固体培养基(PDA)、马铃薯葡萄糖液体培养基(PDB)、LB液体培养基、LB固体培养基、MD培养基、YPD培养基、BMMY及BMGY培养基,其详细配制参照李晓丽等[18]方法;海藻糖酶酶活性测定所用的DNS试剂配制参照李烨青等[17]方法。
1.3 海藻糖酶基因TreA序列分析
根据嗜酸菌株Bisporasp. MEY-1的全基因组序列测序结果,查找到一个新的海藻糖酶基因TreA,并对其进行生物信息学分析。首先,将序列提交给在线网站Softberry(http://linux1.softberry.com/berry.phtml)预测基因的内含子和外显子,SignalP 4.1 Server (http://www.cbs.dtu.dk/services/SignalP/)预测编码蛋白质信号肽的序列。然后利用Vector NTI Advance 10.0软件将基因翻译为氨基酸序列后,分析其氨基酸序列的长度、蛋白质理论分子质量。最后,将氨基酸序列提交到Expasy.PI(https://web.expasy.org/compute_pi/)预测新海藻糖酶TreA的等电点。
1.4 海藻糖酶基因TreA克隆与毕赤酵母重组载体构建
将嗜酸菌株Bisporasp. MEY-1在PDB培养基、30 ℃培养条件下活化48 h后,转接到以海藻糖为唯一碳源的诱导培养基中,于30 ℃、210 r·min-1震荡培养2~3 d,12 000 r·min-1离心10 min收集菌体。根据真菌总 RNA 提取试剂盒说明书提取菌体的总RNA,然后再利用反转录试剂盒Rever Tra Ace-a-TM kit将RNA反转录为cDNA。根据去除信号肽序列的全长基因设计引物(F: 5′-GCTGAATTCTACGTAGAATTCAAGA-TATATTCGACT-3′;R: 5′-CGAATTAATTCGC-CGGCGGCTCAAATAACCAAGG-3′)(横线标注分别为限制性酶切位点EcoRⅠ和NotⅠ),然后以cDNA为模板进行PCR扩增。扩增产物经EcoRⅠ和NotⅠ双酶切,利用T4 DNA连接酶将其连入载体pPIC9后热击转化Trans-T1大肠杆菌感受态,挑取阳性克隆子送测序公司测序,测序正确的重组载体命名为pPIC9-TreA。
1.5 海藻糖酶TreA在毕赤酵母中的表达及纯化
利用质粒小提试剂盒提取测序正确的pPIC9-TreA质粒,限制性内切酶BglⅡ将其线性化后,回收产物电击转化毕赤酵母GS115感受态细胞。然后将转化子涂布于MD平板,在30 ℃培养箱中静置培养48 h后挑取48个克隆子于含有3 mL BMGY培养基的离心管中震荡培养(30 ℃,220 r·min-1);培养48 h后,4 500 r·min-1离心5 min,将菌体重悬于1 mL含有0.5%甲醇的BMMY培养基中诱导培养48 h。最后,通过酶活性测定获得阳性克隆子进行摇瓶放大培养。
将筛选的阳性克隆子在YPD培养基中活化培养72 h,按1%的接种量接种于400 mL BMGY培养基震荡培养48 h后,4 500 r·min-1离心5 min收集菌体转入200 mL BMMY培养基中诱导培养48 h,每隔24 h补加0.5%甲醇。
诱导培养结束后,12 000 r·min-1离心5 min收集发酵液。将200 mL发酵液经10 kD膜包(Vivascience, Germany)浓缩至15 mL后,发酵液于pH 7.0、10 mmol·L-1柠檬酸-磷酸氢二钠缓冲液中过夜透析。然后,将透析过的酶液通过阴离子纯化柱(HiTrap Q HP)进行纯化,收集不同浓度盐离子洗脱下来的蛋白,并通过SDS-PAGE分析确定纯化的单一条带。
1.6 海藻糖酶TreA酶活性测定
海藻糖酶的酶活性测定采用3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)法检测还原糖生成量进行活性分析。具体方法如下:在pH 4.0、60 ℃条件下,1 mL的反应体系包括100 μL酶液和900 μL 1%的海藻糖底物,反应10 min,加入1.5mL DNS终止反应后沸水煮5 min,540 nm下测定OD值。对照组先加终止液,再加酶液。每组三个平行,一个对照。海藻糖酶活性单位定义:在pH 4.0、60 ℃条件下,每分钟分解海藻糖生成1 μmol还原糖所需要的酶量为1个活性单位(U)。
1.7 海藻糖酶TreA酶学性质测定
测定海藻糖酶最适pH时,将纯化的海藻糖酶TreA分别置于60 ℃,不同pH(1.0~12.0,其中pH 1.0~3.0是0.1 mol·L-1甘氨酸-HCl缓冲液;pH 3.0~9.0是0.1 mol·L-1磷酸氢二钠-柠檬酸缓冲液;pH 9.0~12.0是0.1 mol·L-1NaOH-甘氨酸缓冲液)底物条件下进行酶促反应。测定海藻糖酶TreA的pH稳定性时,纯海藻糖酶TreA酶液在不同pH 1.0~12.0缓冲液稀释后置于37 ℃恒温水浴锅中处理1 h,然后在pH 4.0、60 ℃条件下测定其相对剩余酶活,未进行处理酶活为100%对照。
测定海藻糖酶的最适温度时,纯化的海藻糖酶TreA分别在20~90 ℃范围内,pH 4.0条件下进行酶促反应。测定海藻糖酶TreA的热稳定性时,纯化的海藻糖酶TreA分别在60、65和70 ℃恒温水浴锅中分别处理0、2、5、10、15、20、30和60 min后,在pH 4.0、60 °C条件下测定其剩余酶活力,未进行处理的酶活为100%对照。
分别利用pH 4.0、0.1 mol·L-1磷酸氢二钠-柠檬酸缓冲液配制不同浓度(0.5~10 mg·mL-1)的海藻糖作为反应底物。将纯化的海藻糖酶TreA稀释至合适的浓度分别在不同浓度的底物中反应5 min(pH 4.0、60 ℃),测定其酶活性。将测定的酶活数据利用GraphPad Prism 5.01软件分析,获得海藻糖酶TreA的Vmax和Km。
1.8 不同金属离子或化学试剂对酶活力的影响
在海藻糖底物中分别添加NaCl、LiCl、KCl、AgNO3、HgCl2、MgCl2、MnCl2、Pb(CH3COO)2、NiCl2、ZnCl2、CoCl2、CuCl2、CaCl2、CrCl3、FeCl3、SDS、EDTA,和β-巯基乙醇共18种物质,使其终浓度为5和10 mmol·L-1。然后,在pH 4.0、60 ℃条件下分别测定海藻糖酶在不同底物中的剩余酶活力。以未添加任何物质的反应体系为对照组,计算处理后海藻糖酶TreA的相对剩余酶活,评估各种金属离子和化学试剂对海藻糖酶TreA酶活力的影响,每组反应设计三个重复。
1.9 海藻糖酶TreA的蛋白酶抗性评估
在纯化的海藻糖酶TreA中分别添加胰蛋白酶、蛋白酶K、胶原蛋白酶、糜蛋白酶、枯草蛋白酶和胃蛋白酶,使其终浓度为0.1 mg·mL-1,然后将其置于37 ℃恒温水浴锅中分别处理0、30和60 min。在pH 4.0、60 ℃条件下测定其相对剩余酶活,以未添加蛋白酶的反应体系为对照组,来评估海藻糖酶TreA的蛋白酶抗性。
1.10 数据分析
采用Excel 2018和Origin 2017对测定的数据进行处理、计算和差异性分析。
2 结果与分析
2.1 海藻糖酶基因TreA的克隆与序列分析
从嗜酸菌株Bisporasp. MEY-1的基因组中克隆海藻糖酶基因TreA,其cDNA全长序列为3 075 bp,可以翻译编码1 024个氨基酸。其编码的氨基酸序列经在线网站进行信号肽预测,结果表明N端的前21个氨基酸为信号肽序列。TreA成熟蛋白的理论分子质量为109.6 kD,等电点为4.5,属于糖苷水解酶32家族。海藻糖酶TreA的氨基酸序列经在线网站(http://www.cbs.dtu.dk/services/NetNGlyc/)预测,结果显示其蛋白质表面共有7个潜在N-糖基化位点,分别为Asn117、Asn154、Asn177、Asn217、Asn274、Asn289和Asn564。在氨基酸组成方面,酸性氨基酸(天冬氨酸和谷氨酸)占9.28%;碱性氨基酸(赖氨酸和精氨酸)占4.88%;极性氨基酸(天冬酰胺、半胱氨酸、谷氨酰胺、丝氨酸、苏氨酸和酪氨酸)占34.67%;疏水氨基酸(丙氨酸、异亮氨酸、亮氨酸、苯丙氨酸、色氨酸和缬氨酸)占34.47%。将TreA的氨基酸序列在NCBI数据库中进行序列比对,该基因与Baudoiniapanamericana来源的海藻糖酶序列最为相似,序列一致性为68.9%,显示了海藻糖酶基因TreA的新颖性。
2.2 海藻糖酶TreA重组载体的表达和纯化结果
重组表达载体pPIC9-TreA在毕赤酵母GS1115中成功表达,将筛选的阳性克隆子进行摇瓶表达,获得的发酵液进行后续的纯化处理。粗酶液经浓缩、阴离子纯化柱纯化等一系列处理后,获得单一纯化峰,并将浓缩液、纯化液及Endo H处理的纯蛋白分别进行SDS-PAGE凝胶电泳分析。结果显示,TreA的目的条带大小为135 kD左右(图1),经Endo H脱糖基处理后目的条带降为110 kD左右,与理论分子质量一致。
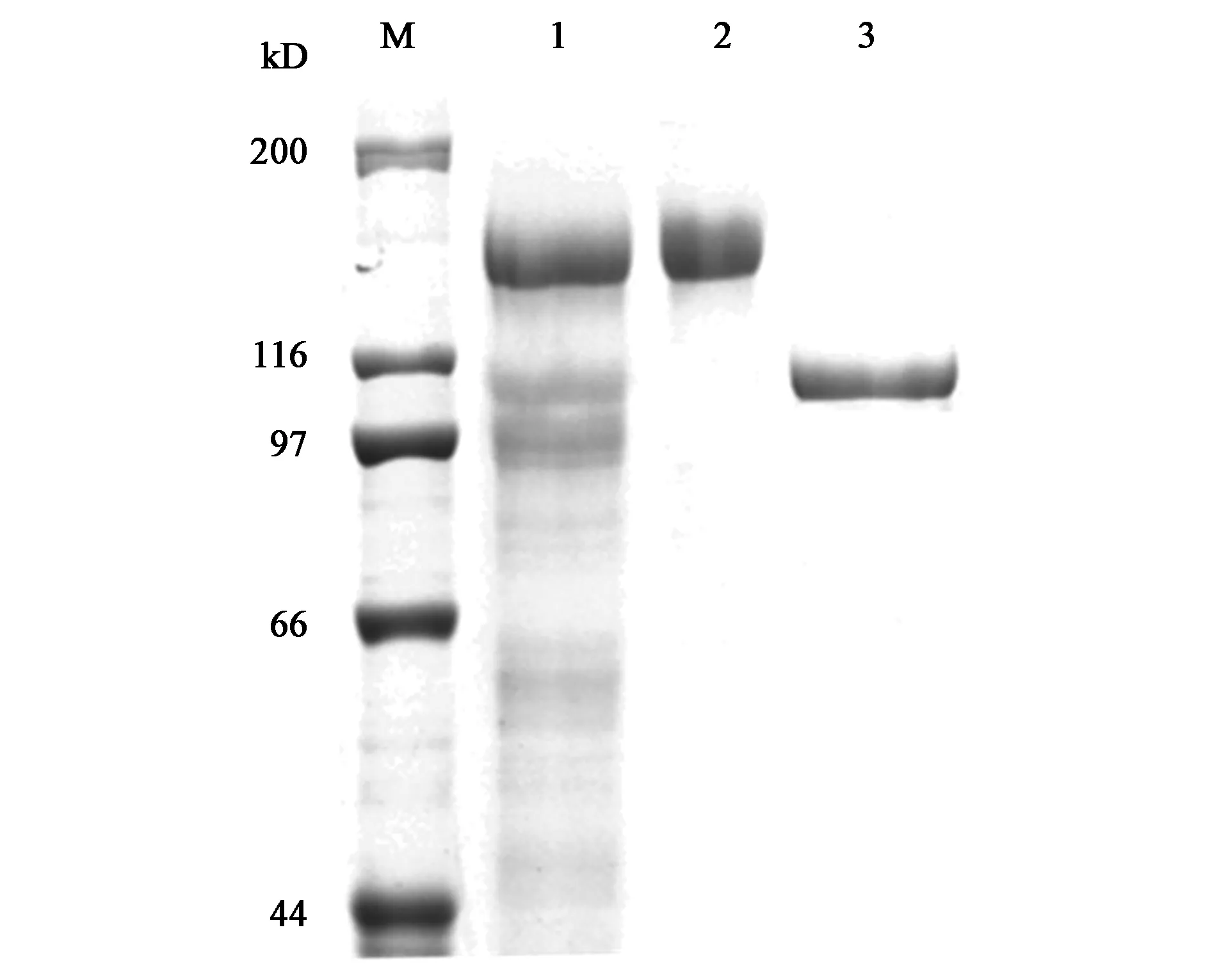
注:M—标准蛋白样品的特定理论分子质量;1—浓缩后TreA; 2—纯化后的TreA; 3—Endo H处理的TreA。Note:M— Molecular weight standard; 1—concentrated TreA with ultrafiltration membrane; 2—Purified TreA; 3—Purified TreA with Endo H.图1 TreA的SDS-PAGE凝胶电泳分析Fig.1 SDS-PAGE analysis of purified recombinant TreA
2.3 海藻糖酶TreA的酶学性质分析
测定重组海藻糖酶TreA在不同pH缓冲液、50 ℃条件下的酶活性,结果表明,TreA的最适pH为4.0,在pH 2.2~5.0范围内,该酶能够维持其60%以上的酶活力,是一种酸性海藻糖酶(图2A)。TreA在极酸条件下(pH 1.0~3.0)可维持100%的剩余酶活性,pH 4.0~7.0可维持100%的剩余酶活性,随着pH条件向碱性方向移动,其稳定性逐渐减弱(图2B)。TreA的最适温度为60 ℃,在40~70 ℃之间都可以剩余50%以上的酶活力(图2C),属于中高温酶。分析TreA的热稳定性,在60和65 ℃下分别处理60 min,测定处理后酶液的剩余酶活分别为85%和70%,在70 ℃下处理30 min,依然能够保持40%的酶活力(图2D)。
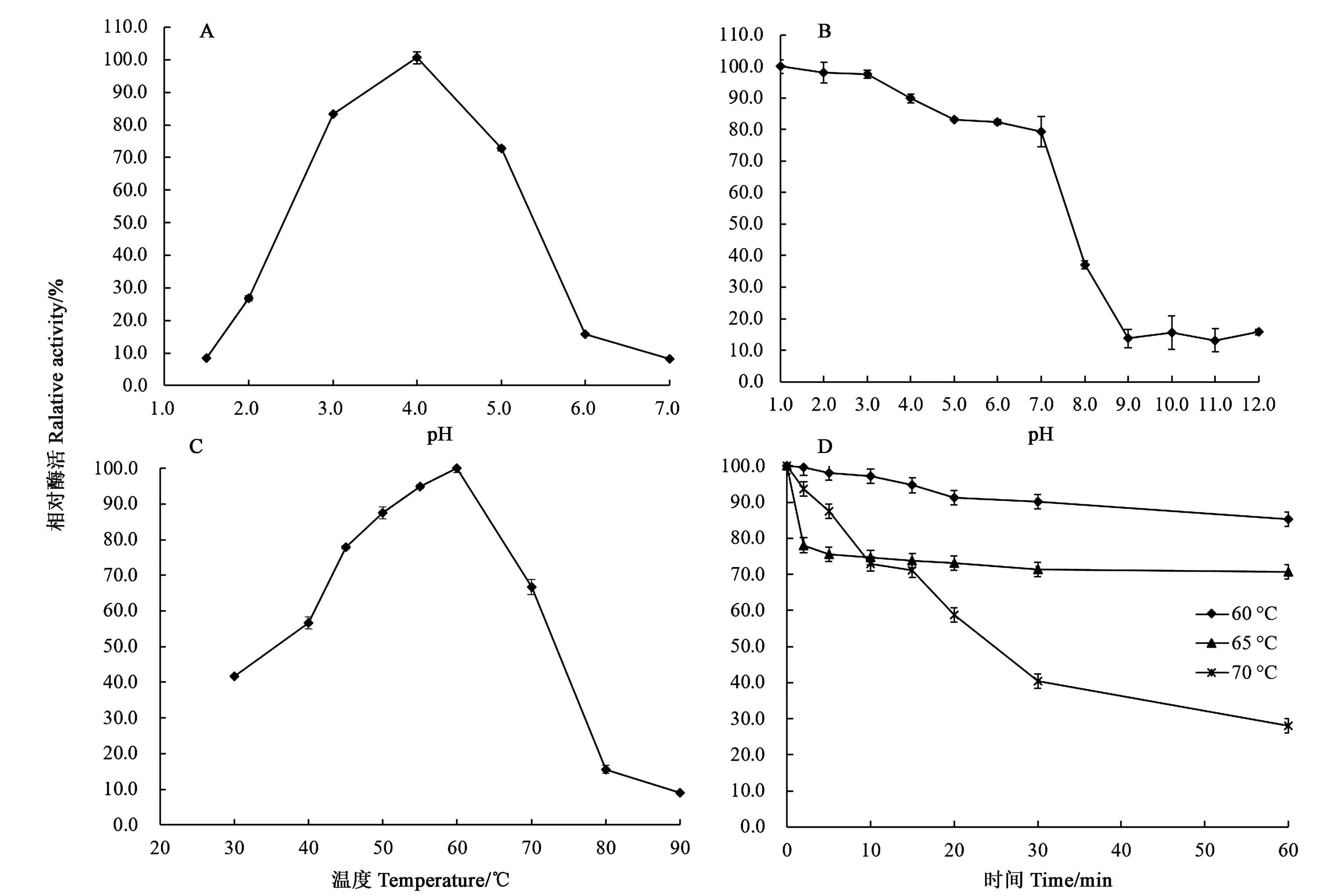
A:最适pH;B:pH稳定性;C:最适温度;D:温度稳定性A: Effect of pH on enzyme activities; B: pH stabilities; C: Effect of temperature on enzyme activities; D: Thermostability图2 重组海藻糖酶TreA的基本酶学性质Fig.2 Characterization of purifird recombinant TreA
以1%海藻糖为底物,在最适条件(pH 4.0、60 ℃)下,重组海藻糖酶TreA的比活为1 913 U·mg-1,动力学参数Km和Vmax分别为0.67 mg·mL-1和119 μmol·min-1·mg-1。
2.4 金属离子及化学试剂海藻糖酶TreA的影响
18种金属离子和化学试剂对重组海藻糖酶TreA酶活性的影响如表1所示。当化学物质浓度为5 mmol·mL-1时,Fe3+、Mn2+、Pb2+和巯基乙醇对海藻糖酶起激活作用;SDS、Ag+和Hg2+对海藻糖酶具有抑制作用;Ag+、Na+、K+、Ca2+、SDS和Hg2+均对海藻糖酶TreA的酶活性具有抑制作用。浓度为10 mmol·mL-1时抑制效果较5 mmol·mL-1时更强,其中对海藻糖酶TreA酶活性影响最大的为Ag+,经5 mmol·mL-1离子处理后其相对剩余酶活性降为43.5%,经10 mmol·mL-1处理后几乎没有酶活性。Co2+、Fe3+和β-巯基乙醇可显著提高海藻糖酶TreA的酶活性,Co2+、Fe3+浓度为5 mmol·mL-1时提高海藻糖酶TreA酶活性效果更加明显,β-巯基乙醇浓度为10 mmol·mL-1时提高海藻糖酶TreA酶活性效果更加明显。
2.5 海藻糖酶TreA对蛋白酶的抗性
测定纯海藻糖酶TreA在含有0.1 mg·mL-1缓冲液中处理后的相对剩余酶活,如图3所示,海藻糖酶TreA对胰蛋白酶、胶原蛋白酶和胃蛋白酶具有非常好的蛋白酶抗性,几乎没有酶活性的损失。然而,海藻糖酶TreA对蛋白酶K和糜蛋白酶的蛋白酶抗性较差,并且随着在蛋白酶中处理的时间越长,酶活性损失越严重:海藻糖酶TreA在0.1 mg·mL-1蛋白酶K和糜蛋白酶中处理30 min后,相对剩余酶活分别为80%、40%;处理60 min后相对剩余酶活分别为60%、20%。另外,枯草蛋白酶对海藻糖酶TreA的酶活性也有轻微的抑制作用。

表1 不同金属离子及化学试剂下纯化重组海藻糖酶TreA活性Table 1 Activities of TreA under different concentration of metal ions and chemical reagents
3 讨论
海藻糖酶作为一种常见的生物催化剂,在生物工业中的应用通常需要在酸性条件下进行,因此挖掘具有嗜酸特性的海藻糖酶基因具有重要意义。在本研究中,通过基因工程技术从嗜酸真菌Bisporasp.-MEY-1中成功克隆了一种酸性、耐中高温的海藻糖酶TreA,其最适pH为4.0,在pH 2.2~5.0范围内,可维持60%以上的剩余酶活力;在pH 1.0~9.0条件下处理1 h,可以维持79%以上的剩余酶活力,在最适pH和pH稳定性上具有明显的优势。目前已有多种真菌来源的酸性海藻糖酶基因被报道,刘迎春[7]利用RT-PCR、RACE等方法成功地克隆了金龟子绿僵菌的酸性海藻糖酶基因;酿酒酵母Saccharomycescerevisiae[19]、白色念珠菌Candidaalbicans[20]、构巢曲霉Aspergillusnidulans[21]中也分离鉴定出酸性海藻糖酶基因。此外,在NCBI数据库中还报道了另外两种丝状真菌烟曲霉Aspergillusfumigatus和埃默森篮状菌Talaromycesemrsonii来源的酸性海藻糖酶疑似基因序列[7],Bisporasp.-MEY-1来源的酸性海藻糖酶基因TreA与上述几种来源的海藻糖酶基因的同源性在25%~50%之间。
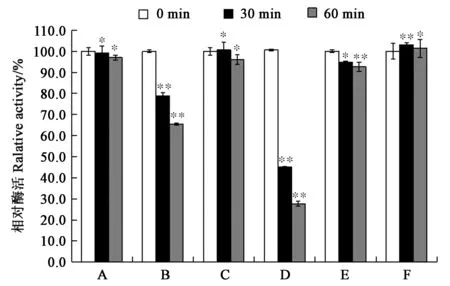
注:A—胰蛋白酶;B—蛋白酶K;C—胶原蛋白酶;D—糜蛋白酶;E—枯草蛋白酶;F—胃蛋白酶。 * 和**分别表示在P<0.05和P<0.01水平具有显著性。Note:A—Trypsin; B—Proteinase K; C—Collagenase; D—Chymotrypsin; E—Subtilase; F—Pepsase. * and ** indicate significant difference at P<0.05 and P<0.01 levels, respectively.图3 蛋白酶对纯化重组海藻糖酶TreA的活性影响Fig.3 Effect of protease on enzyme activities of recombinant TreA
上述真菌来源的酸性海藻糖酶中,已有酶学性质报道的为来源于金龟子绿僵菌的海藻糖酶ReATM1p,NCBI登录号为EF190950,与本研究中的酸性海藻糖酶TreA的序列一致性为47.8%。ReATM1p的最适pH为6.0,在pH 4.5~7.5范围内具有较高的酶活力,但是当反应体系pH超过7.5时,酶活力迅速下降。与本研究中的酸性海藻糖酶TreA的最适pH和pH稳定性相比,ReATM1p的酸适应性明显较差[7]。Enfert等[21]发现,为减少酸性条件下正电荷的积累,许多嗜酸蛋白表面进化出大量酸性氨基酸残基。但是,已有晶体报道的海藻糖酶与本研究的酸性海藻糖酶TreA序列一致性最高的仅为22%,不满足同源建模的条件,无法实现TreA三维结构的可视化分析。分析TreA和ReATM1p的一级结构时,发现TreA的氨基酸序列中酸性氨基酸(天冬氨酸Asp和谷氨酸Glu)占9.28%,ReATM1p的氨基酸序列中酸性氨基酸占8.4%。因此,推测可能是TreA中酸性氨基酸的比例明显高于ReATM1p,导致TreA的酸适应性明显高于ReATM1p。
酸性海藻糖酶在酵母中以一种单体形式发挥活性,在构巢曲霉A.nidulans中以二聚体形式发挥酶活性。酸性海藻糖酶Km的范围为0.8~5 mg·mL-1[19],本研究中的酸性海藻酶TreA的Km为0.67 mg·mL-1,对海藻糖的底物特异性极强。酸性海藻糖酶另一特点是具有大量潜在的N-糖基化位点,这也解释了TreA纯化蛋白经EndoH脱糖基处理后在SDS凝胶条带变小的原因[19-23]。此外,分析酸性海藻糖酶TreA的氨基酸序列,发现其N端1~18位的短肽为信号肽,这进一步验证了酸性海藻糖酶可通过分泌途径的靶向转运,解释了这类蛋白定位于细胞表面的原因[21]。
综上所述,本研究成功获得了一个新颖的酸性海藻糖酶基因TreA,并在毕赤酵母中实现了高效表达。该酸性海藻糖酶具有良好的酸适应性,同时对海藻糖有专一、较好的水解活力。这些特点表明酸性海藻糖酶TreA在生物工业中具有极大的应用价值。

