养殖模式对草鱼池塘底泥微生物群落结构影响的分析
章海鑫,余智杰,张燕萍,张爱芳,王 生,李 涵,付辉云
(江西省水产科学研究所,南昌 330039)
草鱼(Ctenopharyngodonidellus)是我国淡水养殖面积最大、产量最高的品种,产量占淡水养殖总产量的18.4%[1]。水产养殖(特别是草鱼养殖)是为国民提供优质蛋白的主要方式之一,但养殖过程中存在着因养殖密度大而导致水体富营养化、病害增多、品质低下等问题。因此,发展既能保证产量又能提高产品质量,减少环境污染的养殖技术是草鱼养殖面临的迫切问题,而生态养殖是解决这一问题的重要发展方向。
水产养殖生态系统是由养殖动物、水体、底泥等因素构成的相互影响、相互作用的综合生态系统。其中,底泥中的微生物对系统中有机质、无机盐等物质循环代谢的强度直接影响水体富营养化水平[2]和水体理化环境[3];水体中水生植物对微生物分解形成的无机盐的吸收和利用又反过来影响着微生物的群落结构和养殖动物的生长;养殖动物所产生的营养物质对于微生物的结构组成又有一定选择作用。所以,底泥微生物在整个养殖系统中扮演着重要的角色,它不仅参与养殖系统中有机质、无机盐等物质的循环代谢及能量代谢[4],本身又受到水体环境因子、水体植物和养殖动物的影响。
许多研究者认为通过提高养殖系统微生物的分解能力,平衡好水生植物和浮游植物对无机盐的吸收和其为养殖动物提供饵料的关系是生态养殖发展的方向[5],也是解决养殖过程中高产量与污染相矛盾的重要手段[6]。在不同的养殖方式下,养殖池塘底泥、水体和养殖动物肠道中的微生物均有所不同,从而表现出多样性,微生物群落结构的多样性同样会影响着不同养殖条件下水体理化环境因子和养殖效果。因此,从底泥微生物群落结构角度出发,探讨分析不同养殖方式底泥微生物群落结构的特点及其与水体的理化环境的关系可以为开发生态养殖技术提供理论支持,也能够为探讨不同养殖模式之间的优劣提供一个依据,具有重要生态和经济意义。目前,草鱼池塘养殖中常见的模式有鱼菜共生模式[6-7]、山塘模式[8]和传统精养模式,本实验比较了具有代表性的两种草鱼生态养殖模式——鱼菜共生模式和山塘模式与传统精养模式条件下池塘底泥的微生物群落结构的四季变化,并分析微生物群落结构与水体环境因子的关系,旨在为评估和判断草鱼养殖环境的变化情况,发展草鱼生态养殖提供理论支持。
1 材料与方法
1.1 养殖模式构建
在江西省南昌县蒋巷镇三洞村选取一个面积为30 000 m2的草鱼养殖塘,改造成3个面积均为10 000 m2,深度为2.5 m的标准养殖塘。选取其中一个塘采用山塘模式养殖(投喂草,投喂率10%),另一个塘采用鱼菜共生模式养殖(使用PVC管构建浮床,种植水空心菜,种植面积占水面10%,投喂配合饲料,投喂率3%~4%),剩余一个塘采用传统精养模式(投喂配合饲料,投喂率3%~4%)。其中山塘放养草鱼3 740尾/ 10 000 m2,规格为16~24尾/kg,精养池塘和鱼菜共生池塘均放养草鱼12 000尾/1 0000 m2,规格为16~24尾/kg。放养时间均在2018年12月,水深常年保持在2~2.2 m。
1.2 样品采集与预处理
1.1.1 底泥样品采集与预处理
三种养殖模式条件下样品的编号情况见表1。每次采样用彼得森采泥器在池塘四个角离岸边25 m处和池塘中点采集表层底泥样品各50 g,然后混匀成一个样并编号,每个池塘按季度采四次。采集的底泥暂时保存于干冰采样箱中,24 h内带回实验室,冻存于-80 ℃冰箱中。
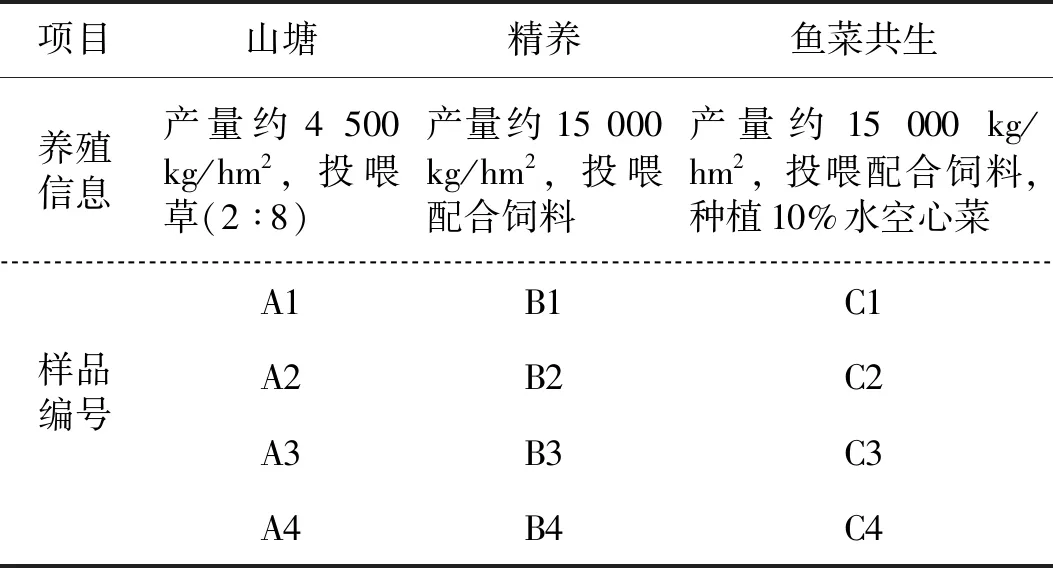
表1 养殖信息及样品编号Tab.1 Information of culture models and sample number
1.1.2 水样采集与处理

1.2 底泥样品DNA 提取、扩增和测序
将每季度混合后的底泥样品充分混合后称取2 g用土壤 DNA 提取试剂盒(OMEGA Soil DNA Kit,D5625)提取。16S rRNA基因V3-V4区扩增引物为:341F(5′-CCTACGGRRBGCASCAGKVRVGAAT-3′) 和 805R(5′-GGACTACNVGGGTWTCTAATCC-3′)。PCR产物使用2%浓度的琼脂糖凝胶进行检测后,利用凝胶回收试剂盒(QIAGEN)对目的条带进行回收。然后使用TruSeq®DNA PCR-Free Sample Preparation Kit 建库试剂盒(Illumina)进行文库构建,构建好的文库经过 Qubit 和 Q-PCR 定量,使用 HiSeq2500(诺禾致源公司,北京)进行测序。
1.3 数据处理与分析
1.3.1 序列处理
参照陈琼等[9]的方法采用Miseq测序平台得到原始数据(raw data),然后进行拼接和质控,得到有效数据(clean data),再进行嵌合体过滤,得到可用于后续分析的有效数据(effective tags)。 然后基于有效数据进行 OTUs(operational taxonomic units)聚类。
1.3.2 统计分析
水质数据采用Excel 2010处理数据,用SPSS19.0进行方差分析和相关性分析。
序列数据利用 Uparse 软件对所有样品的全部effective tags进行聚类,以97% 的一致性将序列聚类成OTUs。根据OTUs聚类结果对每个OTU的代表序列做物种注释,得到对应的物种信息,根据物种注释结果,选取每个样品在门和属2个分类级别上最大丰度排名前10的生成相对丰度柱形累加图。对所有样品的OTUs进行丰度分析、Alpha多样性分析等。使用Qiime 软件(Version 1.7.0)计算Alpha多样性指数,包括丰富度指数(Richness)、ACE 指数(ACE Index)和Chao1 指数(Chaos1 Index)。不同模式间进行UPGMA(unweighted pair-group method with arithmetic means)聚类分析和Anosim(analysis of similarities)分析。
2 结果
2.1 水体环境因子

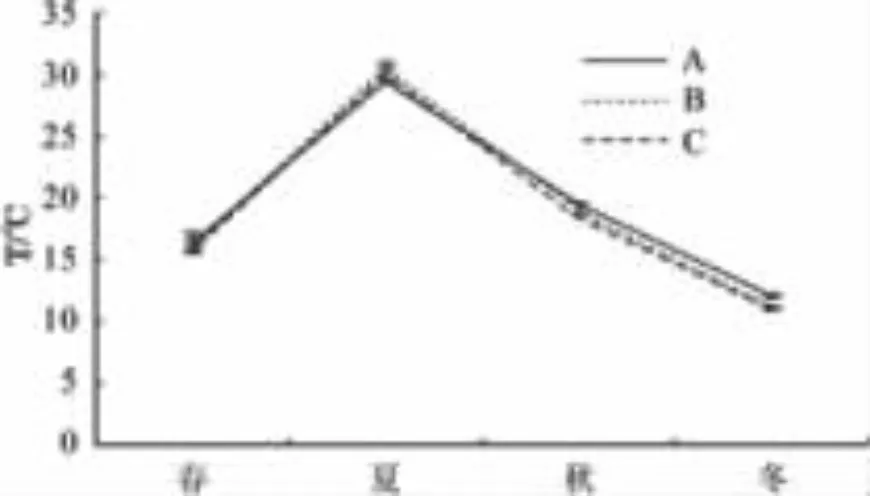
图1 水温变化情况Fig.1 Changes of water temperature in four seasons
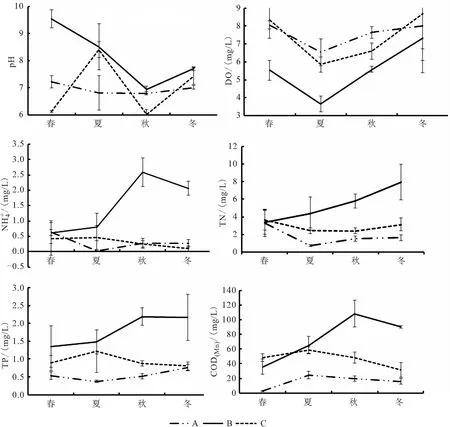
图2 pH、溶解氧、氨氮、总氮、总磷和CODMn四季变化情况Fig.2 Changes of and CODMn in four seasons
2.2 高通量测序数据
测序得到的序列经过拼接和质控,然后过滤掉嵌合体后得到Effective Tags条数为54 775~68 965。去除无法聚类到OTUs和没有注释的序列条数后得到可构建OTUs的序列(taxon tags)条数为48 067~63 071(表2)。以97%的一致性,将每个样品所得的序列聚类成 OTUs(表2)。
2.3 微生物群落结构
2.3.1 物种相对丰度
样品中相对丰度最高的10个OTUs在门水平上分布见图3,可以看到三种养殖模式条件下四季底泥样品中丰度最高的均是变形菌门(Proteobacteria)。但A组变形菌门占比四季是下降趋势;其余占比超过10%的门是拟杆菌门(Bacteroidetes)和酸杆菌门(Acidobacteria),其中酸杆菌门四季占比逐渐升高。B组变形菌门占比先降后上升;拟杆菌门和酸杆菌门占比均超过10%,而且拟杆菌门占比逐渐下降和酸杆菌门占比逐渐上升;绿弯菌门(Chloroflexi)占比也超过10%。C组除了变形菌门外,拟杆菌门占比也超过10%;但绿弯菌门、酸杆菌门、硝棘菌门(Nitrospinae)、螺旋体门(Spirochaetes)等占比均比较高;而且每个门占比四季存在一定的波动。
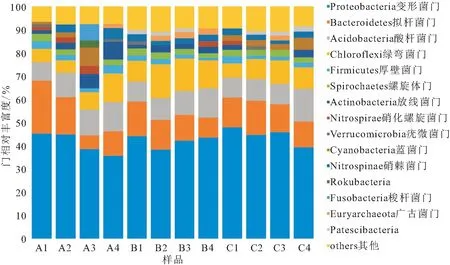
图3 样品在门水平上物种相对丰度Fig.3 Relative abundance of samples at phylum level
2.3.2 核心群落相对丰度
样品在属水平上相对丰度最高的10个OTUs为核心群落,可以反映群落物种的基本结构情况。从图3和表3中可以明显看到三组中没有鉴定的种类在四季中占比均最高,有未培养(uncultured)、未培养细菌(uncultured-bacterium)、未培养土壤细菌(uncultured-soil-bacterium)和未培养原核生物(uncultured-prokaryote)四种。其中地杆菌属(Geobacter)和厌氧粘细菌属(Anaeromyxobacter)在三组中四季均有,其余种属A组与B组和C组相差较大,而且A组的种属四季差异较大。B组和C组重要种属相似,且四季差异较小。具体详见表3。
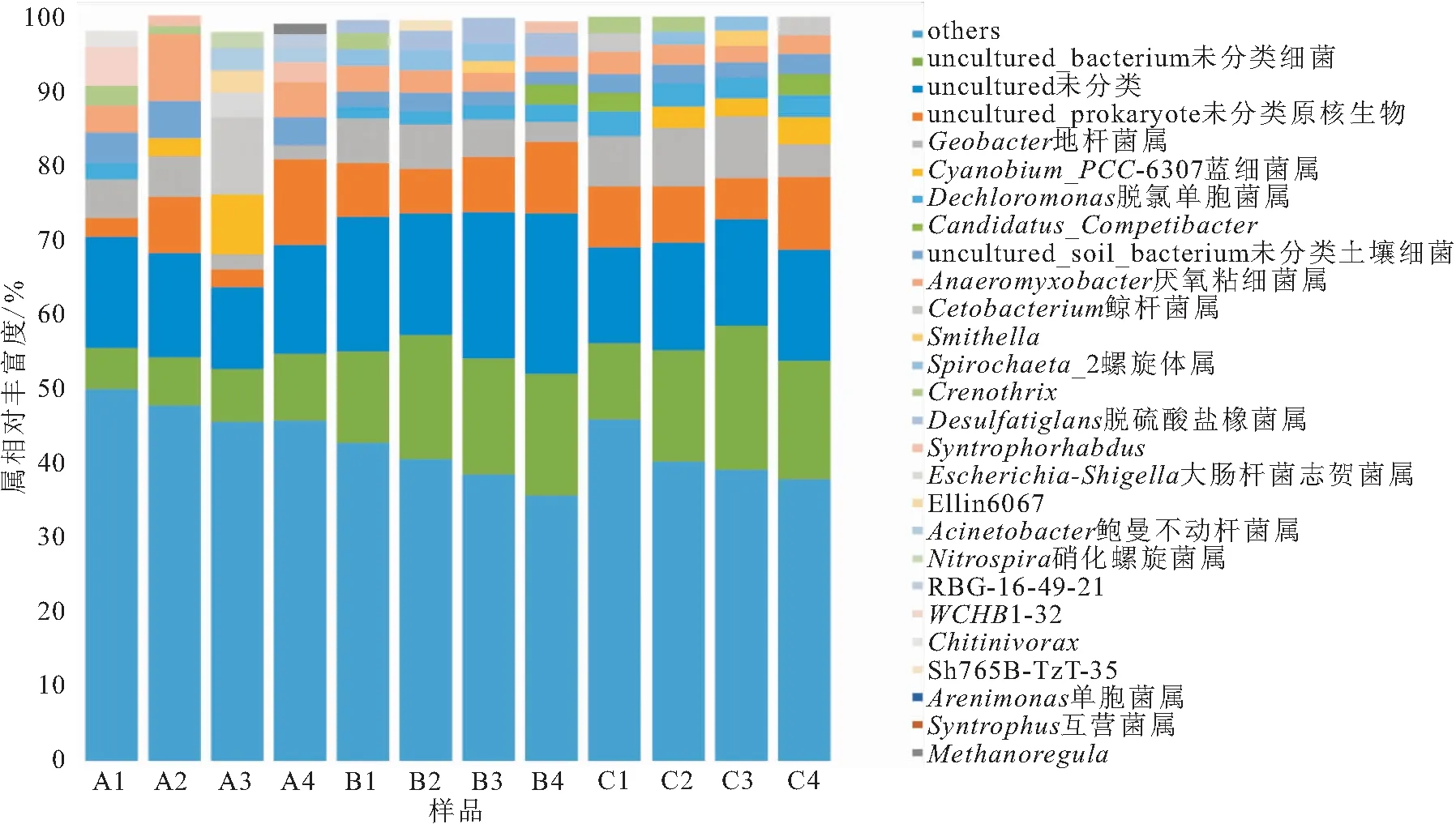
图4 样品在属水平上相对丰度Fig.4 Relative abundance of samples at genus level
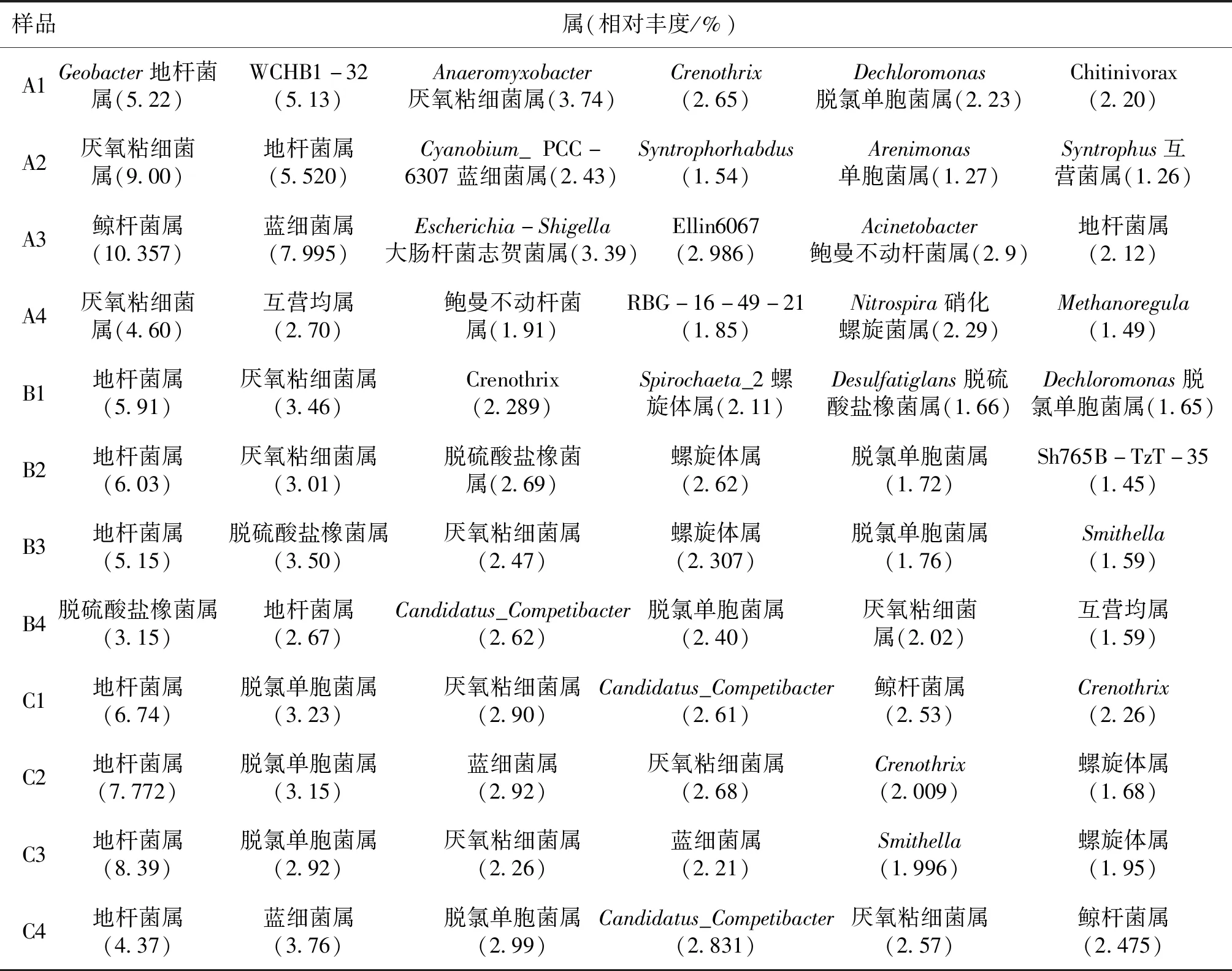
表3 在属水平丰度最高的10个OTUSTab.3 Top ten OTUs with the highest abundance in genus level
2.3.3 微生物群落结构聚类分析
UPGMA聚类分析结果(图5)可知,各样品在时间上的相似性(不同季度)的相似性高于空间上(不同养殖方式)。具体表现为B组和C组相似性接近,而A组与B组和C组相似性低;B组和C组均是夏季和秋季相似度最高,A组是春夏两季相似度高。
对A、B、C三组样品均作了Anosim差异性分析,从图6可以看到A组与C组的组间差异大于A组和B组以及B组和C组,组间差异最小为B组和C组。A、B、C三组的组间差异均显著高于组内差异。
2.4 多样性分析
三组多样性方差分析结果见图7,从图7可以看到A组ACE指数显著低于B组和C组,B组和C组间差异不显著,但Shannon指数三组差异不显著。从表4中可以看到三种养殖模式条件下丰富度指数ACE各季度差异也大。其中A组四季丰富度指数变化差异大,其次是B组,C组丰富度指数四季最稳定;除A组外,B组和C组丰富度均是先增后降的趋势。Chao1指数的变化情况与ACE类似。多样性指数Shannon A组冬季多样性高于B组和C组,但夏季和秋季低于B组和C组,C组除了冬季外其他时间多样性均小于B组;各组内四季也存在一定的变化;A组除了秋季外,其余三个季度均比较稳定; B组和C组类似,均是夏季> 春季>秋季>冬季。
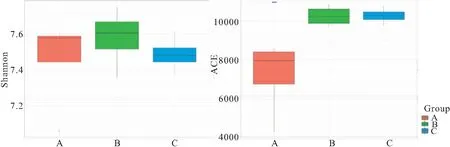
图7 ACE指数和Shannon指数差异分析图Fig.7 Differentiation analysis of richness(ACE) and Shannon indices in groups***表示差异极显著
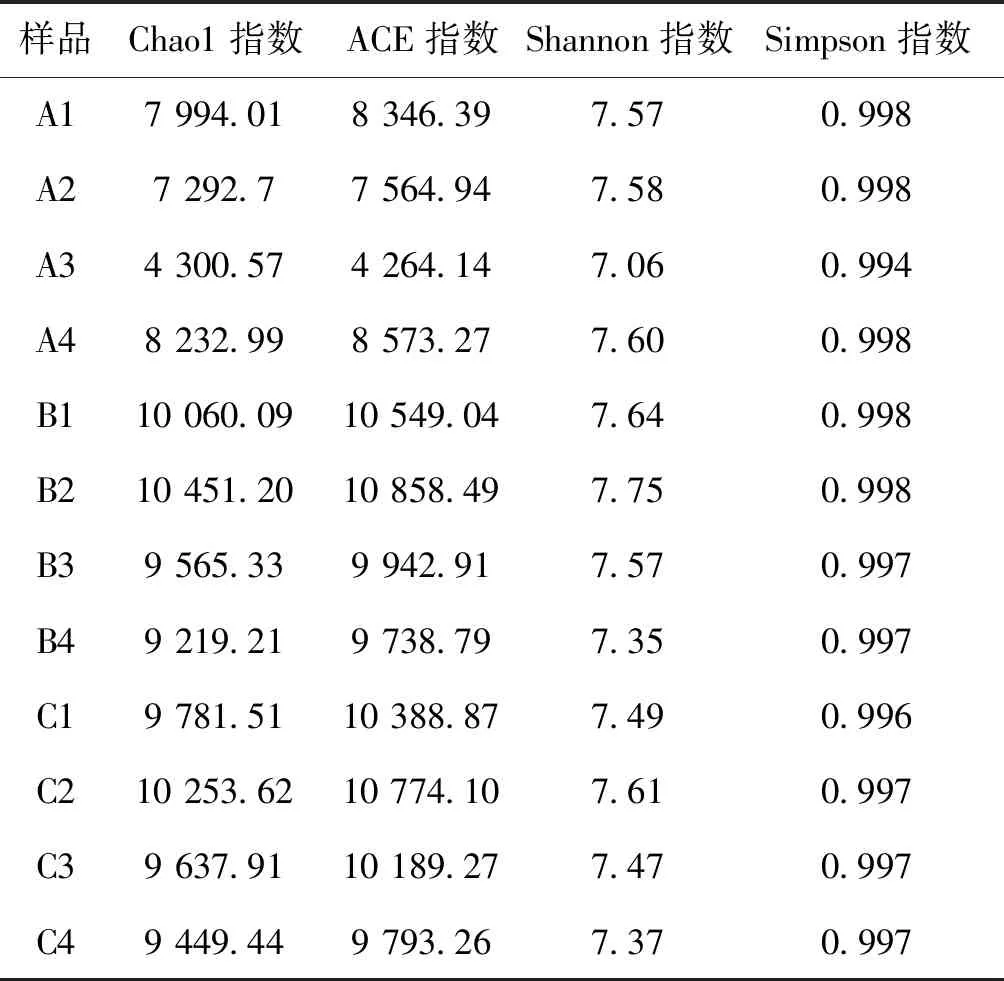
表4 基于OTUs的多样性指数的 Alpha 多样性分析Tab.4 Alpha diversity of bacteria samples associated with the number of observed OTUs
2.5 环境因子与微生物群落多样性相关性分析
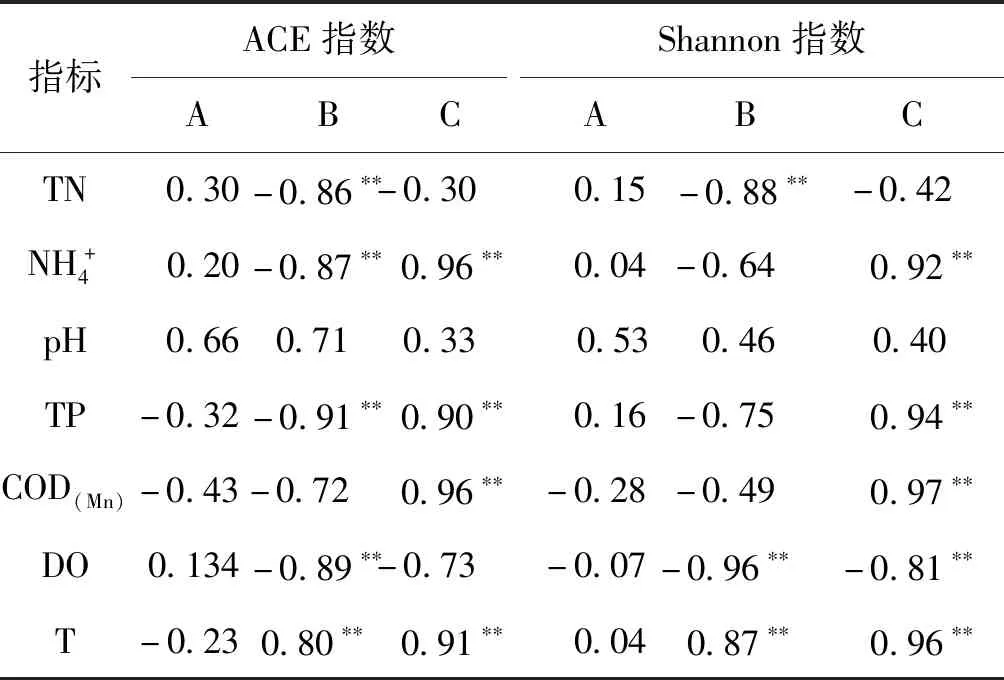
表5 环境因子与多样性相关性分析Tab.5 Correlation analysis between environmental factors and the microbial community diversity
3 讨论
3.1 三种养殖模式条件下底泥微生物群落结构多样性分析
3.2 三种养殖模式条件下底泥微生物核心群落
底泥微生物核心菌群的种类和分布情况可以一定程度上反应底泥微生态情况,比较三种养殖模式各季度微生物群落在属水平的分布情况可以发现,A组主要属在丰度上和种类上四季均有很大差别。春夏季以进行还原作用的厌氧或兼性厌氧菌属的地杆菌属、WCHB1-32属、厌氧粘细菌属、Crenothrix属和脱氯单胞菌属等[11、13-18]为主。而秋季则出现了进行亚消化作用的硝化螺旋菌属[19-21]和能够在好氧条件下进行固氮,厌氧条件下还原硫的蓝细菌属[22]。同时鲸杆菌属、大肠杆菌志贺菌属、鲍曼不动杆菌属的丰度占比也进入了前10。其中鲸杆菌属的种类通常寄生于水产动物肠道,参与促进和合成蛋白质及碳水化合物和脂肪的代谢[23、24];大肠杆菌志贺菌属和鲍曼不动杆菌属均是环境中的条件致病菌,而鲍曼不动杆菌属中菌能够引起水产动物疾病[25-26]。冬季则是以厌氧粘细菌属、Syntrophorhabdus、地杆菌属等和Methanoregula等占主要地位。说明A组底泥微生物核心菌群结构稳定性差。与之相比,B组虽然主要种属的丰富度在四季存在一定的变化,但种类变化较小,主要以地杆菌属、厌氧粘细菌属、Crenothrix、螺旋体属、脱硫酸盐橡菌属等为主。其中的地杆菌属[11、13]、厌氧粘细菌属[15]、Crenothrix属[18]、脱硫酸盐橡菌属[27]均属于厌氧或兼性厌氧菌,在厌氧条件下还原有机物。说明B组底部溶氧不足和有机物沉积过量,因为有机物的积累能刺激地杆菌属和厌氧年细菌属等菌属的大量富集[14]。对水体理化检测的数据也支持这一结论(DO最低、COD(Mn)及三氮均最高)。C组春季丰度较高的是地杆菌属、脱氯单胞菌属、厌氧粘细菌属和Candidatus-Competibacter属等还原有机物,脱硫及除磷作用的菌属,到夏季开始出现蓝细菌属和Crenothrix,且丰富度上升,Candidatus-Competibacter属丰富度也上升。因为蓝细菌属具有能够在好氧条件下进行固氮,厌氧条件下还原硫的作用[22],Crenothrix[18]具有硝化作用,所以C组池塘夏季底泥中除磷、固氮和硝化菌种类和数量均升高,促使水体COD(Mn)、总磷、总氮和氨氮等理化指标均呈现下降的趋势。同时鲸杆菌属也是C组系统中的主要菌属,鲸杆菌属具有参与促进和合成蛋白质及碳水化合物和脂肪的代谢的作用[23、24],是对养殖水产动物有益的肠道菌群。所以,从核心菌群功能结构、微生物的多样性关系,以及参照水体理化指标来分析,均可以表明鱼菜共生系统底泥菌群结构更为优化。
3.2 水体理化环境对底泥微生物群落结构的影响

4 结论
山塘模式虽然水体DO相对较高,水体理化环境最好(TN、TP等最低),但菌群结构不稳定、四季波动大,加上养殖密度低、产量也低,也存在能引起鱼类病害的条件致病菌的隐患,所以其没有达到充分提高养殖效益的目的。鱼菜共生模式虽然产量与精养模式一样,但水体有机物低于精养模式,水生植物能够吸收无机盐类,提供溶氧,而且底泥微生物群落结构稳定,水环境因子能够促进底泥微生物的生长和繁殖,能够稳定持续的净化水体环境,是一种能够保证产量且提高养殖效益的养殖方式。

