MAL2在乳腺癌中的差异表达及预后分析
何东宁, 周浩东, 刘畅, 谭琦, 谷泽慧, 王亚帝
(1.锦州医科大学 附属第一医院 肿瘤内科,辽宁 锦州 121000; 2.锦州医科大学 附属第三医院 精准医学中心,辽宁 锦州 121000)
乳腺癌(breast cancer, BC)是世界范围内女性最常见的恶性肿瘤之一.现有的流行病学数据显示,乳腺癌的发病率正逐年增加.据统计,2018年全球新诊断的乳腺癌患者估计为268 670人,死亡人数估计为41 400人[1-2].乳腺癌的治疗方法很多,且近年来手术、内分泌治疗、抗HER-2(human epithelial growth factor receptor-2, HER-2)靶向治疗方面均有较大进展,但整体预后仍不乐观,原因在于目前对乳腺癌的发生和发展尚未完全了解.因此,对乳腺癌分子机制的阐明、新预后标志物的寻找和潜在治疗靶点的探索,仍是乳腺癌的研究方向和重点.
T细胞分化蛋白2(mal T-cell differentiation protein 2, MAL2)是MAL蛋白脂质家族的成员,是176个氨基酸残基构成的4次跨膜蛋白.正常状态下,MAL2主要参与胞吞并介导细胞间的物质转运,作用于递送膜结合蛋白的细胞内传输途径,在细胞转移中起重要作用[3-4].前期许多研究表明,MAL2在大肠癌[5]、胰腺癌[6]、卵巢癌[7]、肝癌[8]、乳腺癌[9-10]、脑膜瘤[11]、甲状腺癌[12]及口腔鳞癌[13]中呈过表达,且与转移及预后不良密切相关,但均未对MAL2基因作用机制及其与肿瘤病理特征的相关性进行深入研究.本研究通过生物信息学的方法,分析MAL2在乳腺癌中的表达及其与肿瘤病理特征和预后的关系,以期为乳腺癌的预后评估及发生发展机制的研究提供新的思路.
1 资料与方法
1.1 HPA数据库检索
HPA(The human protein atlas)是基于 RNA 测序分析和免疫组化分析的大型转录组和蛋白组数据库[14](https: //www.proteinatlas.org/),可用于正常组织和肿瘤组织蛋白的差异表达分析.方法为进入网站并在搜索框中输入MAL2,点击search;限定条件为①Gene:gene description、protein class;②RNA:RNA tissue category、RNA Tissue specificity score、RNA FPKM tissue specific;③Organ:breast and female reproductive system.
1.2 Oncomine数据库检索
通过Oncomine数据库(http://www.oncomine.org)进行不同肿瘤间的差异性表达分析.检索条件设定为Gene:MAL2.对 Oncomine数据库中所有与乳腺癌及MAL2基因相关的数据进行荟萃分析,观察两者的相关性.
1.3 Ualcan 数据库检索
Ualcan(http://ualcan.path.uab.edu/)数据库[15]包含来自31种癌症类型的 TCGA3 级 RNA-seq和临床数据,可提供对公开的癌症组学数据的方便访问,包括the Cancer Genome Atlas (TCGA)和MET 500队列(从500个转移瘤病人中获得了转移癌组织中基因组序列和免疫应答的详细数据);允许识别生物标记物或对潜在的感兴趣基因进行电子验证;提供基于基因表达的描述基因表达和患者生存信息的图表;评估乳腺癌和前列腺癌分子亚型中的基因表达,通过启动子甲基化评估基因表达的表观遗传调控;通过连接HPRD、GeneCards、Pubmed、TargetScan、DRUGBANK、Open Targets和GTEx进行泛癌基因表达分析.方法为进入网站后选择Analysis选项,使用Scan by genes检索框,检索条件为①Enter Gene symbol: MAL2;②TCGA dataset:Breast invasive carcinoma;③Explore.
1.4 GEPIA数据库检索
GEPIA 数据库[16](http://gepia.cancer-pku.cn/)是由北京大学张泽民教授团队开发的一种交互式网络服务器,用于分析来自 TCGA 和GTEx项目的9 736个肿瘤和8 587个正常样本的 RNA 测序表达数据,将TCGA 和GTEx数据进行整合,进行全面的表达分析.GEPIA提供网页式交互展示和快速定制功能,包括肿瘤/正常差异表达谱分析、剖面绘制、根据肿瘤类型或病理分期进行分析、患者生存分析、相似基因检测、相关性分析和降维分析等分析模块.方法为进入网站后选择single gene analysis,输入MAL2,选择survival analysis.限定条件为①Methods:overall survival;②Group cutoff:median;③Hazards Radio(HR): Yes;④95% Confidence Interval:Yes;⑤Axis Units:Months;⑥Datasets Selection:BRCA.
1.5 STRING网络分析
STRING version 11.0数据库(https://string-db.org/cgi/input.pl)是分析、预测蛋白质与蛋白质相互调控网络的平台,通过String数据库预测MAL2蛋白的上下游调控蛋白及蛋白间的调控关系.检索条件为①Protein Name:MAL2;②Organism:Homo sapiens;③选择“More”;④点击“Analysis”.
1.6 数据处理
参考林晏廷等[17]的数据处理方法,运用配对t检验进行配对样本的组间比较,采用one-way ANOVA 分析MAL2mRNA 表达与乳腺癌临床病理分期的关系,MAL2基因表达与预后的关系,采用Kaplan-Meier模型进行生存分析.所有数据采用在线统计学分析,P<0.05被认为差异有统计学意义.
2 结果
2.1 与乳腺癌预后相关的基因
运用 HPA 病理学图集中的癌症模块进行分析,获得如下信息:与乳腺癌预后相关的基因共有577个,其中与预后不良相关的基因有210个,与预后良好相关的基因有367个(图1).表1列举了与乳腺癌预后不良相关的前10位基因,其中与乳腺癌细胞转移相关的MAL2基因列于此表,可用于后续分析.
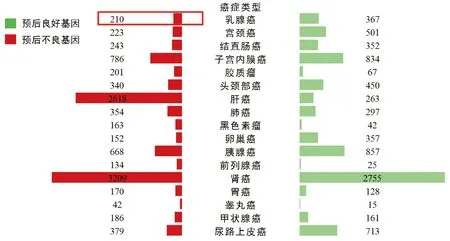
图1 HPA中与乳腺癌预后相关的基因表达情况Fig.1 Expression of genes associated with breast cancer prognosis in HPA表1 与乳腺癌预后不良相关的10个基因Table 1 10 genes associated with poor prognosis of breast cancer

基因简称基因全称预测位置mRNA相对表达量P值TAGLN2转运蛋白2细胞内318.42.06e-5ATP5F1BATP合酶F1亚基beta细胞内236.97.17e-6PTGES3前列腺素E合酶3细胞内102.02.15e-5MAL2Mal, T细胞分化蛋白(基因/假基因)细胞膜98.04.57e-6PGK1磷酸甘油酸激酶1细胞内86.57.05e-8DCTPP1dCTP焦磷酸酶1细胞内36.31.64e-5RAB5BRAS癌基因家族成员细胞内35.92.24e-5TPD52肿瘤蛋白D52细胞内35.61.11e-5PCMT1蛋白质L-异天冬氨酸(D-天冬氨酸)O-甲基转移酶细胞内,细胞膜30.94.02e-7TIMM17A线粒体内膜17A细胞膜23.61.98e-5
2.2 MAL2在BC组织和其他肿瘤组织中的表达
采用Oncomine 数据库分析MAL2在不同肿瘤类型中的差异性表达,检索到MAL2基因相关的癌组织/正常组织差异表达分析结果共308个,其中,MAL2在癌组织中呈显著高表达的有13个结果,呈低表达的有12个结果.其中10个分析结果均显示MAL2基因在乳腺癌组织中呈高表达(图2).
2.3 MAL2基因在BC组织中表达的荟萃分析
在Oncomine数据库中对描述BC的相关研究进行分析,共有10项符合条件的研究,分别对其进行对比分析发现,BC组织中MAL2表达均高于正常对照组织.为进一步明确BC和MAL2表达的相关性,对以上10项研究进行荟萃分析,结果发现,BC组织中MAL2表达水平明显高于正常组织(P<0.05)(图3).
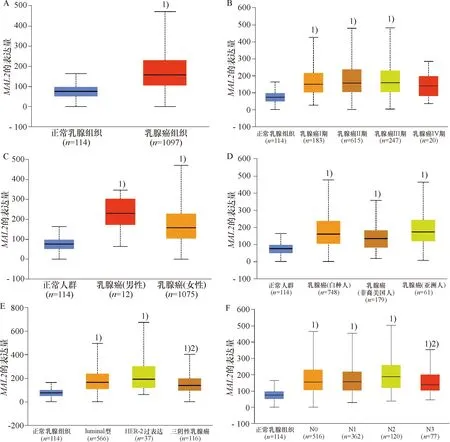
2.4 MAL2 mRNA表达与BC临床病理特征的关系
通过 Ualcan 分析 TCGA 数据库中MAL2在114例正常组织和1 097例BC组织中的差异表达情况.BC组织中MAL2表达高于正常乳腺组织(P<0.05,图4A),不同分期、性别及种族的BC之间MAL2表达无差别(图4B、4C和4D);不同BC分子分型MAL2表达均高于正常乳腺组织,其中Luminal型MAL2表达高于三阴性乳腺癌型(P<0.05,图4E);N2淋巴结转移者MAL2表达高于N3组(P<0.05,图4F);不同激素水平的BC患者MAL2表达无差别(图4G);41~60岁患者组MAL2表达低于81~100岁患者组(P<0.05,图4H);从病理类型方面分析,浸润性导管癌组MAL2表达高于浸润性小叶癌、粘液癌及化

颜色深浅与MAL2基因差异表达排序有关,红色块代表MAL2基因在所有高表达的基因中位于前1%、前5%、前10%; 蓝色块代表MAL2基因在所有低表达的基因中位于前1%、前5%、前10%;白色块代表MAL2基因在所有差异表达的基因中超出前10%.色块上的数字:代表共有几个研究支持此结果.泛癌:一项研究中包含3种以上的癌肿.离群值:基于肿瘤异质性进行的分析,只在肿瘤的某些亚型或特定群体中的异常表达.图2 Oncomine 数据库中MAL2在不同肿瘤中差异性表达Fig.2 Differential expression of MAL2 in different tumors in Oncomine database
生癌组(P<0.05,图4I);在乳腺癌分子亚型中,三阴性乳腺癌的基底样1型MAL2表达高于间充质干细胞型、间充质细胞型及不稳定亚型组(P<0.05),HER-2过表达型MAL2表达高于间充质干细胞型(P<0.05,图4J).

图3 Oncomine数据库中MAL2在不同病理类型乳腺癌与正常组织中的差异性表达Fig.3 Differential expression of MAL2 in different pathological types of breast cancer and normal tissues in Oncomine database
(续上图)
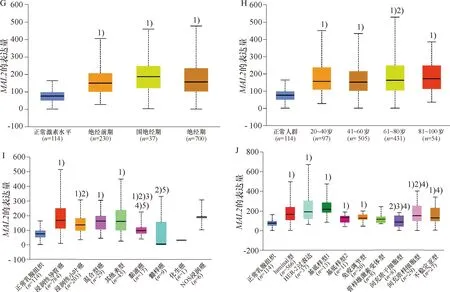
A:乳腺癌组织和正常组织中MAL2的差异性表达;1)与正常乳腺组织组比较,P<0.05;B:不同分期乳腺癌中MAL2的差异性表达;1)与正常乳腺组织组比较,P<0.05;C:不同性别乳腺癌中MAL2的差异性表达;1)表示与正常人群组比较,P<0.05;D:不同种族乳腺癌中MAL2的差异性表达;1)与正常人群组比较,P<0.05;E:不同分子分型乳腺癌中MAL2的差异性表达;1)与正常乳腺组织组比较,P<0.05;2)与Luminal组比较,P<0.05;F:不同淋巴结转移乳腺癌中MAL2的差异性表达;1)与正常乳腺组织组比较,P<0.05;2)与N2组比较,P<0.05;N0:无区域淋巴结转移;N1:1~3个腋窝淋巴结转移;N2:4~9个腋窝淋巴结转移;N3:10个或更多腋窝淋巴结转移;G:不同激素水平乳腺癌中MAL2的差异性表达;1)与正常激素水平组比较,P<0.05;H:不同年龄乳腺癌中MAL2的差异性表达;1)与正常人群组比较,P<0.05;2)与41~60岁组比较,P<0.05;I:不同病理类型乳腺癌中MAL2的差异性表达;1)与正常乳腺组织组比较,P<0.05;2)与浸润性导管癌组比较,P<0.05;3)与浸润性小叶癌组比较,P<0.05;4)与混合型癌组比较,P<0.05;5)与其他类型乳腺癌组比较,P<0.05;J:不同乳腺癌分子分型与TNBC亚型中MAL2的差异性表达.1)与正常乳腺组织组比较,P<0.05;2)与Luminal组比较,P<0.05;3)与HER-2过表达组比较,P<0.05;4)与基底样型1组比较,P<0.05.A: Differential expression of MAL2 in breast cancer tissues and normal tissues;1) Compared with normal breast tissue, P<0.05;B: Differential expression of MAL2 in breast cancers of different stages; 1) Compared with normal group, P<0.05;C: Differential expression of MAL2 in breast cancers of different genders;1) Compared with normal group, P<0.05; D: Differential expression of MAL2 in breast cancer of different races; 1) Compared with normal group, P<0.05;E: Differential expression of MAL2 in breast cancer of different molecular types; 1) Compared with the normal group, P<0.05; 2) Compared with the Luminal group, P<0.05;F: Differential expression of MAL2 in breast cancer with different lymph node metastases; 1) Compared with the normal group, P<0.05; 2) Compared with N2 group, P<0.05; N0: no regional lymph node metastasis; N1: 1-3 axillary lymph node metastases; N2: 4-9 axillary lymph node metastases; N3: 10 or more axillary lymph node metastases;G: Differential expression of MAL2 in breast cancer with different hormone levels; 1) Compared with normal group, P<0.05;H: Differential expression of MAL2 in breast cancer of different ages; 1) Compared with normal group, P<0.05; 2) Compared with 41~60 years old group, P<0.05;I: Differential expression of MAL2 in breast cancer of different pathological types; 1) Compared with normal group, P<0.05; 2) Compared with invasive ductal carcinoma group, P<0.05; 3) Compared with invasive lobular carcinoma group, P<0.05; 4) Compared with mixed cancer group, P<0.05; 5) Compared with other types of breast cancer, P<0.05; J: Differential expression of MAL2 in different breast cancer molecular types and TNBC subtypes;1) Compared with normal group, P<0.05; 2) Compared with Luminal group, P<0.05; 3) Compared with HER-2 overexpression group, P<0.05; 4) Compared with basal-like type 1 group, P<0.05.图4 Ualcan分析TCGA数据库中乳腺癌MAL2 mRNA的表达Fig.4 MAL2 mRNA expression of breast cancer in TCGA database analysis by Ualcan
2.5 MAL2 mRNA的表达与BC预后的关系
进一步运用GEPIA数据库验证TCGA 数据库中BC组织MAL2的差异表达,发现BC中MAL2表达高于正常乳腺组织(P<0.05,图5).运用GEPIA数据库生存分析模块分析MAL2的差异表达及MAL2与BC患者预后的关系,结果发现MAL2低表达及MAL2高表达者各535例.与MAL2高表达组相比,MAL2低表达组的总生存期延长(P=0.000 32,图6),MAL2差异表达对无疾病进展生存期无明显影响(P=0.18,图7).
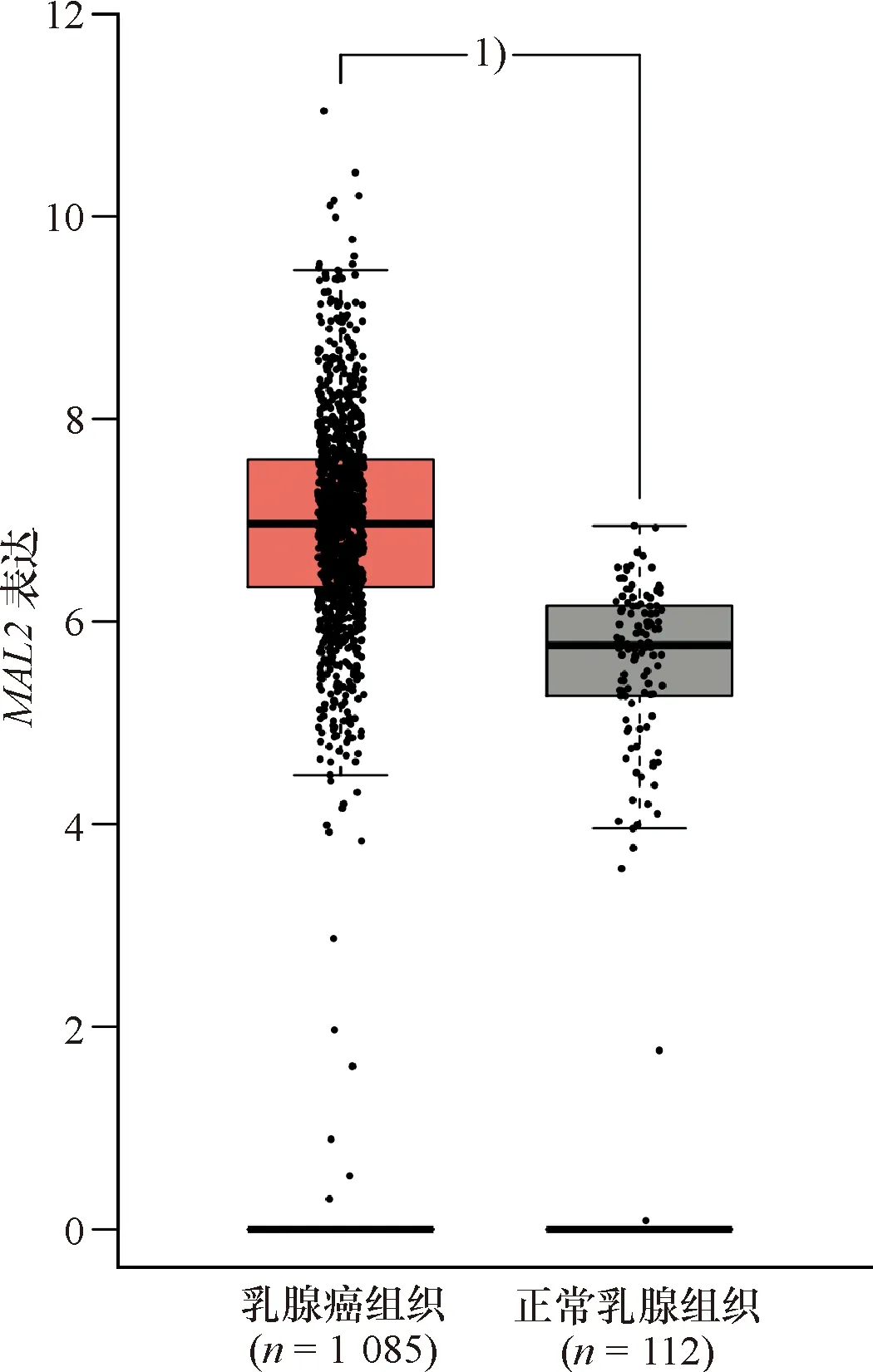
1)与正常乳腺组织比较,P<0.05
2.6 MAL2与上下游蛋白的相互作用
在STRING网站中,分析MAL2与上下游蛋白的关系,可获得相应的关系网络图(图8).PPI富集P值(PPI enrichment p-value)为0.001 746,包含TPD52、TPD52L2、YIF1A、IGR、FRMD4B、SH3YL1、OCLN等与MAL2相互作用的节点数共有22个,其中TPD52、TPD52L2、OCLN与MAL2相互作用(score=0.93).MAL2主要参与补体和凝血级联、细胞间紧密连接、白细胞经内皮细胞迁移等生物学过程(表2、3).
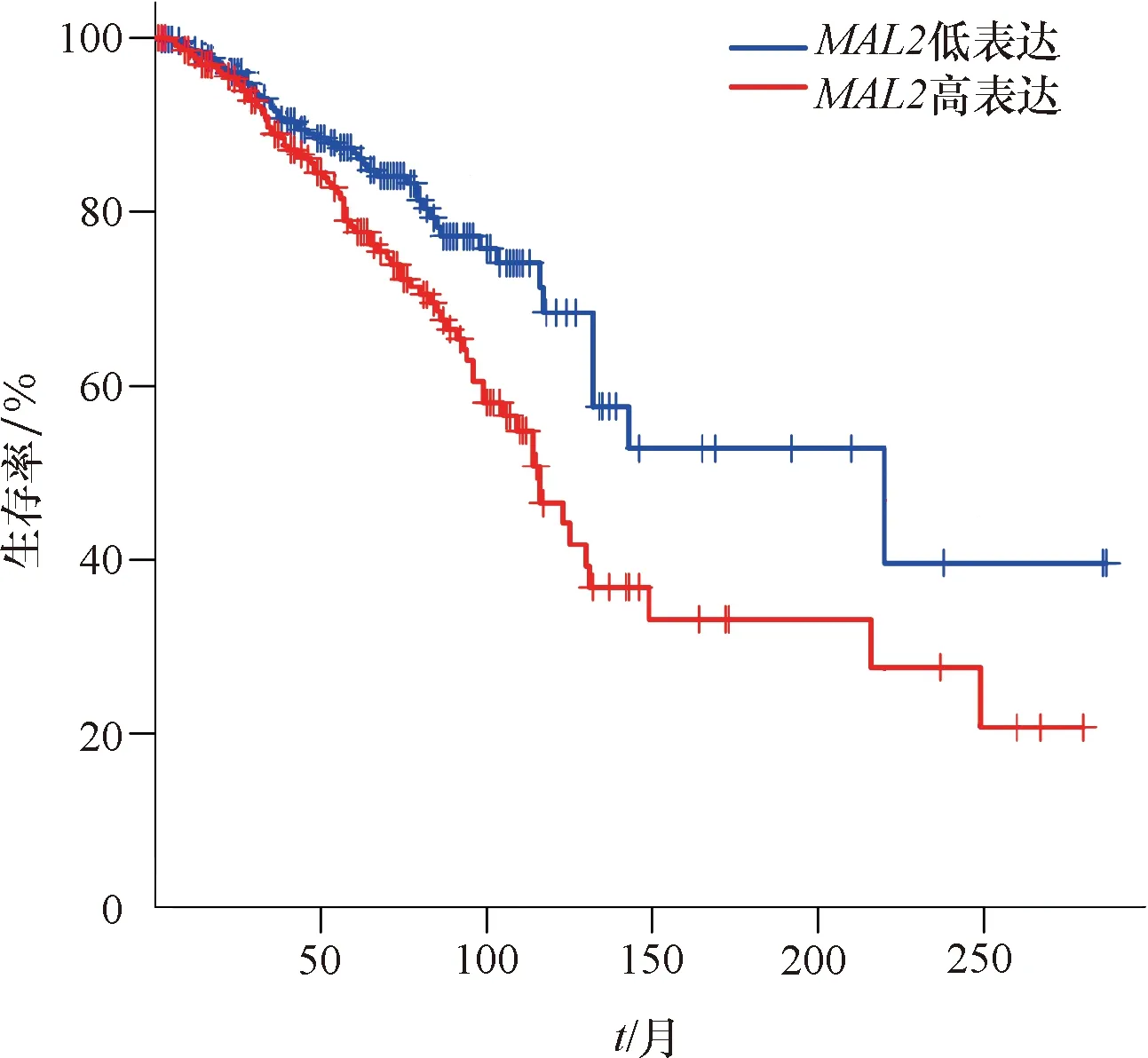
图6 GEPIA 数据库中不同MAL2 mRNA表达水平BC患者的总体生存率Fig.6 Overall survival rate of BC patients with different MAL2 mRNA expression in GEPIA database
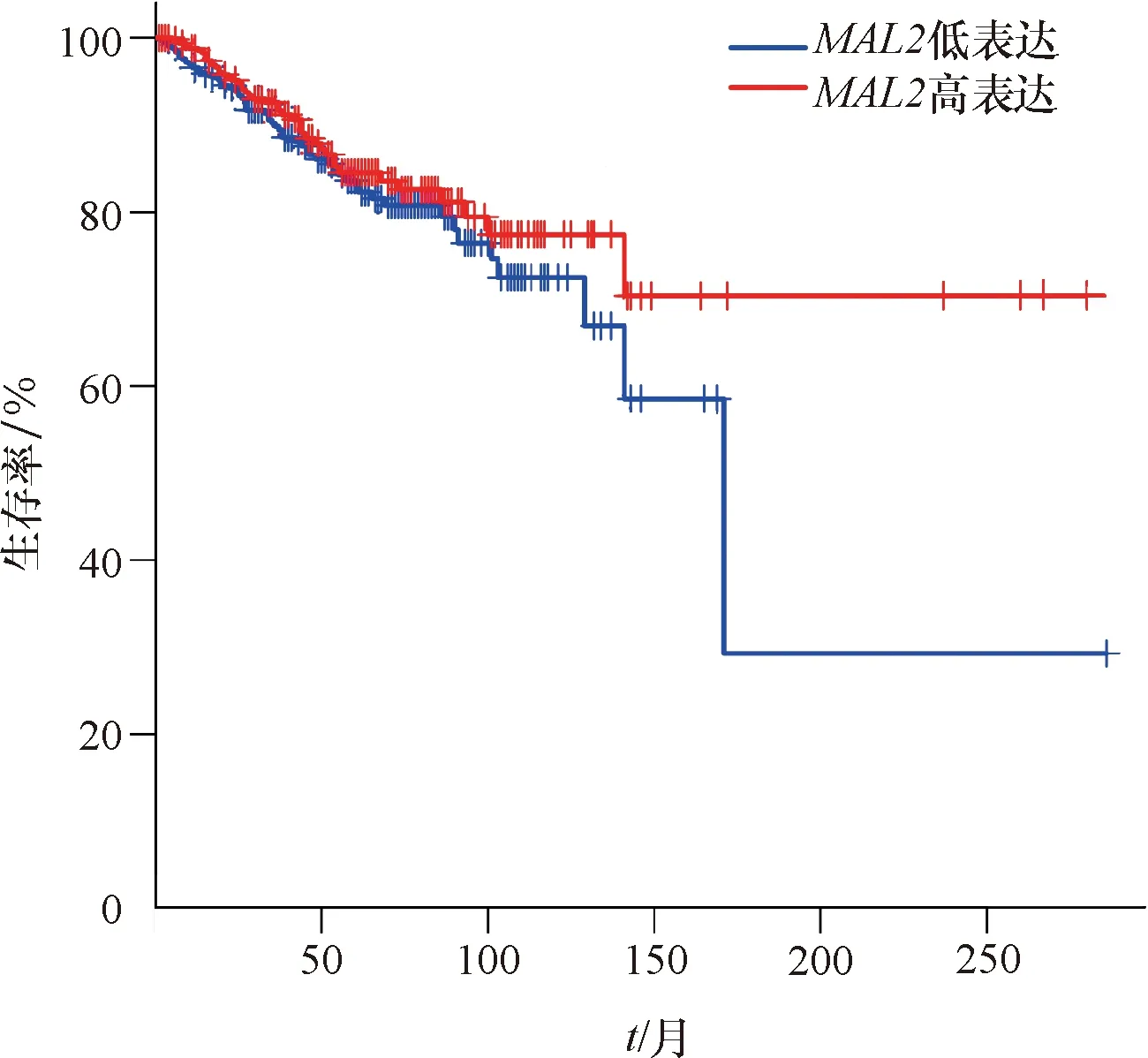
图7 GEPIA数据库中不同MAL2 mRNA表达BC患者的无病生存率Fig.7 Disease free survival rate of BC patients with different MAL2 mRNA expression in GEPIA database
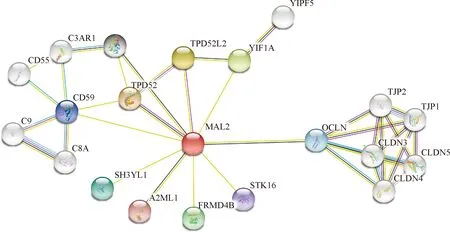
图8 MAL2蛋白交互作用网络图Fig.8 MAL2 protein-protein interaction network map

表2 MAL2生物学过程Top5Table 2 Top5 of MAL2 biological process

表3 MAL2 KEGG信号通路Top5Table 3 Top5 of MAL2 KEGG signaling pathway
3 讨论
目前多项研究已证实MAL2是一种四跨整合膜蛋白,位于染色体8q24,该位置的基因组不稳定性增加,MAL2的表达上调可高达100倍[18],这与多种人类癌症的发生有关,包括肾癌、恶性间皮瘤、神经母细胞瘤、胆管癌等.同时,MAL2的高表达也与结直肠癌或胰腺癌患者的不良预后有关[5-6].虽然MAL2的高表达可能不是癌症发生的驱动因素,但它可能通过改变蛋白质的靶向性导致蛋白质分布失调,从而促进肿瘤转化,最终导致细胞形态、信号转导和迁移能力的变化[19].因此,MAL2可能成为乳腺癌的生物学标志物和有效治疗靶点,这对乳腺癌患者具有重要的临床意义.
本研究通过对Oncomine数据库中的10项研究进行荟萃分析,发现MAL2mRNA在乳腺癌中呈显著高表达.以 GEPIA和Ualcan软件在线分析TCGA数据库中MAL2mRNA的差异性表达,结果与Oncomine 数据库一致,即MAL2mRNA在乳腺癌中高表达.应用Ualcan分析不同临床病理特征乳腺癌中的MAL2mRNA表达,发现乳腺癌组织MAL2mRNA的表达水平与患者年龄、分子分型、淋巴结转移情况及病理分型相关.上述结论说明,在乳腺癌中高表达的MAL2基因,可促进乳腺癌细胞的增殖、侵袭和转移,且与患者不良预后相关[5-6],据此推测MAL2可作为评估乳腺癌预后的重要因素.在进行GEPIA 生存分析时发现,MAL2高表达组总生存时间明显低于低表达组,MAL2表达越高,乳腺癌患者预后越差,这为MAL2作为乳腺癌患者的预后因子提供了依据.三阴性乳腺癌(triple negative breast cancer,TNBC)是乳腺癌的重要亚型,预后较差,可细分为多种分子亚型,但MAL2基因与TNBC分子分型是否相关尚无报道.经Ualcan对TCGA数据库分析发现,TNBC中基底样1型的MAL2表达高于间充质干细胞型、骨髓干细胞型及未特指型,HER-2过表达型MAL2表达高于间充质干细胞型.基底样1型的TNBC和HER-2过表达型的乳腺癌多存在组织学分级高、侵袭能力强的特点[20-21],MAL2在这两种分子分型中均呈高表达,原因可能是MAL2的多次跨膜可通过细胞内转运的协同作用,改变细胞表面蛋白的表达和肿瘤细胞的整体功能[19].为明确MAL2在乳腺癌相关蛋白网络中扮演何种角色及如何调控乳腺癌的恶性进展,Bhandari等[22]进行了相关研究,结果发现MAL2在乳腺癌中具有重要的生物学意义,其异常表达可预测乳腺癌的恶性进展,且MAL2对EMT也具有调节作用.此外,Zhong等[23]通过共表达分析发现了RRM2-let-7a-5p-SNHG16/MAL2信号网络及与乳腺癌预后相关的ceRNA子网络.本研究通过STRING分析发现包含TPD52、TPD52L2、YIF1A、IGR、FRMD4B、SH3YL1、OCLN等与MAL2相互作用节点数共有22个,其中TPD52、TPD52L2、OCLN与MAL2相互作用明显.KEGG及GO分析发现MAL2主要参与补体和凝血级联、细胞间紧密连接、白细胞经内皮细胞迁移等生物学过程,这为MAL2表达调控乳腺癌发展的研究提供了新思路.
本研究采用不同数据库和在线分析工具分析了MAL2在乳腺癌中的差异表达,以及和乳腺癌预后的相关性.分析发现,MAL2在乳腺癌中的表达高于正常乳腺组织,与乳腺癌的不良预后密切相关,且参与乳腺癌恶性发展的调控过程.为验证MAL2可作为潜在的乳腺癌预后因子以及其对乳腺癌的恶性发展具有调控作用,还需进行更多临床及基础机制方面的探索性研究.综上,本研究认为MAL2基因在乳腺癌的发生发展中具有进一步研究的价值,这也为乳腺癌的预后评估及治疗提供了新的思路.

