候选门级辐射类群(candidate phyla radiation)细菌的生理生态与进化
蒋建东,纪彦晗,王保战
(南京农业大学生命科学学院微生物学系 农业农村部农业环境微生物重点实验室,江苏 南京 210095)
候选门级辐射类群(candidate phyla radiation,CPR)细菌是指一类由候选门细菌组成的细菌类群,该概念于2015年由Brown等人提出[1]。CPR细菌不仅细胞体积比普通细菌小(大多数CPR能够通过0.2 μm的微孔滤膜),与之对应的其基因组也普遍较小(平均基因组≤1.25 Mbp,且缺少很多核心代谢途径的编码基因)。因此,推测绝大多数CPR以共生或者寄生的方式存在于各类生态环境中[2]。CPR细菌的16S rRNA基因结构特殊,常常包含大量插入序列,目前常规细菌通用PCR引物无法有效扩增大多数的CPR细菌,因此CPR细菌长期以来一直被忽视[3]。CPR细菌系统发育地位特殊,在生命进化树上能够形成庞大且“独立”的分支[4-5],与细菌域的其他类群显著性地分开(图1)。2016年Hug等从JGI数据库中搜集了30 437个微生物基因组,并从中挑选出属水平的3 083个高质量草图基因组/基因组完成图作为代表基因组,然后分别使用16S rRNA基因序列、16个保守的核糖体蛋白串联序列构建系统发育进化树,同样证明CPR细菌的独特进化地位[4]。此外,2019年Zhu等搜集了10 575个基因组,分别用381个标记基因、30个保守核糖体蛋白序列构建系统发育树,同样也得到了类似的结果[5]。

图1 CPR细菌在生命进化树上的进化地位
CPR细菌在地球上分布非常广泛,存在于地下水[1,6-8]、盐湖[9-10]、口腔[11-14]、海洋[15-17]等各种生境中,在地球生物化学循环中也扮演着重要角色。随着基因组学与生物信息学的快速兴起与发展,截止2018年科学家们发现CPR细菌包含了70多个门级水平的分支[2],2019年科学家们预测CPR细菌可能占据细菌多样性的四分之一[18]。尽管如此,人们对CPR这样一个特殊的细菌类群的了解仍处于初步阶段。人们一直推测CPR细菌以共生或寄生的方式存在于环境中,极难获得纯培养菌株。因此,人们对CPR生理特性、代谢功能与机制等知之甚少。到目前为止,仅有一类CPR细菌(称为TM7)在实验室条件下得到了纯的二元培养物,TM7这类微生物能且仅能依附于宿主进行生长[11,13-14,19]。
基于CPR细菌的重要性以及独特性,本文重点针对CPR细菌的发现历程,细胞形态、基因组学特征和遗传进化,生理代谢潜能,生物地球化学元素循环和实验室纯培养等方面进行综述,为未来CPR细菌的进一步研究提供参考。
1 CPR细菌的研究历程
1996年Rheims等[20]、1998年Philip等[21]在通过16S rRNA基因对环境样品进行细菌检测时,发现 TM7和OP11(以采样地点的缩写命名)分支能在进化树上形成独立的分支(无法定位到已知菌属中),这是第一次发现CPR细菌的存在(图2),但当时并未引起科研界的足够重视。2012年Wrighton等[6]首次使用宏基因组技术研究发现地下水中存在特殊的未培养微生物(BD1-5、OP11、OD1),然而此类微生物的分类地位在当时并未明确。2015年Brown等[1]通过宏基因组测序技术分析了美国科罗拉多河地下水样品,共获得了797个CPR细菌的基因组(metagenome-assembled genomes, MAGs),这些基因组分属于35个细菌门。进一步从这些MAGs中提取16S rRNA基因序列并剔除其中的插入序列后构建系统发育树,发现这些新的CPR细菌门与之前发现的一些候选门(OD1、OP11)聚类在一起,且在系统发育树上形成独立分支。由于这些特殊的细菌在当时都无法在实验室条件下获得纯培养物,Brown等将这类由候选门细菌组成的单系分支命名为候选门级辐射类群(candidate phyla radiation,CPR)。因缺乏对CPR生理代谢等多方面的了解,最初发现的CPR细菌均以微生物学终身成就奖获得者的姓名来命名CPR门(比如以美国著名的微生物学家Carl Woese 命名了Woesebacteria门)。自2015年后,越来越多的CPR细菌类群被发现,截至2018年已发现至少70多个门级水平的CPR分支[2]。然而,目前在实验室条件下能获得纯培养的CPR细菌仅有TM7一个门级分支(已从人体口腔与废水中分离出5株TM7[11,13-14,19])。2013年Rinke等[22]对来自不同环境样品中的未(难)培养微生物进行单细胞测序时发现,OD1、OP11、GN-02三个门的CPR细菌在系统发育树上形成单系分支,为直观认识此3个门之间的关系,Rinke等便提议将由这3个门组成的“超系”命名为佩特斯细菌(Patescibacteria)。2018年Parks等在构建微生物标准基因组数据库时发现Patescibacteria与CPR代表着同一类细菌,于是便提议将CPR细菌重新命名为Patescibacteria,当时包含了至少65个门的CPR细菌分类[23]。截至2021年8月,CPR细菌一共包含了超过75个门级水平的分支(表1)。
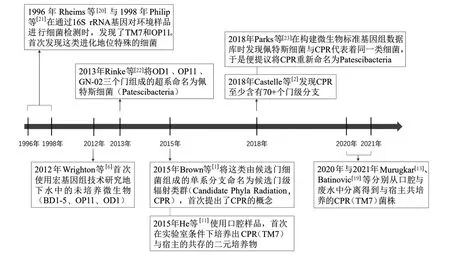
图2 CPR细菌的发现与研究历程

表1 现已知CPR细菌在门水平的成员信息
近年来,针对CPR细菌在自然环境中的分布被广泛报道。相比较于其他生境,目前对CPR细菌的研究主要以水体环境为主,CPR细菌分布在淡水湖[30]、海水[15]、废水处理厂[31]、地下水[1,6-7]等水生态系统中似乎更广泛,且丰度较高。此外,CPR细菌在沉积物[32]、植物根系[33]、土壤[34]中也有分布的报道。有趣的是,在动物[35]与人体[11-13,36]中也检测到了CPR细菌的存在,并被认为可能与人类健康有关。由此可见,CPR细菌在不同的生态系统中广泛分布,其扮演着重要的生理与生态角色。
2 CPR特点介绍
2.1 CPR细胞形态、基因组学特征和系统进化地位
细胞体积小、基因组精简是CPR细菌相比较于普通细菌的一个最显著特征。2015年Brown分别使用1.2、0.2、0.1 μm系列滤膜串联过滤地下水时,通过结合利用16S rRNA基因与宏基因组测序技术发现在0.2 μm与0.1 μm滤膜上存在大量CPR细菌;使用冷冻透射电镜观测到部分CPR细菌能穿过0.2 μm的滤膜,而在0.1 μm滤膜上被富集[1]。2015年He等[11]从人体口腔中分离培养出一株寄生于放线菌的CPR细菌TM7,在电镜下观察到TM7细胞大小在0.2~0.3 μm之间。2021年Batinovic等[19]从废水中分离培养出一株能烈性裂解起泡细菌(Gordoniaamarae)的TM7,电镜观测到的细胞大小在0.3~0.4 μm之间。通过电镜观察,CPR细菌形态主要呈现杆状或者球状[7,19]。
目前获得的CPR细菌的基因组大小约为1 Mbp,与普通细菌、古菌、专性昆虫共生体相比,CPR细菌的基因组更接近于专性昆虫共生体的基因组,显著小于普通细菌和古菌[2]。CPR细菌基因组中存在约50%左右功能未知的基因,同时保守基因也少于我们熟知的普通细菌。Castelle等对1 000多个CPR细菌基因组进行分析比较时发现,大部分CPR细菌的基因组中都存在与同源重组、碱基切除修复和错配修复等相关的基因。因此,Castelle等认为CPR细菌的精简基因组是遗传自祖先的特征,并非是基因组进化过程中发生了缩减[2]。Moreira等比对3个CPR细菌候选门Absconditabacteria(SR1)的基因组后,发现此类CPR细菌只有390个保守基因。同时,Moreira等将SR1门与其他CPR细菌候选门Gracilibacteria和 Peregrinibacteria(PER)对比发现,这3个CPR门可能最近丢失了共同祖先所具有的30%~50%的基因,进而推测这些CPR细菌的基因组内容有活跃的动态进化[10]。CPR精简型基因组特征是源于祖先遗传还是缩减进化存在一定的争论,仍然有待于进一步研究。
Brown等对地下水样品进行宏基因组测序并拼接后,将≥800 bp的16S rRNA基因进行聚类分析,发现31%的CPR细菌的16S rRNA基因中都含有≥10 bp的插入片段,插入位点聚集在16S rRNA基因的可变区和保守区的几个不同的位置。大多数≥500 bp的插入序列编码起催化作用的RNA内含子或者开放阅读框(ORF)[1]。Castelle等[2]发现插入片段现象在一些CPR细菌的23S rRNA基因与tRNA基因中也存在。2016年Eloe-Fadrosh等通过宏基因组学技术,发现使用16S rRNA基因通用引物扩增子检测微生物多样性时会遗漏掉约70%的CPR细菌[3]。由于通用引物的低覆盖率以及CPR细菌16S rRNA基因的特殊性(含插入序列),导致CPR细菌在宏基因组学技术出现前一直被人们所忽略。此外,CPR细菌基因组过度精简,缺少重要的能量代谢基因,推测其普遍通过寄生或共生生长。因此,使用传统的细菌分离培养方法很难获得CPR细菌的纯培养物。2018年Castelle等[2]搜集了1 000多个CPR细菌与DPANN古菌的基因组,用以调查CPR细菌的生物学特性,发现CPR细菌基因组中通常含有很多转氨酶基因,用于转化从环境中获取的氨基酸,并用于自身合成蛋白质,同时将其作为潜在的碳源和能源。CPR细菌基因组中也具有核酸酶基因,能将获得的核苷酸重新转化为DNA 或RNA。CPR细菌基因组中还有许多编码基因参与了IV型菌毛的产生,IV型菌毛可能在DNA吸收[37]、化合物分泌以及与周围细胞的互作中发挥作用[7]。此外,CPR细菌基因组中包含了大量编码糖基转移酶的基因[38],用来生产重组参与细胞表面与胞外环境附着和调节的糖和糖蛋白。更重要的是,与普通细菌通过CRISPR-Cas 来适应性免疫系统防御病毒入侵不同,大多数CPR细菌都通过限制性修饰与顿挫感染(abortive infection)来防御病毒[39]。
2.2 生理代谢特征与潜能
精简的基因组是CPR细菌拥有有限但极为罕见的代谢潜能的重要原因之一。目前的研究表明,大部分的CPR细菌缺乏完整的氨基酸、核苷酸以及脂质合成途径[2],大多数CPR细菌不具备完整的呼吸链,缺失包括NADH脱氢酶和氧化磷酸化复合物Ⅱ-Ⅳ在内的相关基因,完全缺失与三羧酸(TCA)循环有关的基因[2]。然而,在对CPR细菌代谢潜能分析时也有发现在其基因组中存在罕见的代谢相关基因,此代谢通路的上下游基因却不完整,很可能与CPR细菌基因组中存在诸多功能未知基因有关[2]。CPR细菌基因组中常常含有编码大量糖苷水解酶(GHs)基因,能将复杂的含碳化合物降解形成小分子化合物进入糖酵解途径[26]。大多数CPR细菌不具备完整的糖酵解(EMP)途径,缺少催化EMP途径关键酶基因(如磷酸果糖激酶PFK基因等)。CPR细菌基因组中通常含有磷酸戊糖途径(Pentose Phosphate Pathway,PPP)的相关基因[2],因此,推测CPR细菌能通过PPP途径将6-磷酸果糖转化为3-磷酸甘油醛。通常,丙酮酸或者乙酰CoA是CPR细菌中心碳代谢的终产物,CPR细菌一般会先将丙酮酸转化为乙酰CoA,然后进一步将其利用生产短链脂肪酸来平衡碳和电子流[2]。如许多CPR细菌Parcubacteria(OD1)、Dojkabacteria、Microgenomates(OP11)被预测可以利用古菌中常见的ADP-acetyl-CoA合成酶(ADP-Acs)来生成乙酸盐[40]。一些Peregrinibacteria(PER)也可以通过细菌中常见的乙酸激酶(Ack)和磷酸转乙酰酶(Pta)来产生乙酸。除了乙酸盐外,许多CPR细菌还能通过发酵产生乳酸、甲酸或乙醇等产物。在含乙酸盐的地下水中经常发现某些CPR细菌被富集,其中有些CPR细菌基因组编码AMP-乙酰CoA合成酶,表明某些CPR细菌可能具有乙酸盐利用能力[1]。
2018年Castelle等[2]搜集了约1 000个CPR细菌与DPANN古菌的基因组,发现虽然CPR细菌似乎都存在以发酵为基础的生活方式,但不同门水平CPR细菌的生物合成能力有很大差异。某些来自Peregrinibacteria(PER)门的基因组具有相对较多的核心生物代谢能力,似乎都具有合成核苷酸、某些氨基酸和辅因子的能力,但不具备合成脂肪酸的能力。与Peregrinibacteria(PER)门相比,OD1超系成员普遍都缺乏完整的核心生物代谢能力[2]。有趣的是,“CandidatusParcunitrobacternitroensis”[41]不仅具有广泛的代谢能力,拥有基本完整的电子传递链,拥有发酵和呼吸能力以及氮和脂肪酸的代谢能力,最重要的是还含有以氮化合物进行呼吸所涉及的所有酶(亚硝酸盐还原酶、羟胺氧化还原酶和一氧化氮还原酶)的编码序列。这些序列与其他生物体中发现的序列差异显著,表明“CandidatusParcunitrobacternitroensis”以氮化合物进行呼吸的基因不是通过基因水平转移的方式从非CPR细菌中获得的。与代谢能力相对复杂的CPR细菌相比,Katanobacteria、KAZAN和Dojkabacteria这3个门的CPR细菌生物合成与代谢能力最弱,它们缺乏合成核苷酸、氨基酸、脂质、肽聚糖以及各种辅因子的编码基因[2]。因此,我们在关注CPR细菌代谢潜能与生态学功能时,不能因为其共有的一些生物学特征就将整个CPR细菌一概而论,而是应该针对不同门的CPR细菌分门别类地进行研究。
2.3 生物地球化学元素循环
尽管CPR细菌精简的基因组限制了其代谢能力,但CPR细菌在地球生物化学循环中仍然可能发挥着重要而关键的作用。CPR细菌参与碳素循环,具有潜在的固定二氧化碳与降解复杂含碳化合物的能力。2013年Campbell等[42]结合流式细胞仪分选与单细胞基因组扩增技术,从人类口腔样品中分离并获得一支CPR-SR1的基因组,发现其基因组中含有编码1,5-二磷酸核酮糖羧化酶(RubisCO)的基因,具有固定二氧化碳的潜能,与先前研究中发现的Peregrinibacteria(PER)门一样,存在古菌型RubisCO[6]。多项研究表明CPR细菌的RubisCO与古菌十分类似,属于古菌II/III类型和bacterial-like III类型[6,38,42-43]。2016年Wrighton等[43]将Peregrinibacteria(PER)门中的RubisCO基因进行了体外表达,验证了其具有古菌II/III类型的固定二氧化碳的活性。2017年Danczak等[25]将地下水样品宏基因组测序获得的71个CPR细菌基因组与2 000个其他CPR细菌基因组一并分析,确定并统计了135个不同糖苷水解酶(glycoside hydrolase,GH)家族的存在和数量,发现在所有CPR细菌门中,虽然降解底物(如直链淀粉、纤维素)的能力相似,但行使这些能力的糖苷水解酶GH基因不同。尽管几乎所有的CPR细菌基因组都能编码半纤维素侧链降解酶,但具体的糖苷水解酶GH基因在不同的门之间存在差异。2019年Vigneron等[44]使用过滤与宏基因组测序相结合的方法对热岩湖泊中的CPR细菌进行分析,发现每个CPR细菌基因组中平均能鉴定出(29±12)个碳水化合物活性酶(CAZy)基因,表明CPR细菌具有降解复杂含碳化合物的潜力。
不仅如此,在许多CPR细菌基因组中还发现参与反硝化的基因,可见CPR细菌可能还参与了氮素循环。氮素循环是生物圈内基本的物质循环之一,而反硝化作用是氮素循环中重要一环,它与厌氧氨氧化一起将被固定的氮元素以N2形式返还至大气中。Danczak等[25]对地下水样品进行宏基因组测序获得71个CPR细菌基因组,有4个独立metagenome-assembled genomes(MAGs),在其中发现了亚硝酸盐还原酶基因(nirK,反硝化过程中的关键酶基因)分布于2个不同的CPR细菌门(Kaiserbacteria和Harrisonbacteria),这种含铜的亚硝酸盐还原酶能够将亚硝酸盐还原为一氧化氮。同时,将获得的CPR细菌的nirK基因序列与其他nirK基因序列比对并构建系统发育树,发现CPR细菌的nirK基因在进化树上能形成单独的进化支。另有研究发现,CPR细菌中的一个特殊的个体具有一个基本完整的电子传递链,其基因组编码了氮呼吸过程中涉及的所有酶基因(硝酸盐还原酶、羟胺氧化还原酶和一氧化氮还原酶),同样它们的序列与其他生物体内同工酶的序列差异显著[41]。2017年León-Zayas等[45]研究发现CPR细菌中的Parcubacteria也具有硝酸盐还原的能力。综上,多项研究表明CPR细菌中的某些类群可能参与地球生物化学循环中的氮素循环。
硫是生命必需元素,CPR细菌可能也参与硫的氧化还原。2012年Wrighton等[6]通过宏基因组学的方法获得了49个MAGs,发现在CPR细菌OD1与OP11的基因组中含有3种铁氢酶(Fe-hydrogenase)和23种镍铁氢酶(NiFe-hydrogenase)编码基因。进一步通过系统发育分析发现其中17种镍铁氢化酶与古菌热球菌目(Thermococcales)的3b型胞质氢化酶序列高度相似,推测其可能参与多硫化物的还原。2021年He等[8]在比较研究一个农业污染严重的地下水和七个干净的地下水中微生物群落时,共获得746个CPR细菌与DPANN古菌基因组,发现CPR细菌占地下水微生物群落丰度的3%~40%,并且通过检测评估了其参与地球生物化学代谢过程中的关键酶基因,发现在3个CPR细菌基因组中含有硫双加氧酶基因(sdo),以及许多CPR细菌基因组中含有许多硫酸盐还原相关的基因(sat、cysC和cysN),推测CPR细菌在硫循环过程中可能发挥作用。
2.4 CPR的分离培养和生理代谢特征
随着高通量测序技术在微生物学研究领域的广泛应用,越来越多的CPR细菌在各种生态环境中被检测到,但人们目前对于CPR细菌的认识仍大多来自于宏基因组测序拼接得到的MAGs,缺少对纯培养物的研究。在CPR细菌70多个门级分类中,目前仅有TM7分离得到可稳定传代的二元培养物。此外,目前还缺乏可靠的标准化的获得CPR细菌与其宿主纯的二元培养物的方法。
2015年He等[11]通过针对CPR细菌具有链霉素抗性特性,不断提高链霉素浓度获得了一个稳定的TM7与其宿主共培养的富集液,首次在实验室条件下培养出CPR细菌与宿主共存的二元培养物。He等最初也尝试采用传统的平板涂布方法分离CPR细菌TM7x,但一直未能成功,转而经由多个共现性分析发现口腔中的TM7x与放线菌宿主XH001具有一定的相关性,进而不断尝试更换分离培养方法,发现尽管XH001能在平板上形成单菌落,但却无法获得TM7x的单菌落。此外,无论是添加共培养的废液,还是加热杀死XH001都无法让TM7x单独生长,可见CPR细菌TM7x严重依赖宿主XH001菌株。进一步研究发现,在营养丰富条件下TM7x与XH001能共存;但当处于饥饿条件下时,单独培养的XH001能保留活性,而与TM7x一同培养时XH001大部分失活,而TM7x却保留活力。通过透射电镜(TEM)观测,发现XH001表面附着有TM7x(图3a),并且XH001细胞膜出现破损,因此推断TM7x主要寄生于XH001表面,而非共生关系。
2020年Murugkar等[13]总结出前人研究的四点经验:①TM7是一种专性寄生微生物,需要与宿主共存才能生长;②TM7细胞体积小,能够通过0.22 μm滤膜而与其他细菌分开;③TM7与宿主的共培养物在肉汤培养基中能够稳定传代,但在平板上无法形成TM7的单菌落;④推测TM7的宿主来自于放线菌门、厚壁菌门或梭杆菌门。Murugkar等[13]通过过滤收集TM7后,从推测的宿主潜在门中选取潜在宿主,将二者在肉汤培养基中进行共培养,最终成功分离出4株TM7(包含TM7x)。
Gordoniaamarae是最常见的起泡细菌之一,尚未分离出裂解G.amarae的噬菌体,其基因组内存在多种抗病毒机制来抵抗噬菌体感染。2021年Batinovic等[19]在筛选废水处理厂中一株起泡细菌(G.amarae)的噬菌体时,将原位废水通过0.45 μm滤膜过滤后,直接把滤液滴在含起泡细菌单菌落的平板上,意外筛选到一株能烈性裂解起泡细菌的CPR细菌TM7-JR1。TM7-JR1却能够将G.amarae裂解,为将来通过生物方法防治废水处理中的起泡细菌(产生泡沫)提供了一种新的策略。
2021年Moreira等[10]将西欧一个永久咸水湖底采集的微生物垫置于过滤后的原位水体中进行培养,几个星期后观测到大量的光合细菌,同时在光合细菌的表面发现有一个或多个深色的无鞭毛小细胞附着(图3b、c)。研究人员通过显微操作(用毛细管捕捉)搜集附着了小细胞的光合细菌,进行全基因组扩增测序(whole genome amplification,WGA),获得两个基因组,分别属于伽马变形菌门光合细菌与CPR细菌候选门Absconditabacteria(SR1),其中SR1的基因组比较完整,而光合细菌的基因组完整度只有15%左右,推测可能是SR1吸收消耗了光合细菌的DNA。虽然文中并未获得CPR细菌与光合细菌宿主的二元纯培养物,但基本确定了CPR细菌与宿主之间一对一的寄生或者捕食关系。

图3 CPR细菌与宿主细胞共存的显微照片(图片已获再版许可)
综合考虑CPR细菌精简的细菌基因缺少关键核心生物合成与代谢关键基因的特征,以及目前CPR细菌分离培养的经验,可以初步判断绝大多数CPR细菌无法单独生长,需要以其他生物作为宿主营寄生或者共生生长。
3 展 望
近年来急速兴起的组学技术为自然环境以及人工环境中未培养微生物的生态学分布、潜在生理代谢特征、遗传进化等研究提供了强有力的工具,使得CPR细菌等未培养微生物进入我们的研究视野,成为微生物学界关注的焦点。考虑到CPR细菌独特的生命进化地位、生理代谢特征、重要生态学功能、高度的物种多样性和广泛分布的特点,及其与人类健康存在潜在的重要关系,认为未来要在以下几个方向加强对CPR细菌的研究。①CPR细菌物种多样性与生态学分布规律:基于宏基因组和宏转录组等多组学技术手段,在群落和单细胞水平研究CPR细菌在各种自然和人工生态系统以及人体肠道等生境中物种多样性和生态学分布特征,尤其关注CPR细菌与不同潜在宿主间的共现性关系和对生境微生物整体群落的塑造效应。②CPR细菌纯培养和生理代谢特征:在获得CPR细菌基因组学信息的基础上,有目的地优化CPR细菌与宿主共培养条件,并开发相应的高通量培养等技术,实现CPR细菌与宿主的二元纯培养,从而深入探索CPR细菌这一类独特生命体的生理代谢特征。③CPR细菌在地球生物化学循环中的贡献:目前研究发现CPR细菌参与了地球碳、氮、硫、磷等多种元素的生物地球化学循环,未来需要加强多学科交叉,结合生物学、化学、生态学以及地质学等,系统探究CPR细菌在生物地球化学循环中的作用及机制,定量化CPR细菌对地球化学元素循环的贡献。④CPR细菌与人类健康的关系:目前发现CPR细菌对人体健康存在重要影响,然而其作用机理仍然很不清楚。未来需要针对人体口腔和肠道等重要生境CPR细菌的生境溯源、物种多样性和生理代谢机制进行系统研究,解析CPR细菌对人体健康的影响及其作用机制。⑤CPR细菌在生命进化研究中的意义:CPR细菌处于生命进化树的最根部,可能是现存的最古老和最简单的细胞生命体之一,研究其诸多生命形式和进化特征,将对生命起源和细胞结构复杂性演化等重大科学问题产生重要影响。

