海洋多毛类动物的再生演化*
张琳琳 翁洁羊 李 倩
海洋多毛类动物的再生演化*
张琳琳1, 2, 3, 4翁洁羊1, 4①李 倩1, 4
(1. 中国科学院海洋研究所 实验海洋生物学重点实验室 青岛 266071; 2. 中国科学院海洋大科学研究中心 青岛 266071; 3. 青岛海洋科学与技术试点国家实验室 海洋生物学与生物技术功能实验室 青岛 266237; 4. 中国科学院大学 北京 100049)
再生是生物体丢失的组织或器官重新生长和修复的过程, 是生物科学的本质问题之一。再生现象在生物界中广为分布, 但不同生物的再生能力却存在着巨大差异。虽然近年来再生的分子和细胞机制取得了显著的研究进展, 但驱动物种再生能力多样性形成的遗传机制尚不明确。海洋多毛类因其多样且快速演化的再生能力和简单线性的分节结构成为研究再生演化的极佳类群。本文首先从生态和演化生物学的角度综合分析了多毛类再生的特点、进一步总结细胞和分子来源以及再生性状多样性, 然后分析了印太交汇区的海洋多毛类再生性状的特点以及外在多圈层因素对再生性状的潜在驱动效应, 并就未来的发展趋势提出了建议。
再生多样性; 海洋多毛类; 适应性演化; 印太交汇区; 进化发育
再生是指多细胞生物体对衰老的细胞或者组织进行更新替换, 或者对丢失的组织进行修复的过程(Poss, 2010; Birbrair, 2013)。再生一直以来吸引着无数生物学家和临床医生的青睐, 也是《Science》公布的全世界最前沿的125个科学问题中最重要的前25个问题之一。再生是一个古老的性状, 但不同物种的再生能力却存在巨大差异(见图1)。比如水螅、涡虫和多毛类等具有极强全身再生能力, 蝾螈等具有较强的局部再生能力, 小鼠等哺乳动物的某些器官具有再生能力(如小鼠肝脏), 但大部分器官或组织只具有有限的细胞添加或替换能力。
海洋无脊椎动物的典型代表多毛类被称为“海底蚯蚓”, 对底层生态系统的能量流动和物质循环过程具有重要作用, 如营养物的循环、污染物质的分解、扩散和掩埋, 以及次级生产力等(Rhoads, 1982; Solan, 2004; Srivastava, 2014; 张均龙等,2016)。多毛类拥有各式各样的海洋底栖环境适应性性状, 其中的典型代表是“再生”。在适应底栖环境的过程中, 多毛类的头部和尾部演化出了“有”或“无”的再生性状, 位于系统发育树不同演化地位的物种发生多次独立的再生丢失现象。此外, 多毛类的体制为简单线性的分节结构, 实验操作相对容易。以上因素使得多毛类成为再生多样性演化研究的极佳材料。
经典的再生生物学以涡虫、水螅和蝾螈等再生模式生物为主要研究对象。随着高通量测序技术和CRISPR/Cas9基因编辑等技术的突破, 深入研究不同动物类群的再生机制成为可能, 从而催生了一门新的学科“再生演化生物学(EvoRegen)”(Lai, 2018), 从辐射对称的刺胞动物, 到早期两侧对称的无体腔动物, 再到原口动物和后口动物, 每个类群中都涌现出再生演化生物学的代表物种。本文将聚焦于海洋多毛类, 介绍其再生细胞和分子来源的研究进展, 并在此基础上综述世界海洋生物多样性中心印太交汇区的海洋多毛类再生多样性研究进展, 以期为再生演化以及印太交汇区海洋生物多样性中心形成演化机制的研究提供参考。

图1 不同后生动物门间再生能力的分布(引自Bely et al, 2010a)
注: 图中实心圆圈代表具有对应再生能力; 空心圆圈代表无对应再生能力
1 后生动物的再生
按照再生部位不同, 再生分为整体再生和局部再生。局部再生又包括结构水平(肢体、鳍、尾、头、触手等)、器官水平(心脏、肝脏、晶状体)、组织水平(表皮、肠壁)及细胞水平(轴突、肌肉纤维)的再生。依据再生过程的不同, 再生又分为4种方式, 干细胞介导的再生(Stem-cell mediated regeneration)、代偿性再生(Compensatory regeneration)、变形再生(Morphallaxis)和新建再生(Epimorphosis) (Gilbert, 2000)。干细胞介导的再生是指通过干细胞增殖和分化对缺失的细胞或者组织进行更新替换; 例如, 面对损伤, 涡虫干细胞能够增殖、分化成任何缺失的功能性细胞。代偿性再生(比如哺乳动物肝脏)是指分化细胞分裂, 但保持其分化的功能。新的细胞不是来自干细胞, 也不是来自成体细胞的去分化。每个细胞产生与自身相似的细胞, 没有未分化的组织块形成。变形再生指的是通过尚存组织重新进行模式形成和重新建立边界而进行的再生过程, 不需要重新招募细胞, 也不需要大量的细胞增殖。新建再生是指重新长出丢失的组织, 损伤部位的其他组织细胞并不直接介入再生过程, 包括细胞增殖、芽基形成和干细胞或祖细胞的分裂、分化帮助完成缺失部位再生, 比如两栖动物断肢再生(Han, 2005)。新建再生与变形再生相比, 前者会依赖细胞增殖并形成芽基而后者不会。
为什么有些物种具备极强的再生能力, 而另一些物种却只有局限的再生能力?比如: 水螅只用300个细胞就可以完成头部和足部的再生(Bosch, 2007), 整个个体可以从分离的细胞中重组(Hobmayer, 2000; Bosch, 2007); 涡虫的1/279的身体片段可以再生出整个身体(Reddien, 2004; Alvarado, 2006; Robb, 2014); 部分群居海鞘动物可以从只包含血管和血细胞的片段再生整个个体(Brown, 2009); 蝾螈和虾蟹等在断肢之后也能完成再生(Marks, 1970; Mellon, 1978; Sugiura, 2016)。哈佛大学的Srivastava M.实验室通过比较淡水涡虫和阿科尔扁形虫, 发现两个物种再生过程中建立体轴极性的通路均为Wnt和BMP通路, 暗示着在两侧对称动物的祖先中可能有保守的再生体轴信号通路(Srivastava, 2014)。再生演化生物学领域另一个显著的代表成果是2013年来自美国、欧洲和日本的三个独立团队同时在《Nature》上发文指出, 只需要调节Wnt一种信号通路就能够使头部再生部分缺陷的扁虫恢复再生能力, 扁虫是再生模式生物真涡虫的近缘物种(Liu, 2013; Sikes, 2013; Takeo, 2013)。其中的一个例子是马普研究所的Rink J.C.实验室发现在一种头部再生部分缺陷的涡虫()中, RNAi干扰Wnt通路的单个基因就能够使其具备头部再生能力, 暗示着单个基因能够恢复进化上几百万年前丢失的再生程序。
2 海洋多毛类的再生
2.1 海洋多毛类介绍
多毛类是环节动物门最大的一个纲和最为原始的类型。根据分子钟推算, 多毛类起源于5.41—6.35亿年前的埃迪卡拉纪(Han, 2019)。在寒武纪早期5.19亿年前的云南澄江生物群中, 发现了两个已知最古老多毛类新属种——古扇虫()和中华隐刚毛虫()。多毛类共记载物种12529种, 分属于86科(见表1)。其中隐居亚纲5419种, 游走亚纲6306种, 螠虫亚纲173种。不同科的记录物种数量差别较大, 这可能与不同物种的生存环境和取样难度有关。物种多样性列于前五的分别是裂虫科(1075种), 多鳞虫科(900种), 沙蚕科(708种), 龙介虫科(736种)和蛰龙介科(626种)。有27科世界范围内记录物种不足5种, 55科记录物种不足100种。
表1 多毛类分类

Tab.1 The classification of polychaete
注: 表中第1列表示拉丁文科名, 第2列表示中文科名, 第3列表示该科包含的物种数量(数据统计自世界海洋生物名录, www.marinespecies.org)
2.2 多毛类的再生过程
多毛类组织经受到损伤后会启动再生修复过程, 这对再生是十分关键的。海洋多毛类因其多样且快速演化的再生能力和简单线性的分节结构成为研究再生演化的极佳类群。在再生性状的定性描述上, 依据多毛类的再生能力不同可以分为全身再生和局部再生, 全身再生包含, 头部和尾部均能完整再生, 头部不能再生, 尾部能完整再生, 以及头尾部均无法再生三种类型(详见图2) (Zattara, 2016a)。多毛类还具备局部组织的再生能力, 比如像缨鳃虫()的鳃丝和壳盖, 才女虫()的触角, 长手沙蚕的触手等头部分化器官的再生(Mortimer, 2014)。此外根据再生的组织特性, 还可将其分为完全再生和不完全再生, 比如裂虫科的(Aguado, 2015b; Weidhase, 2016)头部能进行部分再生, 但是并不能完全恢复原有的结构, 这就属于不完全再生。
目前, 多毛类再生研究中的典型物种有杜氏阔沙蚕和小头虫两种生物。杜氏阔沙蚕依赖于后端“生长区”不断增加新的体节, “生长区”中有多种干/祖细胞能表达多种特征性分子(Juliano, 2010)。该物种截断后能够再生出尾部, 并依靠“生长区”以产生新体节。它的尾部的再生能够按特定的模式和时间顺序快速完成, 截断1 d后伤口愈合, 2 d后再生芽基形成并且部分组织特化, 截断3 d后后肢生长区形成。并且除了在咽附近进行截断外, 在沿前后轴的其他任意部位截断时再生能力基本相同。反复截断, 杜氏阔沙蚕的再生能力依然存在。基于在杜氏阔沙蚕上建立了先进的分子和遗传工具, 其成为研究再生细胞分子机制的良好模式物种(Williams, 2016)。

图2 多毛类再生模式图
小头虫()在身体截断后能进行尾部再生而不能完成头部再生。截断1 d后伤口愈合, 2 d形成再生芽基。截断后一周芽基内会聚集大量的增殖细胞。截断后7 d即可见明显的神经节。截断后12 d, 通常会形成3—13个新生体节。小头虫具有的快速的再生能力非常利于对再生机制的研究。同时, 在小头虫中成功实现基因编辑, 这将对进一步深入研究再生提供技术支持(Neal, 2019)。
多毛类的再生过程主要包括四个阶段(见图3), 分别是机体损伤、伤口愈合、细胞增殖与模式建成, 以及细胞分化再生长(Licciano, 2012; Özpolat, 2016)。不同物种之间甚至同一物种不同个体之间再生完成的时间有一定差异, 一般来说像小头虫、沙蚕等物种, 再生事件及发生时间为伤口愈合(12—36 h)、芽基出现(1—4 d)、细胞分化与再生长(2—8 d)。
机体损伤和愈合时期 机体损伤是多毛类再生过程的起始, 生命体面对机体损伤或快速起始应激反应, 包括应激基因的表达等。此外, 伤口边缘处的环状肌肉会快速收缩以封闭伤口。有些情况下肠道组织的轻度挤压促进伤口的闭合, 以阻止体液的流失和防止感染, 同时达到伤口愈合的目的(Bely, 2014)。
细胞增殖与模式建成阶段 最显著的特点是再生芽基的生成。此外, 决定头部或者尾部再生的体轴信号也是在此阶段起始的。早期芽基形成伴随着明显的细胞增殖和细胞迁移现象。神经生长进入芽基, 新的神经纤维是从旧腹侧神经索延伸出来的(Müller, 2003; Paulus, 2006)。之后神经纤维会融合形成一个环(吻盲虫Pristina, 双才女虫)或者是三重环(丝鳃虫/)。吻盲虫和丝鳃虫的大脑形成也从这个时期开始, 其中丝鳃虫的神经回路会与新形成的大脑相融合。在吻盲虫和丝鳃虫中, 芽基激活后肌肉纤维才会出现。纵向肌纤维是较早向芽基内部生长的(Zattara, 2011; Weidhase, 2014, 2015)。在隐居亚纲吻盲虫、丝鳃虫以及仙虫()中, 再生纵向肌和环形肌的发生过程都是一致的。
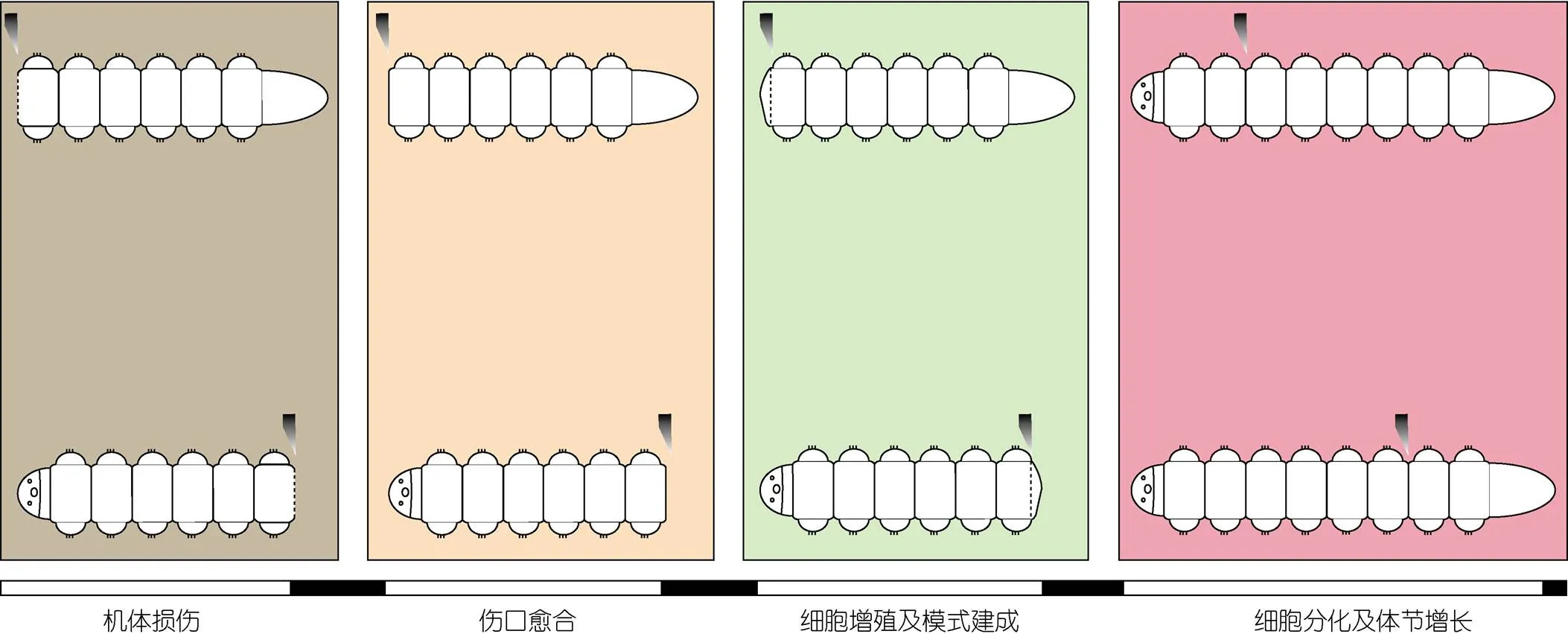
图3 多毛类再生的四个过程
细胞分化再生长时期 这一过程主要包括组织器官的分化, 体节添加区/后端生长区产生出新的体节, 肠道发生变形再生, 体节继续生长, 最终完成再生。一般至此阶段, 环形肌才开始出现。大部分神经系统的再生将在芽基分节前基本完成。比如在游走亚纲的沙蚕()和隐居亚纲的吻盲虫()、丝鳃虫(/Timarete)、小头虫()中, 腹神经索神经节的形成明显晚于外周神经环和大脑的形成。神经索神经节通常在身体明显分节之后才能被检测到(Pfeifer, 2012; Weidhase, 2014; De Jong, 2016)。头部和尾部再生的早期神经发生过程非常相似, 然而后期神经发生过程却有很大的不同。后期神经发育往往具有体轴或组织特异性。虽然伤口愈合和再生通常是同时进行的, 但某些生物的再生不需要此过程。
2.3 多毛类再生的细胞来源
再生芽基主要有两种细胞来源: 干细胞或干细胞样细胞的迁移, 以及局部细胞的去分化(Hill, 1970)。干细胞是一种未特化的细胞, 具备分化为任何其他细胞的能力和自我更新的能力。淡水涡虫和水螅在其一生中都拥有一定数量的全能干细胞, 当受到外界环境的刺激时被激活, 可分化发育成任何一种类型的组织(Reddien, 2004; Alvarado, 2006; Bosch, 2007); 而象蝾螈的肢体再生和斑马鱼鳍再生等, 是通过已分化的成熟细胞的去分化和再分化完成的(Satoh, 2008; Blum, 2015)。
多毛类中只有在少数物种中研究了再生过程的细胞来源, 包括游走亚纲的深海沙虫()和隐居亚纲的沙蚕(), 小头虫()等(Özpolat, 2016)。
在缨鳃虫()的再生过程中, 中胚层、内胚层和外胚层三个组织都对芽基形成有贡献。其中中胚层细胞来源于两部分, 一是来源于肌肉组织的碎片, 另一部分是来源于血管处的细胞(Hill, 1970)。Potswald等在对龙介虫血管处细胞细微结构的研究表明这类细胞具有高度分化的特征(Potswald, 1969)。在杜氏阔沙蚕()和小头虫()再生过程中, 大量细胞向损伤部位迁移。外胚层、中胚层和内胚层来源细胞均检测到增殖(Bely, 2014)。杜氏阔沙蚕尾部再生研究显示再生细胞的来源可能是伤口周围三个胚层的细胞, 伤口和再生芽基表皮细胞快速增殖, 这种增殖可能是新表皮细胞的唯一来源(Planques, 2019)。De Jong利用细胞示踪技术揭示小头虫再生的细胞来源既包括多能干细胞的迁移也含有损伤部位细胞的再分化(De Jong, 2018)。Zattara等也在吻盲虫()中证明细胞来源可以是远距离多能干细胞的迁移(Zattara, 2016b)。目前尚未在多毛类中检测到与涡虫Neoblasts或者水螅i-cells类似的全身分布的多能干细胞。多毛类中还存在再生芽基非依赖性的局部再生, 如管栖多毛类龙介虫()用于进食和呼吸的放射状附属物(触须)和可以关闭管子作为防御的盖体的再生模式并不相同。形成再生芽基是触须再生的重要阶段, 而盖体在没有芽基形成的情况下即可完成再生。值得注意的是, 在龙介虫整个再生过程中, 全身均可见到增殖细胞, 没有发现明显或特别的增殖区域, 这与绦虫相似(Szabó, 2014)。
2.4 多毛类再生的分子机制
多毛类再生性状的分子机制是再生生物学的另一个研究热点。已有的研究主要包括四个方面的生物途径(见表2)。再生过程中, 首先是胞外基质, 细胞迁移等基因参与伤口修复, 比如在杜氏阔沙蚕伤口愈合期有RNA结合蛋白(Pdum-elav)等基因的大量表达。其次, 参与头尾体轴、再生芽基分化体制的基因被激活。细胞增殖和模式建成时期启动了Wnt信号通路以决定机体再生出头部还是尾部。再生相关基因如:基因、等(Takeo, 2010; Pfeifer, 2013; De Jong, 2016)。在细胞分化再生长阶段,基因簇扮演着重要角色。在小头虫和杜氏阔沙蚕中,和基因主要在新发育的腹神经索神经节中表达, 也在后生长带中表达(Pfeifer, 2012)。再生体节形成期间,和基因在新增分节处表达(Prud'homme, 2003), 以及沙蚕中的Hes/Hey家族基因具有类似的作用, 在无脊椎动物神经及分节中发挥功能(Gazave, 2014)。再者, 干细胞样细胞标记基因和在再生早期发挥重要作用(Christen, 2003), 最后, 神经和肌肉等器官分化生长的基因:也在此时期开始表达。详细相关基因汇总见表2(Prud'homme, 2003; De Rosa, 2005; Janssen, 2010; Giani, 2011; Pfeifer, 2012; Novikova, 2013; Pfeifer, 2013; Pruitt, 2014; Szabó, 2014; Kozin, 2015; Özpolat, 2015; De Jong, 2016; Novikova, 2016; Dannenberg, 2018; De Jong, 2018; Kostyuchenko, 2019; Planques, 2019; Andreatta, 2020)。
表2 多毛类再生相关基因
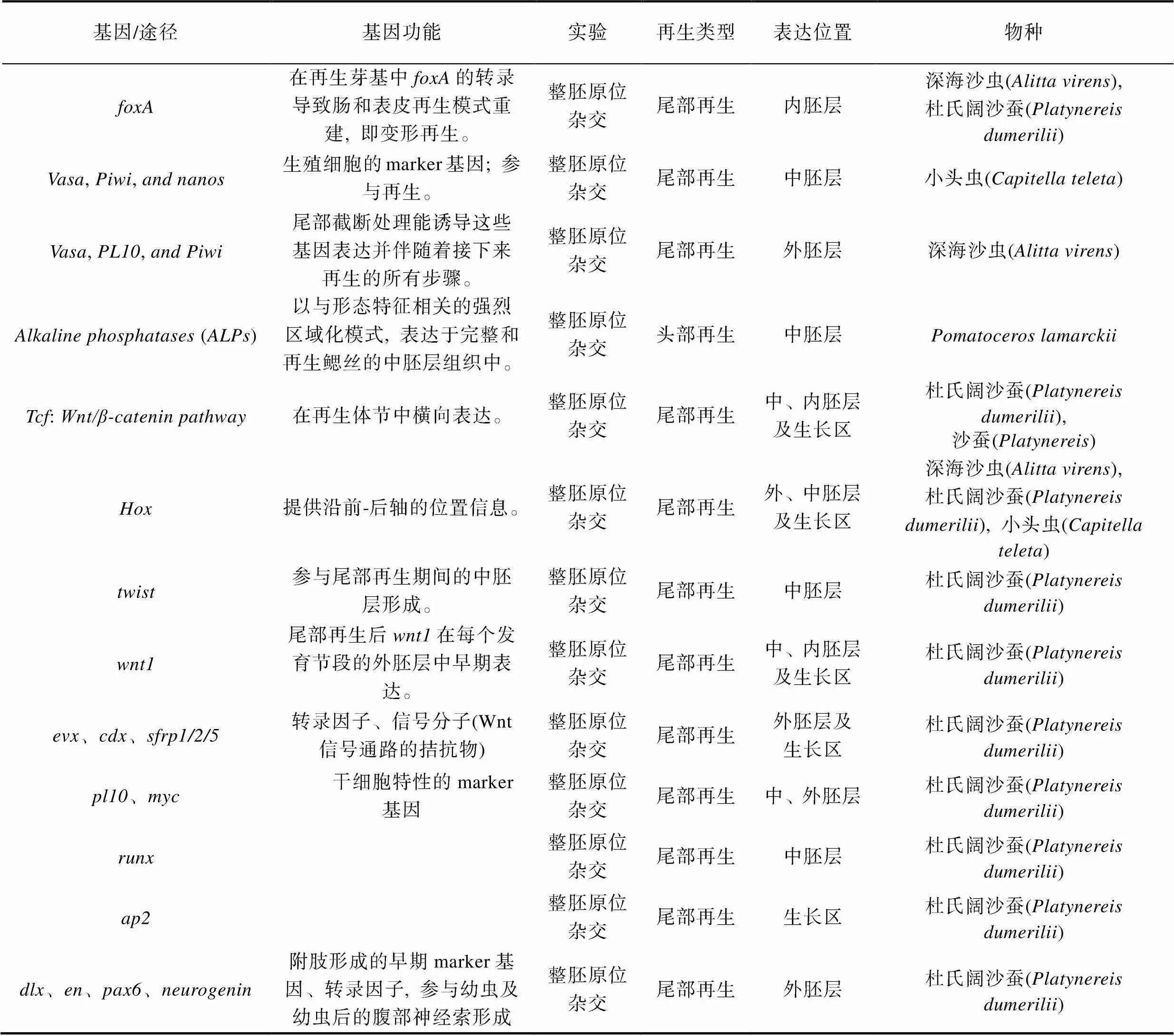
Tab.2 The regeneration related genes in polychaete
2.5 多毛类再生研究的技术手段
多毛类再生实验研究需要对再生能力进行定量分析。通过对新生片段计数、测量再生部位的宽度, 或者通过测量再生部位的长度和宽度计算体积等方法可以对多毛类再生性状进行定量研究以追踪再生引起的形态变化速率。此外, 通过对完成再生所需的时间如伤口愈合时间、实现完全再生所需的天数或者对新生身体修复后的再生体节数目进行统计, 可以对具体物种的再生能力进行测评(Hentschel, 2006; Freitas, 2016; Pires, 2016)。再生过程中组织(肌肉、神经)微观变化时间和数量检测, 可以利用对乙酰化α-微管蛋白的免疫荧光染色来指示神经所在部位(Grimmel, 2016); 可以使用鬼笔环肽(phalloidin)标记肌肉中F型肌动蛋白(Schulze, 2009)。
多毛类再生的研究中不仅需要了解再生过程中形态上的变化, 还需要借助分子及细胞实验手段分析生物体内部的变化。除了宏观数据观测, 还可利用分子手段对再生能力进行评估。对不同再生阶段或不同再生能力的组织进行高通量测序, 如对再生能力不同的两种裂虫进行转录组比较, 筛选差异表达的基因集(Ribeiro, 2019), 可以使我们全面掌握参与再生的关键基因。利用对相关基因的时空表达模式(空间原位杂交, 免疫组织化学等)以确定其是否参与再生过程, 可以对再生关键节点的标记基因进行时空表达检测, 明确基因启动表达的时间段。当然, 解析表型和基因型最直接的手段是利用CRISPR/Cas9对再生相关的基因进行编辑, 通过基因缺失后的再生表型变化来揭示基因功能以及基因与再生之间的关系(He, 2018)。此外, 对于多毛类再生的分子机制研究可以借鉴多组学、基因编辑等其他再生模式物种中成功应用的技术手段。墨西哥钝口螈(), 具有强大的再生能力, 其基因组比人类的更加复杂。2018年, 多个团队共同构建墨西哥钝口螈的全基因组序列, 发现基因组中基因丢失, 而是存在于鱼类和其他两栖动物中的保守基因。作者进一步通过基因编辑技术证实在墨西哥钝口螈中行使的功能, 在蝾螈的发育再生中起到调控作用(Nowoshilow, 2018)。蜥蜴是一种具有羊膜的脊椎生物, 它们能够再生一条正常功能的尾巴。2014年, 有团队对蜥蜴()再生尾巴进行转录组学分析, 发现在蜥蜴尾巴再生过程中激活了326个与创伤反应、发育和修复机制等相关的差异表达基因(Hutchins, 2014)。2019年, 来自美国的学者组装拼接了一种两侧动物祖先物种的全基因组序列, 借助功能基因组学的方法, 作者发现可调节其全身再生的调控因子——EGR, 当身体受到创伤后, EGR开启开关, 调控生物全身再生相关的基因, 开启再生(Gehrke, 2019)。
再生的细胞来源是再生生物学学者最为关心的科学问题, 细胞增殖检测不仅可以帮助理解再生过程中细胞增殖发生的时间还能理清细胞迁移等来源问题。再生过程中检测细胞增殖的方法较多, 比如利用免疫组化技术检测乙酰化组蛋白来显示染色质转录活跃的的组织或细胞; 检测增殖细胞核抗原(PCNA)以观察再生阶段细胞所处的时期(Niwa, 2013)。5-乙炔基-2’脱氧尿嘧啶核苷(EdU)、溴脱氧核苷尿嘧啶(BrdU)标记用于检测处于S期细胞的百分比(Seaver, 2005; Szabó, 2014; Planques, 2019)。此外, 科学家在再生模式生物中通过结合细胞生物学、胚胎发育生物学、遗传学等方法解析再生的细胞来源, 这些方法和研究思路为海洋多毛类再生细胞来源的研究提供了重要的借鉴。多能干细胞是真涡虫(planarian)再生的细胞来源, 但即使这些多能干细胞具有共同的属性, 但仍然是多能干细胞(pluripotent stem cells, PSCs)和谱系祖细胞(lineage progenitors)的复合细胞群。直到2018年, 曾安等人结合单细胞RNA测序分析和单细胞移植技术, 成功地将PSCs和谱系祖细胞分开, 完善了现有的多能干细胞的分类, 也证明多能干细胞是可以被高效单独识别的, 这为研究再生组织的细胞来源提供了非常新颖、有效的方法(Zeng, 2018)。此外, 真涡虫的眼睛在被切掉后也能重新再生出来, 并且与正常的眼睛一样可以行使正常功能。但是视神经是如何重建的, 以及是如何与大脑正确连接以发挥正常的功能仍未有解释。最近有研究, 通过不同细胞标记基因共表达、免疫组织化学、原位杂交、RNAi以及“眼睛移植”等技术, 表明肌肉细胞在模式基因的调控下行使“指路”的功能, 在眼睛神经的再生过程中起到了至关重要的路标作用, 此项研究为神经再生研究引领了新方向(Scimone, 2020)。蝾螈是一种两栖生物, 与其他两栖生物在经历变态发育后便会失去再生能力不同, 蝾螈的四肢可以在整个生命周期中进行再生。2016年, 日本和美国学者以转基因蝾螈为模型, 结合组织移植、免疫组织化学等发现了蝾螈幼体和成体再生过程中的细胞差异来源于一组肌肉纤维细胞, 蝾螈幼体使用干细胞或者祖细胞(stem cells/progenitor cells)再生肌肉, 而成体蝾螈在经历变态后从断肢中再生出肌肉纤维细胞。此结果更进一步解释了蝾螈演变出一种新的再生机制, 保证变态后的成体仍然可以再生完整肢体, 这为其他物种的再生研究提供了借鉴(Tanaka, 2016)。
3 多毛类再生性状多样性
目前多毛类再生演化研究主要聚焦于再生能力强的单一物种, 试图从发育生物学的角度解析这些物种再生中涉及到的细胞和分子机制。不同物种之间的再生能力差异显著, 但针对这些再生能力多样性的研究较少, 仍然停留在物种性状分类和系统演化方面的初步探索, 再生能力演化的分子机制更是领域内的研究瓶颈。
3.1 多毛类再生性状多样性描述
根据多毛类再生可以分为定性和定量, 从定性角度, 多毛类能否最终再生出头、尾的性质, 再生分为3个类型(头尾能再生, 尾部能再生, 头尾都不能再生)。但从再生能力定量角度, 多毛类内不同科之间甚至同一科不同种之间再生能力存在较大差异, 从完全无再生能力的物种至仅靠一个片段就能再生的物种。相较于尾端再生, 头部再生形式更多样化。多毛类头部截断后前端再生的能力大约相当于被切除节段的数量, 这是非常特殊的。最显著的例子是裂虫(), 其被截断的片段数目能够被准确再生(Hyman, 1940)。某些多毛类的头部再生能力会在身体的某些体节处消失(Hyman, 1940)。包括矶沙蚕在内的一些属, 不管在什么部位截断, 头部都可以完全再生, 而在另一些属中, 如果截断位置在身体前端区域, 则不能再生(Hofmann, 1974)。不同的截断位置会产生不同的再生能力。将磷虫()的第13体节分离出来并进行截断, 该节的后半部分能向前和向后再生, 而前半部分仅能向前再生; 而在虫子第13体节中央处截断, 则前半部分能够向后再生(Hill, 1972)。杜氏阔沙蚕头部再生能力相当有限, 只有原口前叶的一部分留在截断面, 口前叶才能再生(Hofmann, 1975)。自裂虫()在第6个体节向任何位置截断尾部都能诱导再生发生, 但在第5、6节之间截断则前部碎片不会再生出尾部, 而将第6节的小碎片保持附着在第5节的后端则会发生尾部再生(Okada, 1938)。当截断裂虫的12或13体节, 通常会产生双头表型, 第二个头出现在第14体节(Okada, 1929)。对巢沙蚕()进行第15刚节截断时, 前端身体无法再生, 后端身体能再生前部, 当超过25刚节时, 前端身体能再生后部而身体后端无法再生(Pires, 2012)。此外, 不同生活史阶段, 再生能力也会不相同。研究表明裂虫()在有性繁殖期间, 前部再生能力会突然下降(Hyman, 1940)。再生还会引起体制改变, 在缨鳃虫(sabellids)和龙介虫(serpulids)中, 头部会在胸部或腹部区域截断之后再生, 相邻的旧身体节段重组为具有典型节段数目和形态的胸部区域(Hyman, 1940)。也有一些特殊类型, 比如原节虫科(Protodrilidae)中没有肠道、口、肛门, 具有与一般多毛类不同的身体体制, 但其也能够前端再生(Jouin, 1979)。智利巢沙蚕()上颌骨会进行周期性的脱落和再生生长, 这与其他物种颌骨持续性生长并不一致(Colbath, 1987)。多毛类内不同科之间同一科不同种之间再生能力定量差异的机制仍不明确。
随着对再生模型生物不断深入的研究, 我们对于再生过程越来越了解, 比较不同再生能力物种之间的再生过程, 将成为推断再生能力演化的重要信息来源(Fröbisch, 2014)。
3.2 多毛类再生性状的起源和演化
再生演化的一个关键科学问题是再生性状是祖先物种的古老起源还是位于系统发育不同位置的物种独立演化而来。目前的研究发现早期动物栉水母具有较强的再生能力(DuBuc, 2014), 暗示再生在后生动物的祖先物种已经存在(Bely, 2010b)。再生性状在演化过程中经历了不同的命运, 包括在哺乳动物中有限的再生能力和涡虫、水螅等物种中极强的再生能力。但因为哺乳动物和涡虫、水螅等物种演化距离相距甚远, 通过比较上述物种的再生过程差异对研究解析再生演化机制有一定局限性。值得强调的是, 仅在多毛类中便出现了丰富的再生性状多样性, 因此多毛类是研究再生多样性研究的极佳类群。在多毛类所属的环节动物门, Zattara等通过对个物种的系统发育树构建和性状溯祖分析, 发现环节动物的再生是个古老的性状, 在环节动物祖先物种已经存在。在环节动物的演化过程中, 头部再生丢失了18次, 尾部再生丢失了5次, 进而形成了现在多毛类的再生性状多样性(Zattara, 2016a)。
在再生性状的多样性研究方面, 目前共发现167种多毛类具备再生能力, 这些物种隶属于31科(Zattara, 2016a)。记载头尾都能再生的具有100种, 遍布23科; 头不能再生而尾能再生的有31种, 分布于16科; 头尾都不能再生的有6种, 分别隶属于好转虫科(Dinophilidae)、矶沙蚕科(Eunicidae)、海蛹科(Opheliidae)、刺螠科(Urchidae)、后螠科(Bonelliidae)、双栉虫科(Ampharetidae), 另外30种只有头或尾再生能力确切的记载。其中, 头部再生能力近期丢失的物种有11种: 尾部再生能力近期丢失的有1种, 这说明在多毛类的演化中发生了多次头部再生能力丢失的现象(也有可能存在头部再生能力的获得), 相对而言尾部再生则持续维持(Bely, 2010)。多毛类在演化过程再生性状多次独立的丢失, 并且很多物种再生性状丢失是快速演化而来, 这对于了解生物再生能力丢失或获得机制提供了丰富的素材。
3.3 多毛类再生性状的遗传多样性
在多毛类的不同物种间再生能力存在差异, 再生性状的遗传性机制并不相同。即使是近缘的物种之间, 再生能力也有差异, 其再生的分子机制也并不相同。裂虫和头部再生能力差异, 前者头部不能完全再生, 后者头部能完全再生。对这两个物种再生过程中转录组比较分析, 在再生过程中差异表达的基因有4771个, 其中再生相关的基因有57个,差异表达的基因有1771个, 其中再生相关的基因有54个。在两个裂虫物种的头部再生过程差异基因包括增殖, 干/多能状态维持、神经系统发育以及位置信息等(Ribeiro, 2019)。中上调的基因是和以及谷氨酰胺合成酶;上调的基因是、和(Weidhase, 2016)。
多毛类再生的细胞来源并不相同。多数毛类再生芽基主要或完全源自截断部位或附近的细胞增殖, 可能是通过细胞去分化而来(Boilly, 1965, 1968, 1969; Bely, 2014), 但在隐居纲的某些类群中, 发现存在干细胞样细胞长距离迁移至再生部位(Myohara, 2012; De Jong, 2018)。沙蚕()中也描述了细胞向截断部位的长距离迁移, 但尚未评估细胞的类型及其对再生的可能贡献(Zattara, 2016b)。再生遗传性状的多样性是生物演化的产物, 是生物生存适应和发展的前提。
3.4 多毛类再生多样性的生态学意义
多毛类是现代海洋中最为繁盛的动物类群之一, 对于维持海洋底栖及浮游生态系统的稳定具有重要作用(张树林等, 2001)。在海洋生态系统的物质循环和能量流动中, 多毛类是有机质的分解者和许多捕食者的饵料。此外, 多毛类幼虫也是经济动物幼体的滤食对象, 其担轮幼虫是对虾幼体的极好食料。沙蚕、齿吻沙蚕、吻沙蚕、矶沙蚕的许多种类, 生殖季节时在月光刺激下能大量群浮于海面, 这一习性会引起鱼类的集群。可见, 多毛类对渔场的分布、渔业资源的状况、鱼类对产卵场的选择都有较密切的关系(张树林等, 2001; 廖秀珍, 2006)。
多毛类与同为底栖生活的海洋贝类相比, 身体柔软, 容易被捕食, 其再生能力有助于多毛类在被捕食者损伤后快速修复, 具有重要的生态学意义。与此同时, 再生也意味着消耗生物体用于自身繁殖和生长等的能量。自身生长繁殖和捕食者两个因素进化权衡, 可能会导致许多物种中再生能力的丢失。另外, 物种本身的改变(如: 躲避捕食者的能力增强, 降低了受伤害的风险)又或者是生态系统中其他因素(如: 捕食者捕食能力的增强, 造成多毛类无法修复的致命伤害), 也会减少再生对环境的适应性(Lahti, 2009)。再例如, 通过疤痕快速愈合伤口的能力被认为与再生有一定冲突, 这可能是哺乳动物再生能力减弱的原因(Bely, 2010)。另一种假说是再生能力的差异也可能是物种生活策略的差异(负选择或间接选择)。生命活动最终是依靠能量, 再生能力也涉及能量分配方式, 能量最佳分配方式将在很大程度上取决于其生活史长短、个体寿命、繁殖模式。
总而言之, 再生丢失可能是自然选择的结果, 也可能是中性性状演化过程中的丢失。科学家们试图通过比较再生能力强和再生丢失的近缘物种, 收集并整合它们的生态、发育、生理等数据揭示再生丢失的生态效应(Bely, 2010)。
4 印太交汇区多毛类的分布及再生能力多样性
印太交汇区(Indo-Pacific Convergence Region)是世界上最大的海洋生物地理区域, 包涵西印度洋红岛至太平洋复活节岛的热带和亚热带水域(Erpenbeck, 2017), 其中物种多样性在珊瑚三角(Coral Triangle, CT)的中心达到峰值, 该区域以菲律宾、马来西亚半岛和新几内亚为边界。由于CT中浅水区海洋物种的高度集中, 该地区通常被称为“海洋生物多样性的庇护所” (Matias, 2018)。印太交汇区的印度太平洋群岛包括2万个岛屿, 从小环礁到大型、地质复杂的热带群岛, 例如新几内亚、苏拉威西岛和婆罗洲。
生物多样性热点是指由于物种形成率、迁移和灭绝率的空间差异而导致区域生物数量和种类异常高的地区(Mihaljević, 2017)。尽管印太交互区是世界海洋生物中心的现象被广泛所知, 但其形成和演化过程尚不明确(Keyse, 2014)。
4.1 印太交汇区多毛类资源
印太交汇区多毛类的生物资源调查相对有限, 大部分物种仅是零散的记录, 缺乏系统的分类和性状挖掘工作。根据海洋生物地理信息系统(Ocean Biogeographic Information System, OBIS), 样品采集位置主要集中在离岸比较近的浅海区。基于目前的生物资源调查情况, 该地区具有丰富多毛类多样性(见图4)。我们初步统计发现该区域多毛类有近1300种, 涵盖69科, 涵盖300多种地区特有种, 从发现的物种数量而言, 该地区低于西太平洋的海域(40—60°N, 10°W—10°E), 由于两者采样数量存在200—300倍的差异, 印太地区采样站点平均记录数量不过1000—2000, 而西太平洋地区采样站点最多的区域记录数量为200000—300000, 我们相信印太地区资源量以及物种多样性远远高于目前的统计数据。物种数量最多的前十个科分别是裂虫科(Syllidae)、沙蚕科(Nereididae)、海稚虫科(Spionidae)、多鳞虫科(polynoidae)、矶沙蚕科(Eunicidae)、蛰龙介科(Terebellidae)、龙介虫科(Serpulidae)、锡鳞虫科(Sigalionidae)、小头虫科(Capitellidae)、缨鳃虫科(Sabellidae), 见表3。其中裂虫科、海稚虫科、缨鳃虫科物种再生能力较强, 多是头尾均可再生物种。

图4 印太交汇区多毛类分布图
注: 数据来自OBIS, www.iobis.org。红点代表在采集到多毛类的站点
表3 部分多毛类印太分布统计

Tab.3 The summary of some polychaete identified in the Indo-Pacific Convergence Region
4.2 印太交汇区多毛类再生性状的特征
印太交汇区丰富的海洋生物资源为研究多毛类再生多样性和再生演化提供了一个极好的平台。我们对印太交汇区海洋生物地理信息系统中描述的多毛类物种及地理位置进行了详细统计, 并与这些物种的再生特点和演化地位进行相关性分析, 结果发现印太交汇区多毛类无论是定性还是定量的再生性状均具有丰富的多样性, 包括同一地理位置的不同生境中发生了再生性状快速演化的物种, 这些可能与其环境适应显著相关, 并可能为进一步回答该地区生物多样性的形成提供典型实例。此外, 我们还发现有若干印太交汇区起源或者独有的多毛类物种, 系统解析这些物种的再生模式也将为回答印太交汇区生物多样性的形成和维持机制提供重要突破口。
基于海洋生物地理信息系统, 发现印太交汇区有28个物种有再生能力的记录(见图5), 其中10个物种隶属裂虫科, 剩余分别是缨鳃虫科(2)、沙蚕科(3)、欧努菲虫科(3)、矶沙蚕科(2)、欧文虫科(1)丝鳃虫科 (1)、仙虫科(1)、燐虫科(1)、海蛹科(1)、叶须虫科(1)、多鳞虫科(1)、海稚虫科(1)。记录到的28个物种中沙蚕科有3个种, 矶沙蚕科有1个种, 多鳞虫科有1个种只能再生尾部, 海蛹科物种是头尾都不能再生, 仙虫科的1个种、欧努菲虫科的1个种, 缨鳃虫的1个种能再生尾部, 头部能否再生未知外, 其余都是两端能够再生的物种。灰色区域代表的是多毛类进化上比较古老的种, 见表4。这些物种主要分布于深度50—100 m之间的浅海区域。多毛类极易受到鱼虾蟹或其他物种的攻击捕食, 本身移动能力以及防御力相对较弱, 再生能力是其保证生存繁育的适应性性状。裂虫科是多毛类中最多样的科之一, 超过70个属, 近1000种。该科特点是地理分布广泛, 几乎存在于所有海洋生境中, 但是发现有一些属却只分布于印度洋、太平洋, 例如、、可能起源于印太地区(Aguado, 2015a)。
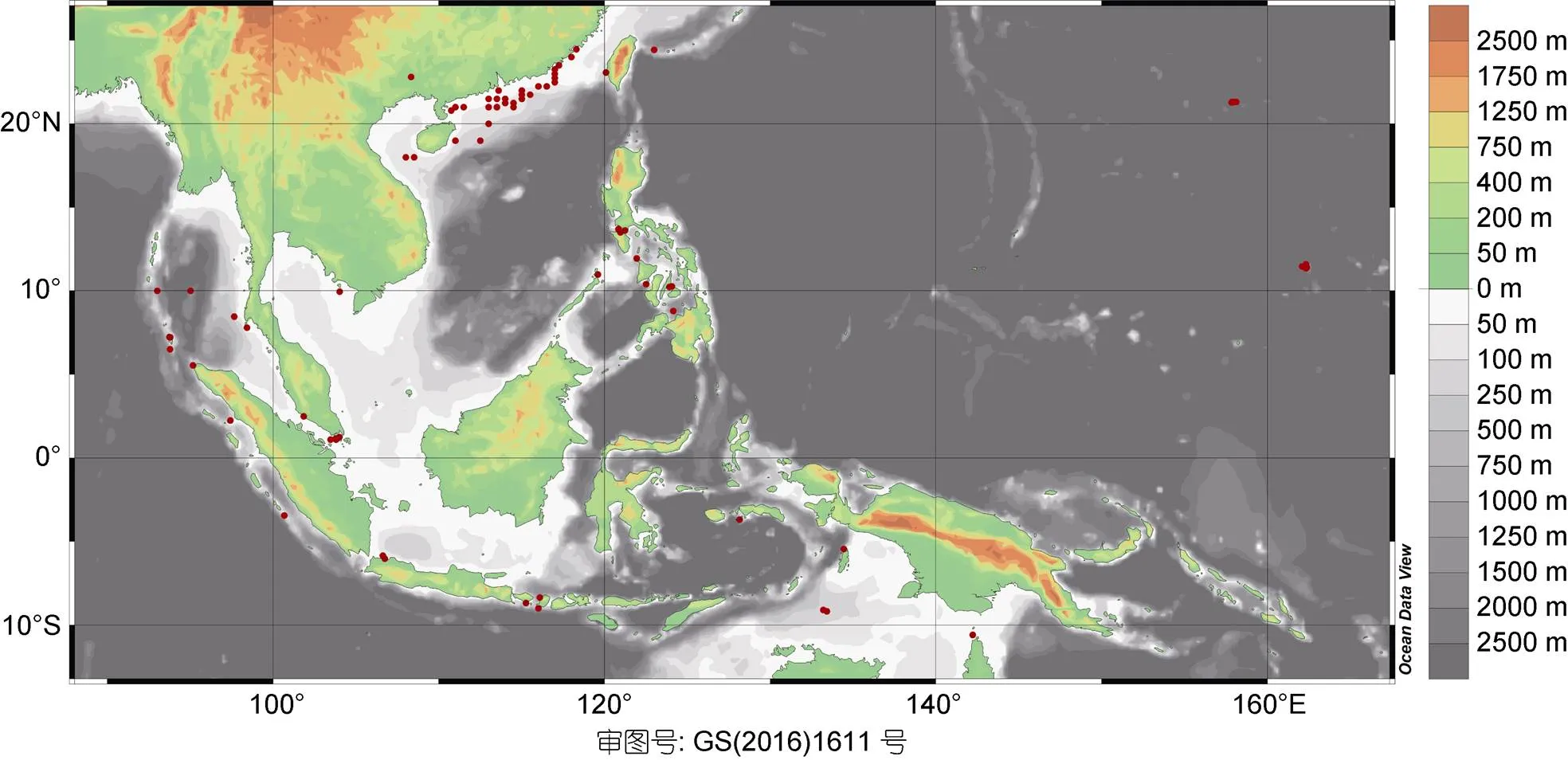
图5 印太交汇区具备再生能力的海洋多毛类分布情况
注: 红点代表在该位置发现多毛类, 右侧不同颜色代表水深/海平面以上高度
表4 印太地区再生物种汇总
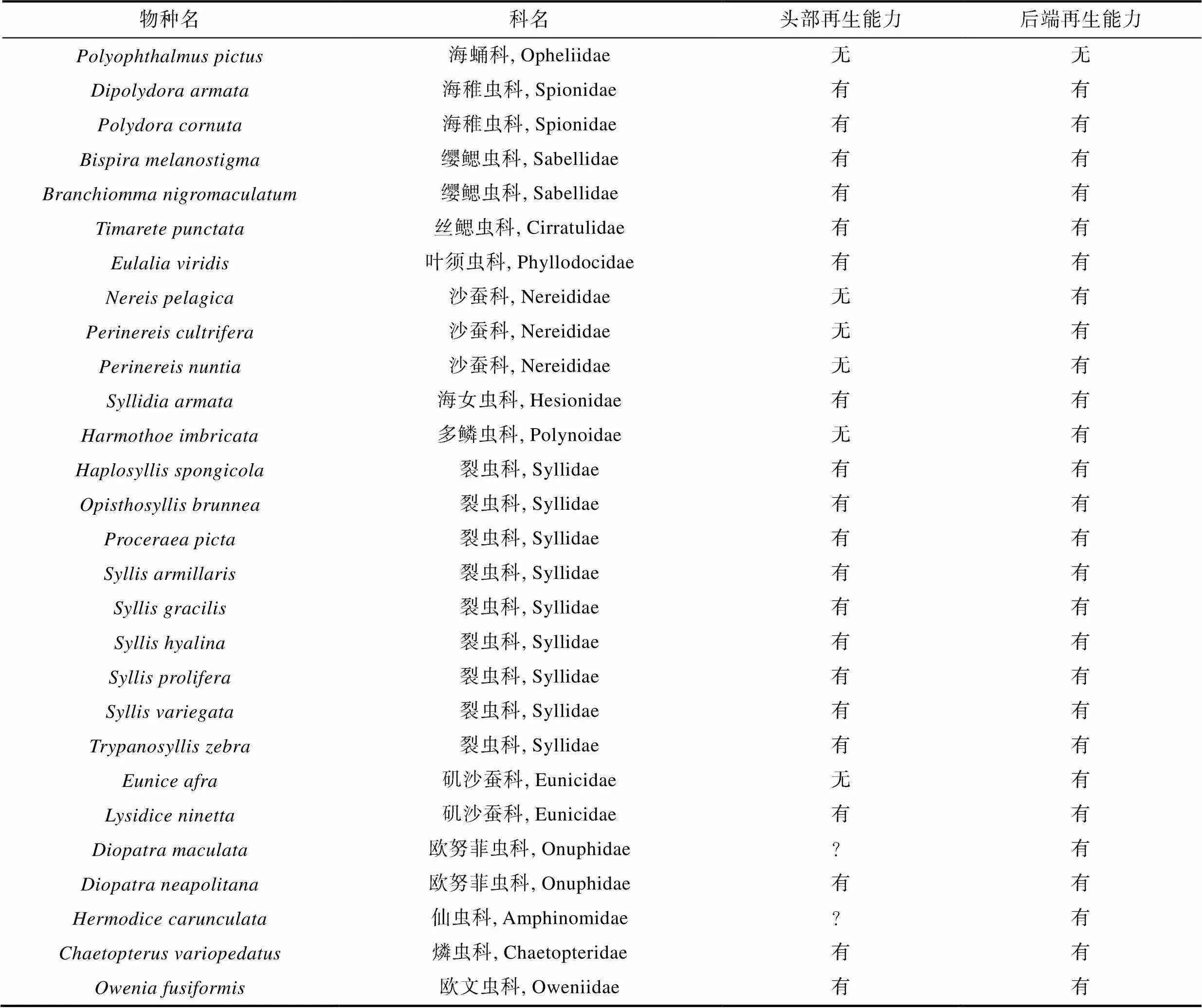
Tab.4 Summary of polychaetes that are capable of regeneration in the Indo-Pacific Convergence Region
注: 表中仙虫科、燐虫科、欧文虫科是多毛类演化上较为古老的类群
海洋生物地理信息系统(OBIS)物种信息分析显示, 印太交汇区分布着不同再生能力的物种, 如海裂虫()、细裂虫()、多育模裂虫()、杂色模裂虫(S)等能够两端再生的物种。复瓦鳞沙蚕()是鳞沙蚕科哈鳞虫属的一种, 分布于印太交汇区的浅海区域, 尾部能再生, 但头部不具备再生能力(Daly, 1973)。它生活于碎石之间的生活习性一定程度上为其提供了庇护所。印太交汇区还生活着许多隶属于同一科但再生能力存在显著差异的物种, 如海稚虫科,可头尾两端再生, 而头尾两端均不能再生(Zajac, 1995)。从它们的采集站位看, 它们分布的经度相似(120°E), 纬度不同(前者在24°N附近, 后者在10°N附近)。矶沙蚕科的巧言虫()可以头尾两端再生, 而该科的只有尾端能再生, 头部再生能力丢失(Hofmann, 1969; Bely, 2006)。有趣的是, 这两个物种的生物地理格局非常相似, 为探索多毛类再生性状分化的内在和外在因素提供了极好的材料。此外, 该地区也生活着无法再生的多毛类, 比如海蛹科内多眼虫()两端不能再生(Hyman, 1940)。根据OBIS的站点信息显示, 这种多毛类在印太主要分布在水深1200—1250 m之间, 此处相较于浅海区域环境相对稳定, 且被捕食压力较小。总之, 印太地区有着丰富的再生演化多样性, 以及该地区丰富的物种以及复杂的生境, 为了再生演化的提供丰富的实验材料。此外, 我们发现多种多毛类是印太地区特有的或者可能起源于印太交汇区(Aguado, 2015a)。
4.3 印太交汇区多毛类再生性状形成的环境影响因素
再生对生物适应环境具有重要的生态学意义, 研究内在遗传和外在环境因素对多毛类再生能力多样性形成的驱动机制, 将为回答印太交汇区海洋生物多样性中心的形成和演化过程提供典型实例。我们初步统计具有不同再生性状的物种在印太交汇区的地理分布格局, 发现头尾均为再生的物种可能更倾向于分布在印太交汇区的北部海域, 而头部不能再生尾部可再生的物种则在某种程度上多分布于印太交汇区的西侧海域。考虑到目前印太交汇区多毛类的生物资源调查尚不完善, 这种再生性状特异性的地理分布格局需要更广泛的生物资源调查数据支持。但再生性状地理分布差异也从另一个方面暗示关键地质事件和海洋气候因素可能是此地区再生多样性形成的重要驱动力。外在的环境因素可能对多毛类再生性状具备不同程度的选择压力, 从而决定了生物再生能力的保留或者丢失。未来应该针对海洋多毛类通过更系统的生物资源调查解析再生性状多样性的地理分布格局, 进一步聚焦印太交汇区不同区域近期演化或者快速演化的再生性状, 从性状和分子的角度解析这些再生性状多样性形成的机制。整合再生性状和及其背后DNA分子的演化生物学分析, 推演再生性状起源、适应和演化的速度和时间节点。并在此基础上, 结合暖池、洋流等关键的地质和环境事件, 揭示大气圈-水圈-岩石圈的多圈层如何互作进而影响生物圈生物再生多样性的形成和演化机制。
此外, 海洋环境中的pH、盐度、温度等非生物因素都会影响多毛类的再生能力, 而构造过程、暖池形成、环流变异及气候变化等古环境过程改变了古海洋环境的pH、盐度、温度等非生物因素, 可能在塑造印太交汇区多毛类再生性状多样性中具有重要作用。在对巢沙蚕()的研究中发现其在较低pH, 以及较低(21)、较高(42)的盐度下再生能力会受到抑制, 温度高于24°C会促进其再生(Pires, 2015)。在比较寄生型短触手才女虫()不同温度下的再生能力时发现高温(24°C)下再生速度更快, 而在低温(14°C)下再生所需时间是前者的两倍多(David, 2012)。热带地区的海洋尤其是太平洋中的“热带辐合带”由于降水丰沛和携带大量淡水的河流的作用, 盐度都比较低。同时, 河流入海会携带丰富的营养物质, 造就了该区域的物种多样性, 同时也为该地区物种再生能力演化提供了物质基础, 印太地区拥有世界上范围最大、海表温度最高的大“暖池”, 是全球热带对流最强、水汽含量最多的区域, 海气相互作用极为强烈(Wang, 2001)。全年温度较高大于28°C这样的环境, 这对温度较高能促进多毛类再生, 由于光和温度直接影响生物量和/或丰度, 也提高了生物体对化学能的利用率。因为修复再生是非常耗能的过程。总之, 在印太交汇区多毛类再生性状的适应性演化中, 应该通过生物学多样性调查, 分析海洋多毛类的再生性状模式, 获得再生性状多样性的分布格局和演化路径。借助功能组学和遗传操作等工具, 解析再生多样性的基因表达调控模式和分子基础, 构建再生演化关键节点的基因调控网络, 阐述再生性状对环境因子的适应机制。
5 结论与展望
再生是生命科学的本质问题之一, 是《Science》公布的全世界最前沿的125个科学问题之一。再生现象在生物界广为存在, 但不同物种的再生能力却存在巨大差异。再生能力多样性是生物多样性的重要组成成分, 是生物不断适应环境的结果。海洋多毛类作为典型的底栖生物, 在底栖生态系统的能量流动和物质循环中发挥着重要的作用, 是整个海洋生态系统不可或缺的组成成分。海洋多毛类因其多样且快速演化的再生能力和简单线性的分节结构成为研究生物再生能力多样性的极佳类群。印太交汇区是世界海洋生物多样性中心, 孕育了千姿百态的多毛类, 为我们研究生物再生多样性提供了极佳的研究平台。更为重要的是, 该地区多毛类再生多样性的形成是受到暖池、洋流等关键地质和环境事件驱动形成, 为揭示大气圈-水圈-岩石圈的多圈层如何互作进而影响生物圈生物多样性中心形成和演化机制提供了典型实例。
目前我国多毛类再生多样性研究仍存在许多不足之处, 尚且缺乏系统性的生物资源调查和分类研究。当然, 这也与多毛类普遍个体较小, 以及栖息环境采样困难相关。此外, 目前上尚无开发成功的多毛类实验模式种, 限制了该类群再生多样性的深入机制研究。最近小头虫中CRISPR/Cas9基因编辑等功能基因组学平台的成功建立为多毛类再生多样性的功能研究提供了可行性(Hsu, 2014; Neal, 2019)。我们相信随着我国海洋生物资源调查能力和功能基因组学等海洋生物学实验技术的飞速发展, 科学家们也能够在再生多样性研究方面取得长足发展, 从一定程度上解析印太交汇区再生性状多样性背后的分子机制, 并阐述关键地质及海洋环境等外在因素塑造再生多样性的驱动效应。
张均龙, 史本泽, 赵 峰等, 2016. 中国海洋底栖生物学发展回顾与展望. 海洋科学集刊, (1): 194—204
张树林, 周一兵, 李永函, 2001. 多毛类生活史的研究综述. 天津农学院学报, 8(2): 35—37, 61
廖秀珍, 2006. 海洋底栖多毛类的分子系统发育学研究. 厦门: 厦门大学硕士学位论文, 1—10
Aguado M T, Glasby C J, 2015a. Indo-Pacific Syllidae (Annelida, Phyllodocida) share an evolutionary history. Systematics and Biodiversity, 13(4): 369—385
Aguado M T, Helm C, Weidhase M, 2015b. Description of a new syllid species as a model for evolutionary research of reproduction and regeneration in annelids. Organisms Diversity & Evolution, 15(1): 1—21
Alvarado A S, 2006. Planarian regeneration: its end is its beginning. Cell, 124(2): 241—245
Andreatta G, Broyart C, Borghgraef C, 2020. Corazonin signaling integrates energy homeostasis and lunar phase to regulate aspects of growth and sexual maturation in. Proceedings of the National Academy of Sciences of the United States of America, 117(2): 1097—1106
Bely A E, 2006. Distribution of segment regeneration ability in the annelida. Integrative and Comparative Biology, 46(4): 508—518
Bely A E, 2010. Evolutionary loss of animal regeneration: pattern and process. Integrative and Comparative Biology, 50(4): 515—527
Bely A E, 2014. Early events in annelid regeneration: a cellular perspective. Integrative and Comparative Biology, 54(4): 688—699
Bely A E, Nyberg K G, 2010a. Evolution of animal regeneration: re-emergence of a field. Trends in Ecology & Evolution, 25(3): 161—170
Bely A E, Sikes J M, 2010b. Acoel and platyhelminth models for stem-cell research. Journal of Biology, 9(2): 14
Bely A E, Sikes J M, 2010c. Latent regeneration abilities persist following recent evolutionary loss in asexual annelids. Proceedings of the National Academy of Sciences of the United States of America, 107(4): 1464—1469
Birbrair A, Zhang T, Wang Z M, 2013. Role of pericytes in skeletal muscle regeneration and fat accumulation. Stem Cells and Development, 22(16): 2298—2314
Blum N, Begemann G, 2015. Osteoblast de- and redifferentiation are controlled by a dynamic response to retinoic acid during zebrafish fin regeneration. Development, 142(17): 2894—2903
Boilly B, 1965. Origine du mésoderme dans la régénération postérieure chezQuatrefages (Annélide Polychète). Comptes Rendus Hebdomadaires Des Seances De L Academie Des Sciences, 261(6): 1561—1564
Boilly B, 1968. Origine des cellules de régénération chezClap. (Annélide Polychète). Arch d’Anat Microscop Morphol Experiment, 57: 297—308
Boilly B, 1969. Origine des cellules régénératrices chezO.F. Müller (Annélide Polychète). Wilhelm Roux’ Archiv für Entwicklungsmechanik der Organismen, 162(3): 286—305
Bosch T C G, 2007. Why polyps regenerate and we don’t: towards a cellular and molecular framework forregeneration. Developmental Biology, 303(2): 421—433
Brown F D, Keeling E L, Le A D, 2009. Whole body regeneration in a colonial ascidian,. Journal of Experimental Zoology Part B: Molecular and Developmental Evolution, 312B(8): 885—900
Christen B, Beck C W, Lombardo A, 2003. Regeneration-specific expression pattern of three posterior hox genes. Developmental Dynamics, 226(2): 349—355
Colbath G K, 1987. Evidence for shedding of maxillary jaws in eunicoid polychaetes. Journal of Natural History, 21(2): 443—447
Daly J, 1973. Segmentation, autotomy and regeneration of lost posterior segments in(L.) (Polychaeta: Polynoidae). Mauri Ora, 1: 17—28
Dannenberg L C, Seaver E C, 2018. Regeneration of the germline in the annelid. Developmental Biology, 440(2): 74—87
David A A, Williams J D, 2012. Asexual reproduction and anterior regeneration under high and low temperatures in the sponge associate(Polychaeta: Spionidae). Invertebrate Reproduction & Development, 56(4): 315—324
De Jong D M, Seaver E C, 2016. A stable thoracic hox code and epimorphosis characterize posterior regeneration in. PLoS One, 11(2): e0149724
De Jong D M, Seaver E C, 2018. Investigation into the cellular origins of posterior regeneration in the annelid. Regeneration, 5(1): 61—77
De Rosa R, Prud'homme B, Balavoine G, 2005.and-skipped in the annelidand the ancestry of posterior growth. Evolution & Development, 7(6): 574—587
DuBuc T Q, Traylor-Knowles N, Martindale M Q, 2014. Initiating a regenerative response; cellular and molecular features of wound healing in the cnidarian. BMC Biology, 12(1): 24
Erpenbeck D, Aryasari R, Benning S, 2017. Diversity of two widespread Indo-Pacific demosponge species revisited. Marine Biodiversity, 47(4): 1035—1043
Freitas R, Pires A, Velez C, 2016. Effects of seawater acidification on(Polychaete, Onuphidae): biochemical and regenerative capacity responses. Ecological Indicators, 60: 152—161
Fröbisch N B, Bickelmann C, Witzmann F, 2014. Early evolution of limb regeneration in tetrapods: evidence from a 300-million-year-old amphibian. Proceedings of the Royal Society B: Biological Sciences, 281(1794): 20141550
Gazave E, Guillou A, Balavoine G, 2014. History of a prolific family: the-related genes of the annelid. EvoDevo, 5(1): 29
Gehrke A R, Neverett E, Luo Y J, 2019. Acoel genome reveals the regulatory landscape of whole-body regeneration. Science, 363(6432): eaau6173
Giani V C Jr, Yamaguchi E, Boyle M J, 2011. Somatic and germline expression ofduring development and regeneration in the marine polychaete annelid. Evodevo, 2(1): 10
Gilbert S F, 2000. Developmental Biology. USA: Sinauer Associates, 693—723
Grimmel J, Dorresteijn A W C, Fröbius A C, 2016. Formation of body appendages during caudal regeneration in: adaptation of conserved molecular toolsets. Evodevo, 7(1): 10
Han J, Morris S C, Hoyal Cuthill J F, 2019. Sclerite-bearing annelids from the lower Cambrian of South China. Scientific Reports, 9(1): 4955
Han M J, Yang X D, Taylor G, 2005. Limb regeneration in higher vertebrates: developing a roadmap. Anat Rec B New Anat, 287B(1): 14—24
He S N, Del Viso F, Chen C Y, 2018. An axial hox code controls tissue segmentation and body patterning in. Science, 361(6409): 1377—1380
Hentschel B T, Harper N S, 2006. Effects of simulated sublethal predation on the growth and regeneration rates of a spionid polychaete in laboratory flumes. Marine Biology, 149(5): 1175—1183
Hill S D, 1970. Origin of the regeneration blastema in Polychaete Annelids. American Zoologist, 10(2): 101—112
Hill S D, 1972. Caudal regeneration in the absence of a brain in two species of sedentary polychaetes. Journal of Embryology and Experimental Morphology, 28(3): 667—680
Hobmayer B, Rentzsch F, Kuhn K, 2000.signalling molecules act in axis formation in the diploblastic metazoan. Nature, 407(6801): 186—189
Hofmann D K, 1969. Untersuchungen über die regeneration des prostomiums und des hinterendes beim polychaetengray. Zoologischer Anzeiger - A Journal of Comparative Zoology, 33: 253—260
Hofmann D K, 1974. Maturation, epitoky and regeneration in the polychaeteunder field and laboratory conditions. Marine Biology, 25(2): 149—161
Hofmann D K, 1975. Prostomial regeneration in the polychaete(Audouin et Milne-Edwards) (Annelida). Wilhelm Roux’s Archives of Developmental Biology, 177(4): 309—322
Hsu P D, Lander E S, Zhang F, 2014. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 157(6): 1262—1278
Hutchins E D, Markov G J, Eckalbar W L, 2014. Transcriptomic analysis of tail regeneration in the lizardreveals activation of conserved vertebrate developmental and repair mechanisms. PLoS One, 9(8): e105004
Hyman L H, 1940. Aspects of regeneration in annelids. The American Naturalist, 74(755): 513—527
Janssen R, Le Gouar M, Pechmann M, 2010. Conservation, loss, and redeployment of wnt ligands in protostomes: implications for understanding the evolution of segment formation. BMC Evolutionary Biology, 10(1): 374
Jouin C, 1979. Description of a free-living Polychaete without gut:n. gen., n. sp. (Protodrilidae, Archiannelida). Canadian Journal of Zoology, 57(12): 2448—2456
Juliano C E, Swartz S Z, Wessel G M, 2010. A conserved germline multipotency program. Development, 24(137): 4113—4126
Keyse J, Crandall E D, Toonen R J, 2014. The scope of published population genetic data for Indo-Pacific marine fauna and future research opportunities in the region. Bulletin of Marine Science, 90(1): 47—78
Kostyuchenko R P, Kozin V V, Filippova N A, 2019.expression pattern in two polychaete species,and: Examination of the conserved key regulator of the gut development from cleavage through larval life, postlarval growth, and regeneration. Developmental Dynamics, 248(8): 728—743
Kozin V V, Kostyuchenko R P, 2015.,, andgene expression during caudal regeneration of the polychaete annelid. Development Genes and Evolution, 225(3): 129—138
Lahti D C, Johnson N A, Ajie B C, 2009. Relaxed selection in the wild. Trends in Ecology & Evolution, 24(9): 487—496
Lai A G, Aboobaker A A, 2018. EvoRegen in animals: time to uncover deep conservation or convergence of adult stem cell evolution and regenerative processes. Developmental Biology, 433(2): 118—131
Licciano M, Murray J M, Watson G J, 2012. Morphological comparison of the regeneration process inand(Annelida, Sabellida). Invertebrate Biology, 131(1): 40—51
Liu S Y, Selck C, Friedrich B, 2013. Reactivating head regrowth in a regeneration-deficient planarian species. Nature, 500(7460): 81—84
Marks E P, Leopold R A, 1970. Cockroach leg regeneration: effects of ecdysterone in vitro. Science, 167(3914): 61—62
Matias A M A, Riginos C, 2018. Revisiting the “centre hypotheses” of the Indo-West Pacific: idiosyncratic genetic diversity of nine reef species offers weak support for the coral triangle as a centre of genetic biodiversity. Journal of Biogeography, 45(8): 1806—1817
Mellon D Jr, Stephens P J, 1978. Limb morphology and function are transformed by contralateral nerve section in snapping shrimps. Nature, 272(5650): 246—248
Mihaljević M, Korpanty C, Renema W, 2017. Identifying patterns and drivers of coral diversity in the central Indo-Pacific marine biodiversity hotspot. Paleobiology, 43(3): 343—364
Mortimer K, Mackie A S Y, 2014. Morphology, feeding and behaviour of British(Annelida: Magelonidae), with discussions on the form and function of abdominal lateral pouches. Memoirs of Museum Victoria, 71: 177—201
Müller M C M, Berenzen A, Westheide W, 2003. Experiments on anterior regeneration in(“Polychaeta”, Amphinomidae): reconfiguration of the nervous system and its function for regeneration. Zoomorphology, 122(2): 95—103
Myohara M, 2012. What role do annelid neoblasts play? A comparison of the regeneration patterns in a neoblast-bearing and a neoblast-lacking Enchytraeid Oligochaete. PLoS One, 7(5): e37319
Neal S, Jong D M D, Seaver E C, 2019. CRISPR/CAS9 mutagenesis of a single r-opsin gene blocks phototaxis in a marine larva. Proceedings of the Royal Society B: Biological ences, 1904(286): 20182491
Niwa N, Akimoto-Kato A, Sakuma M, 2013. Homeogenetic inductive mechanism of segmentation in polychaete tail regeneration. Developmental Biology, 381(2): 460—470
Novikova E L, Bakalenko N I, Nesterenko A Y, 2013. Expression of hox genes during regeneration of nereid polychaete()(Annelida, Lophotrochozoa). Evodevo, 4(1): 14
Novikova E L, Bakalenko N I, Nesterenko A Y, 2016. Hox genes and animal regeneration. Russian Journal of Developmental Biology, 47(4): 173—180
Nowoshilow S, Schloissnig S, Fei J F, 2018. The axolotl genome and the evolution of key tissue formation regulators. Nature, 559(7712): E2
Okada Y K, 1929. Regeneration and fragmentation in the Syllidian Polychaetes. Wilhelm Roux’ Archiv Für Entwicklungsmechanik der Organismen, 115(3): 542—600
Okada Y K, 1938. An internal factor controlling posterior regeneration in syllid polychaetes. Journal of the Marine Biological Association of the United Kingdom, 23(1): 75—78
Özpolat B D, Bely A E, 2015. Gonad establishment during asexual reproduction in the annelid. Developmental Biology, 405(1): 123—136
Özpolat B D, Bely A E, 2016. Developmental and molecular biology of annelid regeneration: a comparative review of recent studies. Current Opinion in Genetics & Development, 40: 144—153
Paulus T, Müller M C M, 2006. Cell proliferation dynamics and morphological differentiation during regeneration in(Polychaeta, Dorvilleidae). Journal of Morphology, 267(4): 393—403
Pfeifer K, Dorresteijn A W C, Fröbius A C, 2012. Activation ofgenes during caudal regeneration of the polychaete annelid. Development Genes and Evolution, 222(3): 165—179
Pfeifer K, Schaub C, Wolfstetter G, 2013. Identification and characterization of aortholog in the polychaete annelidreveals mesodermal expression of. Development Genes and Evolution, 223(5): 319—328
Pires A, Almeida Â, Correia J, 2016. Long-term exposure to caffeine and carbamazepine: impacts on the regenerative capacity of the polychaete. Chemosphere, 146: 565—573
Pires A, Figueira E, Moreira A, 2015. The effects of water acidification, temperature and salinity on the regenerative capacity of the polychaete. Marine Environmental Research, 106: 30—41
Pires A, Freitas R, Quintino V, 2012. Can(Annelida: Onuphidae) regenerate body damage caused by bait digging or predation? Estuarine, Coastal and Shelf Science, 110: 36—42
Planques A, Malem J, Parapar J, 2019. Morphological, cellular and molecular characterization of posterior regeneration in the marine annelid. Developmental Biology, 445(2): 189—210
Poss K D, 2010. Advances in understanding tissue regenerative capacity and mechanisms in animals. Nature Reviews Genetics, 11(10): 710—722
Potswald H E, 1969. Cytological observations on the so-called neoblasts in the serpulid. Journal of Morphology, 128(2): 241—259
Prud'homme B, de Rosa R, Arendt D, 2003. Arthropod-like expression patterns ofandin the annelidsuggest a role in segment formation. Current Biology, 13(21): 1876—1881
Prud’homme B, de Rosa R, Arendt D, 2003. Arthropod-like expression patterns ofandin the annelidsuggest a role in segment formation. Current Biology, 13(21): 1876—1881
Pruitt M M, Letcher E J, Chou H C, 2014. Expression of thegene complement in a spiral-cleaving embryo and trochophore larva. International Journal of Developmental Biology, 58(6—8): 563—573
Reddien P W, Alvarado A S, 2004. Fundamentals of planarian regeneration. Annual Review of Cell and Developmental Biology, 20: 725—757
Rhoads D C, Boyer L F, 1982. The effects of marine benthos on physical properties of sediments - a successional perspective. In: McCall P L, Tevesz M J S eds. Animal-Sediment Relations. Boston, USA: Springer, 3—52
Ribeiro R P, Ponz-Segrelles G, Bleidorn C, 2019. Comparative transcriptomics in Syllidae (Annelida) indicates that posterior regeneration and regular growth are comparable, while anterior regeneration is a distinct process. BMC Genomics, 20(1): 855
Robb S M C, Alvarado A S, 2014. Histone modifications and regeneration in the Planarian. Current Topics in Developmental Biology, 108: 71—93
Satoh A, Bryant S V, Gardiner D M, 2008. Regulation of dermal fibroblast dedifferentiation and redifferentiation during wound healing and limb regeneration in the Axolotl. Development, Growth & Differentiation, 50(9): 743—754
Schulze A, Rice M E, 2009. Musculature in sipunculan worms: ontogeny and ancestral states. Evolution & Development, 11(1): 97—108
Scimone M L, Atabay K D, Fincher C T, 2020. Muscle and neuronal guidepost-like cells facilitate planarian visual system regeneration. Science, 368(6498): eaba3203
Seaver E C, Thamm K, Hill S D, 2005. Growth patterns during segmentation in the two polychaete annelids,sp. I and: comparisons at distinct life history stages. Evolution & Development, 7(4): 312—326
Sikes J M, Newmark P A, 2013. Restoration of anterior regeneration in a planarian with limited regenerative ability. Nature, 500(7460): 77—80
Solan M, Cardinale B J, Downing A L, 2004. Extinction and ecosystem function in the marine benthos. Science, 306(5699): 1177—1180
Srivastava M, Mazza-Curll K L, van Wolfswinkel J C, 2014. Whole-body acoel regeneration is controlled by wnt and bmp-admp signaling. Current Biology, 24(10): 1107—1113
Sugiura T, Wang H, Barsacchi R, 2016. MARCKS-like protein is an initiating molecule in axolotl appendage regeneration. Nature, 531(7593): 237—240
Szabó R, Ferrier D E K, 2014. Cell proliferation dynamics in regeneration of the operculum head appendage in the annelid. Journal of Experimental Zoology Part B: Molecular and Developmental Evolution, 322(5): 257—268
Takeo M, Chou W C, Sun Q, 2013. Wnt activation in nail epithelium couples nail growth to digit regeneration. Nature, 499(7457): 228—232
Takeo M, Yoshida-Noro C, Tochinai S, 2010. Functional analysis of, a novel gene required for mesodermal cell proliferation at an initial stage of regeneration in(Enchytraeidae, Oligochaete). International Journal of Developmental Biology, 54(1): 151—160.
Tanaka H V, Ng N C Y, Yu Z Y, 2016. A developmentally regulated switch from stem cells to dedifferentiation for limb muscle regeneration in newts. Nature Communications, 7(1): 11069
Wang C Z, Enfield D B, 2001. The tropical western hemisphere warm pool. Geophysical Research Letters, 28(8): 1635—1638
Weidhase M, Beckers P, Bleidorn C, 2016. On the role of the proventricle region in reproduction and regeneration in(Annelida: Syllidae). BMC Evolutionary Biology, 16(1): 196
Weidhase M, Bleidorn C, Helm C, 2014. Structure and anterior regeneration of musculature and nervous system incf.(Cirratulidae, Annelida). Journal of Morphology, 275(12): 1418—1430
Weidhase M, Helm C, Bleidorn C, 2015. Morphological investigations of posttraumatic regeneration incf.(Annelida: Cirratulidae). Zoological Letters, 1(1): 20
Williams E A, Jékely G, 2016. Towards a systems-level understanding of development in the marine annelid. Current Opinion in Genetics & Development, 39: 175—181
Zajac R N, 1995. Sublethal predation on(Polychaeta: Spionidae): patterns of tissue loss in a field population, predator functional response and potential demographic impacts. Marine Biology, 123(3): 531—541
Zattara E E, Bely A E, 2011. Evolution of a novel developmental trajectory: fission is distinct from regeneration in the annelid. Evolution & Development, 13(1): 80—95
Zattara E E, Bely A E, 2016a. Phylogenetic distribution of regeneration and asexual reproduction in Annelida: regeneration is ancestral and fission evolves in regenerative clades. Invertebrate Biology, 135(4): 400—414
Zattara E E, Turlington K W, Bely A E, 2016b. Long-term time-lapse live imaging reveals extensive cell migration during annelid regeneration. BMC Developmental Biology, 16(1): 6
Zeng A, Li H, Guo L H, 2018. Prospectively isolated tetraspanin+neoblasts are adult pluripotent stem cells underlying planaria regeneration. Cell, 173(7): 1593—1608.e20
THE EVOLUTION OF REGENERATION IN MARINE POLYCHAETE
ZHANG Lin-Lin1, 2, 3, 4, WENG Jie-Yang1, 4, LI Qian1, 4
(1. Key Laboratory of Experimental Marine Biology, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China; 2. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China; 3. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China; 4. University of Chinese Academy of Sciences, Beijing 100049, China)
Regeneration, the ability to re-grow and repair lost tissues or organs, is one of the essential issues of biological sciences. Regeneration is widely distributed in animals but varies dramatically within or among animal phyla. Recent findings have identified key cellular and molecular principles underlying animal regeneration. However, the genetic mechanism driving the formation of species regeneration diversity is still unknown. Due to their diversity of regeneration ability, rapid gain and loss on phylogeny, and relatively simple segmented body plan, marine polychaete has been recognized as one of the excellent models to study the evolution of regeneration. We first summarize here recent advances in the regeneration evolution of marine polychaete in the context of ecology and evolution biology. We further review the cellular and molecular basis of regeneration, as well as the diversity of regeneration characteristics. In addition, we analyze the characteristics of marine polychaete regeneration traits in the Indo-Pacific Convergence Region, the potential driving effects of multi-circle factors on regeneration traits, and put forward suggestions on future trends.
regeneration diversity; marine polychaete; adaptative evolution; Indo-Pacific Convergence Region; Evo-Devo
* 中国科学院B类战略先导科技专项, XDB42000000号; 国家自然科学基金面上项目, 41976088号; 中国科学院海洋大科学研究中心重点部署项目, COMS2019R01号; 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室引进人才支持计划项目, YJ2019NO01号。张琳琳, 博士, 研究员, E-mail: linlinzhang@qdio.ac.cn
翁洁羊, 研究助理, E-mail: wongjieyang@qdio.ac.cn
2020-07-13,
2020-09-06
Q951+.3
10.11693/hyhz20200700207

