海洋动物适应性演化与多样性研究进展*
张琳琳 李勇男 翁洁羊 李 杰 王 月 李 倩
海洋动物适应性演化与多样性研究进展*
张琳琳1, 2, 3, 4李勇男1, 2, 3①翁洁羊1, 4李 杰1, 4王 月1, 4李 倩1, 4
(1. 中国科学院海洋研究所 实验海洋生物学重点实验室 青岛 266071; 2. 中国科学院海洋大科学研究中心 青岛 266071; 3. 青岛海洋科学与技术试点国家实验室 海洋生物学与生物技术功能实验室 青岛 266237; 4. 中国科学院大学 北京 100049)
海洋生物多样性是海洋生态系统的重要组成部分, 了解海洋生物多样性的形成演化过程不仅是生命科学的本质问题, 而且对海洋生物资源的保护和开发利用具有重要的指导意义。目前海洋生物多样性的形成演化机制尚不明确, 而性状多样性的起源和适应性演化为回答海洋生物多样性的内源性机制提供了重要突破口。本文从演化发育生物学的角度综述了海洋动物适应性性状、其研究方法和新兴模式生物的研究进展, 重点阐述了印太交汇区的海洋动物适应性演化研究进展, 探讨了该区域生物多样中心形成的驱动力, 并就未来海洋生物多样性与适应性演化研究提出了建议。
海洋动物多样性; 适应性演化; 印太交汇区; 演化发育生物学
海洋的生物资源十分丰富, 在全球生物多样性中占有重要位置。生物多样性是指地球上生物圈内所包含的所有生物及其生存环境, 主要包括物种多样性、遗传多样性和生态多样性(蔡立哲, 2006)。生物多样性中心是现代全球生物多样性格局的突出特征。在过去的五千万年中, 伴随着主要板块构造事件的变化, 海洋生物多样性中心由西特提斯地区穿越近半个地球迁移到了印太交汇区(Renema, 2008)。这种迁移凸显了环境变化与生物多样性模式的联系, 对于解释海洋生物多样性中心起源和演化提供了新的切入点。
东印度洋-西太平洋是一个涵盖热带和亚热地区的海洋领域, 西起红海、波斯湾经波利尼西亚东至复活节岛(Erpenbeck, 2017), 复杂的水文、地质和气候环境, 孕育了全球最大的海洋生物多样性中心。在过去的五千万年中, 出现过三个海洋生物多样性中心。在新生代时期, 位于特提斯地区的海洋生物多样性中心迁移到阿拉伯地区, 在中新世早期(23—16 Ma)到达了印度洋-太平洋, 即如今的Indo-Malay Archipelago (IMA)地区(Renema, 2008; Hui, 2016)。其中, 印太交汇区(Indo-Pacific Convergence Region)浅海的珊瑚礁大三角(Coral Triangle)是全球海洋生物多样性最高的区域, 该区域从菲律宾北部开始延伸, 西南穿过马来半岛, 向西延伸到新几内亚以外, 其海洋物种丰富度是热带西部大西洋的两倍至几倍(Russell, 2016), 孕育了全球76%的造礁珊瑚物种, 有超过 3000 种鱼类, 数千种软体动物、甲壳类和棘皮动物等(Briggs, 2007), 被称为海洋多样性的“珠峰”, 是研究海洋生物多样性起源和演化的典型区域。目前该地区生物多样性中心的形成演化机制尚不明确, 海洋生物的适应性性状作为生物多样性的基本元素, 其起源和演化机制无疑为回答生物多样性的形成和演化问题提供了重要突破口。
适应性演化是指生物在外界环境的选择下做出适应性改变的动态演化过程(Mayr, 1982)。在遗传变异, 漂变和自然选择的相互作用下, 自然界演化出千姿百态的适应性性状, 而这些性状是生物多样性的基本元素, 对于我们深入探讨生物多样性的起源和演化具有重要的意义。因此, 本文将从演化发育生物学的角度在海洋动物适应性性状以及性状多样性形成演化的外在、内在驱动力等方面进行综述, 希望为印太交汇区生物多样性中心形成演化机制的研究提供借鉴。
1 海洋环境的显著特点
海洋特殊复杂的环境是海洋生物多样性形成和演化的重要驱动力。过去的6亿年中, 在全球地质演化的推动作用下, 洋底地形发生了重大变化, 形成了一种复杂而又规律的独特地貌(Poulton, 2011)。根据板块构造理论, 地球是由许多板块构成的, 它们漂浮在地幔表面, 可以彼此分散、汇聚或滑动。当两个板块分开时, 岩浆从地幔中上涌并冷却, 生成新的地壳, 形成洋脊; 而当大洋板块和大陆板块汇聚时, 大洋板块俯冲到大陆板块之下, 相互摩擦, 形成长长的“V”字型海沟。作为海洋生态系统的重要组成部分, 除洋脊和海沟外, 洋底还有平原、海山、热液和冷泉等多种复杂环境, 孕育和形成了独特的生命过程和生态系统, 推动了海洋生物多样性的形成(李新正等, 2019)。
海水覆盖了地球表面的70%, 其物理和化学性质会随着纬度、深度、靠近陆地程度和淡水输入量而变化, 可以反映岩石和沉积物的侵蚀、火山活动、与大气的气体交换、生物的代谢以及降雨等过程, 对于海洋生物的生命活动具有重要的影响。海水的3.5%是由溶解的化合物组成, 而另外96.5%是纯水。海水中已发现的元素近80种, 绝大部分呈离子状态, 主要有氯、钠、镁、硫、钙、钾、溴、碳、锶、硼、氟、氮和磷等, 是海洋生物所必需的元素(Gupta, 1993)。此外, 溶解氧也是海洋生命活动不可缺少的物质, 其来源主要是大气和浮游植物的光合作用(Keeling, 2010)。在化学过程、物理过程和生物过程的多重影响下, 氧在海洋中呈垂直分布, 这种分布特征进一步影响了生物的分布和活动。光照也是海洋环境的重要生态因素, 它既可以通过影响海洋有机物的产生, 又可以凭借其自身的分布和变化特点来直接或间接的影响海洋生物的生存、分布、数量、行为以及一些特殊的适应性表现。一般水的深度越大, 穿透的光线就越少, 当达到一定深度时, 就没有任何光线了。这片漆黑的区域占据了海洋的大部分, 被称为无光带, 其上方依次是弱光带和透光带。生物根据其习性和独特的适应能力生活在不同的光带区。
作为一种流体, 海水处于不停地运动之中。洋流(海流)是海水的一种重要运动形式, 指表层海水在外力作用下常年大规模沿某一定方向稳定地流动。洋流是地球能量交换的主要调节者, 对海洋中多种生物过程都具有重要的影响与作用。在洋流交汇的海区, 海水受到扰动, 把下层丰富的营养物质带到表层, 并且形成“水障”, 促进了小生命有机体的繁殖和聚集, 为其他生物提供了丰富的食物, 吸引了众多生物在此处聚集、生存和繁殖, 丰富了海洋生态系统的生物多样性(Garrison, 2012)。
2 海洋动物的适应性性状
海洋中生活着各种各样的生物, 它们是不断适应环境演化而来的。动物界里32个门类中, 海洋生态系统中就有31个, 其中14个门类是海洋所特有的(李新正, 2000)。海洋中生物分布的范围很广, 从赤道到两极水域, 从海水表层到超深渊, 从潮间带的海岸到超深渊带的海沟, 生物无处不在。按照生存环境海洋生物可大致分为浮游生物、游泳生物和底栖生物(王颖, 2013)。这些海洋生物与其独特的生存环境相互作用, 相互影响, 相互适应, 组成了深海平原、极地地区、珊瑚礁、热液冷泉和红树林等生态系统, 形成了物种多样、功能多样和遗传多样的生物格局, 演化出诸多的适应性性状, 主要分为形态学性状、行为学性状、生理学性状和繁殖模式(沈国英等, 2008)。
2.1 形态学性状
除海洋本身特性及海水的光照、温度、盐度和溶解氧等理化环境外, 捕食者是对海洋动物尤其是海洋无脊椎动物影响最大的因素之一。而不同的海洋动物在面对捕食者时, 都有着自己的应对策略, 并在长期的进化过程中演变出丰富的适应性性状。海洋无脊椎动物的代表多毛类, 则表现出典型的“再生性状”, 其触手、触角、触须、鳞片及其他柔弱的部分, 失去后可以很快的再生出来。多毛类如小头虫、沙蚕、燐虫和螠虫等大多栖息于海底淤泥中, 由于海底淤泥可以缓冲海水的物理胁迫, 所以捕食者(鱼、虾、蟹和贝)就成为了多毛类底栖生活的主要选择压力(Tita, 2000)。在长期的适应过程中, 多毛类演化出了极强的再生能力, 可以快速修复被捕食者损伤的身体, 维持自身的生命活动, 但不同的物种其再生能力也存在一定差异。如燐虫()仅留一个体节就能再生成完整的虫体(Berrill, 1928), 而小头虫()仅能尾部再生(de Jong, 2018)。缨鳃虫()则可以变形再生(Jones-Knight, 1984), 当其触手冠和胸部被鱼食去后, 再生体节不超过3个, 其余的则由腹部体节变形改造而成。而对于一些底栖贝类, 如牡蛎、贻贝、扇贝、蛤和螺等, 尽管他们身体柔软, 但拥有厚重的壳, 可以抵御捕食者的侵袭。此外, 鱼类的体态结构也是受到了捕食者的选择压力。生活在海水里的三刺鱼(), 具有完整的骨板和完整的鳍刺, 以此抵御海洋中捕食者(食鱼性鱼类); 而生活在淡水中的三刺鱼, 其天敌主要为鸟类和部分水生无脊椎动物, 因此其骨板和鳍刺数目减少, 增强自身机动性和灵活性, 躲避捕食者(Colosimo, 2005)。
此外, 还有一些海洋动物, 演化出可变体色或与生存环境相似的颜色, 通过伪装来躲避捕食者。如粗吻短带鳚(), 是一种生活在印太海区的小型鱼类, 根据它想混入的鱼群不同, 其体色可在橄榄色、橙色、黑色或蓝色中切换, 以此躲避捕食者或获取食物。拟态章鱼()更是将传统章鱼的喷水和奔跑能力发挥到了极致, 它不仅可以改变自身颜色来躲避捕食者, 更可以通过改变自己的身体来伪装成如狮子鱼、海蛇、比目鱼和水母等15种更具威胁的海洋生物, 以此吓跑捕食者。异铠虾()又被称为羽毛星蹲龙虾或椰子虾, 它们生活在海百合的臂中, 模拟宿主的颜色进行伪装, 躲避捕食者(Cabezas, 2011)。
除捕食者外, 外界物理生存环境也是影响海洋动物适应性演化的一个重要因素。多毛类在海洋中分布广泛, 为适应不同的生存环境, 其在生活方式、体型和身体结构等方面都做出了诸多适应性改变。例如根据不同的环境特征, 多毛类演化出了不同的生活方式: 爬行或底栖(沙蚕、裂虫和叶须虫)、浮游(眼蚕和盲蚕)、穴居(吻沙蚕、海蛹和蛰龙介)、管栖(矶沙蚕、巢沙蚕和磷虫)、穿孔(才女虫和钙珊虫)、共栖和寄生等(杨德渐等, 2005)。此外, 多毛类的外形和身体结构也发生了较多改变(图1)。例如多毛类的疣足在演化过程中发生了特化, 行使了运动、呼吸和排泄等多种功能。燐虫是一种特殊滤食性管栖动物, 其虫体结构高度变异, 具有3个圆盘形扇叶, 分布于身体中部, 像活塞式抽水入管。在摄食过程中, 翼状叶弯曲成环不断分泌黏液扩展成滤食袋。纤毛背沟和纤毛杯状器将黏液膜聚集在一起, 并将其卷成球形颗粒(Berrill, 1928; Flood, 1982)。在印太交汇区特有分布的羽须鳃沙蚕 (), 其背须特化为鳃, 与其他沙蚕科物种相比, 形成了一种独特的环境适应性形态性状(Gowda, 2008; Hsueh, 2019)。这些特殊的身体结构均可能是适应生存环境而演化形成的。
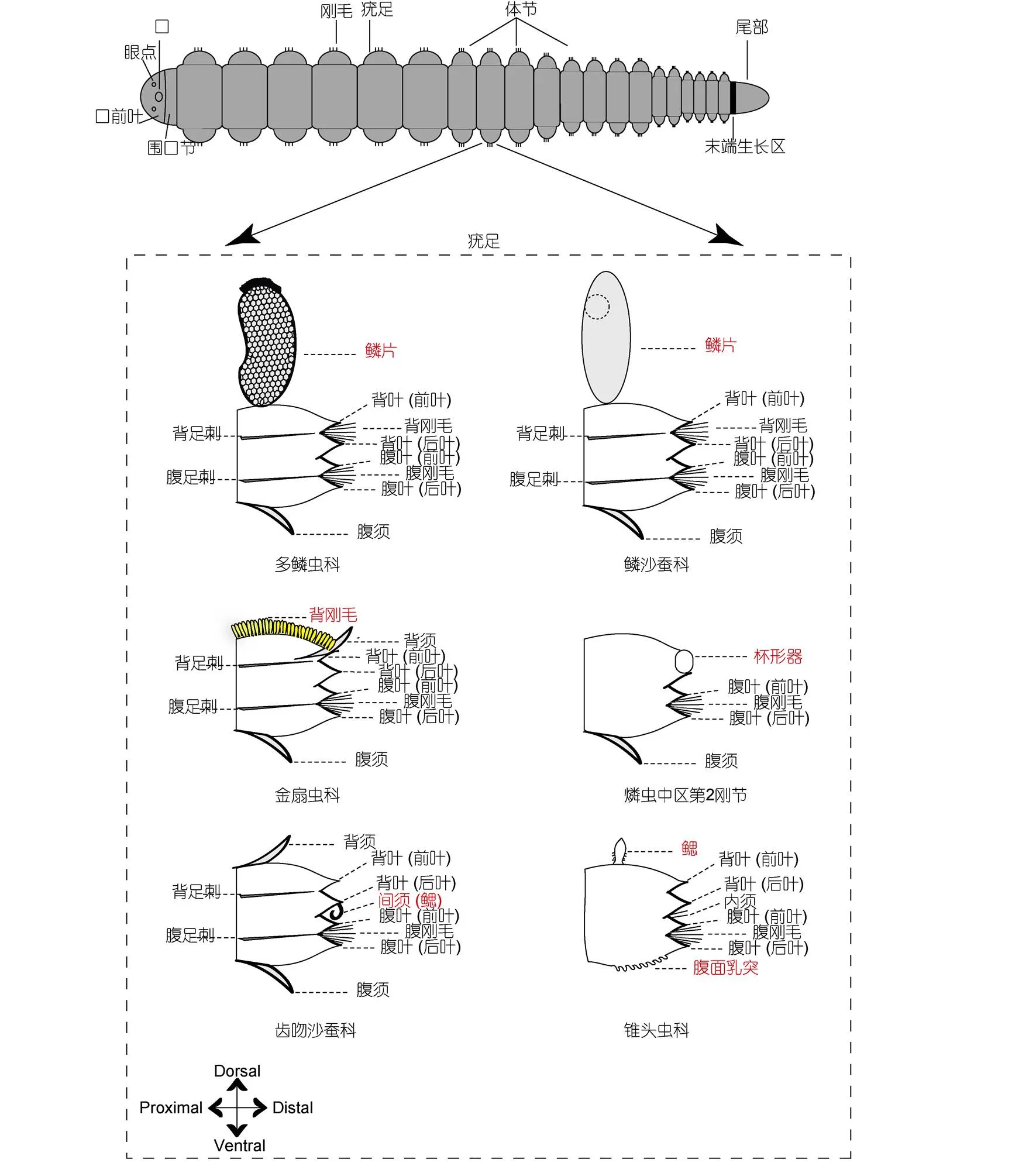
2.2 行为学性状
幼虫的扩散是海洋动物特别是无脊椎动物的一个典型行为学性状, 与其栖息环境相适应, 受到发育模式、光、溶解氧等因素的影响。
许多底栖海洋无脊椎动物在其生命周期中有两个截然不同的阶段: 浮游幼虫阶段和底栖阶段(Cannuel, 2006)。浮游幼虫可以在海洋中扩散, 寻找新的栖息地, 避免持久的近亲繁殖和物种内部食物竞争。在营养丰富的环境中, 生物多为浮游营养型发育模式, 幼虫浮游时间长、浮游能力强, 扩散距离长; 在营养贫乏及特殊环境下, 生物多为直接或者卵黄营养型发育模式, 幼虫不浮游或浮游时间极短, 扩散能力弱, 距离短。例如, 生活在热带和亚热带浅水双壳类多为浮游营养型发育模式, 幼虫浮游时间长、扩散距离长; 而生活在洞穴中的双壳类, 由于缺乏食物, 非浮游营养(卵磷脂营养或直接)的发育占主导地位, 幼虫扩散能力很弱(Cannuel, 2006)。生活在浅海区的海胆可以产生扩散能力强的浮游营养幼虫, 而生活在缺乏食物的较深海区的海胆, 产生扩散能力弱的卵黄营养幼虫(Lawrence, 1994)。不同的生存环境也同样会造就多毛类小头虫不同的幼虫发育模式, 更好的适应环境。如希腊的浅水热液喷口的小头虫为浮游营养型, 德国潮间带的小头虫既有浮游营养型又有直接发育型, 英国小头虫既有浮游营养型又有卵黄营养型(Méndez, 2000)。此外, 捕食者的威胁也会令生物演化出不同的行为学性状。例如在印太交汇区广泛分布的浮蚕科(Tomopteridae), 其疣足特化成桨状附肢用以游泳。该科种类众多, 每个种都有着独特的行为方式。比如, 遇险时有的会蜷缩成球; 有的种类会制造巨大的卵囊进行护卵; 有的种类能够释放发光黏液以此迷惑敌人等, 这些特殊的行为方式是浮蚕为应对捕食者而演化出的特殊性状(Dales, 1971; Gouveneaux, 2017)。
幼虫对光刺激的反应也同样会影响其扩散和定居。定居在潮间带的物种如牡蛎幼虫具有很好的感光性, 使它们在光的诱导下接近海洋的表层, 进入到潮间带的岩石和沙滩等有限的适宜环境中。苔藓虫、多毛类和纽虫等几个属的物种, 在浮游幼虫的早期阶段对光照的积极反应会使它们游到海洋表层, 随着表层洋流迅速扩散。在幼虫的晚期阶段, 他们对光的反应变成消极背光, 便于沉降到较深的水层选择合适的栖息地(Thorson, 1964)。
溶解氧在海洋中垂直分布, 随着海洋深度的增加而降低, 对于幼虫的扩散具有重要的影响(Tomasetti, 2018)。表层水中溶解氧含量最高, 浮游植物多, 营养丰富, 促使幼虫向海洋表层移动, 获得更好的营养条件; 同时氧气充足的环境相比缺氧的环境, 幼虫的游泳速度更加快速, 有利于幼虫在海洋中的扩散。
2.3 生理学性状
复杂多样的生存环境既给海洋动物带来了巨大挑战, 同时也丰富了海洋动物的适应性性状。过去三千万年间, 南极鱼亚目鱼类随着其生存环境的变化而快速演化, 由温暖海域的底栖祖先物种分化成寒冷海域中多样性最高的鱼类类群(许强华等, 2014)。居住在极地地区的海洋硬骨鱼为了适应极寒的环境, 产生各种抗冻蛋白, 以确保在海水冰点以下保持体液正常流动, 避免机体冷冻损伤(Scott, 1986; Bagwell, 2019)。在和五种螺的温度适应性研究中发现, 苹果酸脱氢酶(cMDH)蛋白构象及氨基酸组成的变化可以帮助其更好地适应从–1.9°C(南极洲)到55°C(中国南部海岸)不同温度的环境(Dong, 2018)。氧气是生物生存不可缺少的物质, 但热液喷口和冷泉区域氧气稀少, 存在大量的硫化物和二氧化碳, 这些都对生活在附近的海洋动物带来了巨大挑战。为了适应热液口的环境, 在和spp.的进化过程中, 其体液中高氧亲和力的血红蛋白含量大大增加, 可以很好地从低氧环境中提取氧气, 并且一些双壳类和环节动物, 其血红蛋白还具有结合硫化物的能力(汪青等, 2011)。在.中, 形成了一个内部气体传输系统, 允许其在条件良好时储存氧气, 并在环境氧浓度低时将氧释放到血管系统中, 更好地适应低氧环境(Hourdez, 2002)。分布在印太交汇区深海地带的西伯加虫科(Sibogliidae), 是一种没有嘴巴和消化管的非寄生性动物, 能与化能无机营养菌或甲基营养菌形成共生。为了适应深海低氧的环境, 西伯加虫演化出了一种独特的循环系统, 并且体内具有多种类型的血红蛋白, 能够吸收和运输大量的氧气, 来保证自身的生存(Hilário, 2011)。
此外, 共生也是一个典型的适应性生理学性状。在共生关系中, 一方为另一方提供有利于生存的帮助, 同时也获得对方的帮助。海洋中的共生关系种类繁多, 比如深海冷泉贻贝与多毛类(Sui, 2017)、囊螂科蛤类与其体内的共生细菌(Zheng, 2017; Kong, 2018), 宿主细胞内的共生藻, 小丑鱼与共生海葵, 珊瑚与共生藻类, 鱼类与寄生虾等。许多多毛类也会与其他无脊椎动物类群形成共生关系。其中, 约55%的已知共生物种属于多毛类(Eckelbarger, 2005), 如共生枝裂虫与深海六放海绵, 龙介虫与刺围脊贝, 吸口虫与棘皮动物, 胃鳞虫与海参等。其中, 胃鳞虫(), 借助其保护性着色和拟态(蠕虫的附属物模仿海参的乳头)特征, 隐藏在海参的表面, 以此保护自己(Britayev, 1996)。
2.4 繁殖模式
外部环境是影响海洋无脊椎生物繁殖模式的重要因素。在外界环境的选择压力下, 海洋动物演化出多种多样的繁殖模式。例如, 成熟的雌、雄海马在求爱和交配时身体颜色会更明亮, 且雌性产卵到雄性的育儿袋内由雄体孵育(Narberhaus, 2012; Lin, 2016)。其次, 外界环境对海洋生物的性别决定也具有很大影响。例如温度可以决定海龟的出生性别(Ge, 2018; Weber, 2020)。在温暖的环境中, 可以孕育出更多的雌性小海龟, 而在寒冷的条件下则会有更多的雄性小海龟(Weber, 2020)。进一步研究发现, 海龟的后代性别与埋藏在巢穴中的受精卵受到太阳光照的程度有关。此外, 在一种绿色后螠中, 其幼虫的性别与所处环境有很大关系, 若幼虫在海滩上孵育, 则可能发育成雌性个体; 但若幼虫与雌性的吻有接触, 则会发育成雄性个体(杨德渐等, 1998)。印太交汇区的珊瑚()是首次被证明单性生殖的珊瑚, 并且在后续的研究中也发现其存在混合繁殖的现象, 在同一群落中同时产生有性和无性的幼虫, 其中大部分的幼虫是孤雌生殖的(94%), 而有性幼虫的比例会随着群落大小, 周期和日历天的变化而显著变化(Combosch, 2013)。
在多毛类动物的演化中, 其繁殖模式也产生了多样性(Wilson, 1991), 如断裂生殖(钙珊虫、制端虫、叶磷虫)、出芽生殖(星状钻裂虫、枝裂虫)、再生生殖(齿裂虫、叉毛裂虫)、有性生殖等(杨德渐等, 1998)。并且, 有些多毛类繁殖时会有一些特殊的生殖行为。例如, 某些沙蚕具有特殊的群浮和婚舞等生殖行为。以双齿围沙蚕()为例, 每到繁殖时节的月圆晴朗之夜, 雌雄个体便会爬出洞穴, 群浮到海面, 雄性个体便会围绕一个或多个雌性个体排放精卵, 完成交配。此外, 月相对沙蚕的婚舞也是一个重要因素, 有的沙蚕偏好在满月下完成婚舞(喜光生殖), 有的则喜欢在新月下进行(避光生殖)(Hauenschild, 1956)。有的沙蚕更为聪明, 巧妙借助自身可再生的优势, 来进行繁殖, 进而大大减少群浮过程中可能遭受的伤害。例如太平洋萨摩亚的绿矶沙蚕()在进行生殖群浮时, 雌性个体并不会亲自前往, 而是把性成熟的体后部断区, 让此部分浮到水面, 进行交配(杨德渐等, 1998)。此外, 裂虫的再生繁殖模式也是令人惊奇, 厚多链虫(cf.)具有令人惊讶的繁殖能力, 它可在第30刚节生出1个头部, 以后约7—15刚节又生出一个头, 以此逐渐形成数个个体(杨德渐等, 2005)。而其他的多毛类如小头虫、毛轮沙蚕、才女虫和管沙蚕科的(Paxton, 2015)等物种为了避免外部因素对子代的伤害, 提高繁殖率, 会在产卵前形成一条孕管, 将卵产在孕管内, 为卵的生长发育提供一个安全稳定的环境, 从而保证子代的成活率。
3 海洋动物适应性演化研究方法
海洋动物的适应性性状是丰富多样的, 近年来其背后的遗传和分子机制也取得了一些进展, 形成了一些相对成熟的研究方法, 主要包括生物地理学、系统发生学、群体遗传学和多组学联合分析的方法(图2)。以下将对在海洋动物适应性演化的研究进程中所采用的方法进行简要综述。

图2 海洋动物适应性演化研究方法
3.1 生物地理学
生物地理学是结合生物学和地理学, 用于研究生物的时空分布、演化格局及其形成过程的学科, 其特点是能够从宏观上揭示生物多样性的产生、维持、辐射和演化机制, 如物种的形成、扩散、相互作用等。Briggs等利用不同海洋浅水区珊瑚礁鱼类和软体动物的分布情况、形态和遗传差异, 进行地理谱系分析并构建了生物多样性模型, 发现热印太交汇区的间隔性地理屏障促进了珊瑚三角区生物多样性的形成, 并指出印太交汇区经历了从物种积累到生物多样性形成, 并向外辐射扩散的变化历程(Briggs, 2013)。Cowman等重建了五大热带海域的隆头鱼科Labridae、雀鲷科Pomacentridae和蝴蝶鱼科Chaetodontidae的祖先分布区, 并评估了不同地理隔离对物种变异程度的影响, 证明了地理隔离对于物种演化具有强烈影响(Cowman, 2013)。He等通过对印太交汇区96个地点的石首鱼科进行线粒体COI基因的系统发育分析, 划分了石首鱼科的分布格局, 并指出地理隔离是导致石首鱼科遗传多样性增加的重要原因(He, 2018)。
3.2 系统发育学
系统发育学是一门研究物种形成、进化历史, 以及物种间演化关系的学科, 其主要的研究方法有古生物学、形态学和分子系统学, 研究结果往往以系统发生树和分子钟相结合的方式表示。系统发生分析在研究生物适应性演化中得到了大量应用, 例如Corrigan等采用系统发生重建、多样化速率转变分析等方法, 探究了深海端足类21个种群食腐性口器的适应性演化机制(Corrigan, 2014)。Ingram等以眼睛形态为模型对生活在不同海水深度的多个岩鱼群体进行系统发生分析, 发现其眼睛的大小与栖息环境密切相(Ingram, 2011)。Knope等对26种杜父鱼()的鳞片特征进行系统发生分析, 发现杜父鱼的鳞片数目的改变与栖息地环境的改变息息相关(Knope, 2013)。这些研究事例为“栖息地转变通过生态机会变化驱使适应性物种形成”假说提供了支撑。
除形态特征外, 分子特征也是进行系统发生分析的重要数据基础。Bely等使用马尔可夫最大似然法和贝叶斯MCMC方法, 对401个多毛类物种再生能力进行系统发生重建, 推测出环节动物最近共同祖先具有前后端再生能力(Zattara, 2016)。研究人员比较分析了深海贻贝()和浅水贻贝()的基因特征, 结合进化分析发现现代的深海贻贝是由浅海贻贝在1亿1千万年迁移到深海而来的。这种迁移帮助他们度过了约5700万年前因全球温度上升而造成的海洋底部缺氧导致的大灭绝事件(Sun, 2017)。Yang等选取了生活在热液口的两种甲壳动物(,)的线粒体基因组进行系统发生分析, 并与其他亲缘关系较近的物种进行比较, 发现线粒体基因组大小和组成没有显著差异, 表明这些物种的演化历程具有同步性, 为热液生态系统灭绝/重生假说提供了支撑(Yang, 2012)。
3.3 群体遗传学
群体遗传学是研究生物群体遗传结构变动的学科。该学科主要结合数学和统计学的方法, 探究群体基因频率和基因型频率的变化, 以及基因突变、环境选择和遗传漂变等因素对这些变化的影响, 以此阐述生物演化的机制。
遗传标记是群体遗传学常用的研究方法。以SNP位点作为遗传标记对在不同CO2水平下培养的紫海胆()幼虫进行分析, 发现在CO2浓度升高后, 种群的遗传结构发生了显著变化, 而幼虫的发育和形态却没有明显改变, 说明海洋酸化可能对一些没有外在表型变化的物种已经产生了分子水平的强烈影响。Shen等(2016)利用线粒体COI、16S和Cyt基因对南海冷泉深海和冲绳海槽热液偏顶蛤和深海龙虾的群体遗传结构进行研究, 发现群体不存在显著的遗传分化, 而在南海区域内的中, Xu等(2018)却发现了显著的种群遗传结构分化, 李新正等(2019)认为这说明种群历史动态、扩散能力、基因流水平、地理隔离和环境适应能力是造成物种群体遗传结构差异的主要原因。
3.4 多组学联合分析
多组学联合分析是指利用基因组、转录组和蛋白组等多种组学手段, 结合分子生物学和细胞生物学技术, 对生命有机体复杂生命活动进行深入研究, 以此揭示其背后分子遗传机制的研究方法。Scimone等(2020)使用单细胞测序、荧光原位杂交、细胞移植和RNAi等技术, 揭示了涡虫(planarian)视觉系统再生的分子和细胞机制。Gehrke等(2019)采基因组测序、ATAC-seq和RNA-seq多组学联合分析的方法, 筛选并构建了以egr为核心的再生分子调控网络, 揭示其参与再生的分子机制。此外, Chan等(2010)以SNP位点作为遗传标记构建全基因组关联图谱, 并结合转基因技术对三刺鱼骨板形成和丢失现象进行了研究, 发现这一性状的改变是由基因上游增强子(PEL)决定的, 说明某些调控元件的缺失或获得可能有助于自然群体中该基因控制的表型的平行进化。Li等(2019)比较分析了深、浅海和淡水端足类线粒体基因组, 发现深海sp.线粒体基因中存在更高的非极性氨基酸含量, 且其进化速率较低, 推测这些变异可能增强了sp.对极端环境的适应性。Lan等(2017)测定了马里亚纳海沟深海端足类的转录组, 并选取四种浅海端足类进行比较转录组分析, 发现适应深海极端环境的能力主要来自于氨基酸的替换和某些基因家族的扩张。Wang等(2019)分析了马里亚纳海沟深海狮子鱼()的基因组和转录组, 发现其体内丢失了大量与视觉和色素相关的基因, 且多个具有稳定细胞膜和蛋白结构功能的基因发生了突变, 进而增强了狮子鱼对深海极端环境的适应能力。Cheng等(2019)利用比较转录分析了在冷泉和热液两种环境下的基因表达情况, 发现大量环境适应相关基因不仅存在表达量的差异, 并且受到了选择作用, 推测这些基因的适应性演化和表达模式的差异可能是适应不同深海极端环境的主要原因。
随着CRISPR/Cas9基因编辑技术的发展, 非模式生物反向遗传学研究成为可能。除了上述四种研究策略, 结合多组学技术和基因编辑技术解析动物适应性性状的分子机制极大地推动了演化发育生物学的发展。比如利用CRISPR/Cas-9和RNAi技术探究并验证了Hox基因在海葵()(He, 2018)和钩虾(Martin, 2016)等海洋动物身体模式建成中的功能。
4 适应性演化研究中的新兴模式生物
近年来, 随着高通量测序技术和基因编辑技术的发展, 演化生物学家开发了若干新兴模式生物, 使得适应性演化领域的深入研究成为可能。
4.1 海胆
海胆隶属于棘皮动物门海胆纲, 是较早被使用的模式生物, 其幼虫发育模式更是研究生物适应性进化的极佳模型。在20世纪70年代, 科学家通过观察直接发育型海胆()的发育过程, 记录了其不同于间接发育型海胆()的发育特点, 包括卵黄营养幼虫骨架和肠道的缺失(Williams, 1975)。发育模式不同的海胆具有不同的栖息地。分布在食物资源缺乏的低潮海区岩石缝隙中的海胆是直接发育型模式, 产生卵黄营养幼虫; 分布在海岸和岛上食物资源较多、捕食者较多的平坦岩礁区的海胆是间接发育型模式, 产生浮游营养幼虫。海胆的直接发育模式是通过减少繁殖力和扩散为代价, 以此来降低幼虫死亡率的一种环境适应性策略(Thorson, 1950; Lawrence, 1994; Davidson, 2019)。
为探究造成两种海胆发育模式差异的原因, Wary和Raff首先利用荧光标记和显微镜观察的方法, 追踪了两种海胆原肠胚发育过程中细胞迁移过程, 发现其主要区别在于胚胎发育过程中左体腔形成和成体发育的时间。这些过程在直接发育型海胆中的发生时间要早于间接发育型海胆, 从而使得卵黄营养幼虫的幼虫期大大缩短; 同时, 在直接发育型海胆中, 原肠不能分化成有功能的幼虫肠道或口(Wray, 1991; Smith, 2009), 所以卵黄营养幼虫仅能依靠来自母体的能量完成自身的生长发育, 以此适应食物资源缺乏的环境。此外, 直接发育型海胆的卵黄营养幼虫还表现为幼虫骨骼的缺失。随后, Israel等(2016)对两种海胆的早期发育阶段进行了转录组测序, 构建了发育调控网络, 并利用比较转录组分析了卵黄营养幼虫和浮游营养幼虫不同发育阶段的基因表达情况, 发现发育调控网络中、、、和等相关基因的表达在两种幼虫中存在较大差异, 推测其可能与发育模式和后期形态结构差异有关。研究人员还借助原位杂交技术检测了上述部分基因在海胆幼虫发育过程中的表达模式, 并通过RNA干扰实验探究了基因在骨骼形成中的功能, 发现干扰的表达会影响间接发育型海胆骨骼的形成, 而对直接发育型海胆无影响(Zhou, 2003)。为进一步探究海胆发育模式差异的机制, Wang等(2020)又对不同发育类型的海胆和杂交产生的F1代进行了转录组测序, 比较分析发现调控网络中基因的差异表达, 主要是受到了其早期胚胎阶段顺式反应元件的影响, 进而造成发育模式和骨骼的差异。此方面的研究以海胆幼虫典型的发育模式为模型探究了海洋生物适应演化背后的遗传分子机制, 为生物多样性研究提供了理论基础。
4.2 三刺鱼
三刺鱼是一种小型的硬骨鱼类, 因其背鳍前具有3根游离鳍刺而得名。三刺鱼的祖先是生活在海洋中, 自一万年前冰河时代结束后, 三刺鱼被广泛分布到了新形成的湖泊和河流等淡水环境中, 并在形态结构、行为和生活习性方面演化出丰富的多样性, 成为了生物适应性演化的明星物种(Peichel, 2001)。三刺鱼不具有鱼鳞, 而是拥有体侧骨板。生活在海洋里的三刺鱼一般都具有约35个骨板和完整的鳍刺, 用来抵御海洋中大量的食鱼性鱼类的伤害(Colosimo, 2005)。而对于淡水三刺鱼来说, 它们所面对的威胁主要是鸟类和部分水生无脊椎生物, 所以它们减少了自身体侧骨板和鳍刺的数目来保持较高机动性。此外, 相对于海洋宽阔的生存环境来说, 淡水中往往具有较多的水草, 会限制三刺鱼的游动, 所以保持身体的灵活性对于逃生极为重要(Shapiro, 2004)。造就三刺鱼这种多样性的, 不是基因交流, 而是环境压力。淡水三刺鱼骨板数目的多少与该环境内食鱼性鱼类的丰富程度密切相关, 并且三刺鱼体侧骨板数目的性状是可以遗传的, 具有适应性的, 并且这个性状在没有与海水三刺鱼进行基因交流的情况下, 可以通过迅速的演化来适应自然环境的选择。
为探究造成三刺鱼骨板性状差异的原因, 科学家(Chan, 2010)首先利用遗传学的方法, 进行三刺鱼杂交繁育, 并比较分析了子代三刺鱼的基因差异, 定位到了差异基因和位置。随后采用了分子生物学实验方法进行了功能验证, 揭示了三刺鱼骨板性状差异的机制。首先, 研究人员将具有骨板差异的两种三刺鱼进行杂交繁育, 获得子代杂交三刺鱼(Chan, 2010)。通过对子代三刺鱼进行全基因组关联图谱和数量性状定位发现, 三刺鱼骨板性状的差异可能与(pituitary homeobox transcription factor)基因有关。随后在对两种三刺鱼的基因序列分析发现, 其基因的编码序列没有显著变化, 而是调控区域发生了突变, 并且定位到了的一段增强子序列。然后研究人员构建了该增强子的报告载体, 借助转基因技术, 将增强子转入无骨板的三刺鱼中, 发现该增强子可以令骨板丢失的三刺鱼重新长出骨板。以上结果说明三刺鱼增强子序列受到了外界环境的选择压力, 产生了突变, 进而造成了三刺鱼的骨板差异(Colosimo, 2005; Wray, 2007; Chan, 2010)。此外, 该研究也表明外界环境的选择压力也可以作用于生物关键基因的调控区, 通过调控基因的表达, 来产生利于自身生存的新的适应性性状。这为海洋生物适应演化机制研究提供了新的切入点。
4.3 钩虾
钩虾隶属于端足目、钩虾亚目, 是一种小型甲壳动物, 身体呈拱形故称钩虾。钩虾身体分节, 具有复眼、外骨骼、附肢等结构, 共有19对附肢, 分别行使感觉、摄食、爬行、游泳、生殖等功能, 个体变化多样, 与其生活环境相互适应。
钩虾凭借易饲养、胚胎大、附肢形态结构丰富多样等特点, 成为研究基因功能、身体模式建成和再生的优质生物材料(Sun, 2019)。在对钩虾附肢多样性性状的研究中, 首先利用原位杂交和免疫组化技术检测了家族基因的表达模式, 发现在钩虾胸部取食附肢中没有检测的表达, 而其余的胸部附属物中具有的表达。随后利用RNA干扰技术降低了的表达, 导致了钩虾胸部运动附肢向摄食附肢的转变, 证明了基因在钩虾附肢多样性中的作用(Liubicich, 2009)。此外, Bruce等(2020)还利用CRISPR/Cas9技术敲除了钩虾的腿间隙基因distalless ()、、dachshund ()、extradenticle ()和 homothorax (), 探究了其在身体模式建成中的功能, 并与果蝇等其他昆虫进行比较, 发现昆虫翅膀并不是新生的结构, 而是由甲壳动物已有的古老结构演化而来的。同时, Vargas-Vila等(2010)和Martin等(2016)利用CRISPR/Cas9技术证明了和基因在钩虾DV轴和AP轴建立中的功能, 揭示了该基因与甲壳动物身体模式建成的关系。此外, 研究人员还利用荧光标记和活体成像技术, 在单细胞水平上持续观察了钩虾附肢再生过程中的细胞活动, 发现钩虾外胚层和中胚层再生细胞机制存在差异, 该研究成果为生物再生机制多样性研究提供了新视角(Konstantinides, 2014; Sun, 2019)。
4.4 小头虫
多毛纲是环节动物门最大的一个纲和最为原始的类型。大多数多毛纲物种具有身体分节现象, 表现出较强的再生能力, 以此来适应多变的环境。其中, 典型的代表种是小头虫() (Seaver, 2016)。
小头虫生活在腐殖质丰富的海洋底层环境中, 以翻吞泥沙和消化其中的有机碎屑和微小生物为生, 是世界性公认的海洋环境污染指示种(Seaver, 2016)。小头虫作为底栖生物, 其摄食、爬行、建管和避敌等生命活动, 能够改变生存环境的理化性质, 从而影响污染物的迁移、转化等环境行为, 进而对海洋环境修复产生促进作用(李井懿等, 2017)。小头虫的体制为简单的线性分节体制, 身体柔软细长, 尾部是胚胎后末端生长和繁殖器官所在的区域, 易受到外界环境和捕食者的损伤。在适应底栖爬行环境、自身生长繁殖和捕食者等多个因素的相互平衡下, 小头虫演化出非常独特的“再生性状”: 尾部具有极强的快速再生能力, 但头部再生能力丢失(Seaver, 2016; de Jong, 2018)。在对小头虫再生性状的研究中, 研究人员利用形态观察、原位杂交和免疫组化的方法, 详细的记录了小头虫再生过程中组织形成和再生关键基因的时空表达模式, 并通过Edu-Brdu细胞示踪实验发现小头虫再生细胞来源既包括干细胞样细胞的迁移也含有损伤部位细胞的再分化, 推测其再生机制可能与涡虫等再生模式物种并不完全相同(de Jong, 2016, 2018)。因此, 在研究动物对海洋底层环境的响应和适应方面, 小头虫再生性状是一个理想的研究模式。
此外, 在小头虫胚胎发育方面的研究中发现, 小头虫大脑发育过程中神经前体细胞会发生迁移, 这与果蝇和脊椎动物的神经发生过程存在明显的不同(Meyer, 2009; Seaver, 2016)。并且小头虫与软体动物躯干中胚层的起源不同, 前者是2 d细胞, 而后者是4 d细胞。同时二者在关键早期细胞信号通路方面也存在差异, 在小头虫身体模式形成中发挥重要作用的通路是Nodal信号通路, 而不是BMP通路。这些发现使小头虫成为研究两侧对称动物身体模式进化的重要物种(Seaver, 2016; Lanza, 2018)。并且, 伴随着小头虫高质量基因组的获得, 运用CRISPR/Cas9技术敲除了感光基因opsin, 并成功获得了表型, 标志着小头虫基因组编辑技术的成功建立(Neal, 2019)。这与其他海洋多毛类生物相比, 小头虫具有更好的功能验证平台优势, 是可以帮助我们深入理解生物体制建成和再生机制的极佳动物模型。
综上, 在海胆、三刺鱼、钩虾和小头虫适应性性状的研究中, 充分利用了多组学和反向遗传学技术手段, 取得了一定的进展, 可以帮助我们在一定程度上了解海洋生物适应性性状起源和演化的分子机制。
5 印太交汇区海洋动物适应性进化的研究进展
印度洋和太平洋交汇区作为世界上生物多样性最丰富的海区, 拥有着广泛的浅海区域、漫长的海岸线、众多的岛屿和丰富的生物, 是研究生物适应性演化的典型模式区域。
5.1 印太交汇区生物多样性中心介绍
目前, 印太交汇区高海洋生物多样性的成因尚不明确, 已有的假说包括物种中心、物种积累、物种生存、物种重叠等。其中, 物种中心和物种生存假说认为物种优先起源于热带区域, 然后向两极扩散。而在地球的冰河时期, 位于热带的珊瑚礁大三角起到了保存海洋物种的作用, 使得此区域的物种得以保存(Jokiel, 1992; Briggs, 1999; Bellwood, 2001; Briggs, 2003)。物种积累假说则认为, 分布于珊瑚礁大三角外围的生物, 随着板块运动和洋流扩散, 长期缓慢汇集于此(Jokiel, 1992)。物种重叠假说则认为, 珊瑚礁大三角的海洋生物多样性是印度洋和太平洋的不同海洋生物区系在此重叠的结果(Woodland, 1983; Gaither, 2013)。在上新世和更新世冰期, 珊瑚礁大三角出现地理屏障, 两洋物种隔离分化。冰期结束后, 两洋物种在珊瑚礁大三角区交汇重叠, 生物多样性得以增强。目前来说, 高丰度的生物多样性是由地质历史、构造过程、气候事件、海洋环境与生命过程等多圈层、多界面相互作用形成的结果, 仅凭一种理论或假说并不足以解释这一现象, 人们越来越多地意识到需要联合多种假说来解析印太交汇区生物多样性中心的形成演变(Matias, 2018)。综合上述假说的其中一个关键因素在于评估该地区生物多样性获得与丢失的速率, 并推演生物多样性获得或丢失的时间(Jablonski, 2006)。生物多样性产生和演化的历程是典型性状获得与失去的过程, 这些性状及其变异是生物适应环境(气候变化等)和长期进化的结果, 也是生命多样性得以产生的本质(Vol’Kenshteĭn, 1984; Mergeay, 2012; Liu, 2018)。探索适应性性状起源演化过程从一定程度上是研究不同假说、外在环境和生物内源因素驱动印太交汇区高海洋生物多样性中心形成的重要突破口。
5.2 印太生物多样性形成的外在驱动力和内源性机制
5.2.1 印太交汇区生物多样性中心形成和演变的外源环境因素 复杂的水文、地质和气候环境, 孕育了印太交汇区成为全球最大的海洋生物多样性中心。起初, 特提斯山脉覆盖了今天的阿尔卑斯山、青藏高原和印度-西太平洋岛屿。在始新世, 欧亚大陆与非洲/印度的碰撞导致特提斯山脉向西撤退。最终导致曾经的特提斯洋彻底消失, 继而形成东太平洋、西大西洋、东大西洋、地中海和印度-西太平洋五大海域。中新世中期, 特提斯群岛的关闭隔离了大西洋/地中海和印度-西太平洋, 将欧亚大陆和非洲连接起来(Liu, 2018)。欧亚板块、菲律宾板块、太平洋板块和澳大利亚板块相撞, 有效地关闭了印度洋-太平洋赤道海道(Zhao, 2017), 限制了西太平洋的海洋环流, 于中新世晚期启动形成了印度-太平洋暖池。在此期间碳酸盐体系转变, 主要贡献者从有孔虫转移到珊瑚, 导致在中新世珊瑚丰度突然增加, 珊瑚礁广泛发育, 且印太交汇区内适宜珊瑚礁生存的浅水海洋生物栖息地出现频率的增加与高生物多样性的出现相吻合(Hillebrand, 2018)。
总之, 从始新世到中新世, 印太交汇地区经历了碳酸盐崩溃、气温变冷、短暂冰期以及暖池形成(Dekens, 2007)等标志性地质历史事件, 造成了气候条件变化和环流变异, 影响了海洋生物的活动。到上新世时, 印太交汇区的地理区系已形成今天的大致格局, 主要岛屿之间彼此分离或由廊道相连。在后续的冰期和间冰期反复更替下, 印太交汇区的陆海结构发生巨大变化, 造成海洋生物的隔离、分化和变迁, 推动了印太生物多样性中心的形成。
5.2.2 印太交汇区生物多样性中心形成和演变的生物内源因素 除了外在的环境选择之外, 生物本身的演化能力, 即有机体内在的发生变异的能力, 也决定着生物的演化和生物多样性的产生。从生物多样性产生的内源机制角度上看, 生物的某些遗传特征(如功能选配)使得生物在不影响其基本生存适应的条件下, 以较小的遗传改变而获得显著的表型改变和进化(Melo, 2016; Verd, 2019)。比如 “模块性”假说, 该假说认为核心模块在演化上是高度保守的, 模块的微小改变能够促进生物适应性性状的产生和生物多样性的形成(Slotine, 2001)。例如在印太地区广泛分布的芋螺科(Conidae), 为适应复杂多变的环境, 其成熟毒素段序列拥有了高突变性, 造就了芋螺毒素的多样性, 进而影响了其进食行为(Abalde, 2018)。节肢动物和环节动物体节附肢的发育和适应性分化也为揭示生物多样性产生的内源机制提供了一个很好的例子(Williams, 2001)。动物的体节是重复的形态建成模块, 每一个体节具备独立性, 能够在不影响其他体节的前提下发生变异。因此, 在同一物种不同体节以及不同物种同源体节上的附肢, 具有相似的形态建成模式, 也有体节和物种适应性演化的性状(von Dassow, 2000)。如分布在印太和大西洋等温暖海域的博比特虫(), 是一种夜行的多分节虫子, 营底栖生活, 其身体长期栖于沙子之中, 而头部特化出用于感知环境的触须和具有锋利下颚的口器, 以此满足捕食需求。印太广有分布的巧言虫()是一种生活在多岩石潮间带的海洋多毛类动物, 身体结构简单, 是肉食性的捕食者, 能够捕食较大的猎物(贻贝、藤壶、腹足类和其他环节动物)。其头部演化出具有触须的吻, 当与猎物接触时, 巧言虫吻外翻, 这些触须就会暴露出来, 迅速释放粘液和毒素, 麻痹和捕食猎物。总之, 模块化的体节性状及其背后模块化的DNA分子机制影响生物本身的演化能力, 并于外在的环境选择条件下形成演化热点, 从而驱动生物多样性的形成。
5.3 适应性性状是生物多样性形成的本质和最基本的元素
适应性性状是指生物在环境适应过程中所演化出的变异性状。适应性性状的产生和演化最终是由基因型的改变引起的。近年来, 科学家借助基因的功能选配理论(co-option)从分子的水平上解释生物多样性形成的模块化假说。决定生物多样性新性状的基因可能是从头产生, 也可能是从古老基因演化获得新功能而来, 后者被称为功能选配。基因组测序技术的发展积累了大量数据, 通过比较全基因组序列, 生物学家发现在系统发育树中广泛的存在着保守蛋白以及功能结构域, 暗示这些共有的元件在生物多样性形成过程中广泛地通过功能选配进化出新的功能, 以适应变化的环境。True等(2002)提出了基因功能选配的两种主要分子机制, 一是通过古老基因复制产生的新基因导致新性状产生。基因复制产生的新位点可能会产生新功能化、亚功能化以及假基因化等不同的命运。比如中华白海豚()是鲸类的一员, 分布于印太区域, 已完全适应近岸水域。通过比较基因组分析表明, 中华白海豚演化出494个物种特异性基因家族, 涉及免疫, DNA修复和与潜在适应机制相关的感觉系统(Ming, 2019)。雀鲷科(Pomacentridae)的变色雀鲷是最丰富和最典型的印太珊瑚礁鱼类家族之一。这个家族表现出丰富的颜色模式以及各种与性别相关的性状, 包括雌性异体、雌雄同体, 主要是由其异染色质片段大小差异所致(Getlekha, 2016)。种群基因组调查表明, 高纬度珊瑚种群存在耐热性相关的等位基因, 但是在太平洋南端水温相对较低区域的珊瑚种群中出现的频率很低。但在全球气候变暖的选择压力下, 该地区热耐受和抗氧化的基因出现快速演变, 以此适应更高的海水温度, 帮助种群持续繁衍(Bay, 2017)。另一种机制是通过改变基因顺式作用元件等调控区, 以调控基因的时空和剂量表达模式(而不是改变基因的编码区), 进而指导新性状和生物多样性形成。典型的例子是表达调控区域反应元件的变化, 造成海水和淡水三刺鱼骨板差异的事例。此外, 在对平颏海蛇的研究中发现, 其内部有108个非编码元件发生快速演变, 这可能是保证其能够在较短时间适应海洋环境生活的重要原因(Peng, 2020)。综上, 许多复杂的生物新性状是通过对古老基因的修饰和改造完成的。因而探索基因功能选配的分子机制调控下的性状变异是理解生物多样性形成的一个重要基础。
5.4 海洋底栖生物的多样性
印太交汇区作为全球生物多样性中心, 拥有着丰富的海洋底栖生物资源。海洋底栖生物是指那些全生活史或大部分时间生活在海洋地表或沉积物中的全部生物总称, 按其大小可分为微型底栖生物、小型底栖生物和大型底栖生物。底栖生物在海洋生态系统中占有重要地位, 不仅占据着海洋食物网中不同的生态位置, 还在海洋生态系统的能量流动和物质循环过程中发挥着重要作用(张均龙等, 2016)。同时, 海洋底栖生态系统比水体具有更高的生物多样性, 其中90%的海洋无脊椎动物为底栖生物, 其在浅海区域单位体积沉积物中的丰富度要高于水体一至几个数量级, 是维持海洋生物多样性和海洋生态结构不可缺少的一部分(徐奎栋, 2011)。大多数的海洋底栖生物长期生活在海洋底表或底层沉积物中, 具有区域性强、迁移能力弱等特点, 为海洋生物资源调查取样提供了便捷(张均龙等, 2016)。并且, 底栖生物对于生存环境的变化具有较弱的回避能力, 只能做出适应性的改变, 而不同的底栖动物对环境变化的适应性和响应方式也存在着差异, 进而演化出丰富多样的适应性性状, 成为研究适应性演化的极佳动物类群(徐奎栋, 2011)。
多毛类(环节动物门)是海洋大型底栖生物中物种资源最为丰富的类群之一, 具有80余科, 12000多个已知种。从多毛类全球分布来看, 从极寒到热带都有分布, 不同的类型的栖息地都有其身影(图3), 说明其环境适应能力强, 这得益于其体制建成模式。环境适应过程中, 多毛类头部、躯干部及疣足都会引起相应的形态改变, 并且演化出包括分节、再生、繁殖模式和生活方式等丰富多样的环境适应性状, 是研究生物适应性演化的极佳生物类群。从全球的物种多样性分析来看, 印太交汇区是生物多样性的高峰(Tittensor, 2010)。我们根据OBIS数据(https://obis.org/taxon/883)统计发现, 在印太交汇区的有限野外采样数据(少于大西洋采集数据几十倍)下, 鉴定到的多毛类物种就达到近1300种(图3), 具有60余科。该地区特有多毛类物种比例接近40%, 印太交汇区鉴定的豆缨虫科(Fabriciidae)、不倒翁虫科(Sternaspidae)、西伯加虫科(Siboglinidae)、燐虫科(Chaetopteridae)、笔帽虫科(Pectinariidae)、双栉虫科(Ampharetidae)、小头虫科(Capitellidae)、长手沙蚕科(Magelonidae)、海蛹科(Opheliidae)、真鳞虫科(Eulepethidae)、海刺虫科(Euphrosinidae)、囊须虫科(Saccocirridae)的物种超过一半是印太区特有的。此外还有一些物种在印太分布占有优势, 如目前全球记录到的盘首蚕(), 就有超过80%生活在印太交汇区, 以及约一半的星虫科种也集中在此区域(Giangrande, 2004)。
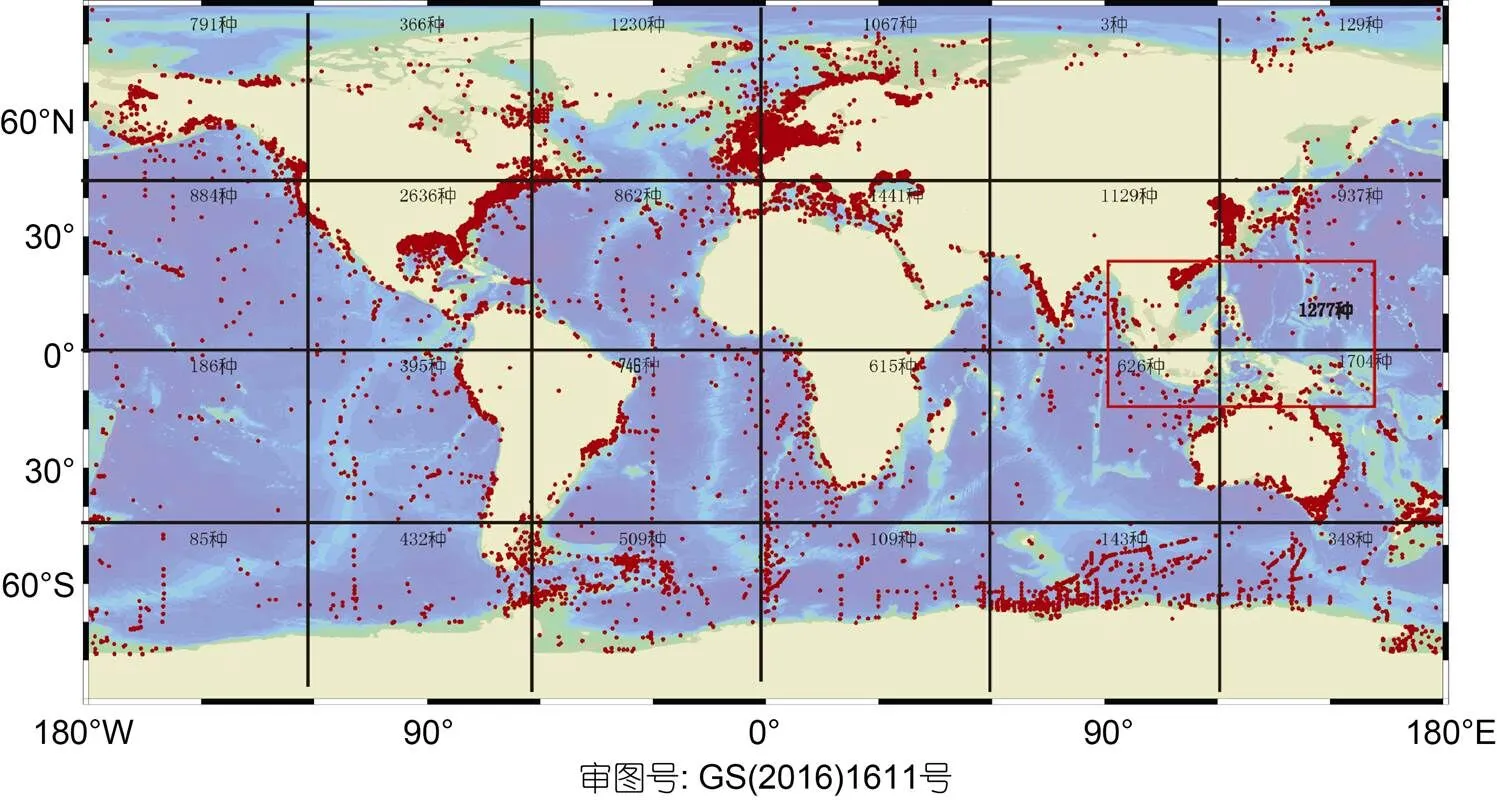
图3 多毛类多样性全球分布
在对印太交汇区温差、溶解氧含量和叶绿素含量等环境因子的分析中发现, 印太交汇区东侧海域形成了低温、少氧的环境。并且, 该区域内记录到包含单指虫(Cossuridae)、欧文虫(Oweniidae)和鳃沙蚕()在内的具有特殊鳃结构的多毛类。其中, 鳃沙蚕是沙蚕科比较特殊的一种, 全世界只有5个种, 其体节长有发达的鳃, 且该性状是由多毛类疣足特化而来的。目前, 已有报道显示该属只分布在印度-西太平洋的热带地区, 零星分布于我国南海海域, 且物种记的录分布区域溶氧含量较低, 而鳃性状极有可能是其为适应生存环境所演化出的一种环境适应性性状。因此, 了解印太交汇区多毛类鳃性状多样性的形成机制对于我们深入解析印太交汇区生物多样性中心形成和演变具有重要的帮助。
印太交汇区复杂的地理环境孕育了丰富的多毛类生物资源, 同时我们相信印太地区的多毛类资源丰富度远比目前发现要多得多, 具有进一步探究的必要性。此外, 借助印太多毛类资源优势, 以其典型适应性性状为模型探究演化机制, 更有助于加深我们对印太地区海洋生物多样性格局形成和演化的认知。
6 展望
海洋生物多样性是海洋生态系统的重要组成部分, 而了解生物多样性中心的形成演化过程不仅是生命科学的本质问题, 而且对于我们开发、利用和保护海洋生物资源具有重要的指导意义。印太交汇区作为全球生物多样性中心, 拥有着丰富的适应性性状, 是研究海洋生物多样性起源和演化的典型模式区域。因此, 在后续的生物多样性研究中, 通过聚焦印太交汇区的典型生物类群和典型性状, 借助海洋生物资源调查, 分析典型生物的适应性性状, 获得适应性性状的分布格局和演化路径, 筛选和建立具有典型适应性性状的实验室模式种。并进一步借助实验海洋生物学的研究手段, 解析性状多样性背后的分子基础, 阐述生物内源机制在适应性性状演化中的驱动力, 以及评估海洋环境外源因素对生物内源演化热点的塑造作用, 为认知印太交汇区典型生物的起源、适应和演化机制提供科学阐述, 加深对海洋生物多样性的整体认识。
王 颖, 2013. 中国海洋地理. 北京: 科学出版社, 1—910
许强华, 吴智超, 陈良标, 2014. 南极鱼类多样性和适应性进化研究进展. 生物多样性, 22(1): 80—87
李井懿, 贾新苗, 田胜艳, 2017. 海洋底栖动物的环境修复作用. 海洋信息, (3): 41—47
李新正, 2000. 浅谈我国海洋生物多样性现状及其保护. 见: 生物多样性保护与区域可持续发展——第四届全国生物多样性保护与持续利用研讨会论文集. 北京: 中国林业出版社, 8—14
李新正, 董 栋, 寇 琦等, 2019. 深海大型底栖生物多样性研究进展及中国现状. 海洋学报, 41(10): 169—181
杨德渐, 孙世春, 2005. 环节动物门. 见: 孙世春. 海洋无脊椎动物. 青岛: 中国海洋大学出版社, 294—295
杨德渐, 孙瑞平, 1998. 什么是海洋多毛环虫? 见: 孙瑞平. 来自海洋的疑问(海洋动物篇). 青岛: 青岛海洋大学出版社, 1—183
汪 青, 项荣花, 包永波等, 2011. 动物血红蛋白研究进展. 宁波大学学报(理工版), 24(2): 20—24
沈国英, 施并章, 2008. 海洋生态学. 北京: 科学出版社, 1—446
张均龙, 史本泽, 赵 峰等, 2016. 中国海洋底栖生物学发展回顾与展望. 海洋科学集刊, (51): 194—204
徐奎栋, 2011. 海洋微型底栖生物的多样性与地理分布. 生物多样性, 19(6): 661—675
蔡立哲, 2006. 海洋底栖生物生态学和生物多样性研究进展. 厦门大学学报(自然科学版), 45(Z2): 83—89
Abalde S, Tenorio M J,Afonso C M L, 2018. Conotoxin diversity in(Born, 1778) and the convergent origin of piscivory in the Atlantic and Indo-Pacific cones. Genome Biology and Evolution, 10(10): 2643—2662
Bagwell S N, Ricker J V, 2019. Antifreeze proteins: effective adaptations of organisms for low temperature survival. BIOS, 90(3): 158—170
Bay R A, Rose N H, Logan C A, 2017. Genomic models predict successful coral adaptation if future ocean warming rates are reduced. Science Advances, 3(11): e1701413
Bellwood D R, Hughes T P, 2001. Regional-scale assembly rules and biodiversity of coral reefs. Science, 292(5521): 1532—1535
Berrill N J, 1928. Regeneration in the polychaet. Journal of the Marine Biological Association of the United Kingdom, 15(1): 151—158
Briggs J C, 1999. Extinction and replacement in the Indo-West Pacific Ocean. Journal of Biogeography, 26(4): 777—783
Briggs J C, 2003. Marine centres of origin as evolutionary engines. Journal of Biogeography, 30(1): 1—18
Briggs J C, 2007. Marine longitudinal biodiversity: causes and conservation. Diversity and Distributions, 13(5): 544—555
Briggs J C, Bowen B W, 2013. Marine shelf habitat: biogeography and evolution. Journal of Biogeography, 40(6): 1023—1035
Britayev T A, Zamishliak E A, 1996. Association of the commensal scaleworm(Polychaeta: Polynoidae) with holothurians near the coast of South Vietnam. Ophelia, 45(3): 175—190
Bruce H S, Patel N H, 2020. Knockout of crustacean leg patterning genes suggests that insect wings and body walls evolved from ancient leg segments. Nature Ecology & Evolution, 12(4): 1703—1712
Cabezas P, Macpherson E, Machordom A, 2011.(Decapoda: Galatheidae): a monospecific genus of squat lobster? Zoological Journal of the Linnean Society, 162(2): 245—270
Cannuel R, Beninger P G, 2006. Gill development, functional and evolutionary implications in the pacific oyster(Bivalvia: Ostreidae). Marine Biology, 149(3): 547—563
Chan Y F, Marks M E, Jones F C, 2010. Adaptive evolution of pelvic reduction in sticklebacks by recurrent deletion of aenhancer. Science, 327(5963): 302—305
Cheng J, Hui M, Sha Z L, 2019. Transcriptomic analysis reveals insights into deep-sea adaptations of the dominant species,(Crustacea: Decapoda: Anomura), inhabiting both hydrothermal vents and cold seeps. BMC Genomics, 20(1): 388
Colosimo P F, Hosemann K E, Balabhadra S, 2005. Widespread parallel evolution in sticklebacks by repeated fixation of ectodysplasin alleles. Science, 307(5717): 1928—1933
Combosch D J, Vollmer S V, 2013. Mixed asexual and sexual reproduction in the Indo‐Pacific reef coral. Ecology and Evolution, 3(10): 3379—3387
Corrigan L J, Horton T, Fotherby H, 2014. Adaptive evolution of deep-sea amphipods from the superfamily Lysiassanoidea in the North Atlantic. Evolutionary Biology, 41(1): 154—165
Cowman P F, Bellwood D R, 2013. The historical biogeography of coral reef fishes: global patterns of origination and dispersal. Journal of Biogeography, 40(2): 209—224
Dales R P, 1971. Bioluminescence in pelagic polychaetes. Journal of the Fisheries Board of Canada, 28(10): 1487—1489
Davidson P L, Thompson J W, Foster M W, 2019. A comparative analysis of egg provisioning using mass spectrometry during rapid life history evolution in sea urchins. Evolution & Development, 21(4): 188—204
de Jong D M, Seaver E C, 2016. A stable thoraciccode and epimorphosis characterize posterior regeneration in. PLoS One, 11(2): e149724
de Jong D M, Seaver E C, 2018. Investigation into the cellular origins of posterior regeneration in the annelid. Regeneration, 5(1): 61—77
Dekens P S, 2007. Characterizing upwelling regions and the Indo-Pacific warm pool in the early Pliocene warm period. California, USA: Doctor Dissertation of University of California
Dong Y W, Liao M L, Meng X L, 2018. Structural flexibility and protein adaptation to temperature: molecular dynamics analysis of malate dehydrogenases of marine molluscs. Proceedings of the National Academy of Sciences of the United States of America, 115(6): 1274—1279
Eckelbarger K, Watling L, Fournier H, 2005. Reproductive biology of the deep-sea polychaete(Polynoidae), a commensal species associated with octocorals. Journal of the Marine Biological Association of the United Kingdom, 85(6): 1425—1433
Erpenbeck D, Aryasari R, Benning S, 2017. Diversity of two widespread Indo-Pacific demosponge species revisited. Marine Biodiversity, 47(4): 1035—1043
Flood P R, Fiala-Médioni A, 1982. Structure of the mucous feeding filter of(Polychaeta). Marine Biology, 72(1): 27—33
Gaither M R, Rocha L A, 2013. Origins of species richness in the Indo‐Malay‐Philippine biodiversity hotspot: evidence for the centre of overlap hypothesis. Journal of Biogeography, 40(9): 1638—1648
Garrison T S, 2012. Essentials of Oceanography. 11th ed. USA: Pearson, 1—20
Ge C T, Ye J, Weber C, 2018. The histone demethylase KDM6B regulates temperature-dependent sex determination in a turtle species. Science, 360(6389): 645—648
Gehrke A R, Neverett E, Luo Y J, 2019. Acoel genome reveals the regulatory landscape of whole-body regeneration. Science, 363(6432): eaau6173
Getlekha N, Cioffi M D B, Yano C F, 2016. Chromosome mapping of repetitive DNAs in sergeant major fishes (Abudefdufinae, Pomacentridae): a general view on the chromosomal conservatism of the genus. Genetica, 144(5): 567—576
Giangrande A, Licciano M, 2004. Factors influencing latitudinal pattern of biodiversity: an example using Sabellidae (Annelida, Polychaeta). Biodiversity and Conservation, 13(9): 1633—1646
Gouveneaux A, Flood P R, Erichsen E S, 2017. Morphology and fluorescence of the parapodial light glands inand allies (Phyllodocida: Tomopteridae). Zoologischer Anzeiger, 268: 112—125
Gowda G, Rajesh K M, Mridula R M, 2008. Abundance and vertical distribution of macrobenthos in a mangrove-fringed brackish water pond in Mangalore, India. The Ecoscan, 2(2): 181—186
Gupta B K S, Machain-Castillo M L, 1993. Benthic foraminifera in oxygen-poor habitats. Marine Micropaleontology, 20(3—4): 183—201
Hauenschild C, 1956. Hormonale hemmung der geschlechtsreife und metamorphose bei dem polychaeten. Zeitschrift für Naturforschung B, 11(3): 125—132
He S N, del Viso F, Chen C Y, 2018. An axial hox code controls tissue segmentation and body patterning in. Science, 361(6409): 1377—1380
Hilário A, Capa M, Dahlgren T G, 2011. New perspectives on the ecology and evolution of siboglinid tubeworms. PLoS One, 6(2): e16309
Hillebrand H, Brey T, Gutt J, 2018. Climate change: warming impacts on marine biodiversity. In: Salomon M, Markus T eds. Handbook on Marine Environment Protection. Cham: Springer, 353—373
Hourdez S, Weber R E, Green B N, 2002. Respiratory adaptations in a deep-sea orbiniid polychaete from Gulf of Mexico brine pool NR-1: metabolic rates and hemoglobin structure/function relationships. Journal of Experimental Biology, 205(11): 1669—1681
Hsueh P W, 2019. Two new species of nereidids (Annelida, Polychaeta) from Taiwan. Zootaxa, 4652(3): 544—556
Hui M, Kraemer W E, Seidel C, 2016. Comparative genetic population structure of three endangered giant clams (Cardiidae:) throughout the Indo-West Pacific: implications for divergence, connectivity and conservation. Journal of Molluscan Studies, 82(3): 403—414
Ingram T, 2011. Speciation along a depth gradient in a marine adaptive radiation. Proceedings of the Royal Society B: Biological Sciences, 278(1705): 613—618
Israel J W, Martik M L, Byrne M, 2016. Comparative developmental transcriptomics reveals rewiring of a highly conserved gene regulatory network during a major life history switch in the sea urchin genus. PLoS Biology, 14(3): e1002391
Jablonski D, Roy K, Valentine J W, 2006. Out of the tropics: evolutionary dynamics of the latitudinal diversity gradient. Science, 314(5796): 102—106
Jokiel P, Martinelli F J, 1992. The vortex model of coral reef biogeography. Journal of Biogeography, 19(4): 449—458
Jones-Knight P, Bowden N, 1984. Incubation and scissiparity in Sabellidae (Polychaeta). Journal of the Marine Biological Association of the United Kingdom, 64(4): 809—818
Keeling R E, Körtzinger A, Gruber N, 2010. Ocean deoxygenation in a warming world. Annual Review of Marine Science, 2: 199—229
Knope M L, Scales J A, 2013. Adaptive morphological shifts to novel habitats in marine sculpin fishes. Journal of Evolutionary Biology, 26(3): 472—482
Kong X, Liu H L, Li Y N, 2018. Two novel short peptidoglycan recognition proteins (PGRPs) from the deep sea vesicomyidae clam: identification, recombinant expression and bioactivity. Frontiers in Physiology, 9: 1476
Konstantinides N, AverofM, 2014. A common cellular basis for muscle regeneration in arthropods and vertebrates. Science, 343(6172): 788—791
Lan Y, Sun J, Tian R M, 2017. Molecular adaptation in the world's deepest-living animal: insights from transcriptome sequencing of the hadal amphipod. Molecular Ecology, 26(14): 3732—3743
Lanza A R, Seaver E C, 2018. An organizing role for the TGF-β signaling pathway in axes formation of the annelid. Developmental Biology, 435(1): 26—40
Lawrence J M, Byrne M, 1994. Allocation of resources to body components inand(Echinodermata: Echinoidea). Zoological Science, 11(1): 133—137
Li J Y, Zeng C, Yan G Y, 2019. Characterization of the mitochondrial genome of an ancient amphipodspMT-2017 (Pardaliscidae) from 10,908 m in the Mariana Trench. Scientific Reports, 9(1): 2610
Lin Q, Fan S H, Zhang Y H, 2016. The seahorse genome and the evolution of its specialized morphology. Nature, 540(7633): 395—399
Liu H G, Li S Q, Ugolini A, 2018. Tethyan closure drove tropical marine biodiversity: vicariant diversification of intertidal crustaceans. Journal of Biogeography, 45(4): 941—951
Liubicich D M, Serano J M, Pavlopoulos A, 2009. Knockdown ofrecapitulates evolutionary changes in crustacean appendage morphology. Proceedings of the National Academy of Sciences of the United States of America, 33(106): 13892—13896
Martin A, Serano J M, Jarvis E, 2016. CRISPR/Cas9 mutagenesis reveals versatile roles of hox genes in crustacean limb specification and evolution. Current Biology, 26(1): 14—26
Matias A M A, Riginos C, 2018. Revisiting the “Centre Hypotheses” of the Indo-West Pacific: idiosyncratic genetic diversity of nine reef species offers weak support for the Coral Triangle as a Centre of genetic biodiversity. Journal of Biogeography, 45(8): 1806—1817
Mayr E, 1982. The Growth of Biological Thought: Diversity, Evolution, and Inheritance. Cambridge, USA: The Belknap Press of Harvard University Press, 1—974
Melo D, Porto A, Cheverud J M, 2016. Modularity: genes, development and evolution. Annual Review of Ecology Evolution and Systematics, 47: 463—486
Méndez N, Linke-Gamenick I, Forbes V E, 2000. Variability in reproductive mode and larval development within thespecies complex. Invertebrate Reproduction and Development, 38(2): 131—142
Mergeay J, Santamaria L, 2012. Evolution and biodiversity: the evolutionary basis of biodiversity and its potential for adaptation to global change. Evolutionary Applications, 5(2): 103—106
Meyer N P, Seaver E C, 2009. Neurogenesis in an annelid: characterization of brain neural precursors in the polychaetesp. IDevelopmental Biology, 335(1): 237—252
Ming Y, Jian J B, Yu F, 2019. Molecular footprints of inshore aquatic adaptation in Indo-Pacific humpback dolphin (). Genomics, 111(5): 1034—1042
Narberhaus I, Krause J, Bernitt U, 2012. Threatened Biodiversity in the German North und Baltic Seas: Sensitivities Towards Human Activities and the Effects of Climate Change. Bonn, German: Federal Agency for Nature Conservation
Neal S, de Jong D M, Seaver E C, 2019. CRISPR/CAS9 mutagenesis of a single-gene blocks phototaxis in a marine larva. Proceedings of the Royal Society B: Biological Sciences, 286(1904): 20182491
Paxton H, 2015., a new brooding species of Onuphidae (Annelida) from Japan. Marine Biodiversity, 45(4): 711—717
Peichel C L, Nereng K S, Ohgi K A, 2001. The genetic architecture of divergence between threespine stickleback species. Nature, 414(6866): 901—905
Peng C J, Ren J L, Deng C, 2020. The genome of shaw’s sea snake () reveals secondary adaptation to its marine environment. Molecular Biology and Evolution, 37(6): 1744—1760
Poulton S W, Canfield D E, 2011. Ferruginous conditions: a dominant feature of the ocean through earth’s history. Elements, 7(2): 107—112
Renema W, Bellwood D R, Braga J C, 2008. Hopping hotspots: global shifts in marine biodiversity. Science, 321(5889): 654—657
Russell J M, Vogel H, Bijaksana S, 2016. The towuti drilling project: a new, long pleistocene record of Indo-Pacific climate//EGU General Assembly. Vienna Austria: EGU, EGU2016-2723
Scimone M L, Atabay K D, Fincher C T, 2020. Muscle and neuronal guidepost-like cells facilitate planarian visual system regeneration. Science, 368(6498): eaba3203
Scott G K, Fletcher G L, Davies P L, 1986. Fish antifreeze proteins: recent gene evolution. Canadian Journal of Fisheries and Aquatic Sciences, 43(5): 1028—1034
Seaver E C, 2016. Annelid models I:. Current Opinion in Genetics & Development, 39: 35—41
Shapiro M D, Marks M E, Peichel C L, 2004. Genetic and developmental basis of evolutionary pelvic reduction in threespine sticklebacks. Nature, 428(6984): 717—723
Shen Y J, Kou Q, Chen W T, 2016. Comparative population structure of two dominant species,(Munidopsidae:) and(Mytilidae:), inhabiting both deep-sea vent and cold seep inferred from mitochondrial multi-genes. Ecology and Evolution, 6(11): 3571—3582
Slotine J J, Lohmiller W, 2001. Modularity, evolution, and the binding problem: a view from stability theory. Neural Networks, 14(2): 137—145
Smith M S, Collins S, Raff R A, 2009. Morphogenetic mechanisms of coelom formation in the direct-developing sea urchin. Development Genes & Evolution, 219(1): 21—29
Sui J X, Li X Z, 2017. A new species and new record of deep-sea scale-worms (Polynoidae: Polychaeta) from the Okinawa trough and the South China Sea. Zootaxa, 4238(4): 562—570
Sun D A, Patel N H, 2019. The amphipod crustacean: an emerging comparative model of arthropod development, evolution, and regeneration. Wiley Interdisciplinary Reviews Developmental Biology, 8(5): e355
Sun J, Zhang Y, Xu T, 2017. Adaptation to deep-sea chemosynthetic environments as revealed by mussel genomes. Nature Ecology & Evolution, 1(5): 0121
Thorson G, 1950. Reproductive and larval ecology of marine bottom invertebrates. Biological Reviews, 25(1): 1—45
Thorson G, 1964. Light as an ecological factor in the dispersal and settlement of larvae of marine bottom invertebrates. Ophelia, 1(1): 167—208
Tita G, Desrosiers G, Vincx M, 2000. Predation and sediment disturbance effects of the intertidal polychaete(Sars) on associated meiofaunal assemblages. Journal of Experimental Marine Biology and Ecology, 243(2): 261—282
Tittensor D P, Mora C, Jetz W, 2010. Global patterns and predictors of marine biodiversity across taxa. Nature, 466(7310): 1098—1101
Tomasetti S J, Morrell B K, Merlo L R, 2018. Individual and combined effects of low dissolved oxygen and low pH on survival of early stage larval blue crabs,. PLoS One, 13(12): e0208629
True J R, Carroll S B, 2002. Gene co-option in physiological and morphological evolution. Annual Review of Cell and Developmental Biology, 18: 53—80
Vargas-Vila M A, Hannibal R L, Parchem R J, 2010. A prominent requirement for single-minded and the ventral midline in patterning the dorsoventral axis of the crustacean. Development, 137(20): 3469—3476
Verd B, Monk N A, Jaeger J, 2019. Modularity, criticality, and evolvability of a developmental gene regulatory network. Elife, 8: e42832
Vol’Kenshteĭn M V, 1984. The essence of biological evolution. Soviet Physics Uspekhi, 27(7): 515—537
von Dassow G, Meir E, Munro E M, 2000. The segment polarity network is a robust developmental module. Nature, 406(6792): 188—192
Wang K, Shen Y J, Yang Y Z, 2019. Morphology and genome of a snailfish from the Mariana Trench provide insights into deep-sea adaptation. Nature Ecology and Evolution, 3(5): 823—833
Wang L Y, Israel J W, Edgar A, 2020. Genetic basis for divergence in developmental gene expression in two closely related sea urchins. Nature Ecology & Evolution, 4(6): 831—840
Weber C, Zhou Y J, Lee J G, 2020. Temperature-dependent sex determination is mediated by pSTAT3 repression of. Science, 368(6488): 303—306
Williams D H C, Anderson D T, 1975. The reproductive system, embryonic development, larval development and metamorphosis of the sea urchin(Val.) (Echinoidea: Echinometridae). Australian Journal of Zoology, 23(3): 371—403
Williams T A, Nagy L M, 2001. Developmental modularity and the evolutionary diversification of arthropod limbs. Journal of Experimental Zoology, 291(3): 241—257
Wilson W H, 1991. Sexual reproductive modes in polychaetes: classification and diversity. Bulletin of Marine Science, 48(2): 500—516
Woodland D J, 1983. Zoogeography of the siganidae (pisces): an interpretation of distribution and richness patterns. Bulletin of Marine Science, 33(3): 713—717
Wray G A, 2007. The evolutionary significance of-regulatory mutations. Nature Reviews Genetics, 8(3): 206—216
Wray G A, Raff R A, 1991. Rapid evolution of gastrulation mechanisms in a sea urchin with lecithotrophic larvae. Evolution, 45(8): 1741—1750
Xu T, Sun J, Watanabe H K, 2018. Population genetic structure of the deep-sea mussel(Bivalvia: Mytilidae) in the Northwest Pacific. Evolutionary Applications, 11(10): 1915—1930
Yang J S, Lu B, Chen D F, 2012. When did decapods invade hydrothermal vents? Clues from the Western Pacific and Indian Oceans. Molecular Biology and Evolution, 30(2): 305—309
Zattara E E, Bely A E, 2016. Phylogenetic distribution of regeneration and asexual reproduction in Annelida: regeneration is ancestral and fission evolves in regenerative clades. Invertebrate Biology, 135(4): 400—414
Zhao D, Li Q, Kong L F, 2017. Cryptic diversity of marine gastropod(Trochidae): did the early pleistocene glacial isolation and sea surface temperature gradient jointly drive diversification of sister species and/or subspecies in the Northwestern Pacific? Marine Ecology, 38(4): e12443
Zheng P, Wang M X, Li C L, 2017. Insights into deep-sea adaptations and host-symbiont interactions: a comparative transcriptome study onmussels and their coastal relatives. Molecular Ecology, 26(19): 5133—5148
Zhou N, Wilson K A, Andrews M E, 2003. Evolution of OTP-independent larval skeleton patterning in the direct-developing sea urchin,. Journal of Experimental Zoology Part B Molecular and Developmental Evolution, 300(1): 58—71
REVIEW ON ADAPTIVE EVOLUTION AND BIODIVERSITY OF MARINE ANIMALS
ZHANG Lin-Lin1, 2, 3, 4, LI Yong-Nan1, 2, 3, WENG Jie-Yang1, 4, LI Jie1, 4, WANG Yue1, 4, LI Qian1, 4
(1. Key Laboratory of Experimental Marine Biology, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China; 2. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China; 3. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China; 4. University of Chinese Academy of Sciences, Beijing 100049, China)
Marine biodiversity are important components in marine ecosystem, understanding the generation and evolution of marine biodiversity is of great significance for the conservation and sustainable utilization of marine biological resources. However, understanding of the molecular basis of marine biodiversity remains poor. The evolution of morphological traits and the process of adaptation have provided key information of the evolution in biodiversity. We summarized recent advances in the adaptive evolution and biodiversity in the evolutionary developmental biology perspective. We reviewed classical adaptive traits, studied the strategies and emerging model organisms in this field, with a focus on the advances in the Indo-Pacific Convergence Region. We further discussed the driving forces that generate and maintain the marine diversity center in this region. A major goal for the future will be to determine morphological diversity and characterize its genetic diversity combining both oceanographic and experimental biology strategies. This in turn will help to explain the ocean’s richest biodiversity hotspot in the Indo-Pacific Convergence Region.
marine biodiversity; adaptive evolution; Indo-Pacific Convergence Region; EvoDevo
* 中国科学院B类战略先导科技专项, XDB42000000号; 国家自然科学基金面上项目, 41976088号; 中国科学院海洋大科学研究中心重点部署项目, COMS2019R01号; 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室引进人才支持计划项目, YJ2019NO01号。张琳琳, 博士, 研究员, E-mail: linlinzhang@qdio.ac.cn
李勇男, 博士, 特别研究助理, E-mail: lyn@qdio.ac.cn
2020-07-04,
2020-10-04
Q951+.3
10.11693/hyhz20200700192

