谷子PHR 家族基因的生物信息学及表达模式分析
吴年隆,舒 军,王啸旗,张 银,韩渊怀,赵雄伟,杨致荣
(1.山西农业大学基础部,山西太谷 030801;2.山西农业大学生命科学学院,山西太谷 030801;3.山西农业大学农学院,山西太谷 030801)
磷(P)是植物生长、发育和代谢所必需的矿质营养元素之一[1],植物主要通过根系从土壤溶液中吸收无机磷[2]。然而,磷在酸性和碱性土壤中磷易与铁、钙、铝等离子反应,生成难溶性磷[3],不利于植物吸收和同化。在农作物的农业生产中,需过量施用磷肥以弥补土壤中有效磷,但此过程会造成严重的环境污染。因此,利用分子生物学手段挖掘解析植物磷高效利用的分子机制,对磷缺乏土壤的可持续农业生产具有重要意义。
植物为了适应低磷生态环境,进化出一套复杂的基因调控网络,其中与磷酸盐吸收转运相关成员中最重要的有PHR1(Phosphate Starvation Response1)、IPS1(Induced by Phosphate Starvation1)、miR399(microRNA399)、PHO2(Phosphate2)和PT(Phosphate Transport)[4-5]。在这个复杂的调控网络中,PHR 蛋白是植物磷调控网络中的重要转录因子,在磷酸盐饥饿诱导下起着重要的信号转导调控作用[6]。目前,拟南芥、水稻、玉米等多个物种已在相应的全基因组水平上鉴定了PHR 家族基因[7-9],其中,拟南芥的AtPHR1 基因功能缺失会重塑膜脂代谢、初级代谢和次生代谢以及光合作用等,从而影响到拟南芥根冠的生长速率和花青素的积累等生理过程[10];水稻中含有MYB-CC 型结构域的基因OsPHR1、OsPHR2 和OsPHR3,其中任意一个基因功能的缺失都会抑制水稻根毛的伸长[4],进而影响植物对磷酸盐的有效吸收。
谷子(Setaria italica,2n=18)是起源于我国的一个古老作物,广泛栽培于欧亚大陆的温带和热带地区[11],具有突出的抗旱、耐瘠薄和高光效等特性,已发展为旱生C4 禾谷类模式作物[12]。为了挖掘谷子耐低磷的优异基因,本研究基于已经公布的谷子全基因组序列[13],利用生物信息学方法鉴定了谷子基因组中PHR 基因家族成员,并对其基因结构、顺式作用元件、基因表达模式等进行了分析,以进一步探究谷子、水稻、高粱、玉米和拟南芥PHR 基因家族的进化关系,为谷子PHR 基因的耐低磷调控网络研究提供一定的理论参考。
1 材料和方法
1.1 谷子PHR 基因家族成员的鉴定及其蛋白理化性质分析
以功能已知的水稻OsPHR1[4]转录因子的蛋白序列为靶标序列,在水稻基因组中进行同源序列比对(同源性P<10e-10),得到水稻所有OsPHR 基因后,在利用hmmbuild 软件对检索到的水稻OsPHR基因建立HMM 模型[14],然后在谷子基因组数据中搜索同源基因,下载谷子PHR 家族基因的蛋白序列和编码序列。利用在线网站Pfam(http://pfam.xfam.org/)进行结构域分析,手动去除不含MYB-CC 结构域的序列。最后利用在线网站ExPASy(https://web.expasy.org/protparam/)分析谷子SiPHR 基因的分子量、氨基酸长度和等电点等蛋白理化性质。
1.2 SiPHR 基因家族成员的系统进化关系分析
从Phytozome 数据库(https://phytozome.jgi.doe.gov/pz/portal.html)下载水稻(Oryza sativa)、高粱(Sorghum bicolor)和拟南芥(Arabidopsis thaliana)PHR 家族基因的蛋白序列,从NCBI 数据库(https://www.ncbi.nlm.nih.gov/)下载玉米(Zea mays)的PHR家族基因的蛋白序列。利用MEGA 7.0 软件[15]对谷子、水稻、高粱、玉米和拟南芥的PHR 家族基因的蛋白序列进行同源多序列比对,并采用邻接法(NJ)构建系统发育树,Bootstrap 值为1 000。
1.3 SiPHR 基因家族成员的保守结构域和基因结构分析
使用在线网站MEME(http://meme-suite.org/tools/meme)预测SiPHR 基因的保守基序,Motif(基序)数设置为10 个,并使用在线网站SMART(http://smart.embl-heidelberg.de/)预测Motif 的类型;最后,基于谷子基因组注释,在TBtools 软件[16]上绘制基因结构。
1.4 SiPHR 基因家族成员的启动子顺式作用元件分析
启动子在调控基因的转录和表达方面有着重要的作用。为了了解谷子SiPHR 基因可能存在的调控和响应机制,在Phytozome 数据库上下载谷子PHR 家族各基因上游1 500 bp 的基因组序列,然后递交至PlantCARE 在线网站上(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/),并进行启动子顺式作用元件分析,最后使用TBtools 软件[16]进行可视化操作。
1.5 SiPHR 基因家族成员的组织表达分析
根据Phytozome 数据库(https://phytozome.jgi.doe.gov)下载谷子PHR 基因转录组数据,分别对叶、芽、穗和根中基因表达进行分析。利用Heatmapper(http://www2.heatmapper.ca/)在线网站绘制热图。
1.6 低磷胁迫下根中SiPHR 基因家族成员的差异表达分析
为了进一步筛选根中响应低磷诱导的SiPHR基因,本研究采用实时荧光定量方法(qRT-PCR)检测SiPHR 基因在低磷条件下的表达量。从前期研究中筛选出来的耐低磷品种B376[17],使用水培法[18]对萌发10 d 谷子幼苗进行培养,每4 d 更换一次营养液,温度为28 ℃,相对湿度为50%左右,光照强度30 000 lx,光周期为14 h/10 h(光/暗),正常磷浓度设置为0.25 mmol/L。之后将正常生长21 d 的幼苗进行低磷(磷浓度为0.005 mmol/L)胁迫处理,在第0、1、3、5 天剪取谷子的根系,冲洗干净后保存在-80 ℃中备用。
利用Trizol 法提取不同胁迫时间下谷子根部样品的总RNA,按照Primer Script RT reagent Kit(Takara)试剂盒说明书对提取到的谷子根部RNA进行反转录,得到cDNA。利用Primer 6.0 和DNAMAN 软件设计SiPHR 基因家族成员PCR 引物(表1)。以SYBR Green 为颜料进行qRT-PCR。15 μL 反应体系包括:2×Taq PCR Master Mix(含荧光染料)7.5 μL、引物10 μmol/L 各0.6 μL、ddH2O 3.3 μL 和cDNA 模板3 μL。反应条件为:95 ℃2 min,95 ℃5 s,60 ℃30 s,72 ℃30 s,35 个循环;4 ℃10 min 保存。
以谷子Actin 基因(Si9g37480)为内参(F:5′-ATGGCCACCAAGCAAACT-3′,R:5′-GGATCTCAC GGAGGGCAACAG T-3′),根据2-ΔΔCt计算SiPHR 基因家族成员的相对表达量。以0 d 为对照组,采用t检验法分析不同低磷胁迫时期的SiPHR 基因表达量的显著性。

表1 SiPHR 实时荧光定量PCR 分析引物
2 结果与分析
2.1 SiPHR 基因家族的鉴定及其理化性质分析
以功能已知的OsPHR 蛋白序列为靶序列,利用HMM 模型的同源比对方法从谷子基因组中共鉴定出16 个SiPHR 候选基因。根据谷子PHR 基因家族成员与拟南芥AtPHR1 基因序列的相似性,由近到远分别命名为SiPHR1~SiPHR16(表2)。
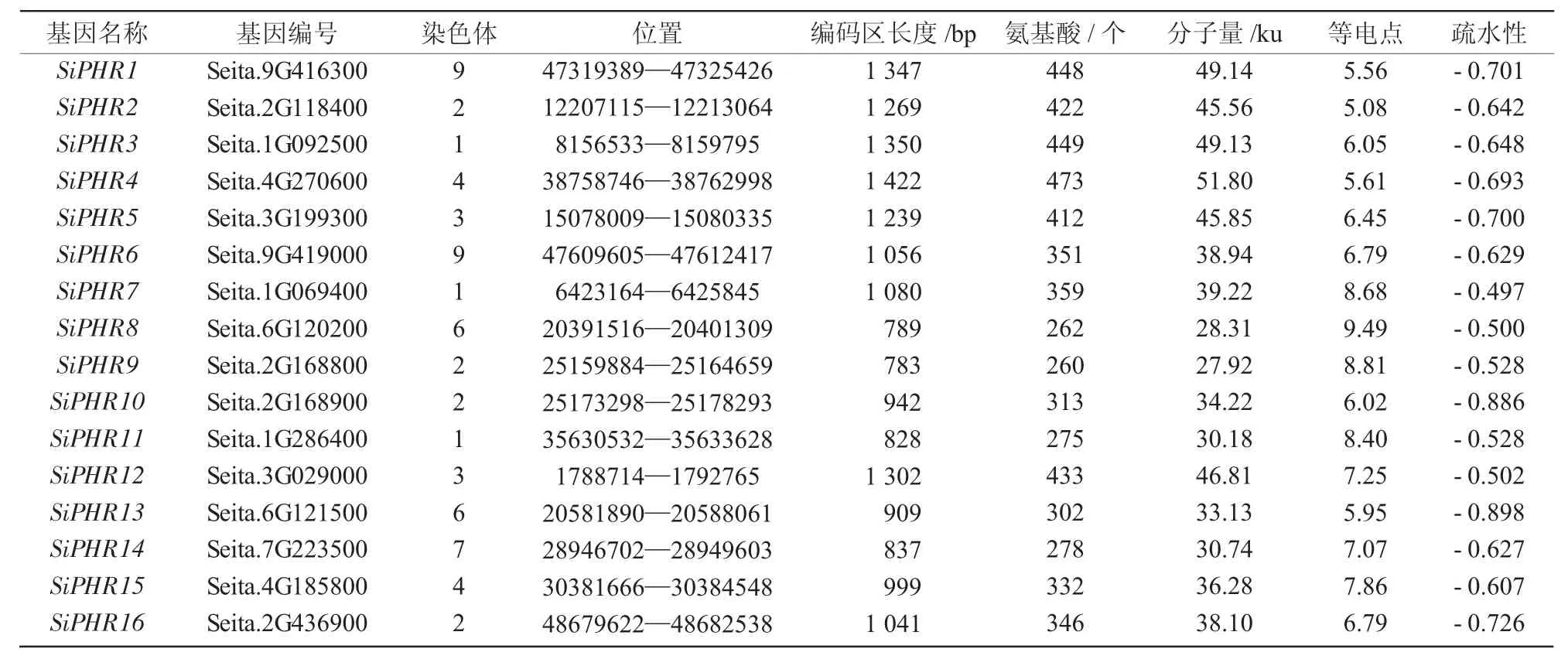
表2 谷子PHR 基因家族成员基本信息
16 个SiPHR 基因分布在7 条染色体上,其中,2 号染色体上分布有4 个;1 号染色体上分布有3个;3 号、4 号、6 号和9 号染色体上各分布2 个;7 号染色体上有1 个SiPHR 基因。16 个SiPHR 基因的编码序列长度在0.7~1.5 kb,编码的氨基酸数量在260~473 个。蛋白的分子量在27.92~51.80 ku;其中7 个SiPHR 蛋白的等电点大于7,呈碱性,9 个SiPHR 蛋白的等电点小于7,呈酸性。SiPHR 蛋白的平均疏水性系数都为负数,均为亲水性蛋白。
2.2 SiPHR 基因家族成员系统进化分析
为了研究谷子PHR 基因家族的系统进化关系,将16 个谷子SiPHR 蛋白序列与18 个水稻Os-PHR 蛋白序列、18 个玉米ZmPHR 蛋白序列、16 个高粱SbPHR 蛋白序列和14 个拟南芥AtPHR 蛋白序列进行比对,并构建系统发育树如图1 所示,将SiPHR 家族成员划分为5 个亚组(ClassⅠ~ClassⅤ)。其中,ClassⅠ有5 个SiPHR 蛋白成员,包括SiPHR1、SiPHR2、SiPHR3、SiPHR4 和SiPHR12;Class Ⅱ和Class Ⅲ均含有2 个成员,分别为SiPHR10 和SiPHR13、SiPHR11 和SiPHR14;ClassⅣ有3 个成员,分别为SiPHR5、SiPHR6 和SiPHR16;ClassⅤ有4 个成员,分别为SiPHR7、SiPHR8、SiPHR9、SiPHR15。

从5 个亚组的聚类情况可以看出,谷子与水稻、玉米和高粱聚在一起,且基本都是成组出现,即每个SiHPR 分别对应水稻、高粱和玉米的1~3 个PHR 同源蛋白。其中,SiPHR5、SiPHR6、SiPHR7、SiPHR8、SiPHR9、SiPHR11、SiPHR13、SiPHR14 分别对应1 个水稻、高粱和玉米PHR 蛋白;SiPHR1、SiPHR2、SiPHR4、SiPHR12、SiPHR16、SiPHR15 对应2 个水稻、高粱和玉米PHR 蛋白;而SiPHR3、SiPHR10 对应3 个水稻、高粱和玉米PHR 蛋白。值得注意的是,SiPHR4 与SiPHR13 分别由SiPHR3与SiPHR10 进化而来。此外,不论在哪个亚组中,拟南芥AtPHR 总是单独聚在一个分支。结果表明,谷子SiPHR 与水稻OsPHR、高粱SbPHR、玉米ZmPHR的亲缘关系明显近于与拟南芥AtPHR 的亲缘关系。因为谷子、水稻、高粱、玉米是单子叶植物,而拟南芥属于双子叶植物,推测这些单双子叶进化时PHR基因在不同植物纲中发生了生物功能上的分化。
2.3 SiPHR 基因家族成员保守结构域和基因结构分析
对SiPHR 基因家族成员保守结构域的分析结果显示(图2-a),所有SiPHR 蛋白中都存在Myb_DNA-binding 和Myb_CC_LHEQLE,而其他Motif 均为功能未知基序,这还有待进一步研究。其中,Motif 3 存在ClassⅠ、ClassⅡ、ClassⅢ亚组中,Motif 6为ClassⅡ亚组特有,Motif 8 和Motif 10 为ClassⅢ亚组特有,Motif 9 为ClassⅣ亚组的2 个SiPHR 特有。值得注意的是,ClassⅤ的SiPHR 蛋白基序类型有所差异,其中,SiPHR3 和SiPHR4 存在4 种Motif,而SiPHR1 和SiPHR2 存在3 种Motif,SiPHR12只含有2 种Motif。这些特殊保守基序可能是不同的SiPHR 参与不同生物学功能的主要因素。
SiPHR 基因家族成员进行基因结构分析发现(图2-b),同一亚组的PHR 成员基因结构类似,不同亚组间PHR 成员的外显子数差异不大。除ClassⅠ的SiPHR9 基因和ClassⅤ的SiPHR 基因均含有7 个外显子外,其他SiPHR 基因均含有6 个外显子。

2.4 SiPHR 基因家族成员的启动子顺式作用元件分析
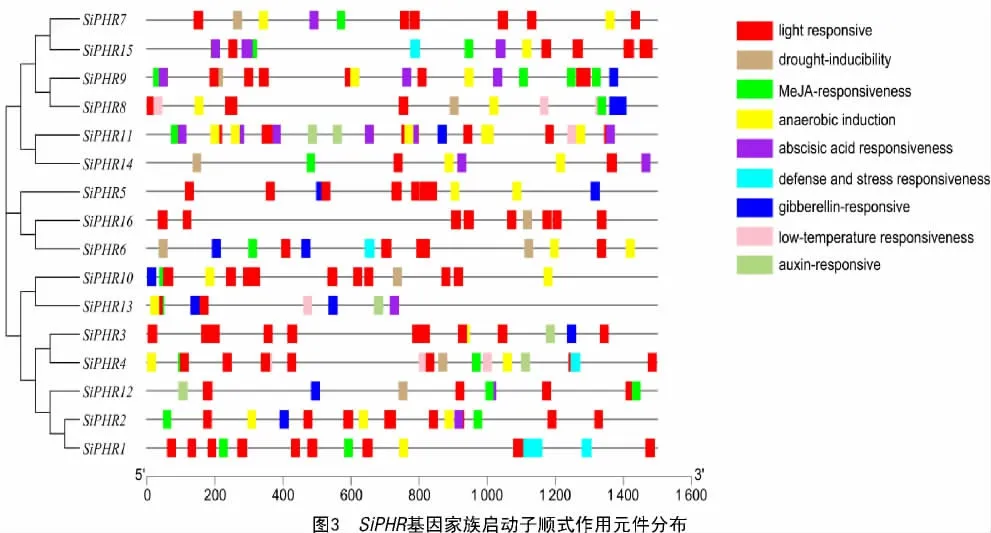
为进一步阐明谷子PHR 家族基因在非生物胁迫中可能存在的调控机制,利用PlantCARE 数据库对启动子序列进行了分析,结果如图3 所示,共鉴定出9 类顺式作用元件,包括光响应、干旱诱导、茉莉酸甲酯、厌氧诱导、脱落酸、抗逆、赤霉素、低温、生长素顺式作用元件;从组成数量上看,SiPHR 基因平均含有4.8 个顺式作用元件,均包含光响应元件,其中,SiPHR11 和SiPHR14 包含的响应元件种类最多(7 种),而SiPHR16、SiPHR3 和SiPHR5 包含的顺式响应元件最少(2~3 种);从组成类型特异性来看,含有低温响应元件的基因有SiPHR4、SiPHR8、SiPHR11 和SiPHR13,含有干旱或茉莉酸甲酯响应元件的基因有SiPHR3、SiPHR5 和SiPHR13,含有生长素响应元件的基因有SiPHR3、SiPHR4、SiPHR11、SiPHR12 和SiPHR13。ClassⅠ、ClassⅡ中除SiPHR8 外其他SiPHR 基因均包含脱落酸,而ClassⅢ、ClassⅣ和ClassⅤ的大部分基因不包括脱落酸,但含有大量的生长素和赤霉素响应顺式作用元件。结果表明,SiPHR 基因的表达不仅受到光诱导的调节,而且在干旱、涝渍(厌氧)等抗逆胁迫中也发挥着作用。其中,SiPHR3、SiPHR5 和SiPHR16 启动子区包含顺式元件较少,仅与光、赤霉素和生长素、厌氧诱导作用有关。
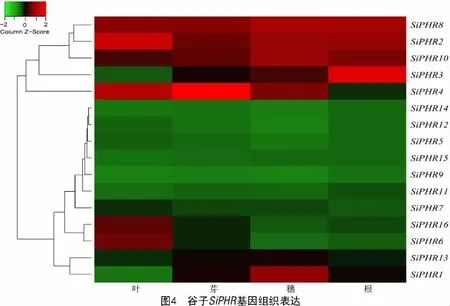
2.5 SiPHR 家族基因的组织表达分析
通过对SiPHR 基因组织表达进行分析发现,除SiPHR9 外,其余15 个SiPHR 基因在叶、芽、穗和根中均有一定的表达(图4)。其中,SiPHR2、SiPHR8和SiPHR10 在4 个组织中均高表达(FPKM均大于13),表明这3 个基因在谷子的各个形态建成方面都发挥了重要作用;SiPHR3 在根中高表达,而SiPHR4 在根中表达量最低;SiPHR6 和SiPHR16 在叶片中有较高表达,结合SiPHR16 启动子顺式元件结构推测,其可能在叶片的光合作用中发挥着重要的作用;SiPHR1 在穗中有较高的表达,其余7 个基因 SiPHR5、SiPHR7、SiPHR11、SiPHR12、SiPHR13、SiPHR14 和SiPHR16 在叶、芽、穗和根中的表达量都较低。由此表明,SiPHR 基因在谷子的各个组织中都发挥了作用,其中,SiPHR1、SiPHR3、SiPHR4、SiPHR6 和SiPHR16 具有明显的组织表达特异性。
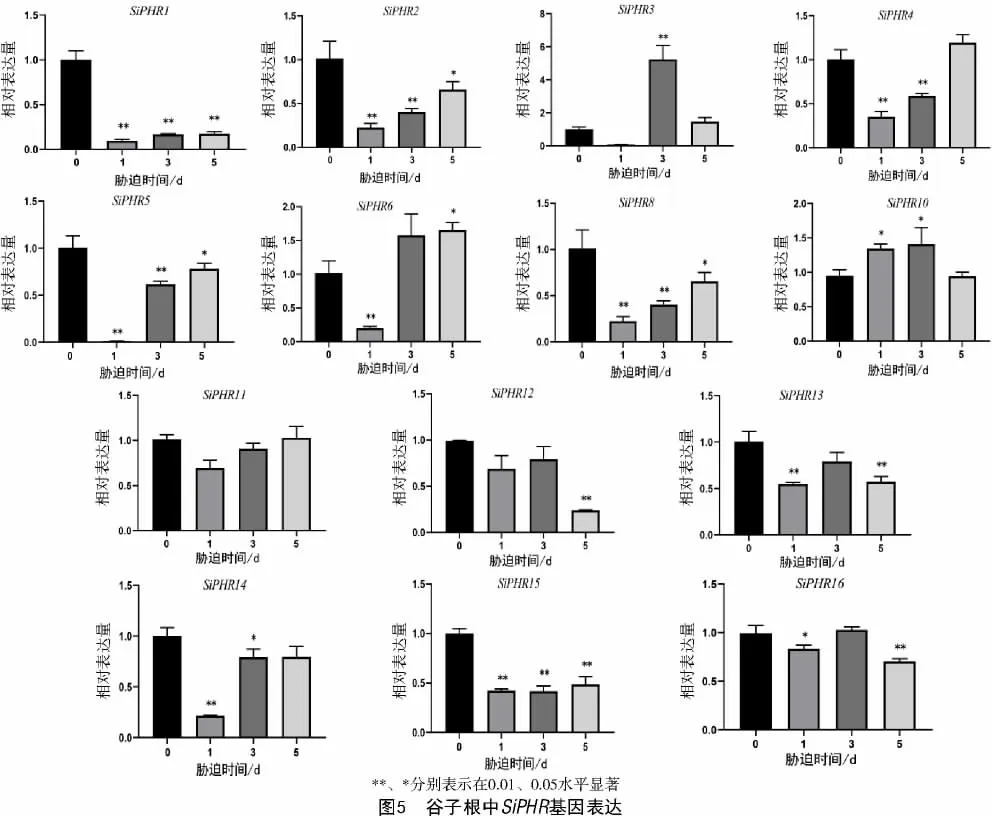
2.6 在低磷胁迫条件下的根中SiPHR 基因的表达模式分析
对低磷胁迫后根系的16 个SiPHR 基因进行差异表达分析显示,除SiPHR7 和SiPHR9 未检测到外,其余SiPHR 基因在低磷处理后,表达量都受到了一定程度的影响(图5),SiPHR1 和SiPHR15 的表达在低磷胁迫后都明显被抑制;SiPHR2、SiPHR4、SiPHR5、SiPHR6、SiPHR8、SiPHR11、SiPHR12 和SiPHR14 在低磷胁迫后表达量持续上升,其中,SiPHR4、SiPHR6、SiPHR11 和SiPHR12 都恢复或超过胁迫前水平。此外,SiPHR3、SiPHR10、SiPHR13、SiPHR16 在低磷胁迫后呈现先上升后下降的趋势。值得注意的是,SiPHR3 的表达量在胁迫后3 d 显著高于胁迫前水平,SiPHR10 的表达量在胁迫后1 d显著高于胁迫前水平,SiPHR16 在低磷胁迫3 d 的表达也恢复到胁迫前的水平。
3 结论与讨论
磷是植物生长发育所必需的营养元素,约占干质量的0.2%。磷缺乏是限制作物产量的一个主要因素,植物已进化出一系列的形态、生理和分子策略来适应磷缺乏的症状[19],这些策略大多数都是通过增强土壤中磷的流动性或者根系对磷的获取,从而提高对磷的利用效率。近年来,调控低磷胁迫的相关基因和蛋白已被陆续发现和鉴定,其中,PHR是一种MYB 型转录因子,在植物磷饥饿反应中起着至关重要的调节作用[20-22]。已有报道,PHR1 和PHR1类基因在拟南芥[23]、水稻[4]、豆类[24]、小麦[20]、玉米[25]和油菜[26]等植物的磷信号调控网络中起着关键作用。此外,拟南芥和水稻的全基因组转录分析表明,大多数磷饥饿响应基因都是被AtPHR1 和OsPHR2 以及其同源基因AtPHL1、AtPHL2、OsPHR1 和OsPHR3诱导而激活[4,27-28]。本研究通过生物信息学的方法共鉴定出16 个谷子PHR 基因,其数量与拟南芥(15 个)、水稻(12 个)、玉米(18 个)和高粱(16 个)相当[29]。但在前人报道中,大豆基因组中存在35 个PHR 基因[24],与本研究数量相差较大的原因除与染色体数目和基因组大小有关外,还与大豆会产生根瘤菌、需要更多的转录因子来促进氮磷协同吸收有关。
对谷子与水稻、玉米、高粱和拟南芥的亲缘关系进行鉴定,16 个SiPHR 蛋白被分为5 个亚组,每个SiHPR 分别对应水稻、玉米和高粱的1~3 个PHR 同源蛋白,主要原因可能是谷子和水稻、玉米、高粱同为单子叶作物,而拟南芥为双子叶作物。此外,谷子SiPHR4 与SiPHR13 分别由SiPHR3 与SiPHR10 进化而来。基因结构分析SiPHR 基因均含有MYB-CC 和Myb_DNA-binding 保守结构域,与拟南芥中PHR 基因家族的结构域特征相同[30]。顺式作用元件通过响应不同的外界信号来调节基因转录过程,进而影响植物的生长发育[31]。已有研究发现,磷酸化信号转导和磷饥饿响应受光、糖、植物激素(生长素、乙烯、细胞分裂素和赤霉素)以及氧和影响[32-35]。如拟南芥中,AtPHR1 的表达会受到光和乙烯的协同调节,且通过AtPHR 基因的启动子实现对磷饥饿响应的调控[36]。本研究中,SiPHR 基因启动子中共鉴定出了9 类顺式作用元件,大量的光响应元件和激素类元件表明,SiPHR 基因的表达和调控受光和激素的影响。
已有报道发现,玉米、水稻和高粱大多数PHR基因在所有组织中均有连续表达,表明它们可能在调节磷摄取和转运方面起着重要作用[4,28,31]。本研究对谷子PHR 家族基因在叶、芽、穗和根中的组织表达进行了分析,发现谷子PHR 家族基因存在组织特异性表达,这与其他禾本科作物PHR 基因的表达情况相似[29]。在组织表达分析中发现,SiPHR3 在根中的表达量极高(FPKM 为37.64),在启动子顺式作用元件中含有与抗逆有关的赤霉素和生长素类响应元件。此外,SiPHR3 在低磷胁迫后第3 天时表达量远远超过了胁迫前的表达水平,故推测SiPHR3 可能与非生物逆境下重塑根部形态有关。
综上所述,本研究在谷子基因组中鉴定了16 个SiPHR 基因,这些基因的氨基酸数量和等电点特性存在很大的差异。此外,SiPHR 在启动子区存在大量的光响应、生物和非生物胁迫(低温、涝渍、干旱等)相关的顺式作用元件。进一步对低磷胁迫后根中SiPHR 基因差异表达分析发现,SiPHR3 和SiPHR10在低磷处理3 d 后显著高表达,此研究将为后续SiPHR 基因的功能研究和谷子新品种选育奠定一定基础。

