基于数据库挖掘:TRIM44蛋白在结直肠癌中的表达差异分析
王鹏宇, 胡圣晨, 陈浙南, 王 彦, 姜 莹
(1.牡丹江医学院,黑龙江省牡丹江市157000;2.南华大学附属第一医院,湖南省衡阳市421000)
结直肠癌(colorectal cancer,CRC)是最常见的恶性肿瘤之一[1]。目前治疗方式仍以手术及放化疗为主,靶向治疗逐渐进入临床并取得一定疗效,但易出现耐药现象,晚期CRC患者预后仍未得到明显改善。因此寻找新的诊断及治疗靶点尤为重要。对CRC相关基因的研究有利于对关键基因进行鉴定或找到新的靶点,以提高CRC的筛查效率、改善晚期CRC患者的预后。
TRIM(tripatite motif)蛋白家族是参与泛素化过程的E3连接酶,介导泛素从E2结合酶到特定靶点上的转移[2]。TRIM44蛋白作为家族成员与细胞增殖、DNA修复、信号转导以及转录等各种生理学过程密切相关[3]。最初研究发现TRIM44可能在神经元细胞的分化和成熟过程中起作用,调节TRIM17的活动,是PAX6表达的负调控因子[4]。目前研究指出TRIM44蛋白在多种肿瘤的发病过程中起着重要作用,可通过Wnt/β-catenin[5]、Akt/p21/p27[6]、mTOR[7-10]、NF-κB[11-12]等信号通路在甲状腺癌、神经胶质细胞癌、肺癌、肝癌等多种癌症中发挥作用,使得TRIM44在肿瘤中的研究逐渐得到重视。
本研究对多个数据库进行挖掘,旨在阐明TRIM44在CRC中的表达及意义,为进一步实验研究奠定理论基础。
1 材料和方法
1.1 数据下载
结直肠癌的原始数据从TCGA(https://portal.gdc.cancer.gov/)数据库下载,检索条件如下:文件数据类型:转录组分析;数据类型:基因表达定量;工作流程类型:HTSeq-FPKM;样本主要位点:Colon;方案:TCGA-COAD TCGA-READ(TCGA Project)。
免疫浸润的原始数据来源于GEO数据库大肠癌基因表达谱研究(GSE113513)中的基于Affymetrix人类基因表达阵列平台(GPL15207)的28个组织芯片。
1.2 基因差异分析
利用Active Perl(5.28.1Build 2801)编写的脚本将count数据(正常样本量32、肿瘤样本量375)与人类基因组注释文件(gtf)进行合并生成单基因样本的mRNA表达矩阵,然后利用R for Windows 4.0.3编写的脚本进行结肠癌(COAD)与直肠癌(READ)的散点差异分析以及配对差异分析。
通过GEPIA数据库中基于TCGA和GTEx的8 587个正常组织样本,9 736个肿瘤组织样本的RNA测序表达数据,分析TRIM44在31种肿瘤中的差异表达。随后,通过Oncomine数据库显示CRC临床标本中过度表达或DNA拷贝数量较多的基因,设置基因TRIM44[P=all,倍数变化=1.5,阈值(按基因排序)=all,数据类型=all],筛选出CRC数据,并设置分析类型为癌-常分析、数据类型为mRNA,再加入限制条件生存状态,得到来自12个数据库的675个正常样本与53例肿瘤样本,对其在不同肿瘤组织中的差异表达情况进行排序。
1.3 生存分析
利用Oncolnc交互式探索生存相关性并下载与mRNA表达数据耦合的临床数据。以P<0.05判定差异有显著性,进行Kaplan-Meier分析并创建生存图。
1.4 基因共表达分析
基于LinkedOmics数据库中来自32种TCGA癌症类型的数据与临床蛋白质组学肿瘤分析协会(CPTAC)生成的基于质谱的蛋白质组学数据。查看TRIM44在CRC中的基因共表达情况,选择癌症队列为COADREAD,设置搜索数据库与目标数据均为样本队列TCGA_COADREAD(样本队列:TCGA_COADREAD,研究机构:UNC,数据类型:RNAseq,平台:HiSeq RNA,数据日期:01/28/2016,研究机构:BI,分析层面:Gene),进行Spearman相关性检验。
1.5 GO分析和KEGG通路分析
导出LinkedOmics数据库中上述共表达数据,分别筛选与TRIM44正相关或负相关的前50个基因,导入David进行GO功能分析与KEGG通路富集分析,筛选最具代表性的前5个通路依次制表。
1.6 肿瘤免疫浸润相关性分析
使用TIMER网络分析工具,设置检索基因TRIM44,限制癌症类型为COAD与READ,探究TRIM44在CRC中B细胞、CD8+T细胞、CD+T细胞、巨噬细胞、中性粒细胞与树突状细胞的免疫浸润情况,以P<0.05判定差异有显著性。
利用GEO芯片数据库筛选符合研究条件的样本,并下载来自GSE113513的矩阵数据与平台文件GPL15207。利用perl进行ID转换分类排序,利用Rlimma包将数据正常化,运行CIBERSORT输出结果并利用perl对其进行过滤(以P<0.05为标准筛选)。然后使用R pheatmap、corrplot、vioplot包与一些自定义函数进行结果的可视化与再分析,并在GEPIA查看免疫细胞相关基因与TRIM44的共表达情况。
1.7 统计学处理
通过R软件4.0.3版进行分析。除非另有说明,默认P<0.05具有统计学意义。
2 结 果
2.1 TRIM44在肿瘤组织和正常组织中的差异表达
通过GEPIA数据库分析TRIM44在31项肿瘤中的表达水平,获得所有肿瘤样品和正常组织配对的基因表达谱,单基因分析结果显示,TRIM44在肿瘤表达的差异性较大,其中在CRC组织中呈现高表达(图1A),COAD组织较正常组织TRIM44表达高出约20%,READ组织较正常组织TRIM44表达高出约26%。随后,通过Oncomine分析TRIM44在肿瘤中的表达情况,从癌-常对比中,可看出TRIM44在CRC中的表达较正常组织较高,并在癌-癌对比中和癌症组织学与多癌症对比中显示出明显差异(图1B)。对TCGA数据库提取的数据进行R语言分析,结果显示TRIM44在COAD的肿瘤组织与正常组织表达有明显差异(P<0.001),配对差异分析显示,COAD组织中TRIM44高表达(P<0.05)(图1C)。READ样本量少,癌-常差异表达(P=0.561)和配对差异分析(P=0.333)均无统计学意义(图1D)。
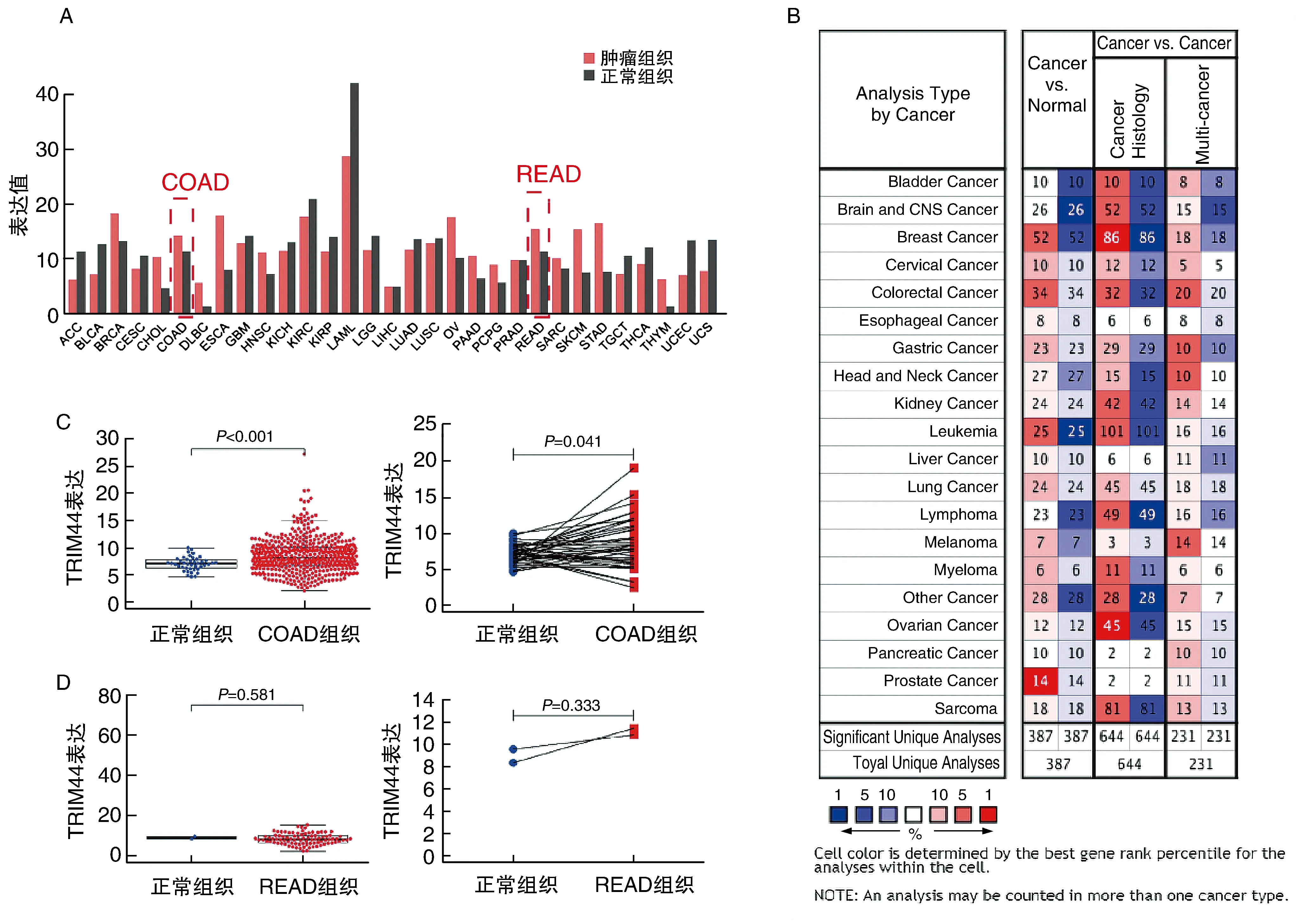
图1 TRIM44的差异表达情况A为GEPIA数据库;B为Oncomine数据库;C和D为TCGA数据库。
2.2 TRIM44表达与结直肠癌患者预后的关系
通过使用Oncolnc工具分析TRIM44 mRNA表达与CRC患者预后的关系,从TRIM44 Cox回归结果发现,22例(5%)COAD肿瘤组织低表达与22例(5%)COAD组织高表达患者预后差异有显著性(P<0.05)(图2)。23例(15%)READ肿瘤组织低表达与23例(15%)READ肿瘤组织高表达患者预后差异有显著性(P<0.05)(图2)。TRIM44 mRNA高表达COAD患者其总体生存率要低于低表达COAD患者,而TRIM44 mRNA高表达READ患者其总体生存率要高于低表达READ患者。
2.3 TRIM44与其相关基因的共表达和富集分析
基因的共表达反映了构成功能关系的常见遗传风险因素,因此本文研究了在结肠中TRIM44与其他基因表达的共存情况。利用LinkedOmics在CRC中检查了其他基因与TRIM44的共表达情况(图3A)。Spearman相关系数检验结果显示,19 828个基因条目中,ASXL2、TAOK1、REST、KIAA0754、FAM168A、TGFBRAP1、PARD3B、RAD54L2、STRN等基因与TRIM44表达呈正相关,MRPL55、KRTCAP2、C9orf142、BLOC1S1、PSMG3、CHCHD1、FAM128B、NDUFC1、WIBG等基因与TRIM44表达呈负相关(图3B)。
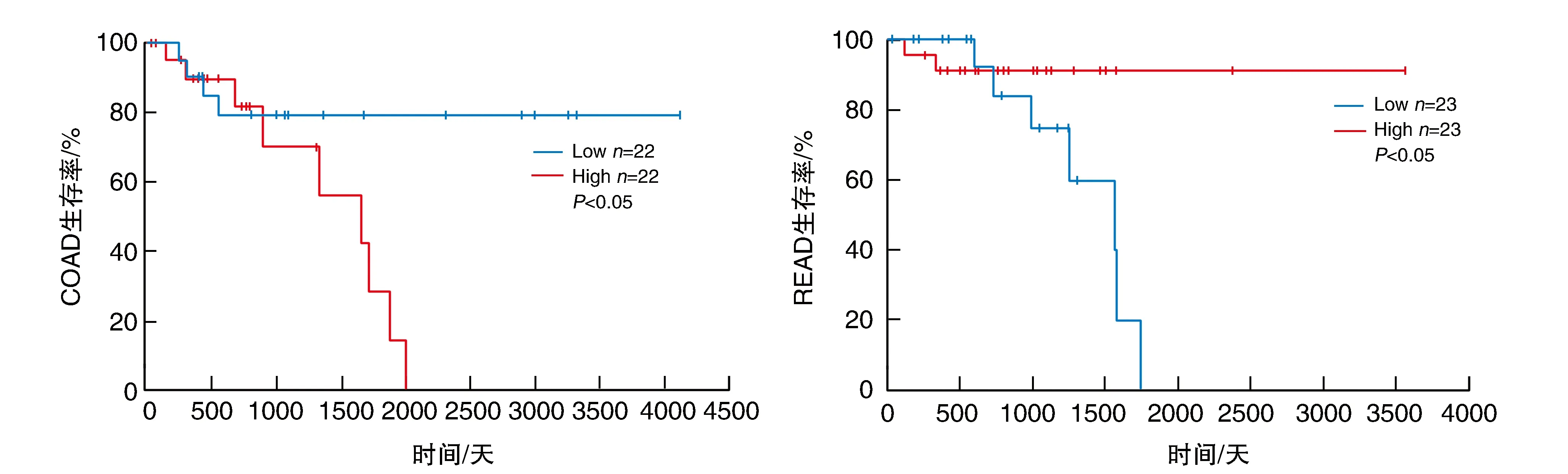
图2 TRIM44表达与COAD和READ患者生存预后的相关性分析
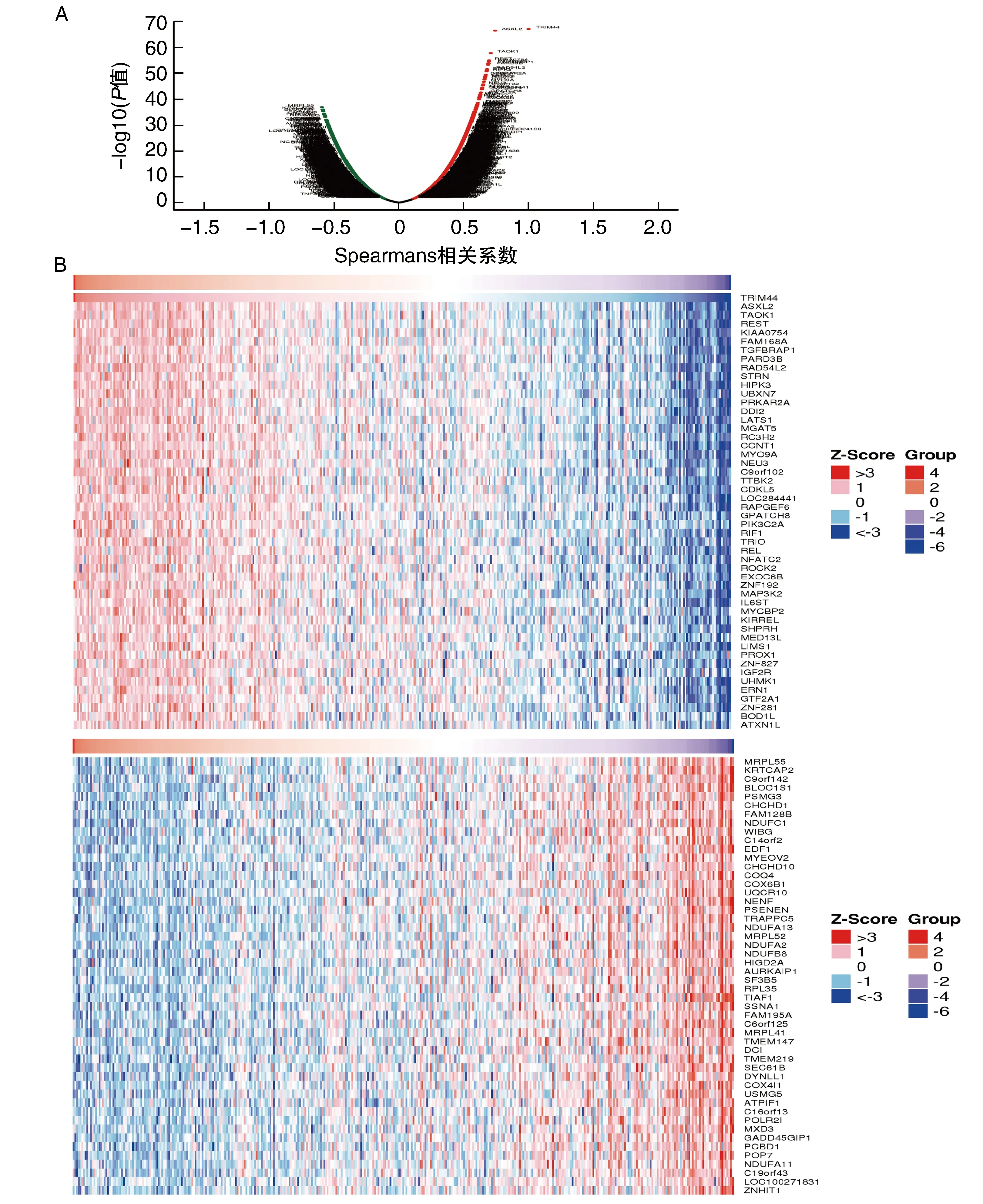
图3 TRIM44共表达基因的相关性分析A为TRIM44共表达基因差异分析;B为TRIM44共表达正相关和负相关基因。
选取与TRIM44正相关或负相关的前50个基因进行GO分析,结果如表1所示,TRIM44相关基因主要富集于以DNA为模板的转录调控、RNA聚合酶Ⅱ启动子的转录、蛋白磷酸化、丝氨酸/苏氨酸的活性等通路。KEGG通路富集分析显示,TRIM44可能参与调节干细胞多能性信号通路并与病毒的致癌作用相关。
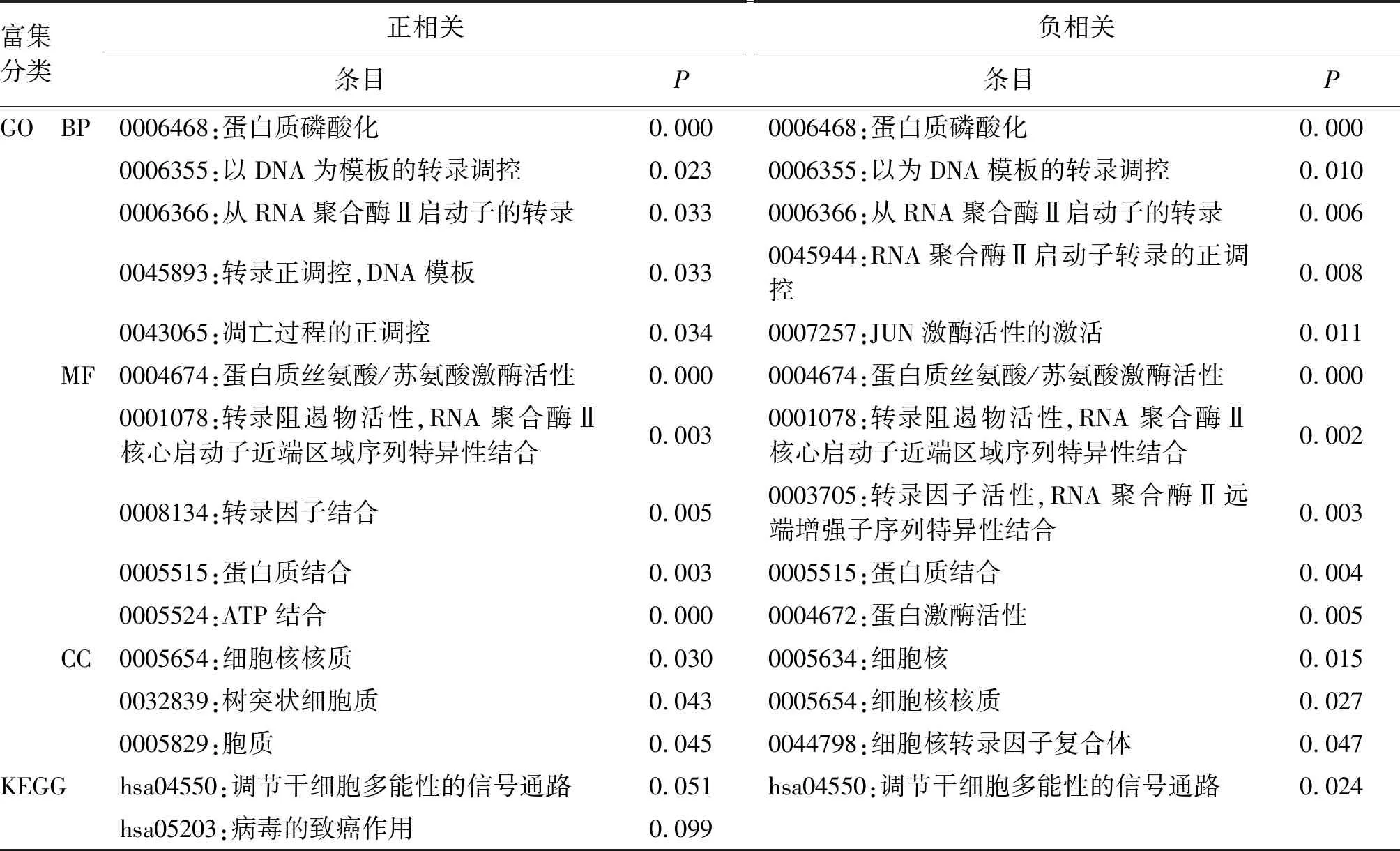
表1 GO功能和KEGG通路分析
2.4 免疫浸润分析
从TIMER数据库分析TRIM44表达是否与肿瘤免疫浸润水平相关。图4结果显示,TRIM44表达在COAD中与肿瘤纯度无显著相关性(P=0.489),与CD8+T细胞(P<0.001)、CD4+T细胞(P<0.001)、巨噬细胞(P<0.001)、中性粒细胞(P<0.001)和树突状细胞(P<0.001)的浸润水平显著相关,与B细胞(P<0.05)浸润水平相关。TRIM44表达在READ中与肿瘤纯度无显著相关性(P=0.306),与CD8+T细胞(P<0.001)、巨噬细胞(P<0.05)、中性粒细胞(P<0.001)和树突状细胞(P<0.001)的浸润水平显著相关,与B细胞(P<0.05)浸润水平相关。

图4 基于TIMER数据库的免疫浸润分析结果
GSE113513芯片样本中免疫细胞比例显示出单核细胞、M0细胞浸润较多(图5A),结直肠癌及正常组织样本中免疫细胞分布多集中在肥大细胞、M0细胞和M2细胞(图5B)。结直肠癌组织样本免疫细胞矩阵显示M0与M2细胞之间相关性较高(图5C)。
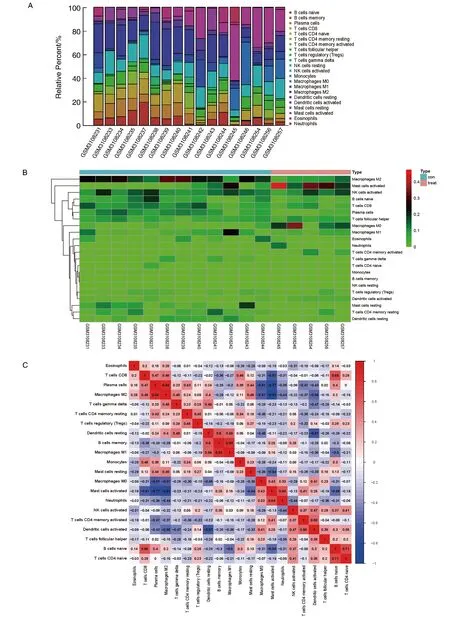
图5 GSE113513芯片样本中免疫组化分析结果A为GSE113513芯片样本中免疫细胞比例;B为结直肠癌及正常样本中免疫细胞分布;C为结直肠癌组织样本免疫细胞相关性。
小提琴图显示M0细胞(P<0.05)与M2细胞(P<0.05)在CRC组织中差异有显著性(图6A)。GEPIA数据库中TRIM44与M0相关基因CCL2(P<0.001)、CD68(P<0.001)、IL-10(P<0.001)以及M2相关基因CD163(P<0.001)、VSIG4(P<0.001)、MS4A4A(P<0.001)显著相关(图6B)。
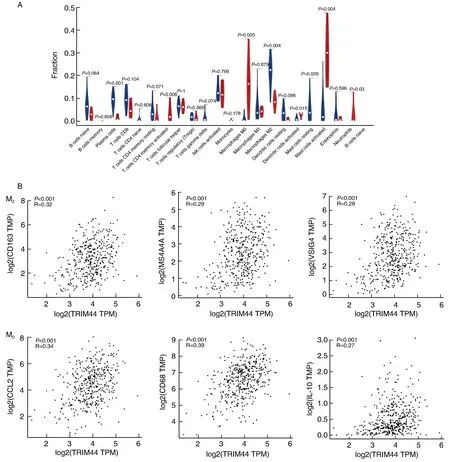
图6 GSE113513芯片样本中免疫组化分析结果A为结直肠癌及正常组织中免疫细胞差异分析,其中蓝色为正常组织,红色为结直肠癌;B为TRIM44与M0相关基因(CCL2、CD68、IL-10)以及M2相关基因(CD163、VSIG4、MS4A4A)的相关性分析。
3 讨 论
随着生活水平的改善,居民饮食比例出现了很大改变,动物蛋白及脂肪摄入的增加使得结直肠癌的发病率和死亡率逐渐上升。2020年,CRC死亡人数估计约5.3万,其中50岁以下约3 640人,占7%[13]。近年来,新确诊患者年龄年轻化,超过50%患者可归因于可改变风险因素,通过筛查和监测来进一步预防,并且在年轻患者中进行更加及时的诊断,尤为重要。目前,筛查CRC最有效的手段包括高灵敏度粪便检测或电子结直肠镜检查,但是目前受限于检测条件与患者依从性,筛查效率与可行性不高,迫切需要寻找新的筛查与判断预后的基因靶点。目前研究表明TRIM44在多肿瘤中有较高表达,主要通过Akt/mTOR信号通路发挥作用[8]。REN等[14]研究表明ELFN1-AS1通过调节miR-4644/TRIM44轴促进CRC的增殖、迁移,可作为CRC的预后指标。Sun等[15]认为LINC00265通过调节miR-216b-5p/TRIM44轴促进大肠癌的糖酵解和乳酸生成。TRIM44在CRC中相关机制渐呈体系,并已有学者利用靶向TRIM44来抑制上皮-间质转化减弱胶质母细胞瘤的增殖和转移[16]。因此通过多数据库宏观查看TRIM44在CRC中的表达可找到相关机制共性,也可进一步证实相关结果的准确性。
不同于既往体外培养和流式细胞术等技术分析肿瘤的模式,多数据库的挖掘分析利于规避小样本量与地理环境等差异导致的不可控变量的影响,可作为临床实验的前瞻研究与重要补充。本研究首先分析TCGA数据库中CRC,探究其癌-常差异表达,并利用GEPIA、Oncomine数据查看TRIM44在CRC中的表达情况,发现TRIM44在CRC中高表达。TCGA数据分析中,READ由于入选TCGA数据中正常组织样本较少,计算统计检验量时偏倚较大,着重关注COAD与READ混合样本产生的结果利于弥补样本量少的问题。Oncolnc结果显示,CRC患者中TRIM44表达与预后相关。通过挖掘LinkedOmics数据库得到CRC中19 828个基因与TRIM44的Spearman相关系数,对正相关前50个基因与负相关前50个基因进行了GO与KEGG通路富集分析,发现TRIM44相关基因主要富集于以DNA为模板的转录调控、RNA聚合酶Ⅱ启动子的转录、蛋白磷酸化、丝氨酸/苏氨酸的活性等通路,显示出TRIM44可能参与调节干细胞多能性的信号通路,并与病毒的致癌作用密切相关。TIMER数据库分析结果显示,TRIM44在CRC中与巨噬细胞显著相关。GEO芯片结果显示M0和M2细胞可能参与CRC细胞的增殖,利用GEPIA探究M0和M2基因与TRIM44的相关性,发现TRIM44与巨噬细胞在CRC中显著相关。以往体外实验表明,肿瘤相关免疫细胞分泌的炎性因子如CCL2、IL-1α等和极化后的M2型巨噬细胞分泌的细胞因子,可促进CRC细胞增殖和肿瘤生长[17]。因为GO分析中TRIM44还在转录调控、蛋白质加工等方面发挥作用,推测TRIM44通过调控CRC细胞中某成分的表达或作用,募集巨噬细胞进入肿瘤免疫微环境激活CCL2基因进而促CRC肿瘤细胞增殖。
本研究通过生物信息学方法分析发现,TRIM44高表达于结直肠癌组织,可作用于M0和M2巨噬细胞,影响转录调控、蛋白磷酸化及丝氨酸/苏氨酸代谢活性,对结直肠癌的发病进行调控,且与患者预后相关,有望成为结直肠癌早期诊断与临床治疗的诊断标志物与治疗靶点,为接下来的实验研究提供理论基础,但本研究基于生物信息学分析,有关结论仍需通过大样本临床研究加以验证。

