黄花蒿中青蒿素生物合成相关转录因子研究进展
李 琦,高晓悦,陈万生,谭何新
黄花蒿中青蒿素生物合成相关转录因子研究进展
李 琦,高晓悦,陈万生,谭何新*
海军军医大学药学院,上海 200433
以青蒿素为基础的联合疗法是疟疾的首选治疗方案,而药用植物黄花蒿是青蒿素的唯一天然来源,也是目前最主要的青蒿素来源,因此培育高产青蒿素的黄花蒿一直是国际研究热点。青蒿素是黄花蒿的次生代谢产物,在植物的次生代谢物合成过程中,关键的转录因子可以调节代谢途径中某个或多个基因的表达,从而调节代谢流的方向和速度,决定着代谢物的产量,因此关键转录因子的表达对于青蒿素的合成非常重要,通过干预转录因子的表达也是提高青蒿素产量的重要手段。综述了黄花蒿中已研究的转录因子功能及调控机制,特别是对筛选获得转录因子基因的方法进行总结,以期为揭示青蒿素合成调控网络奠定基础。
黄花蒿;青蒿素;转录因子;代谢调控;次生代谢物
据世界卫生组织(World Health Organization,WHO)报道,2018年全球共有2.28亿疟疾病例报告,其中40.5万例死亡,2010~2018年疟疾的发病率及死亡率均呈下降趋势,特别是重灾区非洲的疟疾死亡率下降尤为明显,这很大程度上归功于以青蒿素为基础的联合疗法(artemisinin-based combination therapy,ACT)的应用,目前ACT仍为WHO推荐的疟疾首选治疗方案[1]。如何高效获得青蒿素一直是国际研究热点,化学合成方面,早在1983年瑞士科学家Schmid和Hofheinz就提出了青蒿素的化学全合成方案,但反应步骤多、条件苛刻、试剂昂贵、得率低,后续提出的化学合成方案均存在类似问题[2]。合成生物学方面,Keasling团队在青蒿酸生物合成方面的研究堪称合成生物学的典范,然而由于青蒿素的最终合成需要特殊的油性氧化环境,在酵母中难以实现,因此目前较为成熟的方法仍为生物合成青蒿酸[3]。Paddon等[4]提出以青蒿酸为原料的化学半合成方案,最终青蒿素的得率为40%~45%,且存在分离纯化困难的问题。目前最为经济的获得青蒿素的方法仍为从植物中提取,而青蒿素在植株中的含量较低,仅占干质量的0.1%~1%[5],随着青蒿素临床治疗应用的开发,其市场需求会进一步加大,因此需要提高青蒿素的产量。
青蒿素是来源于药用植物黄花蒿L.的次生代谢产物,其生物合成途径属于类异戊二烯途径(图1),由质体中的异戊二烯(meth-ylerythritol phosphate,MEP)途径提供1个异戊烯基焦磷酸(isopentenyl phosphate,IPP)和细胞质中的甲羟戊酸(mevalonic acid,MVA)途径提供1个二甲基烯丙基焦磷酸(dimethylallyl diphosphate,DMAPP)和IPP,然后1份DMAPP和2份IPP在法尼基焦磷酸合成酶(farnesyl diphosphate synthase,FPS)的催化下聚合生成法尼基焦磷酸(farnesyl diphosphate,FPP)[6-7]。从FPP开始进入青蒿素合成特异的下游代谢途径,FPP经紫穗槐二烯合成酶(amorpha-4,11-diene synthase,ADS)催化生成紫穗槐二烯[8];紫穗槐二烯经过3步由细胞色素P450单氧化酶(Cytochrome P450 monooxygenase,CYP71AV1)催化的反应,分别形成青蒿醇,青蒿醛和青蒿酸[9-11]。青蒿醛可在青蒿醛双键还原酶[artemisinic aldehyde delta-11(13)reductase,DBR2][12]和醛脱氢酶1(aldehyde dehydrogenase 1,ALDH1)[13]的催化下先转化成二氢青蒿醛(dihydroartemisinic aldehyde),然后生成青蒿素的直接前体二氢青蒿酸(dihydroartemisinic acid,DHAA)。同时,青蒿醛也会在CYP71AV1 和 ALDH1的催化下生成青蒿酸(artemisinic acid)[11, 13];从DHAA到青蒿素(artemisinin),以及青蒿酸到青蒿素B(arteannuin B)的转化目前认为是非酶促的光氧化反应[14-17]。在植物的次生代谢过程中,转录因子可以调节代谢途径中的一系列基因,对转录因子的干预是一种有效的调控植物次生代谢产物的方法[18]。本文按照获得转录因子基因的不同途径进行分类,综述了黄花蒿中转录因子的研究进展。
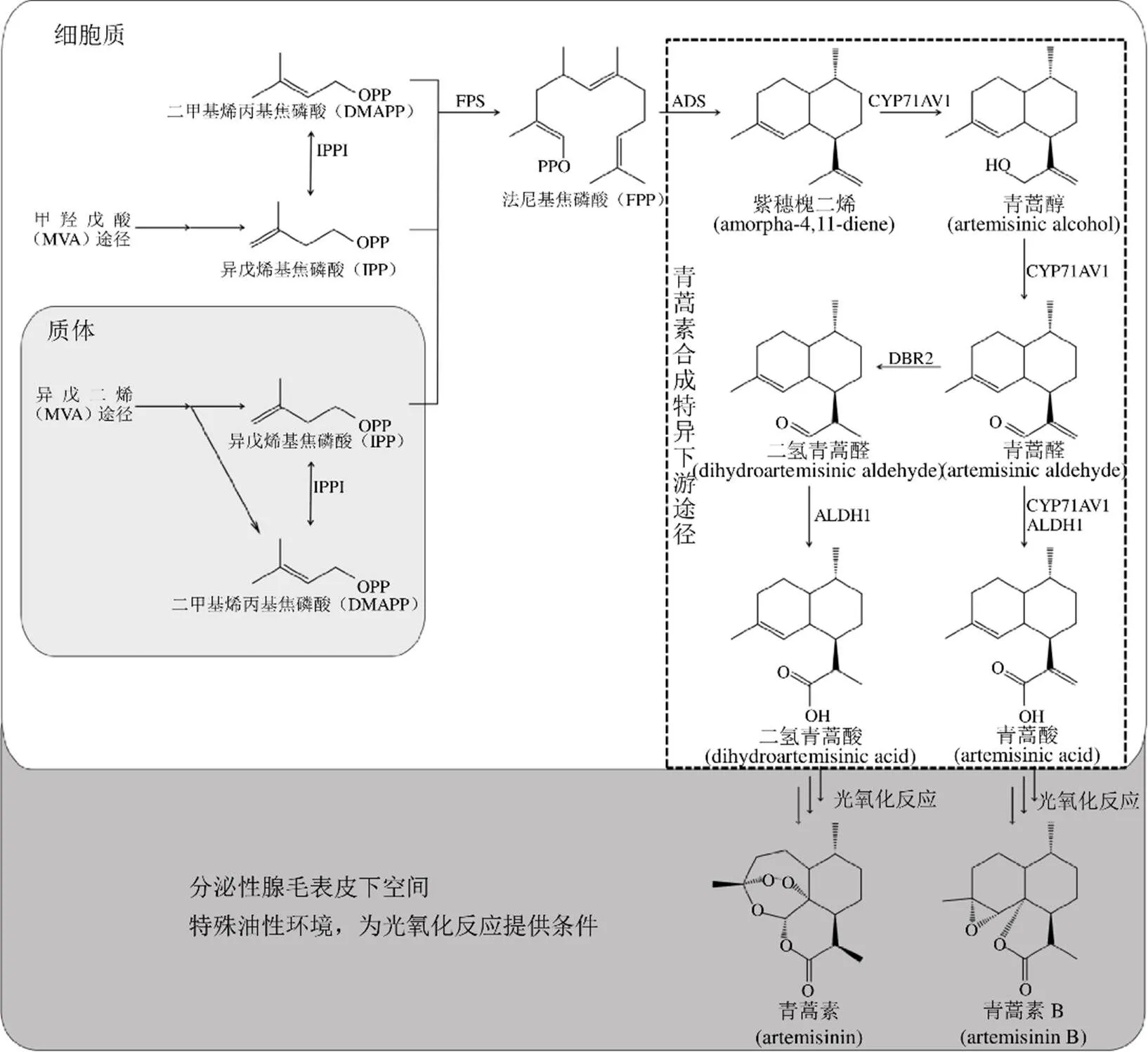
图1 青蒿素生物合成途径
1 激素响应转录因子
1.1 脱落酸(abscisic acid,ABA)对黄花蒿bZip1基因(AabZip1)的影响
ABA的处理能提高青蒿素积累[19],在模式植物拟南芥中A类bZip家族转录因子响应ABA信号[20]。Zhang等[21]通过bZip家族保守结构域查询黄花蒿公共cDNA数据库,得到145个候选基因,其中64个能在分泌性腺毛中表达,通过与拟南芥A类bZip转录因子基因构建系统进化树,筛选得到6个黄花蒿A类bZip转录因子基因,再通过在烟草中进行双荧光素酶实验验证能否激活及的表达,获得目的基因。后续实验表明受ABA、干旱、高盐胁迫的诱导表达,AabZip1通过激活和的表达影响青蒿素的合成,其过表达可使青蒿素和二氢青蒿酸含量分别提高40%~60%、41%~53%。
1.2 茉莉酸(jasmonicacid,JA)和茉莉酸甲酯(methyl jasmonate,MeJA)
1.2.1 黄花蒿基因() bHLH家族的MYC2类转录因子被认为是参与JA信号通路的核心因子[22-23]。Shen等[24]在黄花蒿叶片cDNA文库中发现5个MYC2类转录因子片段序列,其中1条与MeJA处理后的拟南芥中的MYC2基因的表达模式相同,用RACE方法获得其全长序列,命名为。后续研究表明过表达能提高和的转录水平,可使青蒿素和二氢青蒿酸含量分别提高23%~55%、17%~217%。
1.2.2 黄花蒿基因() JA能促进黄花蒿分泌性腺毛起始[25],JAZ蛋白是茉莉酸信号通路的重要调控因子,在拟南芥中能影响腺毛的起始[26],Yan等[27]用AaJAZ8筛选黄花蒿幼叶cDNA文库,发现1个HDZip家族转录因子AaHD1能与AaJAZ8相互作用,且受茉莉酸的正调控。后续研究表明过表达能在不影响植物生长的情况下增加成熟叶片表面分泌性腺毛密度,使青蒿素含量提高50%。
1.2.3 黄花蒿基因(、) AP2/ERF家族转录因子参与植物激素信号响应及次生代谢[28]。Yu等[29]通过在分泌性腺毛cDNA文库中查找AP2/ERF家族转录因子保守结构域,获得7个片段序列,其中2个符合在花中表达、且受MeJA诱导的表达模式,用RACE的方法获得这2条序列的全长,即和。后续研究表明AaERF1、AaERF2均能激活的表达,过表达可使青蒿素和青蒿酸的含量提高19%~67%、11%~76%,过表达为24%~51%、17%~121%,且基因可能通过JA和乙烯信号通路,激活部分防御基因,提高黄花蒿对灰霉病菌Pers.的抗性[30]。
1.3 水杨酸(Salicylicacid,SA)对黄花蒿TGA6基因(AaTGA6)
SA在植物对病原菌的防御响应中起重要作用[31],在拟南芥的SA信号通路中TGA2、NPR1是重要调节因子[32-33]。Lv等[34]分析黄花蒿转录组数据,发现有6条TGA类转录因子序列,通过与拟南芥TGA转录因子基因构建系统进化树,同源性最高的为。后续研究表明的表达能被AaNPR1增强,被AaTGA3抑制,同时AaTGA6能调控基因的表达,过表达可使青蒿素含量提高90%~120%。
2 通过分析启动激活元件获得转录因子
2.1 黄花蒿WRKY1基因(AaWRKY1)
Han等[35]分析基因的启动子发现其含有2个W-box,可能受WRKY家族转录因子调控,通过比对分泌性腺毛的cDNA文库,发现其中有1条转录因子的片段序列,用RACE方法获得全长序列,命名为。后续研究表明AaWRKY1能提高及其它青蒿素合成途径基因的表达,且在腺毛中特异的过表达比在植物中广泛的过表达能更有效的提高青蒿素的积累。
2.2 黄花蒿MYB1基因(AaMYB1)
Matías-Hernández等[36]发现多数青蒿素合成途径基因启动子区域均含有MYB家族转录因子结合位点,推测有MYB家族转录因子参与调控青蒿素生物合成。分析公共黄花蒿分泌性腺毛cDNA文库,发现1个符合R2R3-MYB转录因子特征的片段,用RACE的方法获得全长序列命名为。后续研究表明,的过表达可以提高青蒿素合成途径基因的表达水平,增加分泌性腺毛数量及密度,提高青蒿素的积累。
2.3 黄花蒿bHLH1基因(AabHLH1)
Ji等[37]发现基因启动子区域均含有bHLH家族转录因子结合的E-box,推测可能被bHLH家族转录因子调控,在黄花蒿分泌性腺毛cDNA数据库中分析发现3条可能的bHLH家族转录因子基因片段,用RACE方法能获得了其中2条基因的全长,酵母单杂交实验及EMSA实验证明AabHLH1能与E-box结合,因此选定为目的基因。后续研究表明,AabHLH1能结合的启动子并提高青蒿素合成途径基因的转录水平。
2.4 黄花蒿HD8基因(AaHD8)
Yan等[38]分析的启动子发现含有L1-box,预测可能被HD-Zip家族转录因子结合,利用酵母单杂交、双荧光素酶实验筛选已获得的HD-Zip IV亚家族转录因子基因,获得。后续研究表明AaHD8能激活的表达,从而起始分泌性腺毛的发育,另外AaHD8还能与AaMIXTA1相互作用,调节腺毛发育及角质层形成。
3 与已知功能基因同源的黄花蒿转录因子基因
3.1 拟南芥
3.1.1 黄花蒿基因() 拟南芥ORA59能在植物防御中整合JA和乙烯信号[39]。Lu等[40]通过拟南芥基因序列在黄花蒿分泌性腺毛cDNA文库中查询,得到1个同源性最高的片段,用RACE方法获得其全长序列,即。后续研究表明ABA处理、创伤、寒冷能显著提高的转录水平,MeJA、乙烯处理也能轻微的诱导表达,从而参与植物抗逆。
3.1.2 黄花蒿基因() MYB家族转录因子中MIXTA或MIXTA样转录因子能控制多种植物的细胞形态、腺毛发育和起始等,如拟南芥AtMYB16和AtMYB106调控腺毛分枝及表皮细胞形态[41]。Shi等[42]以拟南芥已知MYB家族转录因子序列在黄花蒿各组织的转录组数据库中查询,获得的序列与拟南芥及其他物种已知的MIXTA或MIXTA样转录因子共同构建系统进化树,筛选获得与拟南芥、同源性最高的序列,即。后续研究表明AaMIXTA1可以调控分泌性腺毛的数量和角质层的生物合成,过表达可以在不影响分泌性腺毛结构的情况下提高青蒿素的积累。
3.1.3 黄花蒿基因() 青蒿素的生物合成量在低温处理后显著升高[43-44],而拟南芥中2个低温相关的转录因子AtICE1和ATICE2均属于bHLH家族[45-46]。Xiang等[47]以bHLH保守结构域在青蒿基因组数据库中查询,得到基因组中205个结果、转录组中122个,按bHLH家族转录因子特征进行分类,其中属于与青蒿素合成相关的V家族转录因子15个,与拟南芥bHLH转录因子共同构建系统进化树,2条序列与拟南芥、同源性最高,其中仅受低温诱导。后续研究表明,AabHLH112能调控的表达,从而提高青蒿素合成途径基因的转录水平,过表达的株系中青蒿素及二氢青蒿酸含量均显著上升。
3.2 其他
3.2.1 黄花蒿基因() 长春花的过表达可以增加萜类化合物的积累[48],Lu等[49]以长春花序列作为查询序列在黄花蒿叶片cDNA文库中比对,获得一条片段序列,用RACE方法获得全长序列,即。后续研究表明在分泌型腺毛中特异性表达,对有正调控作用,过表达青蒿素及二氢青蒿酸含量分别上升40%~53%和22%~35%,同时能提高黄花蒿对灰霉病菌的抗性。
3.2.2 黄花蒿基因() 薄荷与萜类合成相关[50],Kayani等[51]以YABBY家族转录因子保守结构域在黄花蒿基因组数据库中查询,将获得的序列与构建系统进化树,同源性最高的序列即。后续研究表明能受MeJA的诱导,直接结合、的启动子,提高青蒿素合成途径基因当的转录水平,过表达可以使青蒿素及二氢青蒿酸的含量升高。
4 转录组、基因组数据分析
4.1 黄花蒿TAR1基因(AaTAR1)
Graham等[52]对黄花蒿的遗传图谱进行分析的文章中,预测了7个可能与腺毛发育相关的基因,Tan等[53]通过分析黄花蒿转录组数据及同源比对,筛选出其中的1个AP2/ERF家族转录因子,命名为AaTAR1。后续研究表明AaTAR1可以控制蜡质、角质物质的合成,能直接影响分泌型腺毛的形态发育,并通过结合青蒿素合成路径上的基因启动子,来控制青蒿素的合成。过表达能提高青蒿素合成途径基因的转录水平,并提高黄花蒿叶片和花蕾中青蒿素、青蒿酸及二氢青蒿酸的含量。
4.2 黄花蒿SPL2基因(AaSPL2)
在模式植物拟南芥中,有研究表明SPL转录因子参与调控植物的次生代谢[54-55]。Lv等[56]以SPL家族转录因子保守结构域在黄花蒿转录组数据库中查询,得到14个结果,其中含有MicroRNA156识别位点的9条;用JA处理黄花蒿,检测9条SPL转录因子基因的表达水平发现和响应了JA信号,其中的表达模式与青蒿素合成途径基因的表达模式更为接近,因此选择作为目的基因。后续研究表明,基因可激活的转录,从而介导JA对青蒿素合成的调控,过表达能提高青蒿素合成途径基因的转录水平,使青蒿素和二氢青蒿酸的含量提高33%~86%和26%~159%。
4.3 黄花蒿GSW1基因(AaGSW1)
多条WRKY家族转录因子已被证实参与调控青蒿素的生物合成,但它们均非腺毛特异表达转录因子,Chen等[57]以WRKY保守结构域序列在青蒿各组织转录组数据库中比对,共获得122条序列,其中42条能在腺毛中表达,通过与腺毛特异表达基因,如、等,在各组织中的表达模式进行比较,获得表达模式最为接近的1条,命名为。后续研究表明AaGSW1是腺毛特异表达转录因子,受JA信号途径中的AaMYC2和ABA信号途径的AabZip1调控,过表达能提高及的转录水平,使青蒿素含量提高39%~43%。
4.4 黄花蒿NAC1基因(AaNAC1)
NAC家族转录因子在提高植物对灰霉病菌抗性,及调控萜类物质合成方面发挥作用[58-59]。Lv等[60]分析分泌性腺毛转录组数据库,选出RPKM(Reads Per Kilobase per Million mapped reads)值最高的10条NAC家族转录因子基因序列,其中能受SA和MeJA的诱导,作为目的基因进行研究。后续研究发现能受脱水、低温、SA、MeJA的诱导,过表达能使青蒿素和二氢青蒿酸的含量升高,提高黄花蒿对干旱、灰霉病菌的抗性。
5 展望
青蒿素是中医药对世界的重大贡献,随着新适应症的开发,青蒿素的需求量会进一步增大,解析青蒿素的合成路径及调控网络,并利用分子育种的手段获得高青蒿素含量的黄花蒿意义重大。青蒿素的产量与分泌性腺毛的发育情况密切相关,同时黄花蒿也被认为是研究分泌性腺毛发育的潜在模式植物,而转录因子无论在青蒿素的生物合成途径中,还是在腺毛的发育过程中均具有关键的调控作用,因此转录因子是研究的重要靶标。目前对黄花蒿转录因子的研究虽然已经涉及到AP2/ERF、WRKY、MYB、bZip、bHLH等多个家族,但植物转录因子数量庞大,调控网络复杂,当前对黄花蒿转录因子调控作用的认识仍相对有限。随着研究技术手段的发展,特别是转录组、基因组等组学技术及单细胞测序等最新技术开始应用在黄花蒿的研究中,将更有利于发掘重要的功能基因,完善对青蒿素生物合成、分泌性腺毛发育等重要科学问题的认识。
利益冲突 所有作者均声明不存在利益冲突
[1] World Health Organization.2019 [M]. Geneva: WHO, 2019.
[2] Schmid G, Hofheinz W. Total synthesis of qinghaosu [J]., 1983, 105: 624-625.
[3] Ro D K, Paradise E M, Ouellet M,. Production of the antimalarial drug precursor artemisinic acid in engineered yeast [J]., 2006, 440(7086): 940-943.
[4] Paddon C J, Westfall P J, Pitera D J,. High-level semi-synthetic production of the potent antimalarial artemisinin [J]., 2013, 496(7446): 528-532.
[5] Mutabingwa T K. Artemisinin-based combination therapies (ACTs): Best hope for malaria treatment but inaccessible to the needy! [J]., 2005, 95(3): 305-315.
[6] Schramek N, Wang H, Römisch-Margl W,. Artemisinin biosynthesis in growing plants of. A13CO2study [J]., 2010, 71(2/3): 179-187.
[7] Ma D M, Li G, Alejos-Gonzalez F,. Overexpression of a type-I isopentenyl pyrophosphate isomerase ofin the cytosol leads to high arteannuin B production and artemisinin increase [J]., 2017, 91(3): 466-479.
[8] Wallaart T E, Bouwmeester H J, Hille J,.-4,11-diene synthase: Cloning and functional expression of a key enzyme in the biosynthetic pathway of the novel antimalarial drug artemisinin [J]., 2001, 212(3): 460-465.
[9] Wang H Z, Han J L, Kanagarajan S,. Trichome-specific expression of the-4,11-diene 12-hydroxylase (cyp71av1) gene, encoding a key enzyme of artemisinin biosynthesis in, as reported by a promoter-GUS fusion [J]., 2013, 81(1/2): 119-138.
[10] Wang Y, Yang K, Jing F,. Cloning and characterization of trichome-specific promoter of cpr71av1 gene involved in artemisinin biosynthesis inL. [J]., 2011, 45(5): 817-824.
[11] Teoh K H, Polichuk D R, Reed D W,.L. (Asteraceae) trichome-specific cDNAs reveal CYP71AV1, a cytochrome P450 with a key role in the biosynthesis of the antimalarial sesquiterpene lactone artemisinin [J]., 2006, 580(5): 1411-1416.
[12] Zhang Y S, Teoh K H, Reed D W,. The molecular cloning of artemisinic aldehyde Delta11(13) reductase and its role in glandular trichome-dependent biosynthesis of artemisinin in[J]., 2008, 283(31): 21501-21508.
[13] Teoh K, Polichuk D, Reed D,. Molecular cloning of an aldehyde dehydrogenase implicated in artemisinin biosynthesis in[J]., 2009, 87: 635-642.
[14] Lommen W J, Elzinga S, Verstappen F W,. Artemisinin and sesquiterpene precursors in dead and green leaves ofL. crops [J]., 2007, 73(10): 1133-1139.
[15] Lommen W J, Schenk E, Bouwmeester H J,. Trichome dynamics and artemisinin accumulation during development and senescence ofleaves [J]., 2006, 72(4): 336-345.
[16] Brown G D, Sy L K.transformations of dihydroartemisinic acid inplants [J]., 2004, 60(5): 1139-1159.
[17] Brown G D, Sy L K.transformations of artemisinic acid inplants [J]., 2007, 63(38): 9548-9566.
[18] Verpoorte R, Memelink J. Engineering secondary metabolite production in plants [J]., 2002, 13(2): 181-187.
[19] Jing F Y, Zhang L, Li M Y,. Abscisic acid (ABA) treatment increases artemisinin content inby enhancing the expression of genes in artemisinin biosynthetic pathway [J]., 2009, 64(2): 319-323.
[20] Jakoby M, Weisshaar B, Dröge-Laser W,. bZIP transcription factors in[J]., 2002, 7(3): 106-111.
[21] Zhang F, Fu X, Lv Z,. A basic leucine zipper transcription factor, AabZIP1, connects abscisic acid signaling with artemisinin biosynthesis in[J]., 2015, 8(1): 163-175.
[22] Dombrecht B, Xue G P, Sprague S J,. MYC2differentially modulates diverse jasmonate-dependent functions in[J]., 2007, 19(7): 2225-2245.
[23] Zhang H B, Bokowiec M T, Rushton P J,. Tobacco transcription factors NtMYC2a and NtMYC2b form nuclear complexes with the NtJAZ1 repressor and regulate multiple jasmonate-inducible steps in nicotine biosynthesis [J]., 2012, 5(1): 73-84.
[24] Shen Q, Lu X, Yan T X,. The jasmonate-responsive AaMYC2transcription factor positively regulates artemisinin biosynthesis in[J]., 2016, 210(4): 1269-1281.
[25] Maes L, van Nieuwerburgh F C, Zhang Y S,. Dissection of the phytohormonal regulation of trichome formation and biosynthesis of the antimalarial compound artemisinin inplants [J]., 2011, 189(1): 176-189.
[26] Qi T C, Song S S, Ren Q C,. The Jasmonate- ZIM-domain proteins interact with the WD-Repeat/ bHLH/MYB complexes to regulate Jasmonate-mediated anthocyanin accumulation and trichome initiation in[J]., 2011, 23(5): 1795-1814.
[27] Yan T X, Chen M H, Shen Q,. HOMEODOMAIN PROTEIN 1 is required for jasmonate-mediated glandular trichome initiation in[J]., 2017, 213(3): 1145-1155.
[28] Brown R L, Kazan K, McGrath K C,. A role for the GCC-box in jasmonate-mediated activation of the PDF1.2 gene of[J]., 2003, 132(2): 1020-1032.
[29] Yu Z X, Li J X, Yang C Q,. The jasmonate- responsive AP2/ERF transcription factors AaERF1and AaERF2positively regulate artemisinin biosynthesis inL [J]., 2012, 5(2): 353-365.
[30] Lu X, Jiang W M, Zhang L,. AaERF1positively regulates the resistance toin[J]., 2013, 8(2): e57657.
[31] Raskin I, Skubatz H, Tang W,. Salicylic acid levels in thermogenic and non-thermogenic plants [J]., 1990, 66(4): 369-373.
[32] Fan W H, Dong X N.interaction between NPR1 and transcription factor TGA2 leads to salicylic acid-mediated gene activation in[J]., 2002, 14(6): 1377-1389.
[33] Rochon A, Boyle P, Wignes T,. The coactivator function ofNPR1 requires the core of its BTB/POZ domain and the oxidation of C-terminal cysteines [J]., 2006, 18(12): 3670-3685.
[34] Lv Z, Guo Z, Zhang L,. Interaction of bZIP transcription factor TGA6 with salicylic acid signaling modulates artemisinin biosynthesis in[J]., 2019, 70(15): 3969-3979.
[35] Han J L, Wang H Z, Lundgren A,. Effects of overexpression of AaWRKY1on artemisinin biosynthesis in transgenicplants [J]., 2014, 102: 89-96.
[36] Matías-Hernández L, Jiang W M, Yang K,. AaMYB1 and its orthologue AtMYB61 affect terpene metabolism and trichome development inand[J]., 2017, 90(3): 520-534.
[37] Ji Y P, Xiao J W, Shen Y L,. Cloning and characterization of AabHLH1, a bHLH transcription factor that positively regulates artemisinin biosynthesis in[J]., 2014, 55(9): 1592-1604.
[38] Yan T, Li L, Xie L,. A novel HD-ZIP IV/MIXTA complex promotes glandular trichome initiation and cuticle development in[J]., 2018, 218(2): 567-578.
[39] Pré M, Atallah M, Champion A,. The AP2/ERF domain transcription factor ORA59 integrates jasmonic acid and ethylene signals in plant defense [J]., 2008, 147(3): 1347-1357.
[40] Lu X, Jiang W, Zhang L,. Characterization of a novel ERF transcription factor inand its induction kinetics after hormones and stress treatments [J]., 2012, 39(10): 9521-9527.
[41] Oshima Y, Shikata M, Koyama T,. MIXTA-like transcription factors and WAX INDUCER1/SHINE1 coordinately regulate cuticle development inand[J]., 2013, 25(5): 1609-1624.
[42] Shi P, Fu X Q, Shen Q,. The roles of AaMIXTA1 in regulating the initiation of glandular trichomes and cuticle biosynthesis in[J]., 2018, 217(1): 261-276.
[43] Liu W, Wang H, Chen Y,. Cold stress improves the production of artemisinin depending on the increase in endogenous jasmonate [J]., 2017, 64(3): 305-314.
[44] Wallaart T E, Pras N, Beekman A C,. Seasonal variation of artemisinin and its biosynthetic precursors in plants ofof different geographical origin: Proof for the existence of chemotypes [J]., 2000, 66(1): 57-62.
[45] Chinnusamy V, Ohta M, Kanrar S,. ICE1: A regulator of cold-induced transcriptome and freezing tolerance in[J]., 2003, 17(8): 1043-1054.
[46] Fursova O V, Pogorelko G V, Tarasov V A. Identification of ICE2, a gene involved in cold acclimation which determines freezing tolerance in[J]., 2009, 429(1/2): 98-103.
[47] Xiang L E, Jian D Q, Zhang F Y,. The cold-induced transcription factor bHLH112 promotes artemisinin biosynthesis indirectly via ERF1in[J]., 2019, 70(18): 4835-4848.
[48] van der Fits L, Memelink J. ORCA3, a jasmonate-responsive transcriptional regulator of plant primary and secondary metabolism [J]., 2000, 289(5477): 295-297.
[49] Lu X, Zhang L, Zhang F Y,. AaORA, a trichome-specific AP2/ERF transcription factor of, is a positive regulator in the artemisinin biosynthetic pathway and in disease resistance to[J]., 2013, 198(4): 1191-1202.
[50] Wang Q, Reddy V A, Panicker D,. Metabolic engineering of terpene biosynthesis in plants using a trichome-specific transcription factor MsYABBY5from spearmint () [J]., 2016, 14(7): 1619-1632.
[51] Kayani S I, Shen Q, Ma Y N,. The YABBY family transcription factor AaYABBY5directly targets cytochrome P450 monooxygenase (CYP71AV1) and double-bond reductase 2 (DBR2) involved in artemisinin biosynthesis in[J]., 2019, 10: 1084.
[52] Graham I A, Besser K, Blumer S,. The genetic map ofL. identifies loci affecting yield of the antimalarial drug artemisinin [J]., 2010, 327(5963): 328-331.
[53] Tan H X, Xiao L, Gao S H,. TRICHOME AND ARTEMISININ REGULATOR 1 is required for trichome development and artemisinin biosynthesis in[J]., 2015, 8(9): 1396-1411.
[54] Gou J Y, Felippes F F, Liu C J,. Negative regulation of anthocyanin biosynthesis inby a miR156-targeted SPL transcription factor [J]., 2011, 23(4): 1512-1522.
[55] Yu Z X, Wang L J, Zhao B,. Progressive regulation of sesquiterpene biosynthesis in Arabidopsis and Patchouli () by the miR156-targeted SPL transcription factors [J]., 2015, 8(1): 98-110.
[56] Lv Z, Wang Y, Liu Y,. The SPB-box transcription factorpositively regulates artemisinin biosynthesis inL. [J]., 2019, 10: 409.
[57] Chen M H, Yan T X, Shen Q,. GLANDULAR TRICHOME-SPECIFIC WRKY 1 promotes artemisinin biosynthesis in[J]., 2017, 214(1): 304-316.
[58] Bu Q, Jiang H, Li C B,. Role of theNAC transcription factors ANAC019 and ANAC055 in regulating jasmonic acid-signaled defense responses [J]., 2008, 18(7): 756-767.
[59] Nieuwenhuizen N J, Chen X, Wang M Y,. Natural variation in monoterpene synthesis in kiwifruit: Transcriptional regulation of terpene synthases by NAC and ETHYLENE-INSENSITIVE3-like transcription factors [J]., 2015, 167(4): 1243-1258.
[60] Lv Z, Wang S, Zhang F Y,. Overexpression of a novel NAC domain-containing transcription factor gene (AaNAC1) enhances the content of artemisinin and increases tolerance to drought andin[J]., 2016, 57(9): 1961-1971.
Research on transcription factors related to artemisinin biosynthesis in
LI Qi, GAO Xiao-yue, ZHANG Lei, CHEN Wan-sheng, TAN He-xin
School of Pharmacy, Navy Medical University, Shanghai 200433, China
Artemisinin-based combination therapy is the preferred treatment for malaria. The medicinal plantis the only natural source and the main source of artemisinin. It is a hotspot to cultivate a strain ofwith high artemisinin content. Artemisinin is a secondary metabolite of. In the process of plant secondary metabolism, transcription factors play important roles in regulating a series of genes in the metabolic pathway, thereby regulating the direction and speed of metabolic flow. Therefore, the intervention of transcription factor by genetic engineering is an important method to regulate plant secondary metabolism. This article summarizes the functions and regulatory mechanism of transcription factors had been studied in. In particular, the methods for screening these transcription factors genes were summarized in order to provide a reference for the finding of key functional genes.
L.; artemisinin; transcription factor; metabolic regulation; secondary metabolite
R282.12
A
0253 - 2670(2021)06 - 1827 - 08
10.7501/j.issn.0253-2670.2021.06.032
2020-06-03
国家自然科学基金资助项目(81673529);科技部新药创制重大专项(2017ZX09101002-003-002)
李 琦(1992—),男,硕士研究生,研究方向为中药材品质改良。E-mail: liqi0121@163.com
谭何新,女,副教授,博士,硕士生导师,主要从事中药资源研究。Tel/Fax: (021)81871370 E-mail: hexintan@163.com
[责任编辑 时圣明]
——青蒿素

