侵染昆明园林植物的4 个HCRV 分离株核壳体蛋白氨基酸序列分析*
王宇琪 ,吴保为,陶宏征,贾志强,高 雪,王景文,刘雅婷
(1.云南农业大学 农学与生物技术学院,云南 昆明 650201;2.云南农业大学 植物保护学院,云南 昆明 650201)
朱顶红褪绿环斑病毒(Hippeastrum chlorotic ringspot virus,HCRV) 是泛布尼亚病毒科(Peribunyaviridae)番茄斑萎病毒属(Tospovirus)病毒,2013 年在中国云南被首次发现并报道[1],本课题组首次报道了朱顶红褪绿环斑病毒的全基因组序列[2-3]。朱顶红褪绿环斑病毒能系统侵染一点红、番茄、莴苣、旱金莲和豇豆等经济作物,主要症状为同心环纹、叶片畸形、局部褪绿和坏死[4],对农业生产造成了严重的威胁。HCRV 为三分子基因组,分别是L RNA、M RNA 和S RNA。L RNA 仅有1 个开放阅读框,正向编码RNA 依赖的RNA 聚合酶 (RNA-dependent RNA polymerase,RdRp);M RNA 正向编码非结构蛋白(non-structural protein of M RNA,NSm),互补链编码糖蛋白前体(glycoproteins,Gn/Gc);S RNA正向编码非结构蛋白NSs (non-structural protein of S RNA,NSs),互补链编码核壳体蛋白 (nucleocapsid protein,N)[5],N 蛋白序列保守程度较高[6],因此通常被用于进行Tospovirus属病毒的分类检测和鉴定[7],N 蛋白也可能与病毒粒子装配有关[8]。本研究通过RT-PCR 鉴定出葱莲、文殊兰、喜林芋和酢浆草感染HCRV 的4 份样品。为研究核壳体蛋白序列特征,通过RT-PCR、克隆和测序得到样品完整的HCRV S RNA 序列,利用ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder/)获得其N 蛋白序列并进行分析,为HCRV 的后续研究提供基础。
1 材料与方法
1.1 材料采集
2016 年在云南省昆明市主要园林景区进行了调查,采集到八角金盘(Fatsia japonica)、葱莲(Zephyranthes candida)、酢浆草(Oxalis corniculata)、旱金莲(Tropaeolum majus)、蝴蝶兰(Phalaenopsis aphrodite)、文殊兰(Crinumasiaticum)、喜林芋(Philodendronschott) 和鸢尾(Iris tectorum)等疑似感染番茄斑萎病毒属病毒样品(图1),主要表现为褪绿、坏死斑和皱缩等症状,将采集回来的样品用RT-PCR 进行鉴定。
1.2 总RNA 提取
采用去除多糖多酚的TransZol Plant (全式金生物技术有限公司,北京)试剂盒提取RNA,以RNA 为模板,采用PrimeScriptTMⅡ1st strand cDNA Synthesis Kit (宝生物技术有限公司,大连) 进行反转录合成cDNA。
1.3 RT-PCR 扩增、克隆及测序
利用Primer Premier 6.0 软件,以GenBank中公布的HCRV S RNA 序列(序列号:JX-833564.1)设计引物,S RNA 扩增引物为:F:5′-AGAGCAATCGAGGTATAAACAAATAATCATACAC-3′;R:5′-AGAGCAATCGAGGTATAAAACATAAATTCTGAAC-3′。50 μL PCR 反应体系:cDNA 1 μL,上下游引物各1 μL,FastPfu 1 μL,Buffer 10 μL,dNTP 4 μL,ddH2O 32 μL。PCR 反应条件:95 ℃ 1 min;95 ℃ 20 s,50 ℃20 s,72 ℃ 1.5 min,40 个循环;72 ℃ 5 min;4 ℃保存。PCR 反应产物用于1.2%的琼脂糖凝胶电泳检测。将获得的PCR 产物进行纯化以及加A反应。将已纯化的PCR 产物连接在pEASY-T1载体上,将连接产物导入T1 感受态细胞中,经过菌液PCR 鉴定出阳性重组子并送到公司进行测序。
1.4 生物信息学分析
通过ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder/)获得4 个HCRV 分离物的N 蛋白序列,利用DNAMAN 8.0 与分离自云南省蜘蛛兰上的HLS1-2 N 蛋白(登录号:AFX74694)进行序列比对分析;利用MEGA 6.06 软件对NCBI 数据库中已公布的分离自云南、广东、广西和福建地区蜘蛛兰上的HCRV 的N 蛋白以及本研究获得的4 个寄主的N 蛋白进行系统进化分析。

图1 疑似感染番茄斑萎病毒属病毒样品Fig.1 Samples suspected infection with Tospovirus virus
2 结果与分析
2.1 RT-PCR 结果
通过RT-PCR 技术鉴定出葱莲、文殊兰、喜林芋和酢浆草4 份感染HCRV 的样品并扩增出HCRV S RNA 序列,电泳检测条带大小约为2 700 bp (图2)。

图2 HCRV S RNA 扩增结果Fig.2 Amplification of HCRV S RNA
2.2 不同株系HCRV N 蛋白序列比对分析
本研究获得的葱莲YN-ZCH、文殊兰YNCA、喜林芋YN-PS 和酢浆草YN-OC 的N 蛋白,通过DNAMAN 8.0 与首个公布的HLS1-2 N蛋白进行序列比对,一致性达到99.34%。YNZCH 存在6 个突变位点,YN-CA 存在5 个突变位点,YN-PS 存在4 个突变位点,YN-OC 存在6 个突变位点(表1)。
2.3 不同株系HCRV N 蛋白系统进化分析
利用MEGA 6.06 软件对NCBI 数据库中已公布的HCRV 的N 蛋白以及本研究获得的4 个寄主的N 蛋白构建系统发育树,建树方法选择邻接法(neighbor-joining,NJ)。结果显示:本研究获得的4 个HCRV 分离物的N 蛋白与分离自云南省的HCRV N 蛋白聚为1 支,来自广东、广西和福建地区的聚为1 支(图3)。

表1 氨基酸突变位点Tab.1 Amino acid mutation sites
3 讨论
云南省丰富的植物资源和复杂的气候以及丰富的传播介体种类为Tospovirus病毒的发生和流行提供了条件,该属病毒在云南的发生呈逐年上升趋势[9]。自2013 年首次在云南省发现HCRV至今,国内外对其研究较少,目前已知寄主包括蜘蛛兰、烟草、番茄、君子兰[10]和葱莲[11]等,仍有许多寄主尚未被发现。本研究鉴定出葱莲、文殊兰、喜林芋和酢浆草4 个感染HCRV 样品,其中文殊兰、喜林芋和酢浆草是首次报道的HCRV寄主。研究结果表明:4 个HCRV 分离物的N 蛋白与NCBI 首次公布的HLS1-2 N 蛋白有较高的同源性,从系统发育分析可知不同株系的分离物聚类现象与寄主无明显关联,推测可能环境因子对病毒进化的影响强于寄主的影响。
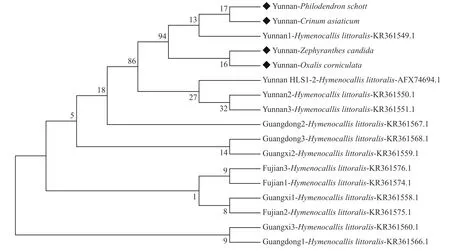
图3 HCRV N 蛋白氨基酸序列系统进化树Fig.3 Phylogenetic tree of HCRV N protein amino acid sequence
4 结论
本研究通过采集疑似感染番茄斑萎病毒属病毒的样本,应用症状学和分子生物学方法鉴定出了葱莲、文殊兰、喜林芋和酢浆草 4 个感染HCRV 样品,对 4 个寄主植物 S RNA 序列中 N蛋白进行序列比对和系统进化分析,结果表明4 个HCRV 分离物 N 蛋白与 NCBI 公布的 HLS1-2 N 蛋白有较高的同源性,HCRV 病毒的地理分布相关性强于寄主相关性。研究结果为 HCRV 的进化研究提供基础,也为进一步探究番茄斑萎病毒属病毒的扩散和进化策略提供参考。

