eDNA技术在极地水环境中的应用进展
刘梦月 赵文玉
研究进展
eDNA技术在极地水环境中的应用进展
刘梦月1,2赵文玉1,2
(1湖南省水生资源食品加工工程技术研究中心, 长沙理工大学化学与食品工程学院, 湖南 长沙 410004;2洞庭湖水环境治理与生态修复湖南省重点实验室, 长沙理工大学水利工程学院, 湖南 长沙 410004)
应用环境DNA(eDNA)技术高效准确地完成极地水生态系统(极区大洋, 近岸海域及湖泊、溪流等)中生物的物种鉴别、多样性分析等研究工作, 对开发极地地区生物资源、预测全球水生态系统变化趋势和平衡稳定和研究极端生境生态学问题等具有重要价值。近年来, eDNA技术发展迅速, 有效地改进了传统方法在水生态系统研究中的缺点, 提高了对水生生物的调查工作的效率。文章综述了eDNA技术对极地水环境中的鱼类、底栖生物、浮游生物、浮游细菌、病毒等水生生物的生物多样性分析、物种鉴定、种群结构分析及生态学等方面研究的应用进展。eDNA技术作为一种新兴的生物调查方法, 有能力加速更新分子时代的现代生物多样性调查的方法, 在研究极地水生生物多样性上具有极大的前景。
eDNA 极地 水环境 水生生物 生物多样性
0 引言
环境DNA(简称eDNA)指直接从环境样品(如土壤、水和沉积物等)获得的遗传物质(DNA片段)[1-2]。eDNA技术是通过从各环境介质中提取出特异性的DNA识别片段, 使用DNA测序技术分析所提取环境DNA的识别片段情况, 定性或者定量地分析生物体在环境介质中的具体分布情况和生态功能特征[1-2]。eDNA对原位采样生境中的生物类群无扰动性, 因此还有可能改善极地水生态系统的环境管理和评估方法[3]。eDNA技术在水生态系统可以进行目标物种的鉴别(如外来入侵物种、濒危物种和珍稀物种)、生物多样性监测与评价等工作。
1 eDNA技术的发展简介
eDNA技术发展离不开DNA测序技术的发展, 自20世纪70年代中期的第一代测序技术诞生以来, DNA测序技术已经取得了重大进展[4-5](图1)。第一代测序技术出现在1977年, 即Maxam和Gilbert发明的化学降解法及Sanger的双末端终止法[6-7]。基于第一代测序技术的发展, 2003年提出的条形码技术通过用特定的短DNA序列将物种进行比对, 实现了对生物的分类鉴定[8]。接下来应运而生的第二代测序技术(next-generation sequencing technology, NGS, 即高通量测序技术), 解决了第一代测序通量低、无法达到大规模应用的问题, 为生物分子研究做出了巨大贡献。eDNA宏条形码技术在二代测序的基础上通过对eDNA片段进行测序, 并将所得序列与标准条形码对比, 鉴定目标生物的种类[9]。1988年, Handelsman等[10]提出的宏基因组学概念为研究微生物提供了新的思路和方法。但直到基于第二代测序技术的宏基因组学分析方法建立才使直接从环境样本中提取基因组DNA后进行测序分析成为可能。宏基因组学技术是基因克隆文库的进一步深化, 避免了微生物培养的繁琐过程, 促进了对极端环境下生物多样性及功能的认识, 在eDNA技术的应用中扮演不可或缺的角色[11]。随着人们对DNA的研究进一步发展, 第三代测序技术已不再依赖聚合酶链式反应(polymerase chain reaction, PCR)技术[12]。
自步入基因组学时代, eDNA技术经过10余年的发展, 从1987年开始在环境微生物学领域应用, 微生物学家Ogram等[13]成功地从湖底沉积物中提取出环境微生物的DNA。2000年, “eDNA”第一次在文献中出现, 该研究中利用细菌人工染色体(BAC)载体构建了基因组DNA文库, 并表明了BAC文库中含有多种环境DNA, 在研究土壤微生物多样性方面有巨大的潜力, 使eDNA技术真正得到认可[14-15]。2008年, eDNA技术第一次运用于水生态系统中, 研究人员利用从水样中提取的DNA去检测一种原产于北美的入侵性两栖动物美国牛蛙是否入侵水域中[1]。之后, eDNA技术开始广泛应用于水生系统生物研究中。2014年, 研究员第一次利用eDNA技术检测萨卡里亚河中4种常见的入侵鱼类, 调查表明了eDNA可以用作监测淡水生态系统中入侵鱼类的重要分子工具[16]。
随着eDNA技术的日益成熟, 其应用由对物种监测的定性研究到现在对生物量评估的相对定量分析, 研究对象由微生物研究到两栖动物、淡水鱼类、海洋鱼类、爬行动物和腹足动物研究等[17]。第二代测序技术的发展促使eDNA技术迅速推广, 在物种鉴定研究上由当初的单个物种鉴定过渡到整个生物群的分析鉴定[18]。比如, NGS技术已经成功用于对无脊椎动物(大样本)的整个群落测序工作中[19-22]。此外, 使用eDNA可监测濒临灭绝的淡水昆虫、甲壳类动物、鱼类和哺乳动物, 并可以通过NGS技术来解释对整个湖区的两栖动物和鱼类的监测情况[23]。发展到如今的第三代测序已经可以不需要经过PCR扩增, 就实现了对DNA分子的测序[24]。
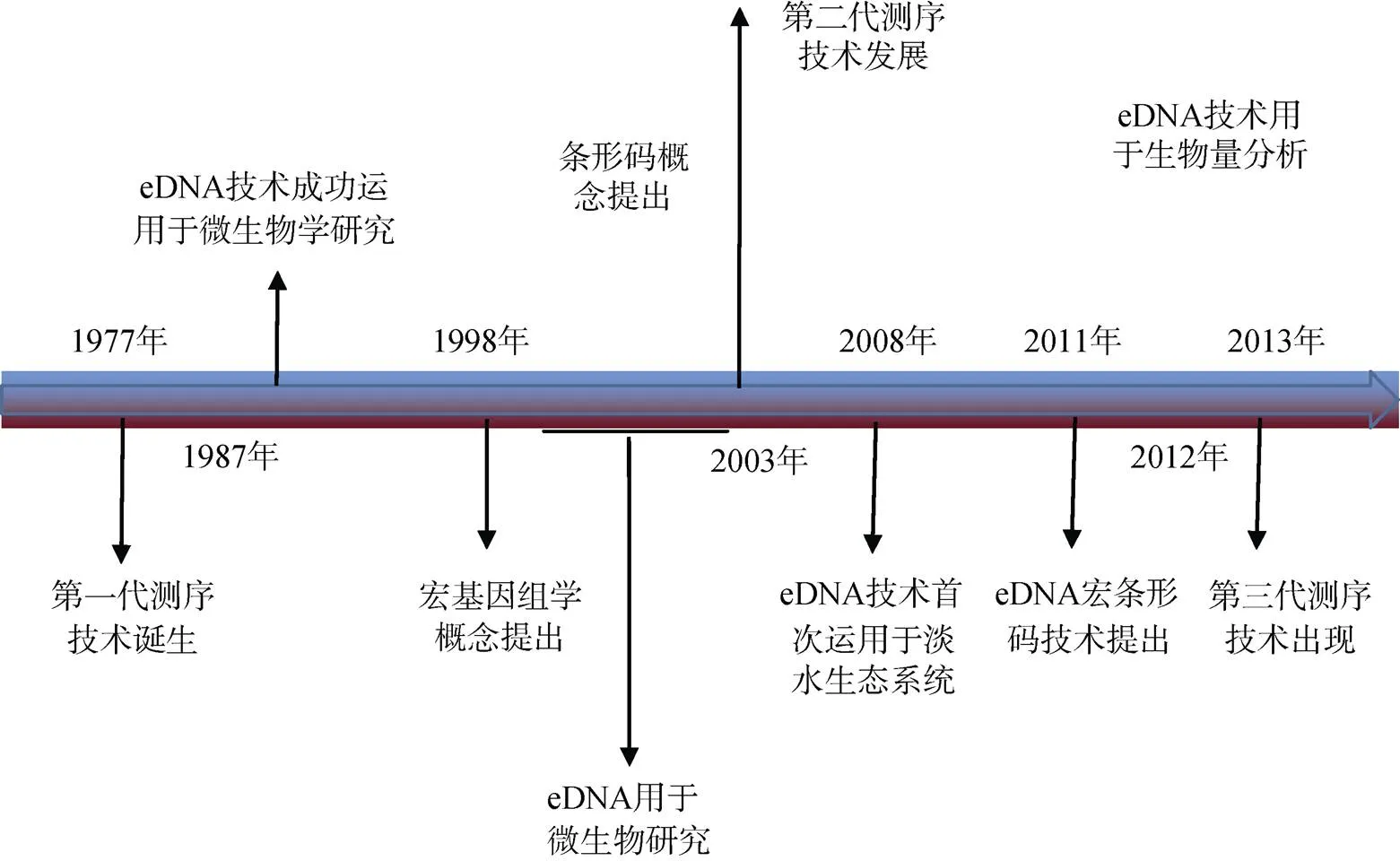
图1 eDNA发展历程导览图
Fig.1. Development path of eDNA
近年来将新一代eDNA技术[24]与机器学习[25]、卫星遥感[26-27]等技术联用, 已经可以大尺度、灵敏、自动地获取生态监测信息, 识别水生态系统中水生生物群落的分布情况[28]。除此之外, eDNA技术还具有以下优点: 优越的物种可检测性和特异性、成本较低且不会造成生态系统干扰、无需获知物种的基本信息即可进行检测、可在无法进行传统调查的地区实施等, 使得环境DNA技术作为一种新兴的监测方法, 虽然仍需要考虑和避免许多陷阱和障碍, 但该方法仍有能力加速更新分子时代的现代生物多样性调查的方法, 在生物多样性监测研究上具有极大的前景[17]。
2 eDNA技术在极地生态系统中的应用
极地地区终年寒冷干燥, 冰雪覆盖, 给预测和改善极地环境的研究工作带来极大的不便, 并且极地生态系统是一个巨大的、潜在的淡水和生物资源库, 是全球生态系统的重要组成部分。极地生物是极地生态系统的物质循环和能量流动中的重要载体, 其中有研究表明极地冰区的海洋鱼类物种形成率明显高于热带海洋地区[29]。随着生物研究技术的不断发展, 从分子和基因组水平对极地生态系统的研究将丰富对其的科学认知。应用传统形态学方法对极地水生生物进行定性和定量的研究有很大的局限性, 如原位采样难度很大, 样本完整性难以预测等。相对而言, 在气候条件极为恶劣的极地地区采用eDNA技术采样受限小、高效省时, 在研究极地水生生物方面有较为突出的优势。eDNA技术现已广泛应用于极地水环境中鱼类、底栖生物、浮游生物、浮游细菌、病毒等不同类群的多样性分析与评估、物种鉴定、群落的生物监测等方面的研究中。
2.1 eDNA应用于南大洋鱼类研究
近年来, eDNA条形码技术被应用于极地鱼类的生物监测研究中, 并实现了物种鉴定的标准化。所依赖的常用条形码基因片段包括细胞色素C氧化酶I(COI)基因、叶绿体基因rbcL、18S核糖体RNA(18S ribosomal RNA, 18S rRNA)基因、内转录间隔区(internal transcribed spacers, ITS)基因、16S rRNA和28S rRNA等。Near等[30]使用线粒体16S rRNA的全基因序列对南大洋类胡萝卜素鱼类Performformes: Notothenioidei进行系统发育研究发现南极鱼亚目鱼类的5个主要进化分支分别是: 裸南极鱼科、阿氏龙䲢科、龙䲢科、鳄冰鱼科、南极鱼科。
李渊等[31]利用形态学鉴定和基于线粒体COI基因的eDNA条形码技术对南大洋Yelcho站周边海域的鱼类进行物种鉴定, 从采集的8尾鱼类样品中成功鉴定出3个有效种, 有7尾是南极鱼科鱼类, 1尾裸南极鱼科。结合两种方法对南极鱼类进行鉴定确保了鉴定结果的准确性和有效性。研究不仅纠正了条形码参考数据库中的错误序列, 一定程度上还反映出了南大洋鱼类的物种组成和生物量均以南极鱼科鱼类为主[31]。
2.2 eDNA应用于底栖生物研究
底栖生物是水生生态系统的重要组成部分, 亦是海洋环境质量的重要指标, 对了解生态系统的结构具有重要意义[32]。按生活方式可分为底栖植物(微藻、大型海藻等)和底栖动物。以底栖蓝藻为例, 蓝藻群落生物量会在极地湖泊和池塘的底栖环境大量生长, 对固碳做出了重要贡献[33]。
在弗雷塞尔湖底栖环境的研究中发现有大量的蓝藻生物量积累。使用形态学和分子方法对南极维多利亚州南部弗雷塞尔湖的天然和人工微生物垫中蓝藻的表型和基因型多样性进行分析[34]。其中分子方法指包括16S rRNA基因克隆库, 变性梯度凝胶电泳(denatured gradient gel electrophoresis, DGGE)和测序[35], 同时结合光学显微镜观察结果, 鉴定出8种形态性; 而分子工具则发现了15种系统型。分子结果表明南极蓝藻多样性远大于单独依托传统显微镜分析出的形态分型。因此, 分子工具能更完整地补充描述南极湖泊底栖中的蓝藻多样性。这也是首次采用PCR引物对16S rRNA基因和对蓝藻序列特异的ITS进行扩增, 并进行蓝藻多样性分析的多相分析的突破[34]。Destombe等[35]采用DNA条形码技术对种在欧洲北大西洋和摩洛哥海岸两种江蓠属大型藻类进行研究, 利用3种独立标记的条形码cox2-cox3间隔区、叶绿体基因rbcL和ITS 2区域对其差异进行遗传分析, 证实了北大西洋存在两个名为的并行分支物种已有200年的历史[35]。研究同时证明多基因条形码能更精确地描绘这两种形态学物种并检测假定的杂交的发生。刘晨临和林学政[36]利用形态学和DNA条形码对白令海海域和冰岛附近海域的褐藻进行鉴定, 并对其来源进行分析。其中采自白令海海域的褐藻为孔叶藻亚种(), 来源于日本北海道, 另一种为瘤状囊叶藻(), 常见于北大西洋沿岸。
Grant和Linse[37]应用DNA条形码技术对南极海洋地区包括韦德尔海、罗斯海在内十几个区域的底栖无脊椎动物进行物种鉴定和遗传研究。研究发现这些水域中的无脊椎动物主要是甲壳纲、环节动物和软体动物等3类, 该研究同时建议南极海洋地区还需要进行更深入的DNA条码研究, 来推动底栖动物的分子生态学研究。
2.3 eDNA应用于浮游生物研究
浮游生物可分为浮游动物和浮游植物。通过eDNA技术, 不仅有助于准确地了解浮游生物生态和功能的多样性、时空分布情况, 同时还可获取物种间遗传进化信息, 寻找新的基因功能。
浮游动物是由原生动物和后生动物组成的生物群落, 主要有原生动物、轮虫、枝角类和桡足类等四类。浮游动物生命周期短, 生长迅速, 对环境变化敏感, 常被用作指示物种。相比而言, 极地海域的浮游动物的种群演替深受海水、海冰变化的影响, 对于环境变动表现得更为敏感强烈, 常被用作研究全球气候变化对极地生态系统影响的重要指标[38]。DNA条形码技术是对极地浮游动物进行物种鉴定的一种常用方法。
在研究极地浮游动物生物多样性分析中, 应用eDNA宏条形码技术可以极大地简化分析步骤。程方平等[40]利用DNA条形码技术验证了南极海域DNA条形码在浮游动物种鉴定的有效性, 研究中系统地比较分析了南极普里兹湾和南极半岛周边海域35种常见浮游动物的124条线粒体COI序列[39-40]。研究同时发现部分物种的种内遗传差异水平较高, 表明DNA条形码还可用作相关物种的种群遗传学研究[40]。此次研究新增的DNA条形码数据以及新提供的兼并引物将推动南极浮游动物环境样品的宏基因组学研究[40]。在北极海域的比较研究中也证实了DNA条形码技术在极地浮游动物种类鉴定及相关研究中的可靠性[41-42]。Chain等[43]在对沿加拿大海岸线(北极)哈德逊湾和哈德逊海峡浮游动物的生物多样性进行大规模的时空评估中用到了eDNA宏条形码技术, 研究表明, 该技术为大规模生物多样性调查提供了一种简化而灵敏的方法。Heimeier等[44]基于16S rRNA、COI和18S rRNA基因扩增的DNA条形码技术结合传统形态学方法对南极罗斯海及附近水域浮游动物(无脊椎动物幼体)进行了物种鉴定, 主要是棘皮动物、软体动物、纽形动物和环节动物等4个门属。研究同时说明了应用分子工具可更好地补充形态学鉴定结果。
与浮游动物相似, 浮游植物也对环境变化极为敏感。浮游植物具有个体小、生长周期短、营浮游生活等特点。极地微型浮游植物较多是低等单细胞藻类, 是极地生态系统重要的初级生产者和食物网的基础。世界上已经发现了超过110多种极地微型浮游植物[45], 人们已经开始应用eDNA技术分析极地微型浮游植物多样性[46]。浮游植物的种群结构和生物量及其多样性能及时地反映极地水域生态环境的变化[47]。传统研究浮游植物的多样性及分布情况的方法是基于显微镜技术的形态学方法, 目前从基因组水平(eDNA分析)对浮游植物进行研究相较于传统方法有明显的优势[48]。Eiler等[49]利用16S rRNA基因作为标记的NGS技术和显微镜技术对南极及周边区域的49个湖泊的浮游植物多样性分析发现: 杂种动物门(phylum)含量最高, 其次是蓝藻(Cyanobacteria)、隐藻(Cryptophyta)、绿藻(Chlorophyta)等。并且NGS技术的分辨率更高。
对环境的变化响应最为直接敏感的一类是广泛分布在海冰、湖水、冰川融水和积雪表层的极地微藻。目前使用基因组学测序对极地微藻的研究仍处于基础阶段[50]。第一个完成全基因组测序的极地微藻是南极拟脆杆藻[51], 研究发现差异等位基因在不同环境下表达的丰度是不同的, 通过这些分析可以估测许多极地微藻的来源与温带藻类不同[50]。
浮游生物的个体大小差异大, 依据不同粒径分级, 浮游生物类群可分为微型以及微微型浮游生物, 它们体积微小, 种类繁多, 难以检测且许多种类缺乏明显的形态学特点。针对北冰洋、南极附近水体中的微微型浮游生物, 研究者使用分子生物学技术对其多样性进行研究, 获取了大量的微微型真核浮游生物的基因序列[52-53]。吴月等[54]利用eDNA技术对楚科奇海海域5个站位22个水层的样品进行微微型真核浮游生物的种群结构和空间分布分析, 研究中通过对扩增的18S rRNA高突变V4区基因片段序列进行高通量测序分析, 得出: 各纲的分布存在明显差异, 相对丰度大于1%的微微型真核浮游生物有6个纲, 分别为不等鞭毛类的金藻纲、沟鞭藻纲、中鼓藻纲、青绿藻cladeII、旋毛纲和银耳纲, 其中自养型微微型真核浮游植物在北冰洋海域约占85.2%, 具有绝对优势。
2.4 eDNA应用于浮游细菌研究
分布在极地海洋各个水层的浮游细菌是极地生态系统的重要组成部分之一, 一定程度上维持着极地海洋生态系统的稳定性和生物多样性。在环境样品中发现新的16S rRNA基因序列, 为研究者们研究微生物多样性提供一个渠道[55]。它是通过确定细菌的系统发育关系进而评价浮游细菌多样性的方法[56]。Dobson等[57]于1993年利用PCR扩增的16S rRNA基因直接测序成功识别出南极高盐湖两个新物种和证实了16S rRNA基因序列分析在系统发育研究上的巨大潜力。
Zeng等[58]对16S rRNA基因进行454焦磷酸测序揭示了北极Kongsfjorden(Spitsbergen)和Chukchi Borderland两个水域的浮游细菌的多样性, 研究发现Kongsfjorden(Spitsbergen)的浮游细菌以γ-变形菌和拟杆菌为主, 而Chukchi Borderland地表海水样品中的以α-变形菌和放线菌为主。同时2014年Zeng等[59]利用同样454焦磷酸测序对南极阿德雷湾及长城湾附近表层海水样品中浮游细菌多样性进行分析, 发现阿德雷湾样品细菌可划分为18个门类, 长城湾样品细菌分属于11个门类, 其中拟杆菌门、变形菌门为优势细菌。
Ghiglione和Murray[60]以标签序列焦磷酸测序方法(基于聚合酶链式反应的毛细管电泳-单链构象多态性方法、PCR-DGGE和DGGE条带测序、PCR标记测序), 即通过扩增浮游细菌的16S rRNA基因的可变V3区、高变V6区序列分析和DGGE分析研究南极沿海浮游细菌多样性和种群结构变化。同时对凯尔盖朗群岛(靠近南极)以及南极半岛浮游细菌进行系统型鉴定, 结合Simpson和Shannon指数预测物种丰富度。研究发现: 两个海域的浮游细菌主要类群比例有明显不同, 但多样性与以往研究相似, 且在南半球夏季的浮游细菌丰度显著高于冬季。Cao等[61]利用基于16S rRNA基因的V1-V3可变区的焦磷酸测序分析研究南极半岛北端表层海域浮游细菌群落结构和群落结构的影响因子。研究中通过所获浮游细菌的覆盖度、丰富度和α多样性指数等来评估水域浮游细菌多样性, 发现最丰富的细菌群是α-变形菌, 其次是γ-变形菌。研究中检测到的环境因素营养盐、叶绿素、原位温度和盐度等指标对浮游细菌多样性和群落变化的影响相对较小[61]。Møller等[62]通过对16S rRNA基因进行焦磷酸测序及对培养菌株进行16S rRNA基因测序, 研究北极高纬度雪中和淡水湖中的细菌群落结构。通过两个区域的细菌多样性分析可知, 在极地冰雪中细菌丰度高于淡水湖, 但是极地雪中和淡水湖中发现的属之间有很强的重叠。
2.5 eDNA应用于病毒研究
病毒构成了地球上最丰富的生物实体并提供了大量的遗传多样性。它是指游离在水体中的各种病毒, 是水生微生物群落和生态系统的一个动态组成部分[63]。浮游病毒分为藻病毒、噬菌体、噬藻体, 除此以外还可发现一些动物病毒和人类病毒等。针对病毒的分子生物技术有PCR-DGGE、脉冲场电泳技术、随机扩增的多态性DNA聚合酶链式反应技术、宏基因组文库、克隆文库等。从核酸水平上研究病毒动态分布和遗传多样性, 极大地促进了浮游病毒群落结构和生态学研究。eDNA技术为极地地区病毒群落结构和遗传多样性提供一个有潜力的新途径。
在北极和南极海冰中都观察到大量的病毒[64]。病毒遗传多样性的研究是以大量的相关遗传信息为背景和基础的, 目前已知的藻病毒、噬藻体等的全基因组序列还较少, 而南极病毒研究的主要限制是缺乏基因组序列数据。Alberto等[65]通过从南极Byers半岛的Limnopolar Lake纯化的2 000万碱基对的病毒DNA进行焦磷酸测序得到89 347个序列, 分析研究病毒群落的高度多样性, 描述了约10 000种病毒基因型和夏季在Limnopolar Lake中发生的病毒的生态演替[66]。研究表明Limnopolar Lake中病毒群落以感染真核生物的病毒主导。同时Limnopolar Lake高度的遗传丰富度表明, 在其他类群的生物多样性较低的系统中, 可以发现较高的病毒多样性[65]。进一步对Limnopolar Lake的深入研究, 在整个光活跃月份和整个年度周期中更频繁地评估病毒种群, 并结合对整个微生物群落组成的季节性动态进行分析, 将病毒与宿主联系起来, 说明病毒在南极水生态系统中发挥着重要作用[66]。
宏基因组技术在研究极地病毒上克服了多数浮游病毒种类无法培养的缺陷, 能直接对环境基因组序列进行分析, 从而获知生物基因序列信息以便进行后续研究[67]。Daniel等[68]通过宏基因组学技术解析北极6个大型水体的病毒DNA, 分析了北极淡水病毒DNA群落结构。在本次研究中提供了来自北极和南极的DNA病毒的深度序列数据, 包括来自6个北极湖泊的病毒群落深度测序数据, 并结合对来自世界各地一系列不同地理位置上已发表的淡水病毒DNA进行了比较分析。结果表明了北极淡水病毒群落由未知和单链DNA病毒主导, 揭示了一些具有双极分布的病毒谱系及全球生物地理和病毒群落的连通性[65]; 北极地区病毒物种丰富度没有降低, 说明病毒可能不遵循纬度多样性梯度, 揭示了南、北两极微生物生态系统之间的差异, 生物地理学和病毒群落的连通性不仅突出了极地环境的独特性, 也突出了两极微生物生态系统之间的差异[68]。Breitbart等[69]根据宏基因组学对从马尾藻海、墨西哥湾、加拿大不列颠哥伦比亚、北冰洋的近岸水域收集到的短序列数据库进行分析, 研究发现浮游病毒群落里含有500~130 000个基因型, 其中很大一部分基因型在不同的地理区域中共同存在[70]。
3 结论
综上所述, eDNA技术已广泛应用于极地水生态系统中鱼类、底栖生物、浮游生物、浮游细菌、病毒的研究, 不限于极地生物群落多样性、物种鉴定、生物监测等课题。尤其基于第二代测序技术的发展, 使用eDNA技术获取数据对极地生物进行监测分析的能力大幅提升, 所获取的eDNA数据对评估一些物种的多样性有重要参考意义, 并且使用eDNA技术能发现很多形态学难以识别的物种, 在分析种群结构及其组成上可以反映出更为精确的结果, 比如物种的遗传多样性。随着eDNA技术的改进, 基本解决了极地环境生态学研究上方法有限、采样空间局限、数据信息处理慢及监测结果不准等一系列问题。总而言之, eDNA技术在认知极地的水生态系统中已扮演重要角色, 应用上已渐趋成熟。
未来发展中, 可将eDNA技术与其他技术相结合, 以有效避免单一技术数据不准确造成的偏差。比如宏基因组文库与克隆文库结合分析能更准确地反映微生物的多样性[67]; 比如抽样检测[71]可尽可能避免eDNA技术在操作过程中出现的污染和陷阱, 提高结果的准确度等。不断扩大eDNA技术的应用范围, 对极地巨大的生物资源进行系统研究统计、完善充实数据库、发展生物信息学工具、增加数据处理的可信度, 可继续拓展eDNA技术在极地生物多样性分析和环境保护中的应用范围, 使其进一步应用到食物网、能量流动、种群遗传信息收集研究等方面[72]。
1 FICETOLA G F, MIAUD C, POMPANON F, et al. Species detection using environmental DNA from water samples[J]. Biology Letters, 2008, 4(4): 423-425.
2 HAILE J, FROESE D G, MACPHEE R D E, et al. Ancient DNA reveals late survival of mammoth and horse in interior Alaska[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(52): 22352-22357.
3 KELLY R P, PORT J A, YAMAHARA K M, et al. Harnessing DNA to improve environmental management[J]. Science, 2014, 344(6191): 1455-1456.
4 董天宇. DNA测序技术[J]. 技术应用于研究, 2018(11): 71-72.
5 徐疏梅. 新一代DNA测序技术的应用与研究进展[J]. 徐州工程学院学报(自然科学版), 2018, 33(4): 60-64.
6 SANGER F, NICKLEN S, COULSON A R. DNA sequencing with chain-terminating inhibitors[J]. PNAS, 1977, 74(12): 5463-5467.
7 MAXAM A M, GILBERT W A. A new method for sequencing DNA[J]. Proceedings of the National Academy of Sciences, 1977, 74(2): 560-564.
8 HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London Series B: Biological Sciences, 2003, 270(1512): 313-321.
9 POMPANON F, COISSAC E, TABERLET P. Metabarcoding a new way to analyze biodiversity[J]. Biofutur, 2011(319): 30-32.
10 HANDELSMAN J, RONDON M R, BRADY S F, et al. Molecular biological access to the chemistry of unknown soil microbes: A new frontier for natural products[J]. Chemistry & Biology, 1998, 5(10): 245-249.
11 COUNCIL N R. The New Science of Metagenomics: Revealing the Secrets of Our Microbial Planet[M]. Washington DC: National Academics Press, 2007:1-170.DOI:org/10.17226/11902.
12 HEATHER J M, CHAIN B. The sequence of sequencers: The history of sequencing DNA[J]. Genomics, 2016, 107(1): 1-8.
13 OGRAM A, SAYLER G S, BARKAY T. The extraction and purification of microbial DNA from sediments[J]. Journal of Microbiological Methods, 1987, 7(2/3): 57-66.
14 RONDON M R, AUGUST P R, BETTERMANN A D, et al. Cloning the soil metagenome: A strategy for accessing the genetic and functional diversity of uncultured microorganisms[J]. Applied and Environmental Microbiology, 2000, 66(6): 2541-2547.
15 JO H. Metagenomics: application of genomics to uncultured microorganisms[J]. Microbiology and Molecular Biology Reviews, 2004, 68(4): 669-685.
16 KESKIN E. Detection of invasive freshwater fish species using environmental DNA survey[J]. Biochemical Systematics and Ecology, 2014, 56: 68-74.
17 单秀娟, 李苗, 王伟继. 环境DNA(eDNA)技术在水生生态系统中的应用研究进展[J]. 渔业科学进展, 2018, 29(3): 23-29.
18 REES H C, MADDISON B C, MIDDLEDITCH D J, et al. REVIEW: The detection of aquatic animal species using environmental DNA–a review of eDNA as a survey tool in ecology[J]. Journal of Applied Ecology, 2014, 51(5): 1450-1459.
19 HAJIBABAEI M, SHOKRALLA S, ZHOU X, et al. Environmental barcoding: A next-generation sequencing approach for biomonitoring applications using river benthos[J]. PLoS One, 2011, 6(4): e17497.
20 GIBSON J, SHOKRALLA S, PORTER T M, et al. Simultaneous assessment of the macrobiome and microbiome in a bulk sample of tropical arthropods through DNA metasystematics[J]. Proceedings of the National Academy of Sciences, 2014, 111(22): 8007-8012.
21 GIBSON J F, SHOKRALLA S, CURRY C, et al. Large-scale biomonitoring of remote and threatened ecosystems via high-throughput sequencing[J]. PLoS One, 2015, 10(10): e0138432.
22 THOMSEN P F, WILLERSLEV E. Environmental DNA: An emerging tool in conservation for monitoring past and present biodiversity[J]. Biological Conservation, 2015, 183(1): 4-18.
23 THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Molecular Ecology, 2012, 21(11): 2565-2573.
24 李明爽, 赵敏. 第三代测序基本原理[J]. 现代生物医学进展, 2012(10): 1980-1982.
25 LIMA-MENDEZ G, FAUST K, HENRY N, et al. Determinants of community structure in the global plankton interactome[J]. Science, 2015, 348(6237): 1262073.
26 TROMAS N, FORTIN N, BEDRANI L, et al. Characterizing and predicting cyanobacterial blooms in an 8-year amplicon sequencing time-course[J].The ISME Journal, 2017, 11 (8): 1746.
27 TORTI A, LEVER M A, JØRGENSEN B B. Origin, dynamics, and implications of extracellular DNA pools in marine sediments[J]. Marine Genomics, 2015(24): 185-196.
28 张飞龙, 杨江华, 杨雅楠, 等. 环境DNA条宏条形码检测水生态系统变化与健康状态[J]. 中国环境监测, 2018, 34(6): 37-46.
29 RABOSKY D L , CHANG J , TITLE P O , et al. An inverse latitudinal gradient in speciation rate for marine fishes[J]. Nature, 2018, 559(7714): 392-395.
30 NEAR T J, PESAVENTO J J, CHENG C H C. Phylogenetic investigations of Antarctic notothenioid fishes (Perciformes: Notothenioidei) using complete gene sequences of the mitochondrial encoded 16S rRNA[J]. Molecular Phylogenetics and Evolution, 2004, 32(3): 881-891.
31 李渊, 张然, 宋普庆, 等. 基于DNA条形码对南极Yelcho站周边海域鱼类的种类鉴定[J]. 极地研究, 2018, 30(2): 192-197.
32 蔡丽萍, 金敬林, 吴盈子. 2016年夏季舟山近岸海域底栖生物调查与研究[J]. 海洋开发管理, 2018, 35(9): 82-87.
33 HAWES I, SCHWARZ A. Photosynthesis in an extreme shade environment: Benthic microbial mats from lake Hoare, a permanently ice-covered Antarctic lake[J]. Journal of Phycology, 1999, 35(3): 448-459.
34 TATON A, GRUBISIC S, BRAMBILLA E, et al. Cyanobacterial diversity in natural and artificial microbial mats of Lake Fryxell (McMurdo Dry Valleys, Antarctica): A morphological and molecular approach[J]. Applied and Environmental Microbiology, 2003, 69(9): 5157-5169.
35 DESTOMBE C, VALERO M, GUILLEMIN M L. Delineation of two sibling red algal species, Gracilaria gracilis and Gracilaria dura (Gracilariales, Rhodophyta), using multiple DNA markers: Resurrection of the species G. dura previously described in the Northern Atlantic 200 years ago[J]. Journal of Phycology, 2010, 46(4): 720-727.
36 刘晨临, 林学政. 两株北极褐藻DNA条形码序列的记录[J]. 极地研究, 2017, 29(2): 228-235.
37 GRANT R A, LINSE K. Barcoding Antarctic Biodiversity: Current status and the CAML initiative, a case study of marine invertebrates[J]. Polar Biology, 2009, 32(11): 1629-1637.
38 HAYS G, RICHARDSON A, ROBINSON C. Climate change and marine plankton[J]. Trends in Ecology & Evolution, 2005, 20(6): 337-344.
39 HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London Series B: Biological Sciences, 2003, 270(1512): 313-321.
40 程方平, 王敏晓, 孙松. 基于mtCOI片段序列的南极海域浮游动物的DNA条形码研究[J]. 极地研究, 2014, 26(2): 212-221.
41 BUCKLIN A, HOPCROFT R R, KOSOBOKOVA K N, et al. DNA barcoding of Arctic Ocean holozooplankton for species identification and recognition[J]. Deep-Sea Research Part I : Topical Studies in Oceanography, 2010, 57(1): 40-48.
42 ZHANG W W, XIE Y W, YANG J H, et al, Applications and prospects of meta barcoding in environmental monitoring of phytoplankton community [J]. Asian Joumal of Ecotoxicology, 2017, 12(1): 15-24.
43 CHAIN F J J, BROWN E A, MACISAAC H J, et al. Metabarcoding reveals strong spatial structure and temporal turnover of zooplankton communities among marine and freshwater Ports[J]. Diversity and Distributions, 2016, 22(5): 493-504.
44 HEIMEIER D, LAVERY S, SEWELL M A. Using DNA barcoding and phylogenetics to identify Antarctic invertebrate larvae: Lessons from a large scale study[J]. Marine Genomics, 2010, 3(3): 165-177.
45 梁晓芸, 魏东, 刘龙军, 等. 极地微藻的研究进展[J]. 海洋科学, 2007,31(4): 92-94.
46 MOCK T, THOMAS D N. Recent advances in sea-ice microbiology[J]. Environmental Microbiology, 2005, 7(5): 605-619.
47 王备新, 杨莲芳, 刘正文. 生物完整性指数与水生态系统健康评价[J]. 生态学杂志, 2006, 25(6): 707-710.
48 EGGE E, BITTNER L, ANDERSEN T, et al. 454 pyrosequencing to describe microbial eukaryotic community composition, diversity and relative abundance: A test for marine haptophytes[J]. PLoS One, 2013, 8(9): e74371.
49 EILER A, DRAKARE S, BERTILSSON S, et al. Unveiling distribution patterns of freshwater phytoplankton by a next generation sequencing based approach[J]. PLoS One, 2013, 8(1): e53516.
50 何美琳, 迟巧云, 王长海. 极地微藻对极端环境的适应机制研究进展研究[J]. 南京农业大学学报. 2019, 42(2): 201-208.
51 FELLER G, GERDAY C. Psychrophilic enzymes: Hot topics in cold adaptation[J]. Nature Reviews Microbiology, 2003, 1(3): 200-208.
52 LOVEJOY C, MASSANA R, PEDRÓS-ALIÓ C. Diversity and distribution of marine microbial eukaryotes in the Arctic Ocean and adjacent seas[J]. Applied and Environmental Microbiology, 2006, 72(5): 3085-3095.
53 CHISHOLM S W, FRANKEL S L, GOERICKE R, et al. Prochlorococcus marinus nov. gen. sp.: An oxyphototrophic marine prokaryote containing divinyl chlorophyll a and b[J]. Archives of Microbiology, 1992, 157(3): 297-300.
54 吴月, 曹叔楠, 何剑锋, 等. 北极楚科奇海微微型真核浮游生物群落结构及其环境相关性分析[J]. 极地研究, 2019, 31(1): 25-31.
55 HUGENHOLTZ P, GOEBEL B M, PACE N R. Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity[J]. Journal of Bacteriology, 1998, 180(24): 4765-4774.
56 薛超波, 王国良, 金珊, 等. 海洋微生物多样性研究进展[J]. 海洋科学进展, 2004, 22(3): 377-384.
57 DOBSON S J, COLWELL R R, MCMEEKIN T A, et al. Direct sequencing of the polymerase chain reaction-amplified 16S rRNA gene of Flavobacterium gondwanense sp. nov. and Flavobacterium salegens sp. nov., two new species from a hypersaline Antarctic lake[J]. International Journal of Systematic Bacteriology, 1993, 43(1): 77-83.
58 ZENG Y X, ZHANG F, HE J F, et al. Bacterioplankton community structure in the Arctic waters as revealed by pyrosequencing of 16S rRNA genes[J]. Antonie van Leeuwenhoek, 2013, 103(6): 1309-1319.
59 ZENG Y X, YU Y, QIAO Z Y, et al. Diversity of bacterioplankton in coastal seawaters of Fildes Peninsula, King George Island, Antarctica [J]. Archives of Microbiology, 2014, 196(2): 137-147.
60 GHIGLIONE J F, MURRAY A E. Pronounced summer to winter differences and higher wintertime richness in coastal Antarctic marine bacterioplankton[J]. Environmental Microbiology, 2012, 14(3): 617-629.
61 CAO S N, HE J F, ZHANG F, et al. Diversity and community structure of bacterioplankton in surface waters off the northern tip of the Antarctic Peninsula[J]. Polar Research, 2019, 38:1-15.DOI:10.33265/polar.v38.3491.
62 MØLLER A K, SØBORG D A, ABU AL-SOUD W, et al. Bacterial community structure in High-Arctic snow and freshwater as revealed by pyrosequencing of 16S rRNA genes and cultivation[J]. Polar Research, 2013, 32(1): 17390.
63 WOMMACK K E, RAVEL J, HILL R T, et al. Population dynamics of chesapeake bay virioplankton: Total community analysis by pulsed-field gel electrophoresis[J]. Applied and Environmental Microbiology, 1999, 65(1): 231-240.
64 COLLINS R E, DEMING J W. Abundant dissolved genetic material in Arctic sea ice Part II: Viral dynamics during autumn freeze-up[J]. Polar Biology, 2011, 34(12): 1831-1841.
65 ALBERTO L B, JAVIER T, DAVID V, et al. High diversity of the viral community from an Antarctic lake[J]. Science, 2009, 326(5954): 858-861.
66 CAVICCHIOLI R, ERDMANN S. The discovery of Antarctic RNA viruses: A new game changer[J]. Molecular Ecology, 2015, 24(19): 4809-4811.
67 王芳. 北黄海藻类DNA病毒遗传多样性研究[D]. 青岛: 中国海洋大学, 2017: 17.
68 DANIEL A C, ALBERTO L B, DAVID A P, et al. Biodiversity and distribution of polar freshwater DNA viruses[J]. Microbial Ecology, 2015,1: e1400127.
69 BREITBART M, FELTS B, KELLEY S, et al. Diversity and population structure of a near-shore marine-sediment viral community[J]. Proceedings of the Royal Society B: Biological Sciences, 2004, 271(1539): 565-574.
70 ANGLY F E, FELTS B, BREITBART M, et al. The marine viromes of four oceanic regions[J]. PLoS Biology, 2006, 4(11): e368.
71 JI Y Q, ASHTON L, PEDLEY S M, et al. Reliable, verifiable and efficient monitoring of biodiversity via metabarcoding[J]. Ecology Letters, 2013, 16(10): 1245-1257.
72 陈炼, 吴琳, 刘燕, 徐海根. 环境DNA metabarcoding及其在生态学研究中的应用[J]. 生态学报, 2016, 36(15): 4573-4582.
PROGRESS OF APPLICATION OF eDNA TECHNOLOGY IN POLAR WATER ENVIRONMENTS
Liu Mengyue1,2, Zhao Wenyu1,2
(1Aquatic Resources Food Processing Engineering Technology Research Center of Hunan, School of Chemistry and Food Engineering, Changsha University of Science & Technology, Changsha 410004, China;2Key Laboratory of Dongting Lake Aquatic Eco-Environmental Control and Restoration of Hunan Province, School of Hydraulic Engineering, Changsha University of Science & Technology, Changsha 410004, China)
Environmental DNA (eDNA) technology has been applied to efficiently and accurately carry out research works involving species identification and diversity analysis of organisms in polar aquatic ecosystems (such as polar oceans, coastal waters, lakes, and streams). This is of great value for the development of biological resources in polar region, prediction of changes in the balance and stability of global aquatic ecosystems, and research on ecological issues in extreme habitats. In recent years, eDNA technology has developed rapidly, effectively addressing the disadvantages of traditional methods in the study of aquatic ecosystems, and improving the investigation of aquatic animals. This review describes progress in the application of eDNA technology in biodiversity analysis, species identification, population structure analysis, and other ecological research on aquatic organisms such as fish, benthic organisms, plankton, and viruses in polar aquatic environments. As a new biological survey method, eDNA technology can drastically accelerate the updating of modern biodiversity methodologies in the current molecular era, and has infinite prospects in polar aquatic biodiversity research.
eDNA, polar, water environment, aquatic organism, biodiversity
2020年1月收到来稿, 2020年2月收到修改稿
湖南省科技创新项目重点研发计划(2018SK2011)、洞庭湖水环境治理与生态修复湖南省重点实验室开放基金(2018DT02)、湖南省水生资源食品加工工程技术研究中心开放基金(2018KJY11)资助
刘梦月, 女, 1997年生。硕士, 研究方向为水环境生态学。E-mall: lmycs2020@163.com
赵文玉, E-mall: wenyuzh@csust.edu.cn
10. 13679/j.jdyj.20200004

