南极大型海藻表面可培养琼胶降解菌多样性分析
桂媛媛 谷晓倩 李江 张培玉
研究论文
南极大型海藻表面可培养琼胶降解菌多样性分析
桂媛媛1谷晓倩2李江2张培玉1
(1青岛大学环境工程学院, 山东 青岛 266071;2自然资源部第一海洋研究所海洋生物资源与环境研究中心, 山东 青岛 266061)
采用选择性培养基、卢戈氏染色法对南极乔治王岛附近海域6株大型海藻表面可培养的琼胶降解菌进行了筛选和分离纯化, 并基于菌株的16S rRNA基因序列进行了分子鉴定和系统发育分析。结果表明, 筛选获得的37株琼胶降解菌分别属于γ-变形菌纲(Gammaproteobacteria), 黄杆菌纲(Flavobacteriia)和放线菌纲(Actinobacteria)。其中γ-变形菌纲包括、、、、、等7个属及Thiotrichaceae科, 黄杆菌纲包括、2个属, 放线菌纲只筛选出1个属优势属为假交替单胞菌属(, 20株), 其次为嗜冷杆菌属(, 4株)。、、和等5个属及Thiotrichaceae科是首次从南极大型海藻表面分离的具有琼胶降解活性的种属。该结果拓展了我们对琼胶降解菌多样性的认识, 为南极微生物资源调查和开发研究提供了科学依据。
南极 琼胶降解菌 多样性 系统发育分析
0 引言
极地地区光照辐射变化极大、光照时间季节性极强、常年水温极低且高盐度等独特的地理和气候特征, 使其形成了酷寒、强辐射和高盐度的自然环境。极地生态系统比普通的生态系统相对简单脆弱, 在极地地区许多极端的低温、高盐度及干燥环境下只有微生物适合生存, 微生物生态可以更加敏锐地反映出极地生态系统的变化, 因此研究极地微生物群落及多样性对认识极地生态环境的变化具有重要作用[1-2]。随着人们对南极地区生态系统的关注度不断提高, 关于微生物多样性的研究也逐渐增多, 但对南极海域大型海藻表面附生菌多样性的研究尚不多见。
海洋微生物在海洋生态系统中扮演着重要角色, 因此研究海洋微生物的种群和功能也越来越受到人们的关注[3]。南极海域分布着丰富的大型海藻资源, 特别是拥有高比例的特有物种。大型海藻在海洋生态系统中占据重要地位, 是海洋生态系统碳循环的重要组成部分。这些大型藻类细胞壁中含有琼脂、卡拉胶、褐藻胶等多种结构复杂的多糖, 且不含有陆生植物难以降解转化的木质素, 因此藻类生物质作为具有明显优势的生物能源的原料, 极易被微生物降解。海洋异养微生物能够消化高分子量的海藻多糖有机物, 将其分解为低分子量物质, 降解后的低分子量寡糖可被海洋生物作为碳源吸收利用[4]。南极海洋环境具有丰富的生物多样性, 且存在大量特有的大型海藻物种, 有望获得新颖的生物资源, 特别是功能微生物及其次级代谢产物。
大型海藻的表面是一个营养丰富、安全舒适的栖息地, 孕育了数量庞大、组成复杂并且充满活力的微生物群落, 每1 cm2大藻表面约包含1.1× 108的微生物[5], 这些微生物在海藻的进化、生理代谢以及海洋生态环境方面都发挥着重要的作用。对于海藻表面大量的附生菌而言, 最具吸引力的无疑是多糖降解菌。多糖降解菌产生的多糖降解酶在海洋生物资源的开发和利用中发挥着重要作用, 此外多糖降解菌在全球的碳代谢以及海藻生物质循环的过程中都扮演着重要的角色, 在海水和海洋沉积物中发现了很多能降解琼胶作为碳源的微生物[6], 海洋环境中不计其数的与海藻多糖降解相关的微生物, 利用自身基因所编码表达的糖苷水解酶(GH)和糖苷裂解酶(PL)参与了重要的碳代谢途径[4], 然而目前有关南极特别是南极大型海藻表面来源的多糖降解菌的研究却鲜见报道。
Tropeano等[7]从乔治王岛波特湾的海洋沉积物中分离到以革兰氏阴性菌为主的南极细菌, 并检测到了琼胶酶活性。Alvarado和Leiva[8]从南极乔治王岛采集的大型海藻表面分离获得30株产色素的琼胶降解菌, 并对其进行多样性分析。但我们对南大洋近岸大量分布的大型海藻表面多糖降解菌的认识还很有限, 因此, 本文通过对南极大型海藻表面具有琼胶降解活性的可培养菌株进行筛选、分离纯化和分子鉴定, 以进一步拓展我们对南极大型海藻表面可培养琼胶降解菌多样性和群落结构的了解和认识。
1 材料与方法
1.1 样品采集
大型海藻样品为2017年12月—2018年2月, 中国第34次南极科学考察期间采集于南极长城站生物湾(SHENGWU COVE, 58°W, 62°S), 具体站位信息如图1所示, 图中红色圆点为大型海藻的采集地点。
此次在南极长城站共采集到6种大型海藻, 按顺序依次编号为DZ-01至DZ-06, 经鉴定分别为[9][10][11][12]、[13]和[14]。其中(DZ-01)(DZ-02)(DZ-03)和(DZ-04)为红藻,(DZ-05)和(DZ-06)为褐藻, 海藻样品图片见图2。
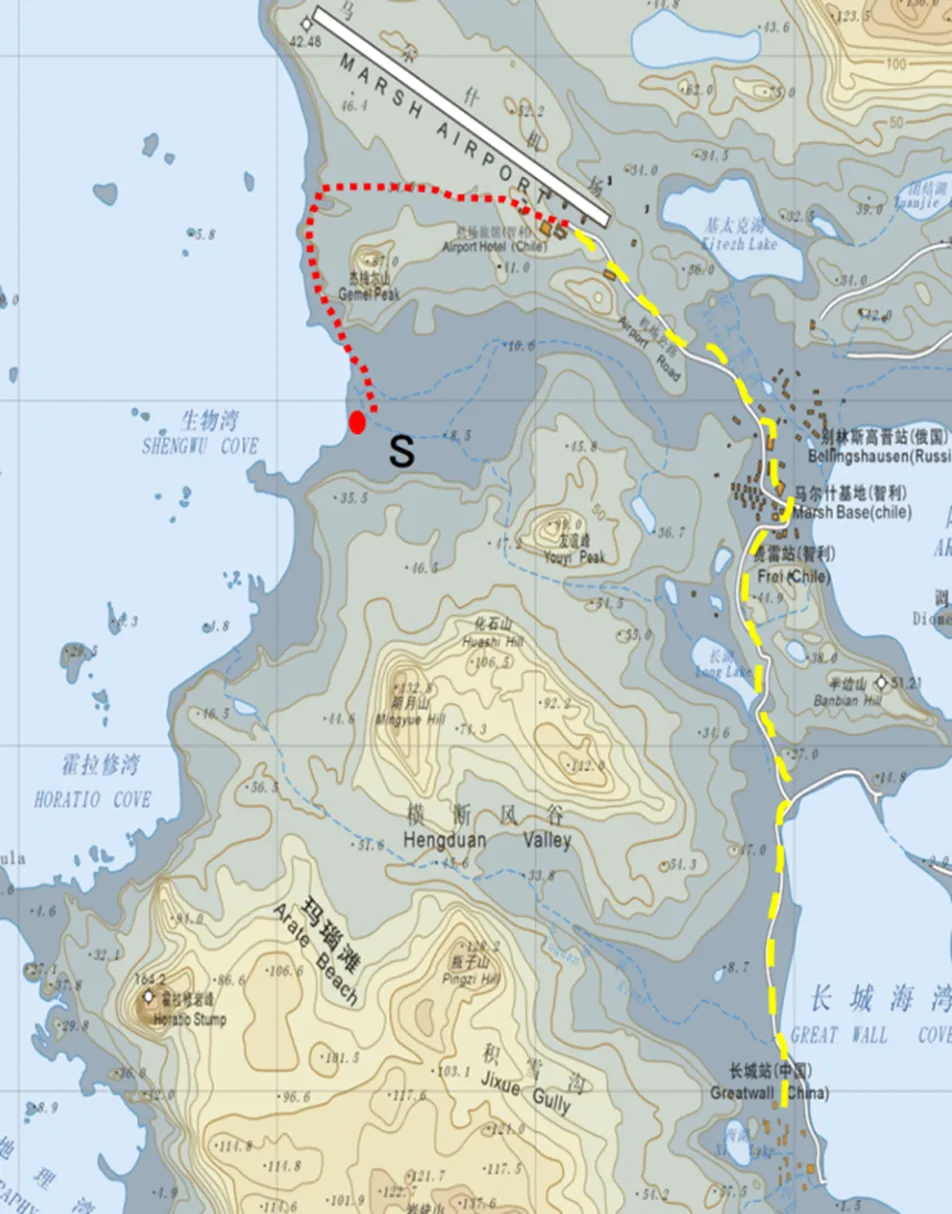
图1 样品采集站位图
Fig.1. Sample collection stations
1.2 样品处理及琼胶降解菌的分离纯化
1.2.1 培养基的配制
采用Zobell 2216E固体培养基对菌株进行分离和纯化; 制备以琼胶作为唯一碳源的固体选择培养基对大藻表面可培养的琼胶降解菌进行富集和筛选; 以海水Zobell 2216E液体培养基对纯化后的细菌进行培养, 各种培养基的配方如下:
(1)固体选择培养基:
胰蛋白胨1 g, 琼脂15 g, 过滤陈海水1 000 mL;
(2)Zobell 2216E液体培养基:
胰蛋白胨5 g, 酵母粉1 g, 陈海水1 000 mL;
(3)Zobell 2216E固体培养基
胰蛋白胨5 g, 酵母粉1 g, 琼脂15 g, 陈海水1 000 mL。

图2 南极大型海藻样品
Fig.2. Sample of macroalgae from Antarctica
各培养基均于1.21×105Pa高压蒸汽灭菌锅灭菌20 min。
1.2.2 卢戈氏染液的配制
卢戈氏染液配方: 碘(I2)5 g, 碘化钾(KI)20 g, 蒸馏水100 mL。
1.2.3 大藻样品处理
将采集的大型海藻样本分别放入无菌封口袋中, 运送到南极长城站实验室4℃冰箱保存。然后将样品带回“向阳红01”综合科考船后立即进行处理, 进行琼胶降解活性菌株的富集和筛选。
首先用孔径大小为0.45 µm滤膜过滤海水并进行灭菌处理, 然后用灭菌的海水冲洗海藻样品表面2~3次, 去除藻体表面吸附的细小杂质、碎屑及杂菌; 将冲洗后的藻体剪成小块(2 cm×2 cm)放入无菌试管, 加入灭菌海水并用涡旋震荡仪震荡1 min, 使藻体表面的附生菌悬于灭菌海水中, 收集细菌悬液备用。
1.2.4 琼胶降解活性菌株的筛选及分离纯化
用灭菌海水将收集的附生菌悬液进行梯度稀释(10−1、10−2、10−3、10−4), 取100 μL不同稀释梯度的菌液, 分别涂布于固体选择培养基, 于10℃倒置恒温培养, 每个样品做3个平行处理。
定期检查菌株生长状况, 直至肉眼可清晰地观察到菌落。对形态上有明显差异的菌落进行编号并进行划线纯化直至获得一种形态的单菌落[15]。同时对纯化菌株的培养基进行卢戈氏碘液染色, 菌落周围形成边缘清晰的水解透明圈即为疑似琼胶降解活性菌株, 纯化后的菌株置于–80℃甘油管中保存备用。
1.3 琼胶降解菌16S rRNA序列测定及系统发育分析
将纯化的菌株分别接种到Zobell 2216E液体培养基(5 mL试管), 于10℃振荡培养箱培养24 h, 取1 mL菌液离心、收集菌体提取DNA。采用细菌DNA提取试剂盒(天根)提取细菌总DNA用于16S rRNA扩增, PCR(Polymerase Chain Reaction)扩增[16-17]采用50 μL体系, 每50 μL PCR反应体系中包括1 µL正向引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)、1 µL反向引物1492R(5′-GGTTACCTTGTTACGACTT-3′)、25 µL Master Mix、2 µLDNA和21 µL超纯灭菌水。PCR扩增反应条件: 95℃预变性5 min, 95℃变性1 min, 55℃复性30 s, 72℃延伸90 s, 30个循环, 最后72℃延展10 min。PCR产物经琼脂糖凝胶核酸电泳检测后, 送到上海派森诺生物科技股份有限公司进行测序和拼接。
根据测序结果, 在NCBI运用BLAST与GenBank数据库已知的细菌16S rRNA基因序列进行相似性比对, 进行同源性分析[18]完成菌种鉴定, 菌株的16S rDNA序列已存储在GenBank数据库中。根据菌落形态差别从中选取具有代表性的21株菌(由于属于的菌株数量较多, 且相似度高达99%以上, 所以我们只选择了其中4株酶解透明圈较大的进行建树), 采用MEGA7.0进行系统发育分析和系统发育树的绘制。应用MEGA 7.0中的Clustal W对测得的序列以及基因库中的相似序列一起进行比对, 然后再用Neighbor-joining模式建树, 选择Bootstrap method, Bootstrap Replications设置为1 000。
2 结果与分析
2.1 菌株的分离纯化
根据菌落形态、颜色、大小等特征不同[19], 共从南极大型海藻样品表面分离纯化获得37株琼胶降解活性菌株, 部分具有代表性的菌株水解透明圈染色结果如图3所示。
2.2 基因序列测定及系统发育分析
2.2.1 基因序列分析
将菌株的16S rRNA测序结果与GenBank数据库进行BLAST对比, 结果表明, 筛选获得的37株琼胶降解菌分别属于γ-变形菌纲, 黄杆菌纲和放线菌纲。γ-变形菌纲包括:、、、、、等7个属及Thiotrichaceae科; 黄杆菌纲包括:、2个属; 放线菌纲仅有1个属。其中优势属为假交替单胞菌属(, 20株), 其次为嗜冷杆菌属(, 4株), 详细信息如表1所示。
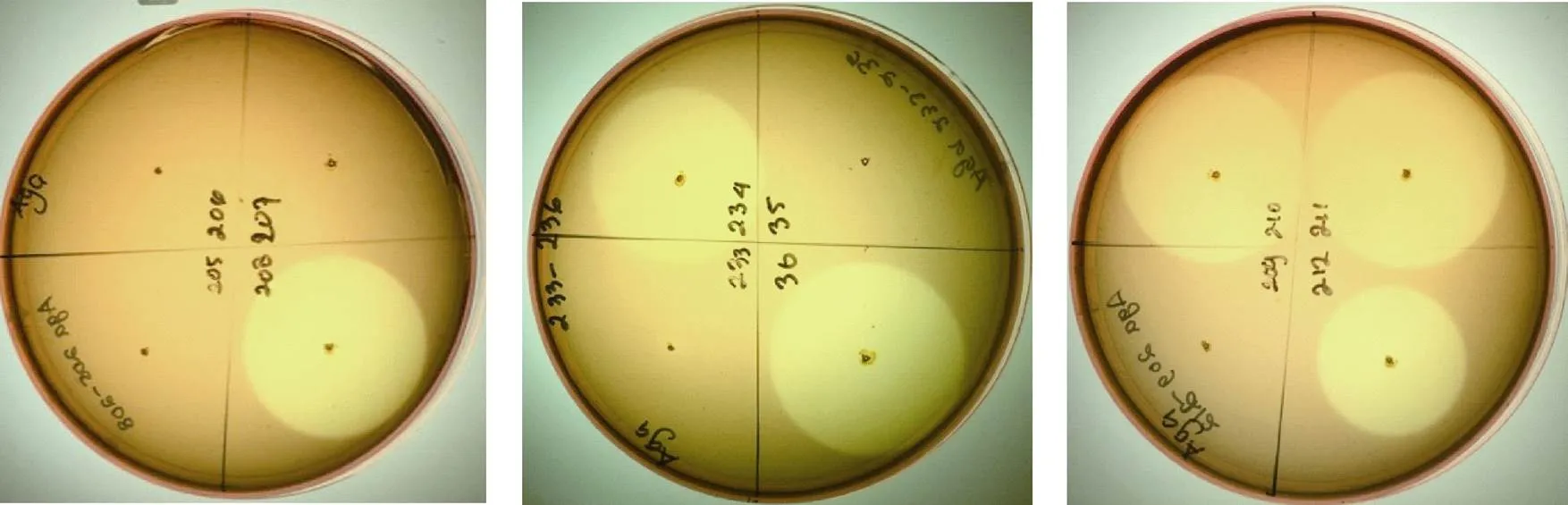
图3 代表性琼胶降解活性菌株的水解透明圈
Fig.3. The hydrolytic transparent circle of representative agar-degrading active strains
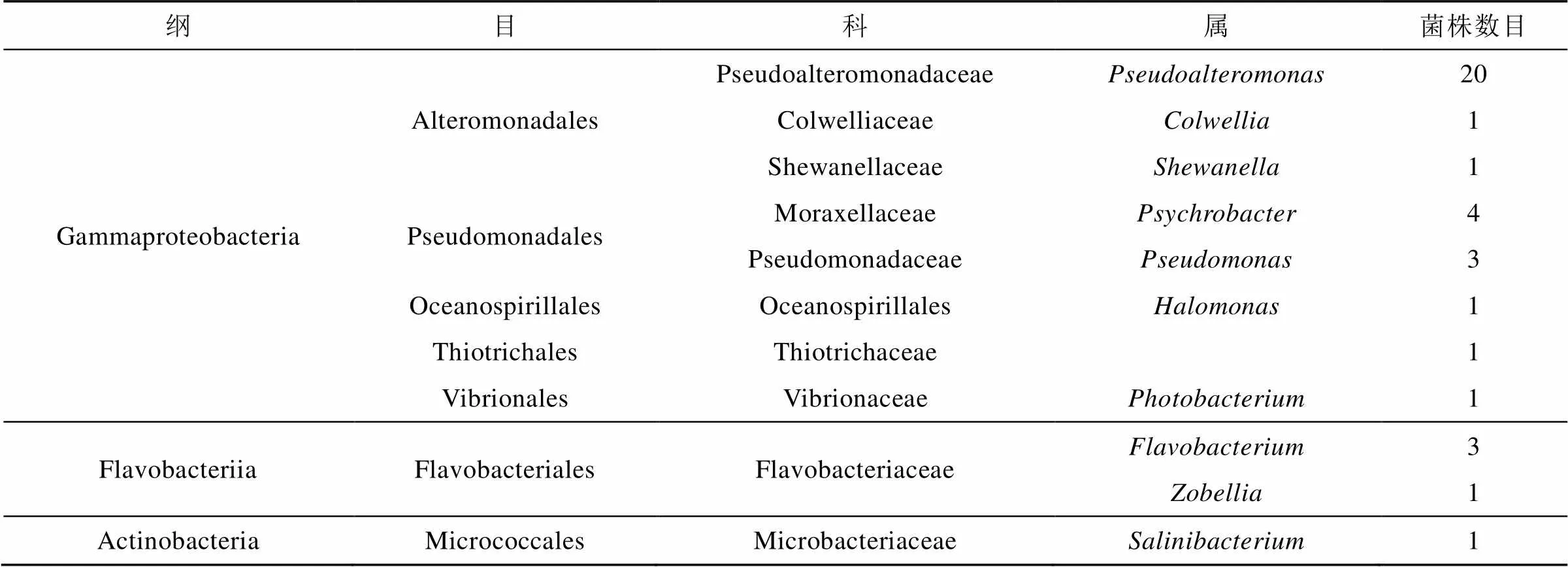
表1 可培育的琼胶降解菌分离鉴定情况一览表
2.2.2 系统发育分析
对筛选出的具有代表性的21株菌株构建系统发育树(图4), 菌株的16S rDNA序列已提交至GenBank数据库, 系统发育分析表明这些菌株的确分别属于γ-变形菌纲、黄杆菌纲和放线菌纲, 该结果与16S rRNA的比对分析结果一致。

图4 系统进化树. 系统进化树中粗体表示实验菌株, 括号内为菌株16S rDNA序列的GenBank数据库登录号
Fig.4. Phylogenetic tree. In the phylogenetic tree, the bolded text is the experimental strain, and the content in brackets is the GenBank number of the 16S rDNA sequence of the strain
3 讨论
本研究从南极乔治王岛附近海域大型海藻表面筛选获得了37株可培养的琼胶降解菌, 对其进行分子鉴定并对其中具有代表性的21株菌株进行系统发育树的构建, 结果表明这些可培养的琼胶降解菌分属于γ-变形菌纲、黄杆菌纲和放线菌纲3个纲。Tropeano等[7]从乔治王岛波特湾的海洋沉积物中分离到的189株可培养细菌分别属于α-变形菌纲、γ-变形菌纲、黄杆菌纲、放线菌纲及杆菌纲。从纲水平上, 本研究结果表明我们采集的大型海藻表面附生菌与其周边环境中微生物种属的多样性组成基本一致。
在属水平上, 我们的研究结果与南极地区已有的相关报道存在明显的差异, Alvarado和Leiva[8]在南极乔治王岛采集的4种南极大型海藻(、、、)表面分离出30株具有琼胶降解活性的菌株, 分别属于、、、、、、、、、和等11个属, 与本研究相比, 除了盐水杆菌属()和卓贝尔氏菌属()外, 其他种属均不相同。Hinojosa等[20]在3种南极大型海藻(、和)表面分离出21株具有琼胶降解活性的菌株, 通过16S rRNA鉴定表明, 这些琼胶降解菌株分别属于、、、、、和等7个属, 与本研究相比, 只有科尔韦尔氏菌属()和假交替单胞菌属()是相同的种属。Lavín等[21]在南极乔治王岛采集的红藻中分离出具有琼胶降解活性的黄杆菌属()。综上可见, 本研究从南极大型海藻筛选获得的、、和等5个属及Thiotrichaceae科是之前未报道的具有琼胶降解活性的南极大型海藻表面附生菌。我们的研究结果与南极地区已有的相关报道存在明显差异的原因可能有2个: 一是用于菌株分离的大型海藻样品种属不同; 二是我们对采集的大型海藻立刻开展了琼胶降解菌的富集和筛选培养, 有利于获得更加丰富的活性菌株。这两个原因都会对菌株的多样性产生可能的影响。
但与在中低纬度近海区域海水、大型海藻表面分离的琼胶降解菌比较发现,和属的琼胶降解菌均有发现。Marjolaine等[22]在褐藻表面分离琼胶降解菌时分离到希瓦氏菌属(); 假单胞菌属()在自然界分布极其广泛, 虽然该属之前没有在南极大型海藻表面分离到的相关报道, 但在近海海水、红藻和褐藻中均分离到假单胞菌属, 且具有较高的琼胶降解活性[23-25]; Azhani等[26]在海藻样品分离提取的菌株中发现盐单胞菌属()具有琼胶降解活性。从目前已有报道看, 琼胶降解细菌主要分布于弧菌属()、假单胞菌属()、假交替单胞菌属()、交替单胞菌属()以及噬琼胶菌属()等属[23]。杜宗军等[23]对青岛近海海水中的琼胶降解细菌进行筛选分离, 得到87株能够降解琼胶的细菌, 其中14株细菌分布在、、、、、、和共8个属中。但本研究获得的可培养琼胶降解菌种属与青岛近海的琼胶降解菌株多样性也存在较大差异, 其中仅有假交替单胞菌属()和假单胞菌属()在南极大藻表面及近海环境中均有分布, 由此可见, 假交替单胞菌属和假单胞菌属的菌株具有很强的适应性, 在全球不同海洋环境中都有广泛的分布, 与该结果一致的是, 假交替单胞菌属()也是我们此次从南极大藻表面筛选获得琼胶降解菌的优势菌株。
本次研究从红藻中分离到的琼胶降解菌株数分别为12株、8株、7株、3株, 褐藻和中分离到的琼胶降解菌株数分别为3株、4株。从4种红藻中分离出的30株琼胶降解菌分别属于(15株)、(1株)、(1株)、(3株)、(3株)(1株)、Thiotrichaceae(1株)、(1株)(2株)、(1株)、(1株)从2种褐藻中分离出的7株琼胶降解菌分别属于(5株)、(1株)和(1株)。之所以红藻表面分离出的琼胶降解菌株比褐藻的数目多且丰富度高, 主要是由于红藻富含琼胶和卡拉胶等成分, 而褐藻中主要成分为褐藻胶。
南极海域分布着丰富的大型海藻及微生物资源, 对南极大型海藻表面琼胶降解菌的筛选和鉴定有助于我们加深对南极海域海洋细菌多样性的理解和认识, 也为南极微生物资源调查和开发研究提供科学依据。
1 林学政, 边际, 何培青. 极地微生物低温适应性的分子机制[J]. 极地研究, 2003, 15(1): 75-82.
2 张锐, 林念炜, 赵晶, 等. 南极阿德雷岛地表沉积物中细菌多样性及对环境的响应[J]. 自然科学进展, 2003, 13(10): 1067-1072. DOI: 10.3321/j.issn: 1002-008X.2003.10.010.
3 MARTIN M, PORTETELLE D, MICHEL G, et al. Microorganisms living on macroalgae: diversity, interactions, and biotechnological applications[J]. Applied Microbiology and Biotechnology, 2014, 98(7): 2917-2935. DOI: 10.1007/s00253-014-5557-2.
4 张恒曦. 主要海藻多糖降解酶活性架构及其降解模式分析[D]. 济南: 山东大学, 2017.
5 CUNDELL A M, SLEETER T D, MITCHELL R. Microbial populations associated with the surface of the brown alga Ascophyllum nodosum[J]. Microbial Ecology, 1977, 4(1): 81-91. DOI: 10.1007/bf02010431.
6 CHI W J, CHANG Y K, HONG S K. Agar degradation by microorganisms and agar-degrading enzymes[J]. Applied Microbiology & Biotechnology, 2012, 94(4): 917-930. DOI: 10.1007/s00253-012-4023-2.
7 TROPEANO M, CORIA S, TURJANSKI A, et al. Culturable heterotrophic bacteria from Potter Cove, Antarctica, and their hydrolytic enzymes production[J]. Polar Research, 2012, 31(1): 18507. DOI: 10.3402/polar.v31i0.18507.
8 ALVARADO R, LEIVA S. Agar-degrading bacteria isolated from Antarctic macroalgae[J]. Folia Microbiologica. 2017, 62(5): 409-416. DOI: 10.1007/s12223-017-0511-1.
9 YARISH C, KIRKMAN H, LÜNING K. Lethal exposure times and preconditioning to upper temperature limits of some temperate North Atlantic red algae[J]. Helgoländer Meeresuntersuchungen, 1987, 41(3): 323-327.
10 ABREU P M, GALINDRO J M. A new polyhalogenated epoxymonoterpene from Plocamium cartilagineum[J].Indian Journal of Chemistry, Section B, 1998, 37B(6): 610-611.
11 GALLARDO T, PÉREZ-RUZAFA I M, FLORES-MOYA A, et al. New collections of benthic marine algae from Livingston and deception Islands (south Shetland Islands) and trinity island (Bransfield Strait) Antarctica[J]. Botanica Marina, 1999, 42(1): 61-69. DOI: 10.1515/bot.1999.009.
12 MOE R L, SILVA P C. Desmarestia antarctica (Desmarestiales, Phaeophyceae), a new ligulate Antarctic species with an endophytic gametophyte[J]. Plant Systematics and Evolution, 1989, 164(1): 273-283.
13 IKEN K, AMSLER C D, AMSLER M O, et al. Field studies on deterrent properties of phlorotannins in Antarctic brown algae[J]. Botanica Marina, 2009, 52(6): 547-557. DOI: 10.1515/BOT.2009.071.
14 FURNEAUX R H, MILLER I J, STEVENSON T T. Agaroids from New Zealand members of the Gracilariaceae (Gracilariales, Rhodophyta): a novel dimethylated agar[J]. Hydrobiologia, 1990, 204/205(1): 645-654. DOI: 10.1007/BF00040300.
15 WANG M P, CHEN L, ZHANG Z J, et al. Screening of alginate lyase-excreting microorganisms from the surface of brown algae[J]. AMB Express, 2017, 7(1): 74. DOI: 10.1186/s13568-017-0361-x.
16 林学政, 陈靠山, 何培青, 等. 种植盐地碱蓬改良滨海盐渍土对土壤微生物区系的影响[J]. 生态学报, 2005, 26(3): 801-807. DOI: 10.3321/j.issn: 1000-0933.2006.03.023.
17 刘杰, 王晓姗, 王能飞, 等. 青岛近海浒苔粘附着细菌16S rDNA系统发育学研究[J]. 科学技术与工程, 2009, 9(8): 2042-2046, 2055.
18 苗婷婷, 邢翔, 杜宗军, 等. 柄海鞘共附生细菌的分离培养与系统发育多样性研究[J]. 海洋科学进展, 2012, 30(1): 111-118.
19 杨晓, 丁慧, 臧家业, 等. 南极菲尔德斯半岛土壤可培养细菌多样性分析[J]. 极地研究, 2016, 28(1): 34-41.
20 HINOJOSA V S, ASENJO J, LEIVA S. Agarolytic culturable bacteria associated with three antarctic subtidal macroalgae[J]. World Journal of Microbiology and Biotechnology, 2018, 34(6): 73.
21 LAVÍN P, ATALA C, GALLARDO-CERDA J, et al. Isolation and characterization of an Antarctic Flavobacterium strain with agarase and alginate lyase activities[J]. Polish Polar Research, 2016, 37(3): 403-419.
22 MARJOLAINE M, TRISTAN B, RENEE M, et al. The cultivable surface microbiota of the brown alga ascophyllum nodosum is enriched in macroalgal-polysaccharide-degrading bacteria[J]. Frontiers in Microbiology, 2015, 6. DOI: 10.3389/fmicb.2015.01487.
23 杜宗军, 赵苑, 李美菊, 等. 青岛近海琼胶降解细菌的筛选和多样性分析[J]. 中国海洋大学学报(自然科学版), 2007, 37(2): 277-282.
24 朱大玲, 唐啸龙, 张宝玉, 等. 一株海藻多糖降解菌的分离鉴定及产酶条件优化[J]. 海洋科学, 2017,41(8): 99-107.
25 欧昌荣, 汤海青, 管斌. 琼胶酶生产菌的筛选、鉴定及其酶学性质的初步研究[J]. 食品科学, 2005, 26(6): 86-90.
26 AZHANI A, NURSYUHAIDA M H, NAZIR B M, et al. Evaluation of disease resistance and tolerance to elevated temperature stress of the selected tissue-cultured Kappaphycus alvarezii Doty 1985 under optimized laboratory conditions[J]. Biotech, 2018, 8(8): 321. DOI: 10.1007/s13205-018-1354-4.
BIODIVERSITY ANALYSIS OF CULTURED AGAR-DEGRADING BACTERIA FROM SURFACES OF ANTARCTIC MACROALGAE
Gui Yuanyuan1, Gu Xiaoqian2, Li Jiang2, Zhang Peiyu1
(1College of Environmental Science and Engineering, Qingdao University, Qingdao 266071, China;2Marine Bioresource and Environment Research Center, First Institute of Oceanography, Ministry of Natural Resources, Qingdao 266061, China)
To analyze the diversity of cultured agar-degrading bacteria from the surfaces of six Antarctic macroalgae samples collected from King George Island, a selective medium and Lugol’s iodine staining method were used to screen and purify the agar-degrading bacteria, and the phylogenetic relationships of these strains were then investigated according to their 16S rRNA sequences. Thirty-seven strains of agar-degrading bacteria were screened, purified, and identified as belonging to the Gammaproteobacteria, Flavobacteriia, and Actinobacteria, respectively. At the genus level, the strains were classified as,,,,,,,,, and, in addition to a strain that was classified as belonging to the family Thiotrichaceae. Among them,andbelong to the Flavobacteriia,belongs to the Actinobacteria, and the remaining genera belong to the Gammaproteobacteria. The most dominant genus of agar-degrading bacteria was(20/37), followed by(4/37).,,,,, and the member of the family Thiotrichaceaeare the first reported taxa with agar-degrading activity isolated from the surfaces of macroalgae in Antarctica. The results not only expand our knowledge of the diversity of agar-degrading bacteria, but also contribute to the investigation and exploitation of Antarctic microbial resources.
Antarctica, agar-degrading bacteria, diversity, phylogenetic analysis
2019年12月收到来稿, 2020年3月收到修改稿
南极重点海域对气候变化的响应和影响(RFSOCC2020—2022)及国家重点研发计划项目(2018YFC1406704)资助
桂媛媛, 女, 1996年生。从事极地微生物研究。E-mail: 1037254782@qq.com
张培玉, E-mail: envbio@163.com
10. 13679/j.jdyj.20190076

