环状RNA hsa_circ_0003568通过miR-140-3p及miR-502-5p调控靶基因CDK6及NFYA在骨肉瘤中的作用
李康路,黄汉记,严国华,谭曼利,黄大计,朱博
(1. 广西医科大学,广西 南宁 530021;2. 广西百色市人民医院,右江民族医学院附属西南医院,广西 百色 533000)
骨肉瘤(Osteosarcoma,OS)是最常见的原发性恶性骨肿瘤之一,多见于儿童、青少年,具有高侵袭性、高转移性、进展迅速和化疗耐药等特点[1 ]。近年来,多药化疗加上手术切除可发现的病灶,已使局限性OS患者的长期生存率提高到65%~70%[2]。然而,目前由于早期诊断不足和缺乏有效的治疗方法,OS患者的5年生存率和预后仍未明显改善,尤其是对多药耐药、复发或肺转移的患者[3]。因此,迫切需要识别新的临床相关预后生物标志物,并探索肿瘤发生的潜在分子机制,以制定更有效的策略来治疗OS。环状RNA(circRNAs)是一种高度稳定、保守的特殊RNA,具有共价闭环特性,不易被内切酶降解,广泛存在于各种组织和器官中[4]。越来越多的研究表明,circRNAs以多种方式广泛参与疾病的发生发展,在癌症中具有重要的作用[5],它们通过靶向许多不同基因或信号通路,并将相关基因紧密连接到调控网络中从而影响肿瘤细胞的增殖、转移、分化和其他生物学过程[6]。然而,在OS中,circRNAs的潜在分子机制尚不清楚。在本研究中,我们主要探讨了环状RNA hsa_circ_0003568在OS中的表达并进行生物信息学研究,以明确其表达和临床意义,为进行OS分子标志物研究提供实验依据,为临床对OS的诊断和治疗提供新的视角。
1 材料与方法
1.1 材料与设备 正常成骨细胞hFOB1.19、OS细胞系SJSA-1和HOS购买于深圳市豪地华拓生物科技有限公司(中国,深圳);胎牛血清购自杭州天杭生物公司;DMEM/F12培养基购自ThermoFisher Scientific公司(美国,马萨诸塞州);TRIzol试剂盒购自Invitrogen 公司(美国,加利福尼亚州);cDNA反转录试剂盒、SYBR green PCR mix购自TAKARA生物技术公司(日本,东京);实时定量 PCR仪(LightCycler 9600)购自罗氏公司 (瑞士,巴塞尔)。
1.2 方法
1.2.1 细胞培养 hFOB1.19细胞用含10%胎牛血清的DMEM/F12 培养基,加入G418(0.3 mg/ml)以及青霉素(100 U/ml)和链霉素(100 U/ml),SJSA-1和HOS细胞系用含10%胎牛血清的DMEM/F12培养基,加入青霉素(100 U/ml)和链霉素(100 U/ml),于5%CO2、37℃恒温培养箱中培养。每隔3 d换新鲜的完全培养基,细胞密度达90%左右时按1∶3比例传代。
1.2.2 RNA 提取和RT-qPCR 采用TRIzol法提取细胞总RNA并利用反转录试剂盒合成cDNA,采用SYBR green PCR mix及hsa_circ_0003568的divergent引物进行RT-qPCR,反应条件如下:预变性95℃ 10 min,变性 95℃ 15 s,退火 60℃ 30 s,45个循环。GAPDH作为内参,采用2-△△Ct法计算hsa_circ_0003568相对表达量。同时,对PCR产物进行Sanger测序分析鉴定,测序由上海生工公司(Sangon Biotech (Shanghai) Co.,Ltd.)完成,引物见表1。

表1 RT-qPCR引物序列
1.2.3 生物信息学分析 通过circRNA的靶miRNA预测工具Circular RNA Interactome(https://circinteractome.nia.nih.gov/index.html)、CircBank Database(http://www.circbank.cn/index.html)和RegRNA2.0(http://regrna2.mbc.nctu.edu.tw/index.html)预测与hsa_circ_0003568相互作用的miRNA,并将其进行交集取3个预测工具同时靶向的miRNA,采用miRNA靶基因预测工具miRDB(http://mirdb.org/)、DIANA TOOLS(http://diana.imis.athena-innovation.gr/DianaTools/index.php?r=microT_CDS/index)、TargetScan(http://www.targetscan.org/vert_71/)和RegRNA2.0(http://regrna2.mbc.nctu.edu.tw/index.html)对交集到的miRNA进行靶基因预测,并将其进行交集取4个预测工具同时预测到的靶基因。通过GEPIA(http://gepia.cancer-pku.cn/index.html)对其交集的靶基因进行生存分析筛选生存预后显著差异的靶基因(P<0.05)。通过筛选的circRNA-miRNA和miRNA-mRNA利用Cytoscape构建circRNA-miRNA-mRNA可视化调控网络。
1.3 统计学方法 采用GraphPad Prism 8对实验数据进行统计学分析,统计检验数据符合正态分布。两组间比较采用t检验,以P<0.05为差异具有统计学意义。
2 结果
2.1 hsa_circ_0003568在OS细胞中的表达 我们通过RT-qPCR在人OS细胞系(SJSA-1及HOS)和人成骨细胞系(hFOB1.19)中检测了hsa_circ_0003568的表达。结果显示,与成骨细胞相比,在人OS细胞系中hsa_circ_0003568的表达显著上调(P<0.05),见图1A,并通过sanger测序验证了hsa_circ_0003568成环的连接点为IST1前体RNA的第6和第7外显子相接而成,见图1B。
2.2 hsa_circ_0003568靶向miRNA的预测 为探索hsa_circ_0003568的高表达在OS中的作用机制,我们通过miRNA预测工具对hsa_circ_0003568靶向的miRNA进行预测。结果显示CircInteractome数据库预测到9个miRNA,CircBank预测到11个miRNA和RegRNA2.0预测到20个miRNA。对其预测的miRNA进行交集,发现3个预测工具同时预测到的2个miRNA为miR-140-3p和miR-502-5p,见图2。
2.3 miRNA靶向基因的预测及分析 研究已经证明miRNA主要是通过调控其靶基因而发挥生物学功能[7]。因此,我们通过miRNA靶基因预测工具分别对miR-140-3p和miR-502-5p进行潜在的靶基因预测。对miR-140-3p靶基因的预测结果显示,miRDB预测到697个靶基因,DIANA预测到881个靶基因,TargetScan预测到495个靶基因和RegRNA2.0预测到228个靶基因。进一步对其预测的靶基因进行交集,发现4个预测工具同时预测到7个潜在的靶基因(ACVR2B、NFYA、CDK6、VKORC1L1、NRIP1、DAZAP2、KIF5A),见图3。对miR-502-5p靶基因的预测结果显示,miRDB预测到457个靶基因,DIANA预测到782个靶基因,TargetScan预测到4120个靶基因和RegRNA2.0预测到74个靶基因。进一步对其预测的靶基因进行交集,发现4个预测工具同时预测到6个潜在的靶基因(ODF4、RAB1B、HOXC8、TNRC6C、NFYA、B3GALT5),见图4。进一步分析发现NFYA基因同时是miR-140-3p和miR-502-5p的潜在靶基因。

注:A:RT-qPCR的结果,*:P<0.05,**:P<0.01;B:has_circ_0003568形成示意图及其PCR产物测序结果。
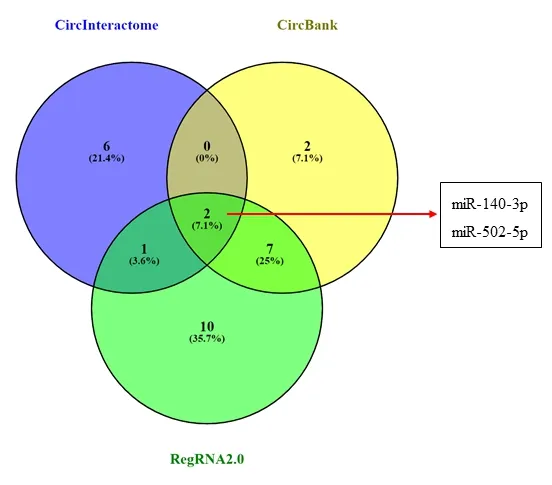
图2 has_circ_0003568靶向的miRNA:CircInteractome、CircBank和RegRNA2.0数据库预测的has_circ_0003568靶miRNA重叠的示意图

图3 miR-140-3p靶向的mRNA:DIANA、TargetScan、miRDB和miRTarBase数据库预测的miR-140-3p靶基因重叠的示意图

图4 miR-502-5p靶向的mRNA:DIANA、TargetScan、miRDB和miRTarBase数据库预测的miR-502-5p靶基因重叠的示意图
2.4 生存分析 通过采用GEPIA数据库对miR-140-3p和miR-502-5p分别交集到的7个和6个靶基因进行生存分析(见表2和表3),结果发现,除了CDF4无生存曲线记录外,其余基因都有相关生存曲线记录。更重要的是,我们发现CDK6和NFYA基因与OS患者的生存预后显著相关,与低表达CDK6的OS病人相比,高表达CDK6的OS病人展现了更差的预后(P<0.001),见图5A;同样,与低表达NFYA的OS病人相比,高表达NFYA的病人生存率更低(P<0.05),见图5B。

表2 miR-140-3p靶基因交集的7个mRNA的生存分析数据
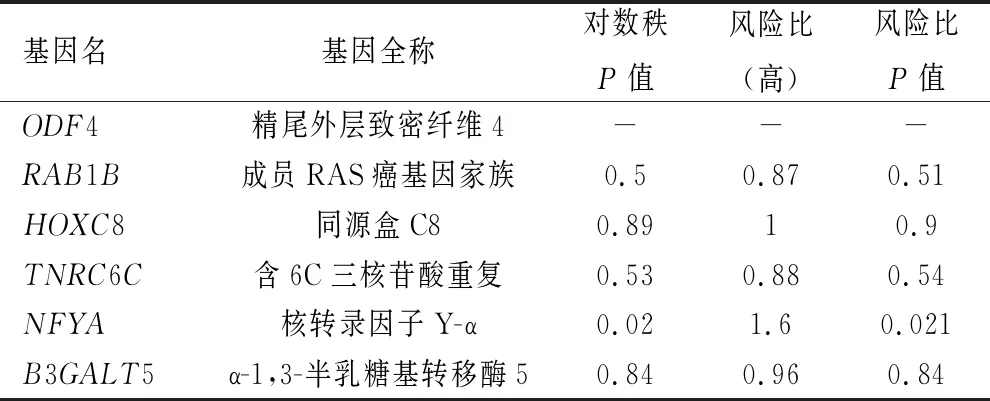
表3 miR-502-5p靶基因交集的6个mRNA的生存分析数据

A:OS标本中CDK6基因高表达组较低表达组预后较差(P<0.001);B:OS标本中NFYA基因高表达组较低表达组预后较差(P<0.05)。
2.5 构建circRNA-miRNA-mRNA网络 我们使用环状RNA hsa_circ_0003568,及其靶向的2个miRNA和13个靶基因用Cytoscape构建了circRNA-miRNA-mRNA可视化网络,见图6A。同时,根据与生存预后显著相关的mRNA,构建了hsa_circ_0003568-miR-140-3p/miR-502-5p-CDK6/NFYA可视化调控亚网络,见图6B。

A:OS中hsa_circ_0003568-miRNA-mRNA相互作用的ceRNA网络。红色代表circRNA,蓝色代表miRNA,黄色代表mRNA,粉色代表CDK6和绿色代表NFYA;B:基于有生存预后潜力的mRNA构建hsa_circ_0003568-miRNA-mRNA亚网络。红色代表circRNA,蓝色代表miRNA,黄色代表mRNA。
3 讨论
OS是一种常见的骨恶性肿瘤,如何治疗OS仍然是困扰人类健康的一大难题。目前OS的治疗主要是手术和化疗,但患者预后仍不理想。尽管先前的研究[8]已经表明许多基因与OS的发生和发展有关,然而,OS的分子机制尚不清楚,阐明OS发展的分子机制仍是当务之急。近年来一些非编码RNA被证实在OS的发病机制中发挥重要作用。circRNAs代表了一类新的保守内源性RNA,通过发挥miRNAs海绵作用调节下游靶基因的表达实现生物学功能[9]。与其他非编码RNA如miRNAs和长非编码RNA (long noncoding RNAs,lncRNAs)相比,circRNAs在哺乳动物细胞中具有高度保守性和高度稳定性[10-11]。这些特性使circRNAs有可能成为理想的生物标记物和潜在的治疗靶点。ceRNA假说描述了转录组中不同类型的编码成员和非编码成员如何通过miRNAs相互通信,竞争与miRNAs结合,然后调控彼此间的表达,从而构建复杂的转录后调控网络,有利于深入了解circRNAs在OS进展中的分子机制[12]。目前,虽然越来越多的研究人员已经开始研究circRNAs的潜在功能,但它们的临床价值在很大程度上仍不为人所知。在本研究中,发现环状RNA hsa_circ_0003568在OS细胞系中高度表达,并通过生物信息学分析预测miR-140-3p和miR-502-5p是hsa_circ_0003568的靶向miRNAs。研究发现miR-140-3p在结肠直肠癌中起到肿瘤抑制的作用,在体内过表达miR-140-3p可抑制结肠直肠癌肿瘤生长[13]。此外,miR-140-3p也被证明是肺癌的抑癌基因[14],在肝癌中可以增强肝癌细胞对索拉非尼的敏感性,抑制肝癌上皮-间质转化、侵袭和转移[15]。研究还发现miR-140-3p通过直接调控三联体基序28的表达来抑制乳腺癌的增殖和迁移[16]。由此说明,miR-140-3p在许多癌症中扮演着抑癌作用。同时,越来越多的研究表明miR-502-5p也是一种肿瘤抑制因子,可以增强细胞凋亡、抑制肿瘤细胞增殖,在许多癌症中发挥重要的抑癌作用,包括乳腺癌、结肠癌、膀胱癌、胃癌、肝癌和非霍奇金淋巴瘤等[17-22]。因此,hsa_circ_0003568在OS中可能通过抑制miR-140-3p和miR-502-5p的表达加速了OS的生长。
本研究还进行了生物信息学分析预测了miR-140-3p和miR-502-5p的潜在靶点。结果表明CDK6及NFYA是miR-140-3p的潜在直接靶点,同时,我们还发现CDK6及NFYA在OS患者的生存分析中具有显著的预后差异。CDK6在细胞周期进程中至关重要,抑制CDK6会导致肿瘤细胞不受控制的增殖,其途径是导致不依赖正常细胞外信号的增殖,或超越确保基因组完整性和稳定性的检查点[23]。CDK6在细胞进展中的作用已经在多种癌症中被发现,包括胶质母细胞瘤、黏液纤维肉瘤和淋巴恶性肿瘤[24-25]。最近的多项研究表明,NFYA在多种癌症中的表达增加与不良预后相关,包括肝癌、前列腺癌、结肠直肠癌等[26-28]。此外,研究发现增加NFYA的表达可促进OS细胞的恶性表型[29]。有趣的是,NFYA同时也是miR-502-5p的潜在靶点,由此暗示hsa_circ_0003568可能通过海绵抑制miR-140-3p和miR-502-5p调控CDK6和NFYA的表达从而参与OS的发生发展过程。
综上所述,本研究验证了hsa_circ_0003568在OS中高表达以及基于生物信息学预测构建了hsa_circ_0003568-miR-140-3p/miR-502-5p-CDK6/NFYA的可视化调控网络,提示hsa_circ_0003568可能是OS潜在的治疗目标和预后的生物标志物,为将来探索其在OS预后判断中的作用提供了新的途径。

