基于高光谱技术的菌落图像分割与计数
李艳肖,胡雪桃,张 芳,石吉勇※,邱白晶
基于高光谱技术的菌落图像分割与计数
李艳肖1,胡雪桃1,张 芳2,石吉勇2※,邱白晶1
(1. 江苏大学农业装备工程学院,镇江 212013;2. 江苏大学食品与生物工程学院,镇江 212013)
在平板菌落计数过程中,菌落与背景区域类似的颜色会干扰菌落的准确计数。为了准确测定细菌数,该研究利用高光谱图像技术捕捉成分差异引起的菌落与背景区域光谱特征,并结合化学计量学方法对平板的菌落进行分割并实现计数。采集枯草芽孢杆菌菌落平板的高光谱图像,提取菌落、背景区域的高光谱信息;利用遗传算法结合最小二乘支持向量机建立菌落区域/背景区域判别模型;随后,将菌落平板高光谱图像中每一个像素点对应的光谱信息代入判别模型以判断属于菌落的区域,模型的识别率为97.22%;最后,利用特征波段下的高光谱图像实现菌落的分割及计数,计数平均相对误差值为4.2 %,用时约为10 min。相比较于计算机视觉计数法,菌落计数法的平均相对误差降低了49.4%,结果表明建立的方法有望成为一类新的准确平板菌落计数方法。
图像分割;遗传算法;最小二乘支持向量机;高光谱图像技术;平板菌落计数;化学计量学;枯草芽孢杆菌
0 引 言
平板菌落计数法可测定食品、农产品的活体微生物数量,是有效评价食品微生物学安全性的国标方法[1]。该方法将食品样品制成包含活体微生物的稀释液并置于无菌的琼脂培养基上,每个活体微生物均可借助吸收琼脂中的营养物质不断繁殖形成菌落,依据菌落与背景(琼脂培养基)的颜色差异对菌落进行分割并实现计数[2-3]。
现有的平板菌落计数方式可分为人工视觉法和计算机视觉法两大类。人工视觉法利用肉眼(必要时借助放大镜)感知菌落与背景的颜色差异来分割、识别平板中的菌落,借助记号笔或菌落计数器实现平板中菌落的计数[4-5]。人工视觉法具有结果准确的优点,但是用眼强度大、菌落识别具有一定的主观性[6],难以实现大量样本的快速计数。计算机视觉法利用相机获取菌落与背景图像信息,利用图像处理算法捕捉菌落与背景的颜色差异特征,可自动分割平板中的菌落并实现计数[7-8]。在计算机视觉法基础上发展了全自动菌落计数仪,可实现菌落的自动计数[9]。建立的检测方法具有客观、速度快的优势[10],但菌落与背景颜色接近时图像处理算法分割菌落的能力下降[11],导致菌落的计数结果出现误差。卫生检验检疫部门的实践操作表明,当菌落与背景区域的颜色非常接近时,不仅计算机视觉法难以有效分割菌落,甚至人工视觉也经常发生误判[12]。为了提高菌落计数的准确度,学者提出了琼脂培养基中添加菌落着色剂氯化三苯甲基四氮唑(Triphenyl Tetrazolium Chloride,TTC)的研究思路,研究结果表明TTC的加入可以增大菌落与背景区域的颜色差异[13-14],从而提高菌落分割的准确度。但相关研究已证实TTC会抑制微生物的生长[4],加入TTC与提高菌落计数准确度的初衷背道而驰。
本课题组研究发现引起菌落与背景产生颜色差异的根本原因在于二者的化学成分不一致[15],因此拟从表征菌落区域与背景区域的成分存在差异这一角度出发,借助高光谱图像数据包含像素点光谱信息的优势[16],利用高光谱图像技术捕捉菌落及背景区域因成分差异引起的光谱特征,并结合化学计量法对平板中的菌落进行分割及计数,期望得到一类新的平板菌落计数法,为食品、农产品的微生物准确检测提供切实有效的方法。
1 材料与方法
1.1 样品的制备
将保藏于冰箱的枯草芽孢杆菌()接种到LB(Luria-Bertani)液体培养基中,37℃恒温培养24 h,镜检无杂菌后,用无菌水以10-2、10-3、10-4梯度稀释涂布于LB固体培养基上[17],在37 ℃恒温培养箱培养24 h后取出立即进行高光谱图像采集和菌落计数。为保证试验数据的重现性,每个稀释度做3个重复。
LB液体培养基配方为:无菌水1 000 mL,蛋白胨10.0 g,酵母粉5.0 g,葡萄糖5.0 g,氯化钠10.0 g;LB固体培养基配方为:无菌水1 000 mL,蛋白胨10.0 g,酵母粉5.0 g,葡萄糖5.0 g,琼脂粉20.0 g,氯化钠10.0 g。2种培养基用5 mol/L的NaOH调pH值至7.0,在210 ℃高压下蒸汽灭菌21 min。
1.2 高光谱图像采集和光谱信息获取
高光谱图像采集系统如图1所示[18],主要由波长范围为400~1 000 nm、光谱分辨率为2.8 nm的图像光谱仪(V10E型,芬兰ImSpector公司)和CCD摄像头(Clara,英国Andor公司)、150 W光纤卤素灯(Fiber-Lter DC950 型,美国DolanJenner公司)、精密电控平移台(SC30021 A型,中国Zolix公司)和计算机等部件组成。高光谱图像数据采集之前,需打开系统预热30 min以上,预热后设置高光谱系统采集的相关参数如电控平台速度、相机曝光时间等;数据采集完成后,对采集到的光谱数据进行黑白校正;其中试验参数设定、采集步骤及校正具体步骤参照Zhu等人建立的方法[19]。
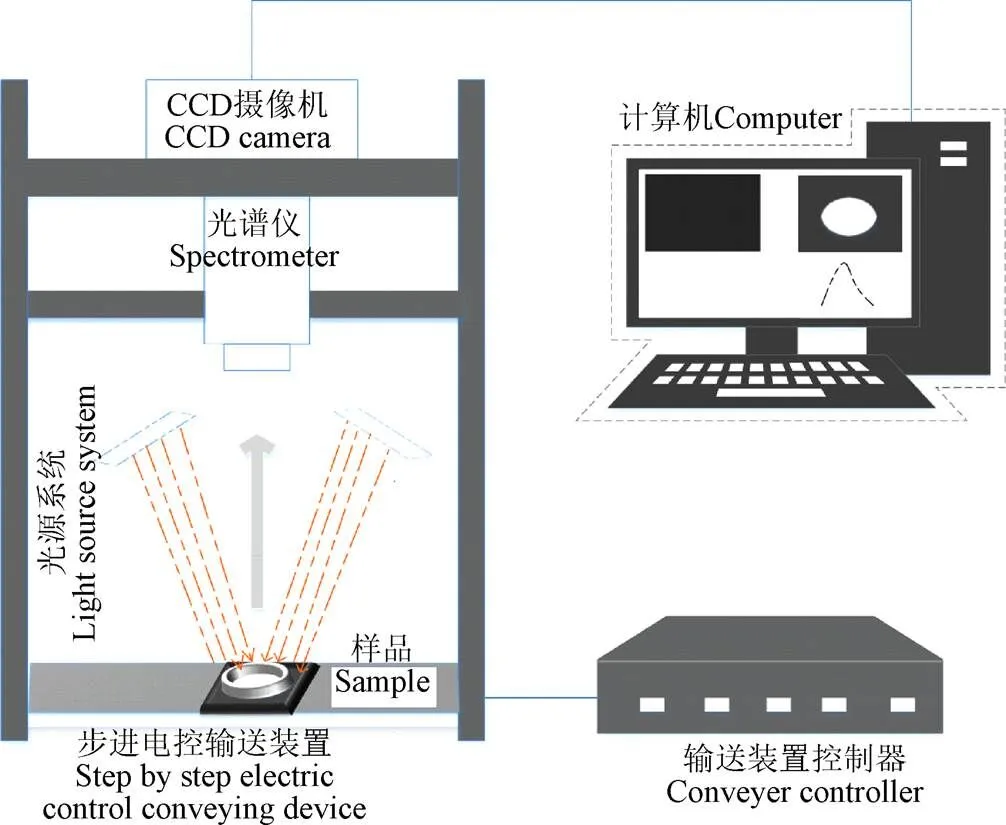
图1 高光谱图像采集系统
本文使用ENVI软件从36个菌落平板高光谱图像中提取36条菌落光谱、36条培养基光谱和36条培养皿边缘的高光谱信息。首先在每个平板菌落图像中随机选择5个菌落感兴趣区域(大小为1×1像素的正方形),然后提取每个感兴趣区域内光谱信息,最后将5个区域的光谱取平均值,得到对应的1条原始光谱,通过处理36个平板,共得到36条菌落光谱。同理,采集得到36条培养基光谱和36条培养皿边缘光谱,共采集108条原始光谱。每条光谱数据包含618个波长下光谱信息,最终得到大小为108×618的原始光谱数据集,其中,108为数据的个数,即样本数,618为每个数据包含的变量数。
1.3 菌落分割模型的建立
为了消除高光谱图像采集中高频随机噪声、基线漂移、样本表面粗糙和光散射等带来的不良影响,首先必须对培养基、培养皿边缘和菌落的原始光谱进行预处理。标准正态变量变换(Standard Normal Variate Transformation,SNV)预处理可以使分散的光谱曲线集中,有效实现对基线的校正,减少样本信号噪声,提高信号的分辨率和灵敏度[20-21]。
为了初步探索菌落与非菌落之间光谱的差异性,研究利用主成分分析(Principal Component Analysis,PCA)对光谱进行聚类分析。根据物质内部基团对光能的吸收具有选择性可知,并不是所有波长下的光谱都同物质内部成分高度相关,故需要对高光谱信息进行特征筛选。遗传算法(Genetic Algorithms,GA)通过选择、交换、突变等遗传算子,随着不断的遗传迭代,淘汰掉较差的变量,保留目标函数值较优的变量,最终筛选出特征光谱信息用于建模[22]。本文采用偏最小二乘法(Partial Least Squares,PLS)的交叉验证均方根误差(Root Mean Square Error Cross Validation,RMSECV)作为评价个体波长优势的依据,用来判断种群个体的优劣程度。
本文利用GA选择的特征变量建立GA-LS-SVM模型,用于菌落平板的菌落、培养基和培养皿边缘的识别。最小二乘支持向量机(Least Square Support Vector Machine,LS-SVM)可以将多维的特征输入映射到高维的核空间,使原来不可分的数据获得新的特征后进行分类,很好地解决非线性分类问题[19]。试验采集了108条光谱(培养基、培养皿边缘和菌落的样本光谱数都为36条),通过随机分组将72条光谱作为校正集,余下的36条作为预测集。模型的优劣以模型识别率为评价指标,模型识别率为菌落正确识别的概率,即正确识别的菌落数量/菌落总数。根据菌落区域和背景区域因成分差异引起的特征光谱建立其GA-LS-SVM判别模型,再利用该判别模型对高光谱图像中每一像素点对应的光谱信息进行判别,识别高光谱图像中菌落区域和背景区域。
1.4 菌落计数
平板菌落计数的主要步骤分为高光谱图像的获取、菌落的分割、粘连菌落的分割以及菌落数的计算。高光谱图像的获取参照1.2节。获取样品的高光谱图像后,将每个像素点的光谱信息代入菌落分割GA-LS-SVM模型中。当该像素点被识别为背景时,将该像素点的灰度值设为0,当该像素点被识别为菌落时,将像素点的灰度值设为1。经过上述步骤后,得到菌落分割二值图,实现待测培养平板高光谱图像中菌落区域的判别和分割。
采用灰度阈值分割方法对特征波段下的高光谱菌落割图像进行粘连菌落的分割,得到粘连菌落分割二值图,菌落区域的灰度值为1,非菌落区域的灰度值为0。结合菌落分割二值图和粘连菌落分割二值图,若2张图片的中像素点的灰度值都为1时,则该像素点的灰度值记为1,此时该像素点为菌落区域。若2张图中像素点的灰度值不全为1时,该像素点的灰度值记为0,此时该像素点为背景区域。由此得到了最终菌落计数图。
采用局部邻域算法中的轮廓跟踪算法对分割出的菌落最终计数图进行计数[18,23],对图像进行扫描将此次标号改为其八连通域内的最小标号,重复执行此过程到不需要做标号标记为止。最终实现对培养平板中的枯草芽孢杆菌菌落计数。
为了评价建立的计数方法的可重复性,对7个菌落平板进行了菌落计数。为了验证建立方法的准确性,利用人工视觉计数和计算机视觉计数验证[1]。
2 结果与分析
2.1 高光谱信息与图像信息的分析
高光谱图像技术采集到的光谱信息主要呈现的是物质内部化学基团合频和倍频吸收情况[24],而菌落和背景区域内部化学组分差异会引起光谱响应值的差异。枯草芽孢杆菌菌落、LB固体培养基和培养皿边缘的感兴趣区域及3种物质的平均光谱曲线如图2a所示,光谱曲线的波长范围为431~963 nm,3种物质的光谱曲线形状及反射率值都存在很大的差异。利用3种物质的特征光谱曲线,可以实现它们的准确区分。本文通过对菌落和背景区域的光谱信息进行主成分分析,来初步探索菌落和背景的光谱差异性。主成分分析可以在不降低光谱差异性的前提下,对光谱数据进行降维处理,将得到的主成分数据特征用散点图表示,每一散点代表1条光谱(图2b)[25]。由图2b的菌落与背景二维主成分分析图可以看出,利用第1主成分(the first Principal Component Analysis,PC1)和第2主成分(PC2)可以明显的区分菌落和背景区域,有良好的聚类趋势。PC1和PC2的贡献率分别为81.05%和4.16%。以上结果表明利用高光谱信息可以对菌落与背景进行定性分类。
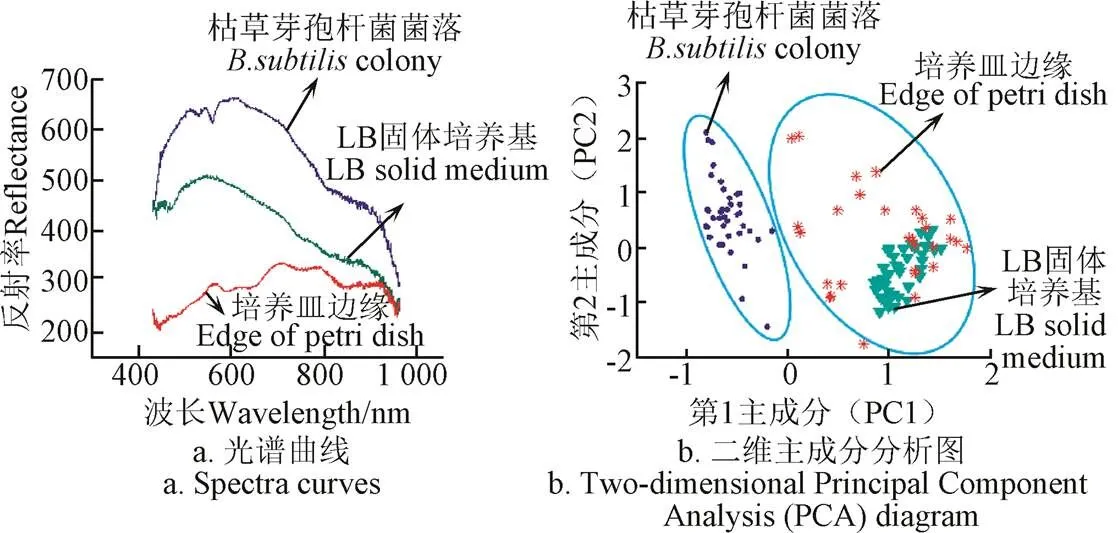
图2 原始光谱曲线及其主成分分析图
2.2 光谱的预处理
本试验首先采用SNV方法对校正集原始光谱(24条菌落区域光谱、48条背景区域的光谱)进行处理,在后续试验中,选用SNV预处理后的光谱数据进行分析和建模。在数据建模过程中,输入的变量越少,建立的模型越稳定性,鲁棒性越好。因此本试验采用GA进行特征波长筛选,以便简化变量信息,在不损失有用光谱信息的前提下,最大程度地减少变量数。按照GA的取值规律,设定控制参数种群大小为108、遗传迭代次数为200、染色体个数为50、基因交换概率为0.95、基因变异概率为0.01,来优化遗传算法。通过该优化的遗传算法,最终从全波段中筛选出能表征菌落和背景的11个特征波长,分别为604、636、790、799、748、683、492、437、558、470和928 nm。所筛选出的特征波长主要与枯草芽孢杆菌在生长繁殖过程分泌大量挥发性脂肪和胞外酶如蛋白酶、乙酸及丁酸等代谢物及培养基成分等中含氢基团C-H、O-H合频和倍频吸收有关。
2.3 菌落的分割
经GA特征波长筛选后,模型自变量由全波段光谱数据的618个降至11个。将菌落区域和背景区域对应11个特征波长下的光谱信息作为输入变量,光谱种类(菌落或背景区域)为输出变量,利用化学计量学方法GA-LS-SVM确立光谱信息和光谱种类的关系,得到菌落的判别模型GA-LS-SVM。LS-SVM建模过程中选择径向基函数(Radial Basis Function,RBF)为核函数,采用交叉验证法与网格搜索法对RBF核参数的参数即正则化参数和对核参数2参数进行寻优计算,寻优过程由粗选和精选2个步骤组成[26]:粗选格点数10×10,搜索步长较大;精选格点数仍为10×10,在粗选基础上,以较小步长更加细致地搜索,以定标集交互验证均方根误差(Root Mean Square Error of Cross Validation,RMSECV)最小为指标,确定最优的(,2)组合。本文设定10-2~102为参数的范围进行网格搜索。经处理得到模型最佳参数组合为2=78.48,=1 311.004,此时GA-LS-SVM模型预测集的正确识别率为97.22%(RMSECV最小值为0.488)。同时,利用全波长的光谱信息建立的LS-SVM模型预测集的正确识别率为94.44%。结果说明GA筛选出的11个特征波长包括菌落和背景区域的大部分光谱信息,可以利用该判别模型进行后续菌落的分割。
把菌落和背景区域在全变量下的高光谱图像(图3a)简化为在特征波长下的图像,逐一提取特征波长下待测培养平板中的高光谱图像中每一像素点的高光谱信息,代入上述已建的GA-LS-SVM判别模型,对菌落平板中的每一像素点进行判别。当像素点灰度值为1时,像素点处被判别为菌落,同时,当像素点灰度值为0时,表明该位置为背景区域,最终实现待测培养平板高光谱图像中菌落的判别和分割。由于高光谱图像技术采集图像信息时易受仪器及环境噪声的影响,使得分割后的菌落区域图像有少量杂散噪声点[6]。可根据先验知识,人机交互设定1个阈值,统计噪声点连通区域的面积,如面积小于设定的阈值,则认为是噪声点,不予计数[18]。经检测本试验菌落图像上噪声点像素面积小于6而菌落的像素点面积超过16,因此设定阈值为6可去除噪声点的干扰。得到菌落图像分割二值图(图3b)。图3b是利用菌落识别方法对平板的菌落进行了分割,此时只是将菌落从背景中分离出来,有些菌落之间存在明显的粘连,粘连菌落的存在往往导致计数结果小于实际的菌落数,利用此图无法对准确计算菌落数量。因此有必要对粘连的菌落进行分割并计数。
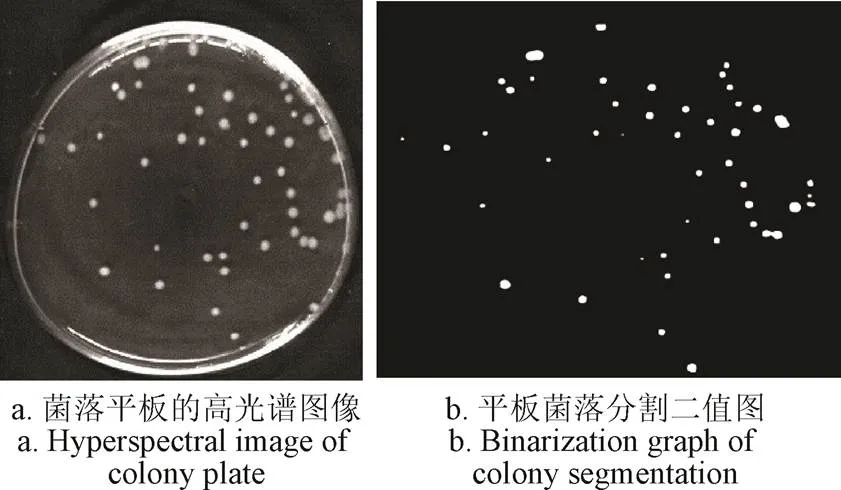
图3 高光谱图像菌落分割结果
2.4 粘连菌落的分割
针对特征波长下高光谱图像中粘连菌落不同部位的像素点灰度值存在较大差异的特点,本文提出采用灰度阈值分割方法实现粘连菌落的分割[26]。对贡献率最大的波长(604 nm)图像进行粘连菌落处理,来实现粘连菌落的分割,得到了粘连菌落分割二值图。具体的操作过程为:从枯草芽孢杆菌在604 nm下的高光谱图像的伪彩色图(图4a)可以看出,单一菌落颜色从中心到边缘依次呈红色、黄色、天蓝色以及蓝色且呈圆圈型变化。对图4a进行三维图形等高线处理,以像素点颜色灰度表示等高线的大小(图4b),并放大粘连菌落的三维图形(图4c)。根据颜色标尺,每个菌落中心点到边缘的灰度值由0.8逐渐减小为0.3。对于粘连菌落,粘连菌落有2个中心点,2个中心呈红色,由中心向边缘延伸时,粘连菌落还有黄色(灰度值约为0.5~0.6)和天蓝色(灰度值约为0.4~0.5)。图4c可观察到粘连菌落有2个明显的峰,顶部的颜色仍为红色,菌落粘连处不仅有黄色(灰度值高于0.5),还有天蓝色(灰度值低于0.5)。这说明该高光谱图像粘连菌落区域的颜色特征和非粘连区域的颜色特征不同,2个菌落并未完全合并。因此将菌落粘连处的天蓝色区域作为分割区域,将分割阈值设为0.5,分割后的粘连菌落如图4d所示。
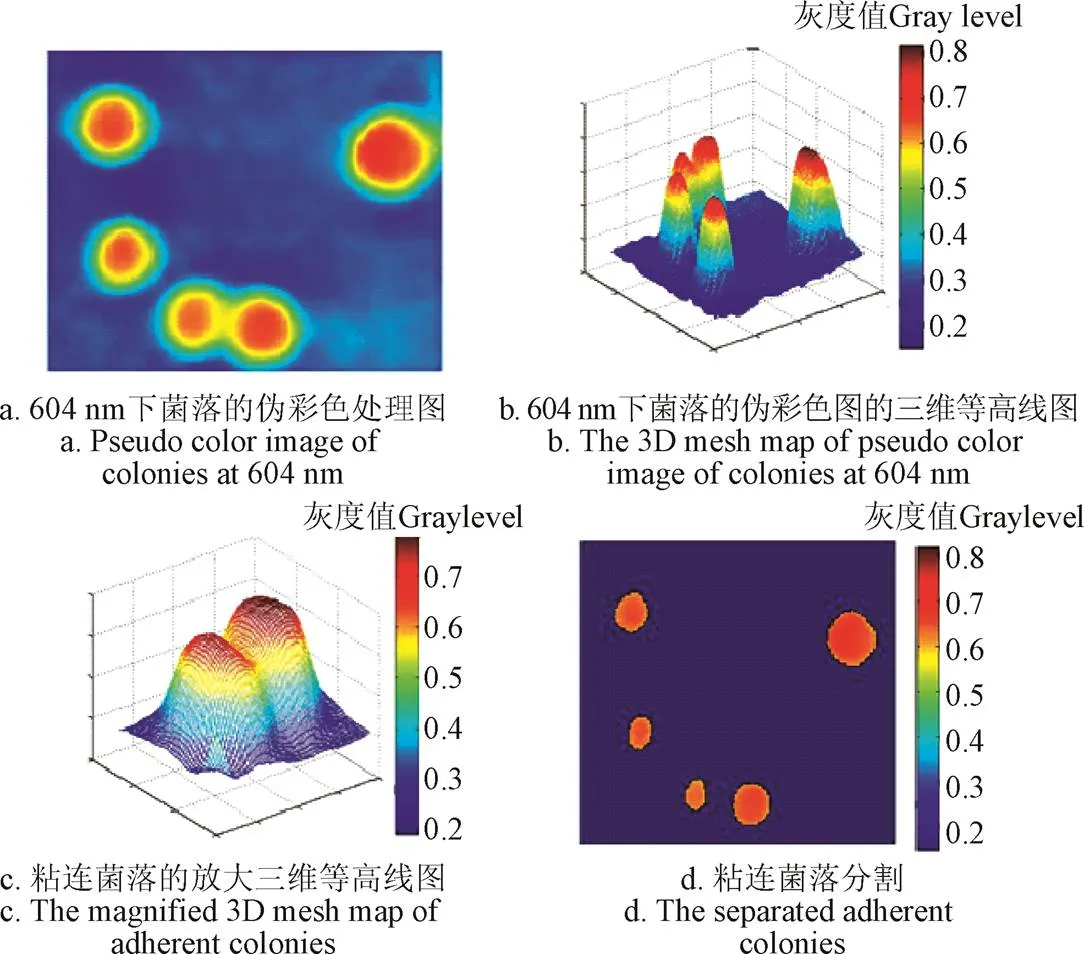
图4 利用604 nm下的高光谱图像特征分割粘连菌落的结果
经过菌落分割和粘连菌落分割等步骤,得到最终菌落计数图(图5),实现了菌落的准确分割。

图5 最终菌落计数图
2.5 菌落计数
采用局部邻域算法中的轮廓跟踪算法对分割出的菌落最终计数图进行计数[27],最终实现培养平板中的枯草芽孢杆菌菌落计数,计数结果为48(图5)。
枯草芽孢杆菌菌落形态呈圆形,其边缘光滑,颜色为浅黄色,本研究培养的枯草芽孢杆菌菌落与典型菌落形态描述一致(图6a)。针对本试验枯草芽孢杆菌菌落的彩色图像特点,本文分别采用人工视觉和计算机视觉技术对培养平板中的枯草芽孢杆菌菌落进行计数。利用人工视觉计数法计算的枯草芽孢杆菌菌落数为46(图6a)。采用灰度阈值分割算法对菌落平板彩色图像(图6a)进行二值化处理,结果如图6b所示。从图6b可以看出,阈值分割法使一部分背景点错分为菌落,特别是右上边部分,菌落和背景连在一起,不利于后续的计数。再对图6b进行处理,以便去除培养皿边缘和分割粘连菌落等的影响,最终得到菌落图像分割图,如图6c所示。从图6c清晰地可以看到有粘连菌落没能被成功分割开,有些菌落被分割成背景等,计算机视觉计数法计算该平板中菌落数为44(图6c)。

注:图b的椭圆区域为平板背景点错分为菌落的区域,图c的椭圆区域为没能被成功分割开的粘连菌落区域。
利用提出的基于高光谱技术的平板菌落计数方法、基于计算机视觉技术的平板菌落计数方法和人工视觉计数法(国标法)分别对同一培养平板中枯草芽孢杆菌菌落进行计数,计数结果依次为48、44和46。为了验证建立方法的准确度和可重复性,研究对7个菌落平板进行检测。将人工视觉计数方法作为国家标准方法,高光谱图像计数法的平均相对误差值为4.2%,优于计算机视觉技术计数结果(平均相对误差值为8.3%),具体结果如表1所示。全自动菌落计数仪是在计算机视觉技术上发展起来的,主要利用图像处理算法捕捉菌落与背景的颜色差异特征,自动分割平板中的菌落,从而实现计数,自动菌落计数的时间一般少于5 min,检测结果精度与计算机视觉技术一致。对于本研究建立的方法,检测平板菌落数的步骤主要包括高光谱图像的获取、菌落分割、粘连菌落分割和菌落数计算,耗费的时间分别为4、2、2和2 min,总耗时约为10 min。比自动菌落计数耗时长。但是其准确度却大大提高,平均相对误差降低了49.4%,结果表明,建立的计数方法有利于食品、农产品中菌落数的准确检测。
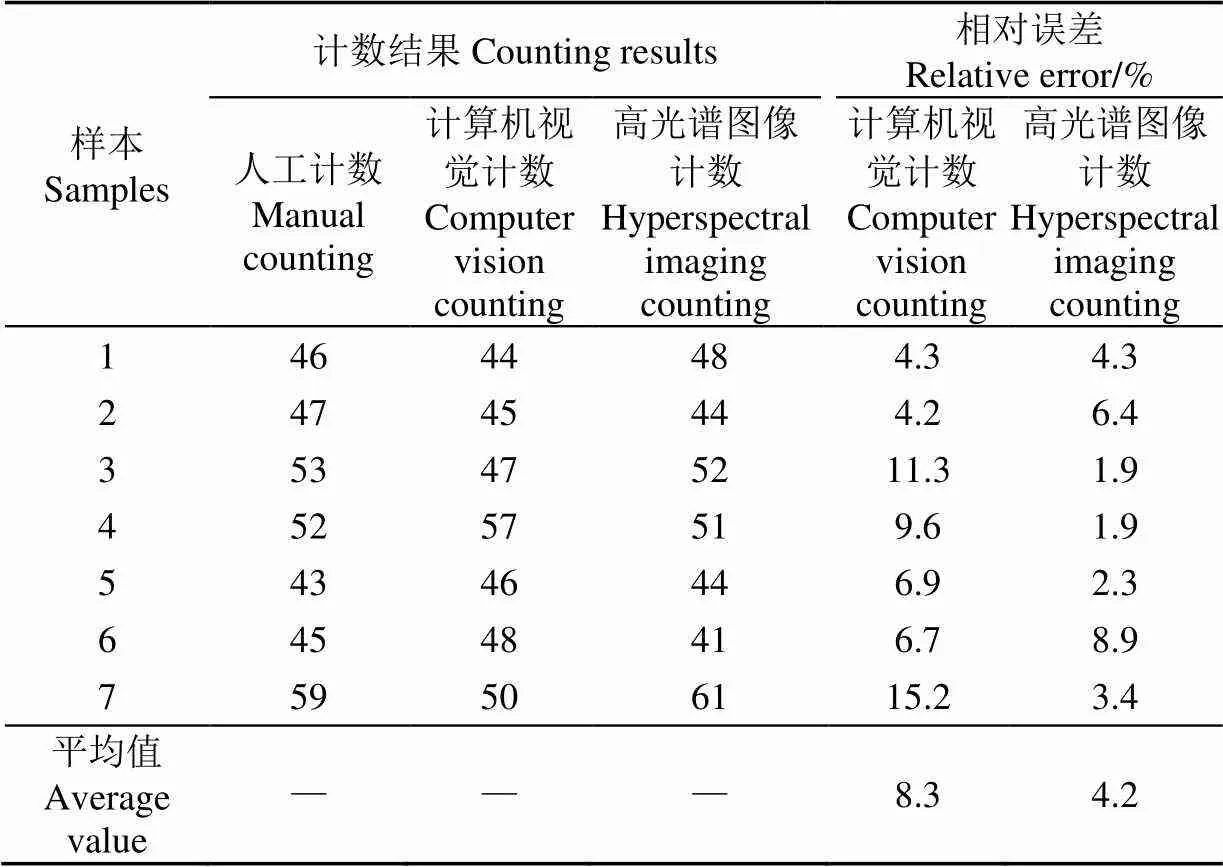
表1 3种计数方法的检测结果
本研究以枯草芽孢杆菌菌落平板为实例,详细介绍了基于高光谱技术的平板菌落计数方法,建立的方法只能用于实际样品中枯草芽孢杆菌的菌落计数。若要检测其他菌株菌落数,需要重新建立适用于目标菌株的计数方法,具体的方法可以参考本研究建立计数法的步骤。混合菌株菌落平板计数是当前研究的难点和热点,当不能根据菌落形貌肉眼区别菌株类别时,利用人工视觉计数和计算机视觉计数法无法实现混合菌株平板中每种菌株菌落的计数。不同菌株菌落具有不同高光谱图像信息,因此将高光谱图像技术和菌落平板计数法结合对混合菌落平板进行计数是课题组接下来的研究目标。
3 结 论
本文利用高光谱图像技术捕捉菌落与背景区域因成分差异引起的光谱特征,并结合化学计量法方法对平板中的菌落进行分割并实现了计数。主要结论如下:
1)利用遗传算法(Genetic Algorithms,GA)结合最小二乘支持向量机(Least Square Support Vector Machine,LS-SVM)建立了菌落区域和背景区域的判别模型,模型识别率为97.22%。
2)利用已建GA-LS-SVM判别模型实现了菌落与背景的分割,利用特征波段604 nm下的高光谱图像实现了粘连菌落之间的分割,最终建立了基于高光谱技术的菌落计数方法。建立的菌落计数法平均相对误差为4.2%,低于计算机视觉法的平均相对误差(8.3%)。
3)建立的菌落计数法耗时约10 min,表明提出的菌落计数方法的有望成为一种新的平板菌落计数方法,为食品、农产品中微生物数量的准确检测提供技术支持。
[1] 国家食品药品监督管理总局,国家卫生和计划生育委员会.食品安全国家标准食品微生物学检验菌落总数测定:GB 4789.2-2016[S]. 北京:中国标准出版社,2016:1-5.
[2] 王忠东,于建焕. 食品卫生微生物学检验菌落总数测定方法的效果分析[J]. 临床医药文献电子杂志,2015,2(20):4285-4286.
Wang Zhongdong, Yu Jianhuang. The analysis of food hygiene microbiology method for determination of total number of bacterial colonies[J]. Journal of Clinical Medicine, 2015, 2(20): 4285-4286. (in Chinese with English abstract)
[3] 张哲,杨峰,李新圃,等. 基于 Photoshop 和计数软件精准计数平板上菌落个数的新方法[J]. 微生物学通报,2016,43(7):1646-1648.
Zhang Zhe, Yang Feng, Li Xinpu, et al. A new couting method for colonies on plating medium based on photoshop and counting software[J]. Microbiology China, 2016, 43(7): 1646-1648. (in Chinese with English abstract)
[4] Matić T, Vidović I, Silađi E, et al. Semi-automatic prototype system for bacterial colony counting[J]. 2016. International Conference on Smart Systems and Technologies (SST), 2016: 205-210.
[5] Cheong J S. Aerobic Bacterial, Coliform, Escherichia Coli, and Staphylococcus Aureus Counts of Randomly Selected Sstreet Foods in Kampar, Perak[D]. Sungai Long: UTAR, 2016.
[6] Yoon S C, Lawrence K C, Park B. Automatic counting and classification of bacterial colonies using hyperspectral imaging[J]. Food and Bioprocess Technology, 2015, 8(10): 2047-2065.
[7] Ferrari A, Lombardi S, Signoroni A. Bacterial colony counting with convolutional neural networks in digital microbiology imaging[J]. Pattern Recognition, 2017, 61: 629-640.
[8] Liu D, Wu X, Yang J, et al. Comparison of detection methods of total bacterial colonies in infant rice flour[J]. Journal of Food Safety and Quality, 2019, 9(23): 6090-6094.
[9] Choudhry P. High-throughput method for automated colony and cell counting by digital image analysis based on edge detection[J]. PloS One, 2016, 11(2): 0148469.
[10] 王振杰,孙晔,顾欣哲,等. 计算机视觉技术对食品腐败检测的研究进展[J]. 食品安全质量检测学报,2015,6(8):2969-2975.
Wang Zhnejie, Sun Ye, Gu Xinzhe, et al. Research progress on computer vision for detection of food putrefaction[J]. Journal of Food Safety and Quality, 2015, 6(8): 2969-2975. (in Chinese with English abstract)
[11] Mao C, Yang Z, Sun X, et al. Fungus bacteria bag density detection system based on image processing Technology[M]. Data Processing Techniques and Applications for Cyber-Physical Systems (DPTA 2019): Advances in Intelligent Systems and Computing, Springer; 2020, 1163-1170.
[12] Mutimawurugo M, Wagara I, Muhinyuza J, et al. Virulence and characterization of isolates of potato bacterial wilt caused by Ralstonia solanacearum (Smith) in Rwanda[J]. African Journal of Agricultural Research. 2019, 14(6): 311-320.
[13] 李光泽. 食品微生物检验菌落总数测定方法的效果分析[J]. 中国卫生产业,2016,13(1):134-135.
Li Guangze. Analysis of the method for the determination of total number of colonies in food microbe test[J]. China Health Industry, 2016, 13(1): 134-135. (in Chinese with English abstract)
[14] Ren D, Chen P, Wang Y, et al. Research and application on the characteristics of compound cold water soluble gel in petrifilm aerobic count plates[J]. LWT-Food Science and Technology. 2017, 82: 335-341.
[15] 石吉勇,吴胜斌,邹小波,等. 高光谱技术融合平板菌落法同步计数酸奶中益生菌[J]. 食品科学,2018,39(24):16.
Shi Jiyong, Wu Shengbing, Zou Xiaobo, et al. A simultaneous counting method for total probiotics in yogurt with mixed strains based on hyperspectral imaging technology and plate colony method[J]. Food Science, 2018, 39(24): 16. (in Chinese with English abstract)
[16] 石吉勇,李文亭,胡雪桃,等. 基于叶绿素叶面分布特征的黄瓜氮镁元素亏缺快速诊断[J]. 农业工程学报,2019,35(13):170-176.
Shi Jiyong, Li Wenting, Hu Xuetao, et al. Diagnosis of nitrogen and magnesium deficiencies based on chlorophyll distribution features of cucumber leaf[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2019, 35(13): 170-176. (in Chinese with English abstract)
[17] 刘宝生,余占桥. 一株高度黏连的枯草芽孢杆菌的活菌计数[J]. 黑龙江畜牧兽医,2011,381(9):19-21.
Liu Baosheng, Yu Zhanqiao. A modified plate count for a strain of highly sticky Bacillus subtilis[J]. Heilongjiang Animal Husbandry and Veterinary Medicine, 2011, 381(9): 19-21. (in Chinese with English abstract)
[18] 董春旺,梁高震,安霆,等. 红茶感官品质及成分近红外光谱快速检测模型建立[J]. 农业工程学报,2018,34(24):306-313.
Dong Chunwang, Liang Gaozhen, An Ting, et al. Near-infrared spectroscopy detection model for sensory quality and chemical constituents of black tea[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2018, 34(24): 306-313. (in Chinese with English abstract)
[19] Zhu Y, Zou X, Shen T, et al. Determination of total acid content and moisture content during solid-state fermentation processes using hyperspectral imaging[J]. Journal of Food Engineering, 2016, 174: 75-84.
[20] 赵杰文. 食品、农产品检测中的数据处理和分析方法[M]. 北京:科学出版社,2012.
[21] 宁井铭,孙京京,朱小元,等. 基于图像和光谱信息融合的红茶萎凋程度量化判别[J]. 农业工程学报,2016,32(24):303-308.
Ning Jingming, Sun Jingjing, Zhu Xiaoyuan, et al. Discriminant of withering quality of Keemun black tea based on information fusion of image and spectrum[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2016, 32(24): 303-308. (in Chinese with English abstract)
[22] 孙俊,靳海涛,芦兵,等. 基于高光谱图像及深度特征的大米蛋白质含量预测模型[J]. 农业工程学报,2019,35(15):295-303.
Sun Jun, Jin Haitao, Lu Bing, et al. Prediction model of rice protein content based on hyperspectral image and deep feature[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2019, 35(15): 295-303. (in Chinese with English abstract)
[23] 石吉勇,吴胜斌,邹小波,等. 基于高光谱技术的酸奶中常见致病菌的快速鉴别及计数[J]. 光谱学与光谱分析,2019,39(4):1186.
Shi Jiyong, Wu Shengbing, Zou Xiaobo, et al. Rapididentification and enumeration of common pathogens in yogurt using hyperspectral imaging[J]. Spectroscopy and Spectral Analysis, 2019, 39(4): 1186. (in Chinese with English abstract)
[24] Altuntas V, Altuntas S, Gok M. Automatic colony segmentation on agar surface by image processing[J]. NISCAIR-CSIR, 2018, 77(8): 451-456.
[25] 邹小波,申婷婷,石吉勇,等. 基于高光谱图像技术的固态发酵中芽孢杆菌的快速识别[J]. 现代食品科技,2016(4):1-7.
Zou Xiaobo, Shen Tingting, Shi Jiyong, et.al. Quick identification of bacillus in the solid-state fermentation based on hyperspectral imaging technology[J]. Modern Food Science and Technology, 2016(4): 1-7. (in Chinese with English abstract)
[26] 胡雪桃,朱瑶迪,邹小波,等. 近红外光谱技术快速预测镇江香醋乙醇脱氢酶活性的研究[J]. 中国调味品,2016,41(2):20-25.
Hu Xuetao, Zhu Yaodi, Zou Xiaobo, et al. Research on alchol dehydrogenase activity of seed vinegar culture during solid-state fermentation of Zhenjiang aromatic vinegar[J]. China Condiment, 2016, 41(2): 20-25. (in Chinese with English abstract)
[27] Bonah E, Huang X, Aheto J H, et al. Application of hyperspectral imaging as a nondestructive technique for foodborne pathogen detection and characterization[J]. Foodborne Pathog Dis. 2019, 16(10): 712-22.
Colony image segmentation and counting based on hyperspectral technology
Li Yanxiao1, Hu Xuetao1, Zhang Fang2, Shi Jiyong2※, Qiu Baijing1
(1.,212013,; 2.,,212013,)
Colony plate counting methods as the national standard method are common and traditional for quantity inspection of living bacteria in food and agricultural products. The colonies are counted by manual counting and computer vision counting methods because of the color difference between colonies and background (such as medium and the edge of petri dish). However, colonies and background with similar color will interfere with the colony segmentation and lead to the deviation in counting results. Considering colonies and background have different spectral information resulting from different chemical compositions, hyperspectral imaging technology can be applied to colony segmentation and adherent colonies separation combined with chemostics. The segmentation method of colony and background, separated method of adherent colonies and calculation method of colony number were developed for colony counting. The hyperspectral images of() colony plate were acquired in the wavelengths from 431 to 963 nm. Spectral information of colonies and background (medium and the edge of petri dish) was extracted after preprocessing hyperspectral images. Genetic algorithms (GA) was used for processing spectral data and eleven characteristic wavelengths were selected (604, 636, 790, 799, 748, 683, 492, 437, 558, 470 and 928 nm). Genetic Algorithms least Square Support Vector Machine (GA-LS-SVM) model was established for distinguishing colonies and background by using the spectral information at the eleven characteristic wavelengths. The identification model with identification rate of 97.22% indicated that the colony and background could be successfully distinguished. The segmentation method of colony and background was developed. The spectral information of every pixel was extracted to identify whether it is colony or background by using the identification model. The binary image of colony segmentation was obtained through the spatial information in hyperspectral images. The location of colony was assigned as 1 and the location of background was assigned as 0, resulting in colony segmentation. The hyperspectral image at 604 nm was used for segmentation of adherent colonies to obtain binary image of adherent colonies segmentation. The segmentation threshold between background and colony was set as 0.5. The results demonstrated that the colonies were successfully segmented from background and adherent colonies could be accurately separated. Finally, the isolated colonies were counted by contour tracking algorithm after colony segmentation from background and adherent colonies separation. For application, acquisition of hyperspectral image, colony segmentation, adhesion colonies separation and colony number calculation were used forcolony counting of real samples. The time required by the developed method was about 10 min. Its average relative error of colony count was 4.2% with the manual counting method as the standard method. In addition, the colony plate counting was performed by using computer vision counting method. The average relative error of colony counting was 8.3% which was higher than the developed method. These results indicated that this method performed better than computer vision counting method though the consuming time was longer than that spent by the automatic colony counter. This method with high accuracy can become a novel plate colony counting method and provide technical support for the detection of microbial quantity in food and agricultural products.
image segmentation; genetic algorithms; least square support vector machine; hyperspectral imaging technology; colony counting; chemometrics;
李艳肖,胡雪桃,张芳,等. 基于高光谱技术的菌落图像分割与计数[J]. 农业工程学报,2020,36(20):326-332.doi:10.11975/j.issn.1002-6819.2020.20.038 http://www.tcsae.org
Li Yanxiao, Hu Xuetao, Zhang Fang, et al. Colony image segmentation and counting based on hyperspectral technology[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2020, 36(20): 326-332. (in Chinese with English abstract) doi:10.11975/j.issn.1002-6819.2020.20.038 http://www.tcsae.org
2020-08-04
2020-10-10
国家重点研发计划(2016YFD0200708);国家自然科学基金(61301239);江苏省现代农业重点项目(BE2019359)
李艳肖,高级实验师,主要从事农产品快速检测研究。Email:5983459@qq.com
石吉勇,教授,博士生导师,主要从事食品、农产品快速无损检测研究。Email:shi_jiyong@ujs.edu.cn
10.11975/j.issn.1002-6819.2020.20.038
O657.3
A
1002-6819(2020)-20-0326-07

