基于物种敏感度分布的概率生态风险评价的Matlab实现
冯永亮
(唐山学院 基础教学部,河北 唐山 063000)
0 引言
物种敏感度分布(Species Sensitivity Distribution,SSD)是一种用于描述不同物种对某个胁迫因素敏感度差异的统计分布模型。SSD的概念最早于1978年由美国环保局(U.S.EPA)在推导全国环境水质标准时提出,随后于1985年确定了以SSD的5%分位数所对应的浓度(HC5)推导环境基准值,并以此代替之前的专家判断法,沿用至今[1]。我国在2017年颁布的《淡水水生生物水质基准制定技术指南》中也推荐使用SSD模型进行淡水水生生物水质基准推导。此外,随着SSD理论的发展,许多学者开始采用SSD与环境监测数据相结合的概率方法评价污染物的生态风险[2-5]。Chen[3]分别采用急性和慢性毒性数据,构建了9种污染物的SSDs曲线,并结合环境监测浓度构建联合概率曲线(JPCs)评价了中国台湾北部基隆河中污染物的生态风险,发现Zn和Cu的风险最高,有机物的风险可忽略。目前SSD模型在许多国家和地区得到了很好的应用[6-7],已经成为污染物环境质量标准制定和生态风险评价的核心方法。
随着SSD理论的发展和广泛应用,一些学者或机构为SSD模型的构建和生态风险评价开发了相应的软件。这些软件大体上可分为两类:一类是基于Excel表格开发的,如USEPA开发的一款用于因果评估的在线应用程序(CADDIS)中的SSD Generator,荷兰国家公共卫生和环境研究所(RIVM)开发的ETX 2.0[8];另一类是基于专业的统计软件R开发的,如澳大利亚联邦科学和工业研究组织(CSIRO)基于Burr III分布开发的软件BurrlizO[9],Kon等开发的网页版工具MOSAIC_SSD[10]。这些软件主要集中于SSD的构建和环境标准(HC5)的推导,存在着可选统计模型较少、未能与环境数据相结合等缺点。本研究结合环境监测数据,首次给出了基于SSD的概率生态风险评价的Matlab函数“PERA”,为污染物的生态风险评价提供技术支持。
1 SSD的原理和假设
不同生物生活史、生理、形态、行为和地域分布的差异构成了生态系统的生物多样性。对于生态毒理学,这些生物多样性意味着不同生物对特定污染物具有不同的敏感性。物种间的这种敏感性差异可以利用特定的概率分布曲线即SSD曲线来描述。
物种敏感度分布的基本假设是不同物种对特定污染物的敏感性能够用一些统计分布模型(如Log-normal和Log-logistic分布等)来描述。生态毒理学的数据可以看成是来自于这些分布模型的样本,并用于SSD参数的估计。SSD模型的基本形式见图1。
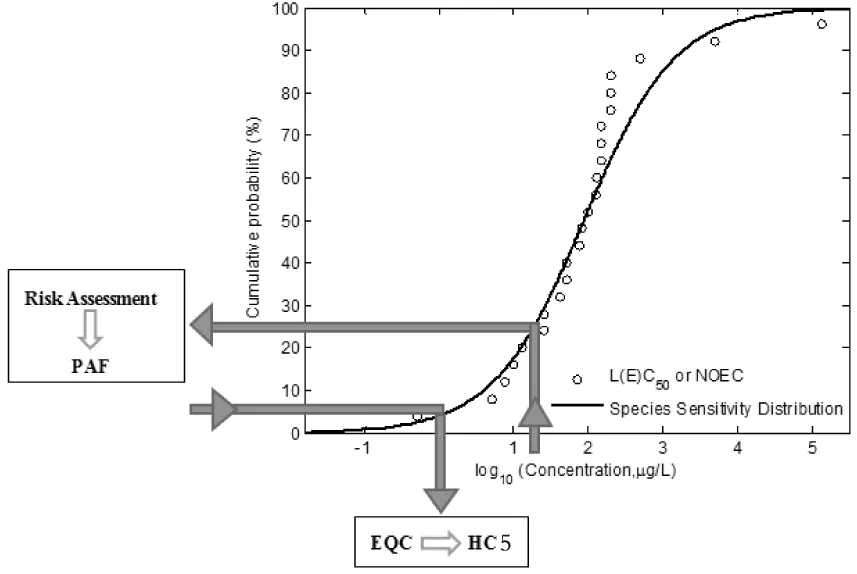
图1 物种敏感度分布曲线示意图
图1中的圆圈为原始毒性数据点,可以是急性(如半数致死浓度,LC50)或慢性(如无观测效应浓度,NOECs)效应浓度。拟合的曲线为毒性数据的累积概率曲线(即SSD)。图中的箭头表示SSD具有正向和反向两个方面的应用。其中反向(箭头Y→X)可应用于污染物的环境质量标准(EQC)的获得,主要是通过曲线计算特定物种损害比例所对应的污染物浓度,通常取HC5作为EQC,从而保护生态系统中95%的生物不受污染物的影响;正向(箭头X→Y)可以用于生态风险评价,主要由SSD曲线计算污染物在特定浓度下(一般为环境监测浓度)导致有害生物效应的累积概率,即受影响物种的比例(PAF),进而定量表征污染物的生态风险。
2 基于SSD的概率生态风险评价
化学物质的生态风险主要是通过环境监测数据与毒性数据进行表征的,表征的方法包括简单的风险商HQs法和相对复杂的概率生态风险评价法。风险商HQs法(即环境监测浓度除以毒性阈值)是简单的单点估计法,具有简单、透明和对数据要求低等优点,但其只适用于污染物筛选的初级阶段[11-12]。为了更加精确详细地表征生态风险,很多学者推荐采用更为复杂的概率方法,即联合概率曲线(JPC)法。JPC方法通过比较环境监测数据和毒性数据的分布特征可以定性和定量地表征生态风险,常被用作细化各种污染物生态风险的高水平方法[13]。下面给出JPC的构建过程和其生态学意义。
设EC为污染物的环境监测浓度,SS为污染物的毒性效应浓度,随机变量X1和X2分别来自于EC和SS的概率分布。
Pr(logEC>logSS)=Pr(X1>X2)。
(1)
式(1)为污染物的环境监测浓度大于其毒性效应浓度的概率,有两种等价的积分形式表达式:

(2)
或
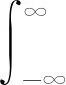
(3)
其中PDFXi,CDFXi(i=1,2)分别表示随机变量Xi的概率密度函数和累积分布函数;式(3)中1-CDFX1(x)表示随机变量X1超过给定数值x的概率,即传统概率论中的生存函数,在环境毒理学中通常将其定义为超越函数EXFX1(x)=1-CDFX1(x)。此时式(3)可等价转化为下式

(4)
由式(2)和式(4)右边的积分表达式可知,Pr(X1>X2)即为污染物环境风险在统计学上的期望值,其在数值上等于Van Straalen[14]给出的生态风险δ和Cardwell[15]给出的期望总风险(ETR)。
基于式(4),以毒性数据的累积概率(即dCDFX2(x))为自变量,环境监测数据的超越概率(即EXFX1(x))为因变量,构造的曲线即为联合概率曲线(具体构建过程见图2)。该曲线上的点表示导致不同物种损害水平的概率[2],曲线与两坐标轴围成的面积即为污染物环境风险的期望值Pr(X1>X2),也有学者称该值为发生有害生物效应的总体风险概率(ORP)[16-17]。
类似地,JPC也可以根据式(2)进行构建,即暴露数据的累积概率(dCDFX1(x))为自变量,毒性数据的累积概率(CDFX2(x))为因变量,曲线与两坐标轴围成的面积为Pr(X1>X2)。
3 概率生态风险评价的Matlab程序
基于上述分析过程,构建用于污染物概率生态风险评价的Matlab函数“PERA”,其程序代码如下所示。
function [HCq,ORP]=PERA(Tdata,q,option,ECdata)
% Tdata为污染物的毒性数据,需为向量形式。
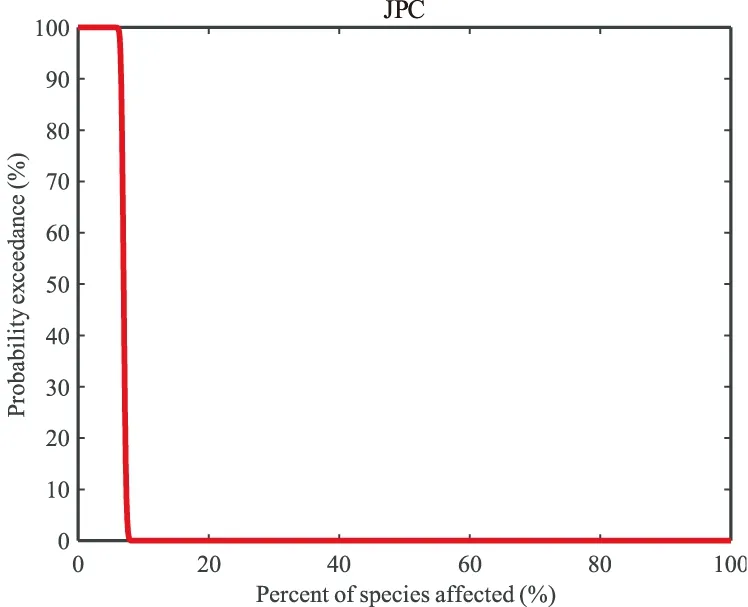
% q为SSD的q分位数HCq,0 % option=1和option=2分别表示基于Log-normal和Log-logistic分布构造SSD曲线。 % ECdata为污染物的环境监测数据,需为向量形式。 % [HCq]=PERA(Tdata,q,option),返回SSD曲线和HCq值。 % [HCq,ORP]=PERA(Tdata,q,option,ECdata),除返回SSD曲线和HCq值外,还返回JPC曲线和ORP值。 if nargin<3 error('输入参数必须为3个或4个'); end data=sort(Tdata); [n,m]=size(data); n=max(n,m); p=zeros(n,1); for i=1:n p(i)=i/(n+1); end data1=log10(data); if option==1 dis='normal'; else if option==2 dis='logistic'; end end [log,logci]=mle(data1,'distribution',dis); p2=icdf(dis,q,log(1),log(2)); HCq=10^p2; N=10000; mm=0.2; mi=min(data1); mi=min(mi)-mm; mx=max(data1); mx=max(mx)+mm; x1=linspace(mi-0.1,mx+0.1,N); p11=cdf(dis,x1,log(1),log(2)); p=p*100; p11=p11*100; MS=6; figure plot(data1,p,'ro','MarkerSize',MS,'MarkerFaceColor','r') hold on LW=1.5; % 图像中线的宽度。 plot(x1,p11,'k-','LineWidth',LW) axis([mi-0.1,mx+0.1,0,101]); FS=12;% 图像中字号的大小。 xlabel('log_{10}(concentration)') ylabel('Potentially Affected Percentage (%)') set(gca,'FontSize',FS,'Fontname','times new Roman') set(get(gca,'XLabel'),'FontSize',FS,'Fontname','times new Roman') set(get(gca,'YLabel'),'FontSize',FS,'Fontname','times new Roman') %%以下程序用于构造联合概率曲线JPC。 if nargin==4 data2=log10(ECdata); [phat,pci]=mle(data2,'distribution','normal'); %拟合环境数据的参数。 y=0:0.00001:1; p22=icdf(dis,y,log(1),log(2)); %求SSD的反累积对数浓度数值。 p33=cdf('normal',p22,phat(1),phat(2)); %求相对于SSD反累积对数浓度数值对应的环境监测数据的累积函数值。 p44=1-p33; %求相对于SSD反累积数值对应的环境监测数据的超越概率。 ORP=trapz(y,p44); %计算JPC曲线与两坐标轴所围区域的面积。 p44=p44*100; y=y*100; figure plot(y,p44,'r','LineWidth',LW) title('JPC','Fontname','times new Roman','FontSize',FS) xlabel('Percent of species affected (%)'); ylabel('Probability exceedance (%)'); set(gca,'FontSize',FS,'Fontname','times new Roman') set(get(gca,'XLabel'),'FontSize',FS,'Fontname','times new Roman') set(get(gca,'YLabel'),'FontSize',FS,'Fontname','times new Roman') end 采用Li[5]对莱州湾西部表层海水重金属Zn的环境监测数据(2016年9月监测)和Zn对海洋生物的慢性毒性数据对本文Matlab程序进行实际运行。 给定Zn的毒性数据Tdata=[20,1000,1144.962,97,1000,2154.395,10000,142.5,313,100,10,5000,400,380,4500,1000,970,200,139,480,230,600,520,124.0718,200,178,2500,630,88.5,15000,170.2,605,55,160,64,10,500,616.4414,80,170.9976,100,270,674,2000,2768.212,960,421.7163,1000,250,160,5900,1944.402]; Zn的环境监测数据ECdata=[39,40.6,39.1,38.6,43.6,40.4,41.2,41.9,41.1,37.8,38.4,36.8,38.4,38.7,39.1,38.5,41.2,40.7,42.4,39.5]; 在Matlab 2017b下运行: >> [HC5,ORP]=PERA(Tdata,0.05,2,ECdata) HC5=29.0852 ORP=0.0698 所得SSD曲线和JPC曲线分别见图3和图4。该结果与Li[5]的研究成果一致。 图4 莱州湾西部表层海水中Zn的联合概率曲线(JPC) 专业的统计软件R是学者们在做关于污染物生态风险评价时使用最多的一个软件平台[7,18-19]。R软件强大的统计软件包(如fitdistrplus)能够为SSD的建立、HC5的估计等提供更多自由全面的选择,但该软件的熟练运用需要使用者具有一定的概率统计学功底。本文给出的Matlab函数“PERA”只需要使用者具有污染物的毒性数据就能够方便地得到SSD曲线和其重要参数HC5,为概率统计学功底较弱的环境工作者提供了技术支持。 目前关于生态风险评价的软件主要集中在SSD的构建和环境标准(HC5)的推导,如CSIRO开发的BurrlizO[9],U.S.EPA开发的SSD Generator和Kon等开发的MOSAIC_SSD[10]等。这些软件虽然可以让使用者方便地得到SSD和HC5,但并没有与污染物的环境监测数据相结合,无法进行污染物的概率生态风险评价。本文给出的Matlab函数“PERA”只需要使用者具有污染物的毒性数据和环境监测数据,就可以方便地得到联合概率曲线JPC和污染物的总体风险概率ORP。虽然RIVM开发的软件ETX 2.0也可以给出JPC和ORP,但该软件中的SSD模型只能选Log-normal[8],而本文所给函数中的SSD模型有Log-normal和Log-logistic两种选择。另外,本文所给Matlab函数的开源代码,使用者可以对其作适当增加或修改(如增加或修改SSD模型的概率分布类型等),较ETX2.0更具灵活性。目前关于SSD的软件主要是基于Excel表格和专业统计软件R开发的,尚未有基于Matlab的程序或函数,本文首次给出了基于SSD的概率生态风险评价的Matlab函数“PERA”,为污染物的生态风险评价提供进一步的技术支持。4 Matlab程序的应用实例
5 讨论

