miRNA与肾脏发育
赵晓琪,敖英,陈海云,汪晖
综 述
miRNA与肾脏发育
赵晓琪,敖英,陈海云,汪晖
武汉大学基础医学院药理学系,发育源性疾病湖北省重点实验室,武汉 430071
MicroRNAs( miRNAs)是一类内源性小非编码RNA (约19~25个核苷酸),主要通过与靶mRNA中的互补靶序列结合,在转录后水平负调节基因表达。miRNA在包括器官发育在内的广泛生物过程中发挥着重要作用。最近研究表明,某些miRNA在肾脏高表达,并与肾脏发育及肾脏疾病密切相关,提示miRNA为肾脏生理学和病理学中的重要调节剂。本综述重点介绍了miRNA在肾脏发育调控中的研究进展,探讨了miRNA在肾脏异常发育的发生发展中起到的作用,为肾脏发育相关疾病的诊断和研究提供参考。
microRNA;肾脏发育;肾脏发育不良;机制;诊断
自发现一种非编码RNA (non-coding RNA, ncRNA)可以特异性沉默秀丽隐杆线虫()的基因功能以来,科学家对ncRNA的研究不断深入。MicroRNA (miRNA)是目前研究最多的ncRNA。到目前为止,已报告有近200种物种中存在超过28,000种miRNA[1]。据估计,有多达1/2的转录本受miRNA调节[2]。miRNA介导的基因表达调控作为一种较为保守的基因调控方式被证实参与大多数生物过程,如细胞分化、细胞凋亡、肿瘤发生和转移等[2]。一些在肾脏中高表达的miRNA被认为在肾脏生理和病理中扮演重要角色,可能成为肾病的一种新的诊断标志物和治疗靶点[3]。目前,一些与肾脏发育相关的研究表明,miRNA在肾脏发育中起关键作用。本文综述了miRNA与肾脏发育相关研究进展,探讨了miRNA在肾脏发育及其相关疾病中的潜在作用。
1 miRNA
miRNA是在真核生物中发现的一类内源性、长度约19~25个核苷酸的非编码单链RNA片段。miRNA具有多样性、进化保守性、组织特异性和时序性,在多种组织和器官的发育过程扮演重要角色。miRNA作为调控因子在真核生物体内广泛存在,通过与靶mRNA结合而在基因沉默和翻译抑制中起作用[2]。
1.1 miRNA的形成和功能
miRNA一般是由基因间DNA序列编码。在细胞核中,基因组DNA在RNA 聚合酶Ⅱ(RNA Pol Ⅱ)作用下产生长度为数千个碱基对的初级miRNA转录本(primary transcripts miRNA, pri-miRNA)。pri- miRNA在细胞核内被RNase Ⅲ核酸酶Drosha和DGCR8蛋白组成的微处理器复合体(DGCR8-Drosha)切割加工,释放为大约70个核苷酸的茎环结构,称为前体miRNA (precursor-miRNAs, pre-miRNA)。继而,pre-miRNA在RNA-GTP依赖的核质/细胞质转运蛋白exportin-5的作用下,形成复合物从核内运输到细胞质中。在细胞质,pre-miRNA被RNase Ⅲ内切酶Dicer识别并切割,释放出长度为19~25个核苷酸的二聚体miRNA:miRNA*(双链miRNA)。后者在RNA解旋酶作用下解聚,生成单链的成熟miRNA[4](图1)。
大多数的miRNA充当基因表达的负调控因子。miRNA通常靶向结合在信使RNA (messenger RNA, mRNA)的3′非翻译区(3′-untranslated region, 3′-UTR),导致基因降解或翻译抑制。内源miRNA的抑制活性取决于其是否加载到RNA诱导的沉默复合物(RNA- induced silencing complex, RISC)中。单链miRNA被载入Argonaute (AGO)蛋白,形成RISC复合物。复合物靶向结合到与互补的mRNA的3′-UTR上,进而调控靶mRNA的表达[3]。miRNA的作用方式与其与靶基因的互补性有关。当miRNA与靶mRNA之间完全配对互补时,可能影响靶mRNA的切割和降解[1]。当miRNA与靶mRNA之间不完全配对时,miRNA可能通过抑制翻译或促进mRNA去腺苷酸化和衰变来抑制蛋白质合成[5]。动物中大部分miRNA与靶mRNA之间不完全配对,故多为此种方式影响蛋白表达水平。然而,在某些情况下,一些miRNA能促进特定靶mRNA的翻译。例如,miRNA可通过与AGO2等蛋白质结合形成特定的复合物,在不同的静态(G0)细胞中激活靶基因的翻译[6]。

图1 miRNA的形成和功能
miRNA基因被RNA聚合酶Ⅱ转录为初级miRNA(pri-miRNA)转录本。微处理器复合物(DGCR8-Drosha)将pri-miRNA加工成前体miRNA (pre-miRNA),然后通过转运蛋白exportin-5将其输出到细胞质中。Pre-miRNA被Dicer切割以产生成熟的miRNA。成熟的miRNA识别各自的靶标mRNA,募集RNA诱导的沉默复合物(RISC),并通过翻译抑制,腺苷酸化和/或增强mRNA降解来介导其靶标的转录后抑制。
1.2 miRNA表达的调控
miRNA的生成和降解受到严格的调控,以保证特定的miRNA能在特定的时间、细胞表达适当的水平。一旦失调,将会引起下游靶基因的失控,进而导致疾病的发生[1]。目前的研究表明,miRNA的表达受到多个层面的调控:
(1)转录水平的调控。位于基因间的miRNA由其独立的启动子启动转录,而位于内含子区的miRNA则可跟随宿主基因一起转录或独立转录。miRNA的启动子也受到转录因子、增强子、沉默元件和染色质修饰等调控[7]。目前,已报道参与 miRNA转录调控的转录因子约有75种,较常见的转录因子有NK-κB、c-Myc、p53和C/EBPα等。
(2)转录后水平的调控。miRNA基因转录后,从 pri-miRNA直到最后加工成熟并组装成RISC的全过程均受到机体精细的调控,其机制主要有RNA 编辑、miRNA微加工复合物的调控和RNA结合蛋白对特异miRNA的调控[2]。miRNA加工成熟过程中的关键分子Drosha和Dicer均需与相应的辅助分子组成复合体以发挥作用,以上分子的表达水平和活性也均受到精细的调控[2,7]。
(3)降解的调控。目前的研究表明,miRNA降解的调控主要包括腺苷或尿嘧啶残基的修饰、形成RNA-蛋白质复合体以及核酸酶的降解等[5]。此外,最近研究发现了一种新兴的miRNA的降解途径:TDMD (target RNA-directed microRNA degradation),即特异性靶RNA与具有广泛互补性的miRNA的结合,可触发结合的miRNA的降解[8]。
(4)表观遗传调控。据估计,约50% miRNA基因与CpG岛相关联,许多miRNA的表达受到DNA甲基化的影响[9]。也有研究表明,许多miRNA可同时接受甲基化和乙酰化的表观遗传学调控。近年研究显示,有些miRNA也可以反馈调节表观遗传机制,体现了miRNA调控网络的复杂性,也增加了基因调控系统的稳固性[9]。
2 miRNA与肾脏发育
2.1 肾脏发育的主要阶段
哺乳动物的肾脏起源于中胚层体节外侧的细胞索,即生肾索。在人类胚胎的第18天(embryonic day 18, E18)/小鼠() E8.5时,肾脏开始出现。按时间顺序,肾脏发育经历前肾、中肾和后肾3个阶段。前肾和中肾是暂时性器官,在胚胎发育过程中相继退化,后肾则发育成为永久性肾脏[10]。
在人类E22/小鼠E9.5时,生肾索头端的生肾节内开始形成前肾管。继而生肾索尾侧开始逐渐形成中肾管。人类E35/小鼠E10.5时,中肾导管尾端向背侧长出输尿管芽(ureter bud, UB)。输尿管芽顶端侵入间充质时,生肾索分化为后肾间充质(metanephric mesenchyme, MM)。UB和MM组成后肾,二者相互诱导,促使后肾发育成熟。UB逐级分支,最终形成完整的泌尿集合管系统。后肾间充质细胞则经历间充质–上皮转变,一部分分化为非上皮化的基质细胞,最终形成平滑肌、基质和肾的微脉管系统;另一部分分化形成肾单位,包括肾小体、近曲小管、髓袢和远曲小管[10]。
2.2 肾脏发育过程中miRNA的表达
尽管关于肾脏中的miRNA的相关研究日渐丰富,但关于miRNA在肾脏发育中作用的数据有限,其相关功能尚不清晰。近年来的测序研究确定了小鼠胚胎肾脏中miRNA的表达谱,促进了对肾脏发育中miRNA的研究[11,12]。Aguilar等[11]发现,在小鼠E12和E13时,胎肾中的、、和表达非常丰富,而在成年肾脏组织中表达较少,这表明在肾脏发育过程中miRNA表达具有时间差异性。此外,从E12至E13,并至小鼠成年的过程中,肾脏中下调的miRNA还有、、、、、、、、、和等;上调的miRNA有、、、、、、、、、和等;而未发生变化的有、、、、、、和等[11]。这些miRNA在发育过程中的时间差异性值得人们进一步探索。
2.3 miRNA对肾脏发育的影响
大量研究提示miRNA在胚胎发育和分化中执行调节发育时机功能[13,14]。最近的一项研究发现,肾脏发育过程中具有时间差异表达的/轴可通过上调生长促进基因重组人胰岛素样生长因子-2 (insulin like growth factor-2, Igf2)来调节肾生成的停止,从而控制小鼠肾脏发育的持续时间[15]。这暗示了上述时间差异表达的miRNA在肾脏发育不同阶段起相应调控作用的潜能。
另外一些研究通过特异性敲除肾脏组织/细胞中的miRNA或miRNA生物合成中的关键组件如Drosha和Dicer等,进而研究miRNA在肾脏发育中所起的作用[16,17]。这些胚胎发展出一系列肾脏缺陷,包括水肿形成、肾上皮分化延迟及肾小球数目减少等,直接或/和间接地说明了肾脏发育过程中miRNA基因调控的重要性(表1)。
2.3.1 miRNA与肾脏发育过程中的关键转录因子
研究表明,miRNA可能通过影响关键转录因子参与早期肾脏发育过程。肾单位祖细胞表达的几种转录因子,包括Six2、Sall1、Pax2和WT1等,对其增殖和存活以及随后的分化是必不可少的[26~28]。一项研究发现,在前肾间充质中消除了Dicer功能后,肾单位祖细胞中的Six2、Sall1、WT1、Pax2和Cited1等明显减少,且后肾间充质中促凋亡蛋白Bim明显增加,最终导致严重的肾发育不良[22]。有研究提出在胚胎干细胞中沉默时,WT1、Pax2和Wnt4被下调[29]。此外,有研究发现,在体外可通过靶向WT1抑制后肾间充质干细胞细胞的增殖,因此其可能在肾脏发育和肾脏相关疾病中发挥重要功能[30]。这些研究提示了miRNA在早期肾脏器官发生期间对调节这些细胞谱系的存活起关键作用。
LIM级联的同源因子(LIM-class homeobox factor, Xlim1/Lhx1)是早期肾管形成和肾单位分化所必需的重要转录因子。它在肾脏发育过程中表现为需要严格控制的动态表达模式[31]。一项对非洲爪蟾前肾发育的研究表明,敲除肾脏中的可导致分化延迟,肾单位减小和增殖减少[16]。研究进一步发现,可靶向抑制Xlim1/Lhx1。在没有的情况下,Xlim1/Lhx1维持在高水平,从而导致肾上皮细胞的终末分化延迟[16]。此外,Lhx1也可与转录共激活因子Fryl协同作用,通过调控和簇的表达来调节早期肾脏发育[32]。这些研究表明,miRNA在调节前肾发育的过程中是不可或缺的。
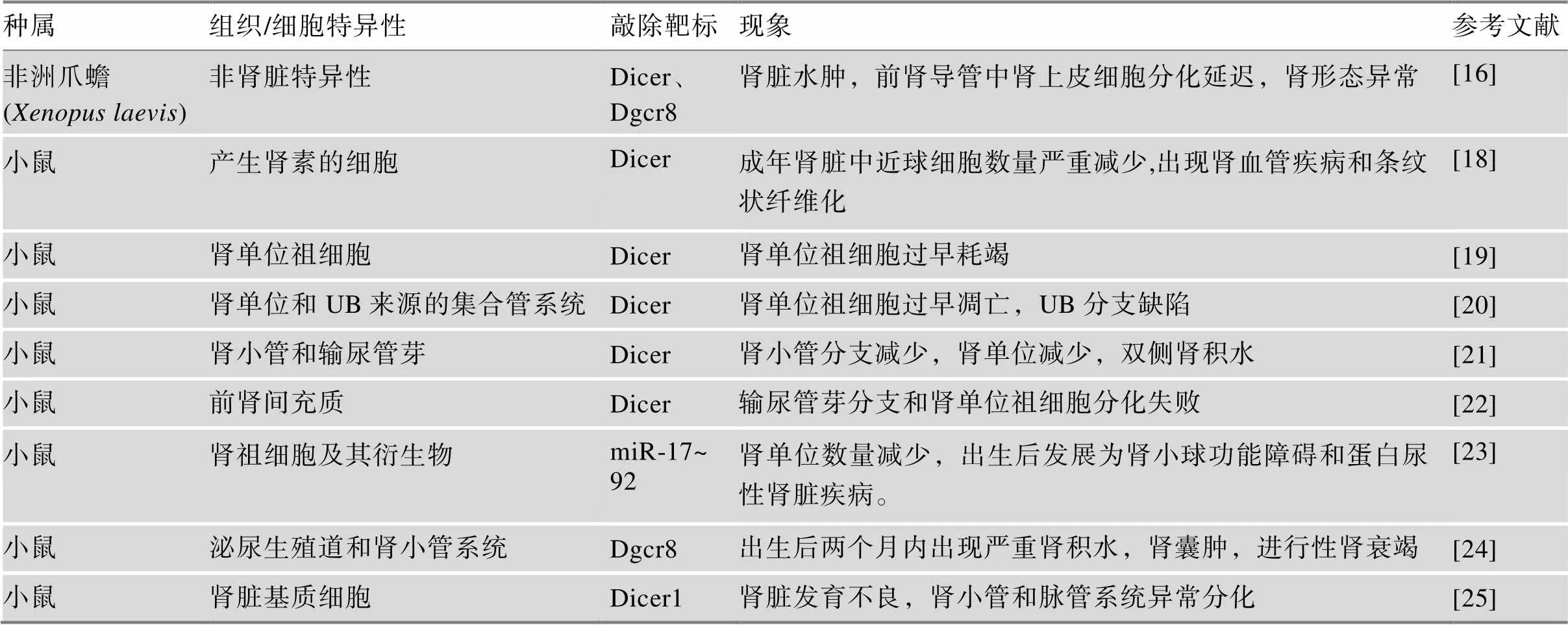
表1 肾脏发育相关的miRNA敲除动物模型
2.3.2 miRNA与肾脏发育过程中的GDNF/Ret信号通路
在后肾发育过程中,输尿管芽的出芽和分支是关键步骤。胶质源性神经营养因子(glial-cell-line- derived neurotrophic factor, GDNF)/c-Ret酪氨酸激酶受体(c-Ret tyrosine kinase receptor, c-Ret)信号通路是输尿管芽分支的主要诱导者[33]。研究发现,在肾单位谱系和输尿管芽来源的集合管系统细胞内特异性敲除Dicer的小鼠中分支形态发生破坏,其表型与输尿管尖端的Wnt11和c-Ret表达的下调相关。因此,可推断Dicer通过影响Dicer依赖性miRNA活性,进而影响GDNF/c-Ret信号通路,在发育中的小鼠肾内起调节作用[34]。先前一些神经系统发育和疾病相关的研究显示,、、和通过与的3'UTR 相互作用抑制了GDNF的表达,并且在体内替换了对GDNF 的3'UTR序列反应较不敏感的miRNA和RNA结合蛋白可导致内源性GDNF表达增加(Gdnfhyper)[35]。最近一项研究发现,这种Gdnfhyper / hyper小鼠的肾脏体积较小且出现畸形[36]。证明了肾脏发育中GDNF的水平和功能受其3'UTR影响。这些研究提示在miRNA可通过影响GDNF/c-Ret信号通路而参与肾脏发育。
2.3.3 miRNA与肾脏发育过程中的TGF-β/BMP信号通路
骨形态发生蛋白(bone morphogenic proteins, BMPs)是转化生长因子-β (transforming growth factor-β, TGF-β)超家族生长因子的成员。肾脏发育过程中正常的输尿管芽和肾单位的发生需要BMP信号诱导,且基因突变可导致肾脏发育不良。最近的许多研究提供了miRNA与TGF-β/BMP信号通路关键基因相互影响的证据。
TGF-β/BMP信号通路调控miRNA水平的一种机制是其下游效应蛋白Smad与Drosha相互作用[37]。在血管平滑肌细胞中,Smad-Drosha相互作用可促进初级转录物加工成成熟的[37]。而在肾脏中也起着重要作用。有文章报道,因其促增殖和抗凋亡而在鱼的肾脏再生中起作用[38]。这些线索提示很可能参与肾脏发育,当然这可能也涉及除了TGF-β/BMP信号通路以外的机制。
许多研究表明,miRNA可通过TGF-β受体2 (TGF-beta receptor type-2, TGFβR2)参与上皮–间质转化(epithelial-mesenchymal transition, EMT)过程的调节。EMT涉及生理过程和各种病理事件的许多方面,不仅在和肾纤维化中起关键作用,也参与胚胎发育。研究证实是靶标。在肾小球系膜细胞中,表达增加可导致表达降低[39]。也是靶向的EMT抑制性miRNA。的过表达可通过上调上皮细胞标志物E-钙粘蛋白来抑制EMT,并且在人肾2 (HK2)细胞系中下调间充质标记物laminin和α-SMA等[40]。另有研究发现通过直接靶向近端小管上皮细胞中的来抑制TGF-β1诱导的EMT[41]。家族也在早期肾脏中高表达[16],这提示高水平的可能在肾脏发育过程中保护肾脏上皮细胞免于自发的去分化。而过表达则可以通过抑制靶标,进而增强TGF-β1诱导的EMT[42]。也被证实可通过导致TGFβR1下调来抑制TGF-β/Smad信号激活[43]。这些研究均提示了miRNA与肾脏发育潜在的联系。
此外,在miRNA对涉及肾纤维化的关键分子的调节的研究中发现,和BMP-7/6处于调节反馈环路中,不仅抑制BMP-7/6表达,而且miR-22本身表达可由BMP-7/6诱导,此研究证明了在BMP信号级联中起关键作用[44]。尽管有大量miRNA与TGF-β/BMP信号传导相互影响的证据,但是在发育中的肾脏中,这些miRNA的功能在很大程度上还不确定,这也为未来miRNA与肾脏发育的研究提供了新的方向。
2.3.4 miRNA与肾脏发育过程中的肾素–血管紧张素系统
肾素–血管紧张素系统(renin-angiotensin system, RAS)是血压和液体/电解质稳态的主要调节器,也在控制正常肾脏发育中起着核心作用[45]。RAS系统主要成分包括:肾素、血管紧张素原、血管紧张素转换酶、血管紧张素I (angiotensin I, Ang I)、Ang Ⅱ、血管紧张素1型受体(angiotensin Ⅱ type 1 receptor, AT1R)和AT2R。RAS的所有组成部分在肾脏发育期间高度表达。Sequeira-Lopez等[18]通过构建在表达肾素的细胞中条件性敲除Dicer的小鼠,从而仅在产生肾素的细胞中选择性抑制miRNA的成熟。Dicer的敲除导致成年肾脏中近球细胞数量严重减少,同时肾素Ren1和Ren2的基因表达下降、血浆肾素浓度下降,并出现肾功能异常和严重的肾血管异常。这表明miRNA对于肾素细胞规格和肾血管正常发育是必需的。此外,一些在成年组织中的研究已证明miRNA可调节RAS中所有环节的蛋白质表达[46]。例如,内皮细胞、血管平滑肌细胞等细胞中的靶向抑制AT1R的表达,从而显着降低Ang II诱导的信号传导[47,48]。这也提示了miRNA在RAS信号传导调控中的重要地位。然而,对于肾脏发育过程中调控RAS组分的特定miRNA仍鲜有报道。
2.3.5 miRNA与肾脏发育过程中的HDAC
染色质修饰是一种表观遗传机制,可影响基因的转录活性。其中组蛋白脱乙酰酶(histone deacetylase, HDAC)在许多细胞过程中起重要作用,包括细胞周期、增殖、分化和细胞死亡[49]。一些对斑马鱼和小鼠的研究均表明了HDAC与前肾和后肾的发育有关。使用HDAC抑制剂处理斑马鱼胚胎中肾祖细胞数量增加,最终因肾祖细胞增生而导致肾功能受损[50]。将E13.5的小鼠肾脏用Scriptaid (I类和II类HDAC的抑制剂)培养后,后肾发育所必需的转录因子表达受到抑制,细胞正常的增殖和凋亡受到影响,最终导致肾脏发育不良[51]。这些研究提示HDAC对调节肾脏发育至关重要。有研究表明,高血糖可通过抑制信号转导从而加剧HDAC4的作用,导致足细胞中蛋白质脱乙酰基及蛋白质降解,最终导致肾功能障碍[52]。也有研究发现,HDAC抑制剂治疗可通过刺激小鼠肾脏和基因的转录来抑制钙转运相关基因Claudin-14的表达,从而减少小鼠尿钙排泄[53]。这提示miRNA与HDAC的相互影响及其对下游靶基因的影响可能在肾脏稳态中起重要作用。虽然很少有报道具体研究它们在肾脏发育中的作用机制,但这些证据提供了miRNA和HDAC之间的互作与肾脏发育的联系,这也是值得我们未来深入研究的一个方向。
3 肾脏miRNA表达改变与肾脏发育异常
如前所述,一些研究通过敲除肾脏局部特定细胞谱系中miRNA生物合成关键酶的方法,探究了miRNA介导的基因调节在肾脏发育中的作用。结果发现,缺乏miRNA动物的肾脏表型出现各种先天性肾脏和泌尿道异常(congenital anomalies of kidney and urinary tract, CAKUT)[21]。那么,miRNA是否在胎儿肾脏发育异常的机制中发挥重要作用?这个问题在近年来引起了越来越多研究者的关注。
3.1 引起肾脏发育异常的因素
近几十年的科学研究使人们对肾脏发育异常的认知越来越透彻。研究表明,遗传变异和胎儿环境的改变是导致胎儿肾脏异常发育的主要因素[54]。
3.1.1 遗传变异
染色体异常、拷贝数变异和单基因遗传异常是导致CAKUT的最常见因素。目前,相关的人群研究和动物研究已证实了多个与CAKUT发病相关的基因,如、、、、和等[55]。其中常染色体显性突变是CAKUT 最常见的单基因病因,常与肾脏发育不全及无功能的多囊性发育不良肾脏有关[56]。另外,的双等位基因失活基因突变与最严重的CAKUT表现即双侧肾发育不全有关[57]。此外,突变或表达异常多见于肾脏发育缺损或发育不良[58];/的突变多与腮–耳–肾综合征有关[58]。
3.1.2 胎儿环境改变
胎儿环境改变是诱发CAKUT和肾脏发育迟缓的另一个重要因素[59]。大量研究表明,孕期暴露于不良环境可影响肾脏发育,导致肾单位数量降低,肾功能下降,并存在成年高血压和慢性肾脏病编程[60]。这些因素包括孕妇营养不良[61]、胎盘供血不足[62]、孕妇糖尿病[63]、糖皮质激素[64]、尼古丁[65]、酒精[66]、维生素A缺乏症[67]以及孕妇用药暴露(例如血管紧张素转换酶抑制剂、抗生素、霉酚酸酯、抗癫痫药物和环磷酰胺)等[60,63,68,69],其影响机制也得到了较为充分研究。
研究表明,母鼠孕期低蛋白饮食(low protein diet, LP)可致子代宫内发育迟缓(intrauterine growth retardation, IUGR),并表现有肾脏发育不良,可能与RAS抑制及Na+-ATP酶活性升高等有关[61]。本实验的系列动物研究也证实,孕期咖啡因、乙醇、尼古丁、地塞米松等外源物暴露均可影响胎儿肾脏RAS相关基因表达,导致子代肾脏发育不良[64,70~72]。此外,我们发现孕期咖啡因暴露也可通过KLF4低表达编程引发子代足细胞发育毒性,进而导致成年肾脏疾病易感[73]。而在孕期乙醇暴露的IUGR动物模型中,“GC-IGF1”轴编程改变也对肾脏发育不良及成年后肾小球硬化易感起到了至关重要的作用[70]。另外也有研究也提出,母亲吸烟可引起肾脏氧化应激、线粒体变化,对后代成年肾结构、血压、尿钠排泄造成影响[63]。此外,孕期地塞米松暴露也可通过影响Wnt4表达,进而影响TGF-β表达,导致细胞凋亡增加、促凋亡基因增加及抗凋亡基因降低,进而造成肾单位数减少[74]。
3.2 miRNA表达的改变可能与参与肾脏发育异常
近年来,大量研究已表明miRNA表达失调与各种生物体和器官系统的发育缺陷表型有关。一些研究也提供了miRNA参与肾脏异常发育发病机制的证据。
3.2.1 遗传变异所致肾脏异常发育中的miRNA
基因测序技术为基因组学的研究提供了便利,推动了miRNA在疾病中的相关研究。目前,只有少数研究在miRNA与特定的遗传变异的肾脏疾病之间建立了明确的联系。Jovanovic等[75]通过对19名CAKUT患者和9名对照的输尿管组织样本中收集的全基因组表达数据进行分析,鉴定出了7种可能在CAKUT中发挥潜在作用的miRNA:、、、、、和。其中被验证在CAKUT患者组织中表达显著增加,且可能与对肾脏和尿道正常发育至关重要的生物学过程有关[75]。但仍需进行进一步的功能分析以揭示这些特定miRNA在肾脏异常发育中的作用。研究发现,簇在胚胎的正常发育中似乎是必不可少的,其缺失可导致人类发育障碍的Feingold综合征,其特征包括肾脏发育缺陷[76]。此外多项研究表明,在多种多囊肾病小鼠模型中簇被上调,而簇的失活减慢了囊肿的增殖[77]。这主要是因为簇靶向抑制囊性肾脏疾病基因,包括、和。另一个被认为与常染色体显性遗传性多囊肾病有关的miRNA是,其在患有多囊肾病(polycystic kidney disease, PKD)的人和鼠的囊肿中表达增加。加剧囊肿生长的潜在机制可能涉及直接抑制促凋亡的肿瘤抑制因子PDCD4[78]。这些研究表明,miRNA是肾脏发育相关疾病发病机制的关键调节剂之一。
3.2.2 环境因素所致肾脏异常发育中的miRNA
在环境因素引起的肾脏异常发育中,miRNA的调控作用也可能起着关键作用。最近一项研究发现,怀孕的母鼠给予miRNA抑制剂后,在子代肾脏等脏器中可检测到miRNA水平持续显着降低。这表明孕期服用的一些可诱导miRNA表达的药物(如基于多西环素的四环素控制的反式激活剂和基于他莫昔芬的雌激素受体系统)可凭借母体-胎盘-胎儿传递,进而影响子代肾脏中miRNA表达[79]。此外,一项关于母体蛋白摄食限制的动物研究发现,LP后代大鼠(Rattus norvegicus)肾小球中一些miRNA显著下调,如(71%)、(50%)、(60%)和(59%)[80]。虽然这些研究未探究miRNA表达失调与子代肾脏发育异常的直接关系,但其表明了miRNA与环境因素所致肾脏异常发育的关联。更多miRNA的具体作用仍需得到更多的关注。
4 结语与展望
近些年来,miRNA作为肾脏发育和疾病中的重要调控分子备受研究者关注。随着研究的不断深入,越来越多的miRNA被发现在肾脏发育中差异表达,它们可通过影响关键生长因子或相关信号通路来参与肾脏发育过程。Drosha或Dicer等敲除的研究及肾脏发育相关疾病中的研究也提示了miRNA在肾脏发育中是必不可少的。但是,仍然存在许多问题需要解决:Drosha或Dicer的敲除可影响全部miRNA表达的改变,而单个miRNA或某miRNA簇参与肾脏发育过程的具体作用机制仍不明确;此外,miRNA在肾脏发育异常相关疾病中的确切作用仍然未知。未来的工作应侧重于阐明肾脏发育过程中单个或一系列miRNA的具体功能,了解其调控生理和在病理过程中的作用的精确机制,并充分利用先进的测序技术来探究在肾脏发育异常相关疾病中起关键作用的miRNA。肾脏发育中miRNA相关基因网络的构建将有助于进一步了解其在肾脏发育及相关疾病中的作用,并有望提出更具临床应用价值的肾脏疾病早期预警标志物及治疗靶标。
[1] Saliminejad K, Khorram Khorshid HR, Soleymani Fard S, Ghaffari SH. An overview of microRNAs: Biology, functions, therapeutics, and analysis methods, 2019, 234(5): 5451–5465.
[2] Lu TX, Rothenberg ME. MicroRNA, 2018, 141(4): 1202–1207.
[3] Ho J, Kreidberg JA. The long and short of microRNAs in the kidney, 2012, 23(3): 400–404.
[4] Ma SY, Bai Y, Han N, Wang JH, Weng XY, Bian HW, Zhu MY. Recent research progress of biogenesis and functions of miRNA*, 2012, 34(4): 383–388.马圣运, 白玉, 韩凝, 王君晖, 翁晓燕, 边红武, 朱睦元. miRNA*生物合成及其功能研究的新发现. 遗传, 2012, 34(4): 383–388.
[5] Valadkhan S, Gunawardane LS. Role of small nuclear RNAs in eukaryotic gene expression, 2013, 54: 79–90.
[6] Vasudevan S, Steitz JA. AU-rich-element-mediated upregulation of translation by FXR1 and Argonaute 2, 2007, 128(6): 1105–1118.
[7] Kawahara Y, Zinshteyn B, Chendrimada TP, Shiekhattar R, Nishikura K. RNA editing of the microRNA-151 precursor blocks cleavage by the Dicer-TRBP complex, 2007, 8(8): 763–769.
[8] Fuchs Wightman F, Giono LE, Fededa JP, de la Mata M. Target RNAs strike back on microRNAs, 2018, 9: 435.
[9] Zhang WT, Duan N, Zhang Q, Song T, Li Z, Zhang CG, Chen X, Wang KZ. DNA methylation mediated down- regulation of miR-370 regulates cell growth through activation of the wnt/β-catenin signaling pathway in human osteosarcoma cells, 2017, 13(5): 561–573.
[10] Schedl A. Hastie ND. Cross-talk in kidney development,2000, 10: 543–549.
[11] Aguilar ALG, Piskol R, Beitzinger M, Zhu JY, Kruspe D, Aszodi A, Moser M, Englert C, Meister G. The small RNA expression profile of the developing murine urinary and reproductive systems, 2010, 584(21): 4426– 4434.
[12] Nagalakshmi VK, Lindner V, Wessels A, u J. microRNA- dependent temporal gene expression in the ureteric bud epithelium during mammalian kidney development, 2015, 244(3): 444–456.
[13] Ambros V. MicroRNAs and developmental timing, 2011, 21(4): 511–517.
[14] Schulman BRM, Esquela-Kerscher A, Slack FJ. Reciprocal expression of lin-41 and the microRNAs let-7 and mir-125 during mouse embryogenesis, 2005, 234(4): 1046–1054.
[15] Yermalovich AV, Osborne JK, Sousa P, Han A, Kinney MA, Chen MJ, Robinton DA, Montie H, Pearson DS, Wilson SB, Combes AN, Little MH, Daley GQ. Lin28 and let-7 regulate the timing of cessation of murine nephrogenesis, 2019, 10(1): 168.
[16] Agrawal R, Tran U, Wessely O. The miR-30 miRNA family regulates Xenopus pronephros development and targets the transcription factor Xlim1/Lhx1, 2009, 136(23): 3927–3936.
[17] Cerqueira DM, Bodnar AJ, Phua YL, Freer R, Hemker SL, Walensky LD, Hukriede NA, Ho J. Bim gene dosage is critical in modulating nephron progenitor survival in the absence of microRNAs during kidney development, 2017, 31(8): 3540–3554.
[18] Sequeira-Lopez MLS, Weatherford ET, Borges GR, Monteagudo MC, Pentz ES, Harfe BD, Carretero O, Sigmund CD, Gomez RA. The microRNA-processing enzyme dicer maintains juxtaglomerular cells, 2010, 21(3): 460–467.
[19] Ho J, Pandey P, Schatton T, Sims-Lucas S, Khalid M, Frank MH, Hartwig S, Kreidberg JA. The pro-apoptotic protein bim is a microRNA target in kidney progenitors, 2011, 22(6): 1053–1063.
[20] Nagalakshmi VK, Ren Q, Pugh MM, Valerius MT, McMahon AP, Yu J. Dicer regulates the development of nephrogenic and ureteric compartments in the mammalian kidney, 2011, 79(3): 317–330.
[21] Bartram MP, Höhne M, Dafinger C, Völker LA, Albersmeyer M, Heiss J, Göbel H, Brönneke H, Burst V, Liebau MC, Benzing T, Schermer B, Müller RU. Conditional loss of kidney microRNAs results in congenital anomalies of the kidney and urinary tract (CAKUT), 2013, 91(6): 739–748.
[22] Chu JYS, Sims-Lucas S, Bushnell DS, Bodnar AJ, Kreidberg JA, Ho J. Dicer function is required in the metanephric mesenchyme for early kidney development, 2014, 306(7): F764–F772.
[23] Marrone AK, Stolz DB, Bastacky SI, Kostka D, Bodnar AJ, Ho J. MicroRNA-17~92 is required for nephrogenesis and renal function, 2014, 25(7): 1440–1452.
[24] Bartram MP, Dafinger C, Habbig S, Benzing T, Schermer B, Müller RU. Loss of Dgcr8-mediated microRNA expression in the kidney results in hydronephrosis and renal malformation, 2015, 16: 55.
[25] Nakagawa N, Xin CY, Roach AM, Naiman N, Shankland SJ, Ligresti G, Ren SY, Szak S, Gomez IG, Duffield JS. Dicer1 activity in the stromal compartment regulates nephron differentiation and vascular patterning during mammalian kidney organogenesis, 2015, 87(6): 1125–1140.
[26] Kreidberg JA, Sariola H, Loring JM, Maeda M, Pelletier J, Housman D, Jaenisch R. WT-1 is required for early kidney development, 1993, 74(4): 679–691.
[27] Rothenpieler UW, Dressler GR. Pax-2 is required for mesenchyme-to-epithelium conversion during kidney development, 1993, 119(3): 711–720.
[28] Dressler GR, Patel SR. Epigenetics in kidney development and renal disease, 2015, 165(1): 166–176.
[29] Viñas JL, Ventayol M, Brüne B, Jung M, Sola A, Pi F, Mastora C, Hotter G. miRNA let-7e modulates the Wnt pathway and early nephrogenic markers in mouse embryonic stem cell differentiation, 2013, 8(4): e60937.
[30] Xue MM, Zhou YR, Liu XY, Ni DS, Hu YX, Long YS, Ju P, Zhou Q. Proliferation of metanephric mesenchymal cells is inhibited by miR-743a-mediated WT1 suppression, 2016, 14(5): 4315–4320.
[31] Dressler GR. The cellular basis of kidney development, 2006, 22: 509–529.
[32] Espiritu EB, Crunk AE, Bais A, Hochbaum D, Cervino AS, Phua YL, Butterworth MB, Goto T, Ho J, Hukriede NA, Cirio MC. The Lhx1-Ldb1 complex interacts with Furry to regulate microRNA expression during pronephric kidney development, 2018, 8(1): 16029.
[33] Majumdar A, Vainio S, Kispert A, McMahon J, McMahon AP. Wnt11 and Ret/Gdnf pathways cooperate in regulating ureteric branching during metanephric kidney development, 2003, 130(14): 3175–3185.
[34] Maheu M, Lopez JP, Crapper L, Davoli MA, Turecki G, Mechawar N. MicroRNA regulation of central glial cell line-derived neurotrophic factor (GDNF) signalling in depression, 2015, 5(2): e511.
[35] Kumar A, Kopra J, Varendi K, Porokuokka LL, Panhelainen A, Kuure S, Marshall P, Karalija N, Härma MA, Vilenius C, Lilleväli K, Tekko T, Mijatovic J, Pulkkinen N, Jakobson M, Jakobson M, Ola R, Palm E, Lindahl M, Strömberg I, Võikar V, Piepponen TP, Saarma M, Andressoo JO. GDNF overexpression from the native locus reveals its role in the nigrostriatal dopaminergic system function, 2015, 11(12): e1005710.
[36] Li H, Jakobson M, Ola R, Gui YJ, Kumar A, Sipilä P, Sariola H, Kuure S, Andressoo JO. Development of the urogenital system is regulatedthe 3'UTR of GDNF, 2019, 9(1): 5302.
[37] Davis BN, Hilyard AC, Lagna G, Hata A. SMAD proteins control DROSHA-mediated microRNA maturation, 2008, 454(7200): 56–61.
[38] Hoppe B, Pietsch S, Franke M, Engel S, Groth M, Platzer M, Englert C. MiR-21 is required for efficient kidney regeneration in fish, 2015, 15: 43.
[39] Faherty N, Curran SP, O'Donovan H, Martin F, Godson C, Brazil DP, Crean JK. CCN2/CTGF increases expression of miR-302 microRNAs, which target the TGFβ type II receptor with implications for nephropathic cell phenotypes, 2012, 125(pt 23): 5621–5629.
[40] Liu TM, Nie F, Yang XG, Wang XY, Yuan Y, Lv ZS, Zhou L, Peng R, Ni DS, Gu YP, Zhou Q, Weng YG. MicroRNA- 590 is an EMT-suppressive microRNA involved in the TGFβ signaling pathway, 2015, 12(5): 7403–7411.
[41] Gong Y, Qin ZX, Zhou BS, Chen H, Shi ZM, Zhang J. MicroRNA-200a inhibits transforming growth factor β1-induced proximal tubular epithelial-mesenchymal transition by targeting β-catenin, 2017, 137(3): 237–249.
[42] Wang JY, Gao YB, Zhang N, Zou DW, Wang P, Zhu ZY, Li JY, Zhou SN, Wang SC, Wang YY, Yang JK. miR-21 overexpression enhances TGF-β1-induced epithelial-to- mesenchymal transition by target smad7 and aggravates renal damage in diabetic nephropathy, 2014, 392(1–2): 163–172.
[43] Choi HI, Park JS, Kim DH, Kim CS, Bae EH, Ma SK, Kim SW. PGC-1α suppresses the activation of TGF-β/Smad signalingtargeting TGFβRI downregulation by let-7b/c upregulation, 2019, 20(20): 5084.
[44] Long JY, Badal SS, Wang Y, Chang BHJ, Rodriguez A, Danesh FR. MicroRNA-22 is a master regulator of bone morphogenetic protein-7/6 homeostasis in the kidney, 2013, 288(51): 36202–36214.
[45] Yosypiv IV. Renin-angiotensin system in mammalian kidney development, 2020.
[46] Butterworth MB. Role of microRNAs in aldosterone signaling, 2018, 27(5): 390–394.
[47] Stankovic A, Kolaković A, Živković M, Djurić T, Bundalo M, Končar I, Davidović L, Alavantić D. Angiotensin receptor type 1 polymorphism A1166C is associated with altered AT1R and miR-155 expression in carotid plaque tissue and development of hypoechoic carotid plaques, 2016, 248: 132–139.
[48] Zheng L, Xu CC, Chen WD, Shen WL, Ruan CC, Zhu LM, Zhu DL, Gao PJ. MicroRNA-155 regulates angiotensin II type 1 receptor expression and phenotypic differentiation in vascular adventitial fibroblasts, 2010, 400(4): 483–488.
[49] Seto E, Yoshida M. Erasers of histone acetylation: the histone deacetylase enzymes, 2014, 6(4): a018713.
[50] de Groh ED, Swanhart LM, Cosentino CC, Jackson RL, Dai WX, Kitchens CA, Day BW, Smithgall TE, Hukriede NA. Inhibition of histone deacetylase expands the renal progenitor cell population, 2010, 21(5): 794–802.
[51] Chen SW, Bellew C, Yao X, Stefkova J, Dipp S, Saifudeen Z, Bachvarov D, El-Dahr SS. Histone deacetylase (HDAC) activity is critical for embryonic kidney gene expression, growth, and differentiation, 2011, 286(37): 32775–32789.
[52] Lin CL, Lee PH, Hsu YC, Lei CC, Ko JY, Chuang PC, Huang YT, Wang SY, Wu SL, Chen YS, Chiang WC, Reiser J, Wang FS. MicroRNA-29a promotion of nephrin acetylation ameliorates hyperglycemia-induced podocyte dysfunction, 2014, 25(8): 1698–1709.
[53] Gong YF, Himmerkus N, Plain A, Bleich M, Hou JH. Epigenetic regulation of microRNAs controlling CLDN14 expression as a mechanism for renal calcium handling, 2015, 26(3): 663–676.
[54] Nicolaou N, Renkema KY, Bongers EMHF, Giles RH, Knoers NVAM. Genetic, environmental, and epigenetic factors involved in CAKUT, 2015, 11(12): 720–731.
[55] Bertram JF, Goldstein SL, Pape L, Schaefer F, Shroff RC, Warady BA. Kidney disease in children: latest advances and remaining challenges, 2016, 12(3): 182–191.
[56] Avni FE, Lahoche A, Langlois C, Garel C, Hall M, Vivier PH. Renal involvement in children with HNF1β mutation: early sonographic appearances and long-term follow-up, 2015, 25(5): 1479–1486.
[57] Skinner MA, Safford SD, Reeves JG, Jackson ME, Freemerman AJ. Renal aplasia in humans is associated with RET mutations, 2008, 82(2): 344– 351.
[58] Weber S, Moriniere V, Knüppel T, Charbit M, Dusek J, Ghiggeri GM, Jankauskiené A, Mir S, Montini G, Peco-Antic A, Wühl E, Zurowska AM, Mehls O, Antignac C, Schaefer F, Salomon R. Prevalence of mutations in renal developmental genes in children with renal hypodysplasia: results of the ESCAPE study, 2006, 17(10): 2864–2870.
[59] Juvet C, Simeoni U, Yzydorczyk C, Siddeek B, Armengaud JB, Nardou K, Juvet P, Benahmed M, Cachat F, Chehade H. Effect of early postnatal nutrition on chronic kidney disease and arterial hypertension in adulthood: a narrative review, 2018, 9(6): 598–614.
[60] Brophy P. Maternal determinants of renal mass and function in the fetus and neonate, 2017, 22(2): 67–70.
[61] Dötsch J, Alejandre-Alcazar M, Janoschek R, Nüsken E, Weber LT, Nüsken KD. Perinatal programming of renal function, 2016, 28(2): 188–194.
[62] Ergaz Z, Avgil M, Ornoy A. Intrauterine growth restriction- etiology and consequences: what do we know about the human situation and experimental animal models?, 2005, 20(3): 301–322.
[63] Corrêa RRM, Pucci KRM, Rocha LP, Júnior CDP, Helmo FR, Machado JR, Rocha LB, Rodrigues ARA, Glória MA, Guimarães CSO, Câmara NOS, Reis MA. Acute kidney injury and progression of renal failure after fetal programming in the offspring of diabetic rats, 2014, 77(3): 440–446.
[64] Li B, Zhu YN, Chen HY, Gao H, He HY, Zuo N, Pei LG, Xie W, Chen LB, Ao Y, Wang H. Decreased H3K9ac level of AT2R mediates the developmental origin of glomerulosclerosis induced by prenatal dexamethasone exposure in male offspring rats, 2019, 411: 32–42.
[65] Stangenberg S, Nguyen LT, Chen H, Al-Odat I, Killingsworth MC, Gosnell ME, Anwer AG, Goldys EM, Pollock CA, Saad S. Oxidative stress, mitochondrial perturbations and fetal programming of renal disease induced by maternal smoking, 2015, 64: 81–90.
[66] Gray SP, Denton KM, Cullen-McEwen L, Bertram JF, Moritz KM. Prenatal exposure to alcohol reduces nephron number and raises blood pressure in progeny, 2010, 21(11): 1891–1902.
[67] Goodyer P, Kurpad A, Rekha S, Muthayya S, Dwarkanath P, Iyengar A, Philip B, Mhaskar A, Benjamin A, Maharaj S, Laforte D, Raju C, Phadke K. Effects of maternal vitamin A status on kidney development: a pilot study, 2007, 22(2): 209–214.
[68] Rosenblum S, Pal A, Reidy K. Renal development in the fetus and premature infant, 2017, 22(2): 58–66.
[69] Luyckx VA, Brenner BM. Birth weight, malnutrition and kidney-associated outcomes--a global concern, 2015, 11(3): 135–149.
[70] Chen HY, Zhu YN, Zhao XQ, He HY, Luo JS, Ao Y, Wang H. Prenatal ethanol exposure increased the susceptibility of adult offspring rats to glomerulosclerosis, 2020, 321: 44–53.
[71] Ao Y, Sun ZX, Hu SS, Zuo N, Li B, Yang SL, Xia LP, Wu Y, Wang LL, He Z, Wang H. Low functional programming of renal AT2R mediates the developmental origin of glomerulosclerosis in adult offspring induced by prenatal caffeine exposure, 2015, 287(2): 128–138.
[72] Sun ZX, Hu SS, Zuo N, Yang SL, He Z, Ao Y, Wang H. Prenatal nicotine exposure induced GDNF/c-Ret pathway repression-related fetal renal dysplasia and adult glomerulosclerosis in male offspring, 2015, 4(4): 1045–1058.
[73] Zhu YN, Chen HY, Zhao XQ, Li B, He HY, Cheng H, Wang H, Ao Y. Decreased H3K9ac level of KLF4 mediates podocyte developmental toxicity induced by prenatal caffeine exposure in male offspring rats, 2019, 314: 63–74.
[74] Sheen JM, Yu HR, Tiao MM, Chen CC, Huang LT, Chang HY, Tain YL. Prenatal dexamethasone-induced programmed hypertension and renal programming, 2015, 132: 41–48.
[75] Jovanovic I, Zivkovic M, Kostic M, Krstic Z, Djuric T, Kolic I, Alavantic D, Stankovic A. Transcriptome-wide based identification of miRs in congenital anomalies of the kidney and urinary tract (CAKUT) in children: the significant upregulation of tissue miR-144 expression, 2016, 14(1): 193.
[76] de Pontual L, Yao E, Callier P, Faivre L, Drouin V, Cariou S, Van Haeringen A, Geneviève D, Goldenberg A, Oufadem M, Manouvrier S, Munnich A, Vidigal JA, Vekemans M, Lyonnet S, Henrion-Caude A, Ventura A, Amiel J. Germline deletion of the miR-17∼92 cluster causes skeletal and growth defects in humans, 2011, 43(10): 1026– 1030.
[77] Patel V, Williams D, Hajarnis S, Hunter R, Pontoglio M, Somlo S, Igarashi P. miR-17~92 miRNA cluster promotes kidney cyst growth in polycystic kidney disease, 2013, 110(26): 10765–10770.
[78] Lakhia R, Hajarnis S, Williams D, Aboudehen K, Yheskel M, Xing C, Hatley ME, Torres VE, Wallace DP, Patel V. MicroRNA-21 aggravates cyst growth in a model of polycystic kidney disease, 2016, 27(8): 2319–2330.
[79] Hönig J, Mižíková I, Nardiello C, Solaligue DES, Daume MJ, Vadász I, Mayer K, Herold S, Günther S, Seeger W, Morty RE. Transmission of microRNA antimiRs to mouse offspringthe maternal-placental-fetal unit, 2020, 24(6): 865–879.
[80] de Barros Sene L, Mesquita FF, de Moraes LN, Santos DC, Carvalho R, Gontijo JAR, Boer PA. Involvement of renal corpuscle microRNA expression on epithelial-to-mesenchymal transition in maternal low protein diet in adult programmed rats, 2013, 8(8): e71310.
The role of miRNA in kidney development
Xiaoqi Zhao, Ying Ao, Haiyun Chen, Hui Wang
MicroRNAs (miRNAs)are endogenous small non-coding RNAs (19–25 nucleotides) that negatively regulate gene expression at the post transcriptional level by binding to complementary target sequences in the target mRNA. miRNAs play an important role in a wide range of biological processes, including organ development. Recent studies have shown that some miRNAs are highly expressed in the kidney and are closely related to kidney development and diseases, suggesting that miRNAs are important regulators in kidney physiology and pathology. This review will focus on the research progress of miRNA in kidney development, and discuss the role of miRNAs in the occurrence and development of renal dysplasia, which will provide a reference for the diagnosis and research of diseases related to kidney development.
microRNAs; kidney development; kidney dysplasia; mechanism; diagnosis
2020-07-16;
2020-09-30
国家自然科学基金项目(编号:81872943, 81220108026, 81430089, 81001466),湖北省卫计委项目(编号:WJ2017M002)和湖北省自然科学基金项目(编号:2017CFB649)资助[Supported by the National Natural Science Foundation of China (Nos. 81872943, 81220108026, 81430089, 81001466), Hubei Province health and Family Planning Scientific Research Project (No. WJ2017M002) ,and the Natural Science Foundation of Hubei Province (No. 2017CFB649)]
赵晓琪,在读硕士研究生,专业方向:肾脏发育毒理。E-mail: xiaoqizhao@whu.edu.cn
敖英,博士,副教授,硕士生导师,研究方向:肾病的发育起源及药物防治,肾脏发育毒理。E-mail: yingao@whu.edu.cn
10.16288/j.yczz.20-112
https://kns.cnki.net/kcms/detail/11.1913.R.20201019.1107.001.html
URI: 2020/10/20 15:09:09
(责任编委: 陈帅)

