藏绵羊BOLL的分子特征及其在睾丸中的表达调控与功能分析
李讨讨,王霞,马友记,2,尹德恩,张勇,赵兴绪
(1甘肃农业大学动物科学技术学院,兰州 730070;2甘肃肉羊繁育生物技术工程实验室,甘肃民勤 733300;3甘肃农业大学动物医学院,兰州 730070)
0 引言
【研究意义】藏绵羊作为我国三大原始绵羊品种之一,是分布于海拔3 000 m以上的青藏高原及其毗邻地区种群数量最多的家畜[1],为当地农牧民提供了极其重要的生活物资和经济收入,并在高寒草甸草地生物多样性及生态系统功能中发挥重要作用。但由于藏绵羊所具有的发育周期长、繁殖力低下、性成熟晚等繁殖生理特点,严重制约了其种群的数量。目前有关雄性藏绵羊睾丸发育及精子发生的分子生物学认识仍十分匮乏。睾丸作为维持雄性动物生殖能力的最关键器官,主要生物学功能是产生雄性配子(精子),而精子发生是精子产生所必需的发育过程,其有序运行是雄性生殖系统建立和维持的前提保障[2]。精子发生过程受到许多基因在多个层面(表观遗传、转录、转录后、翻译、翻译后等)的精密调控[3-4]。研究精子发生相关基因在藏绵羊睾丸发育及精子发生中的表达特征及调控作用,对于理解绵羊及其他高原动物睾丸功能维持的分子调控机理具有重要科学意义。【前人研究进展】BOLL(也称BOULE)作为无精症缺失(Deleted in Azoospermia,DAZ)基因家族中最古老的成员,目前已被广泛报道特异性存在于从昆虫至哺乳动物的性腺中,并且主要在雄性动物睾丸中表达,如锯蝇[5]、果蝇[6]、虹鳟[7]、鸡[8]、小鼠[9]、地鼠[10]、山羊[11]、牛(黄牛、牦牛、犏牛)[12]、猕猴[13]、人[14-16]等。在睾丸发育期间,BOLL在精子发生(尤其减数分裂阶段)中发挥着重要的调控作用,当其表达缺失或功能异常时,可导致精子发生过程停滞,从而因精子生成障碍而造成精液品质不良甚至雄性不育。在BOLL缺失或突变的人和果蝇睾丸中均表现出减数分裂紊乱和停滞,进而导致无精症,并且人的BOLL可以挽救果蝇睾丸中BOLL缺失导致的减数分裂缺陷[14,17],提示BOLL在昆虫和人上具有类似的减数分裂调控作用。反之,在无精症患者睾丸中,也发现BOLL的表达显著降低,并且随着睾丸功能衰竭程度的加深其降低程度愈发明显[18]。在小鼠中,BOLL缺失的睾丸体积明显缩小,虽然精母细胞的减数分裂过程可以正常进行,但减数分裂产生的圆形精细胞丧失进一步发育为成熟精子的能力[9],表明BOLL在精子发生阶段的调控作用因物种而异。此外,在人原始生殖细胞中BOLL的过表达可诱导联会复合体的形成从而进入减数分裂阶段,形成在细胞形态上类似于圆形精细胞的单倍体生殖细胞[15]。与之相类似,在奶山羊雄性生殖干细胞中过表达BOLL也可启动并促进其减数分裂进程[11]。【本研究切入点】当前研究表明,BOLL尽管在雄性动物的精子发生过程中扮演不可或缺的角色,且在很多物种精子发生的减数分裂阶段起关键调控作用,但不难发现,其所执行的生物学功能因物种的不同存在差异,并且目前在藏绵羊睾丸中有关BOLL的序列特征、表达模式及调控作用尚不清楚。【拟解决的关键问题】本研究以藏绵羊作为试验动物,旨在解读BOLL的分子特征及在绵羊睾丸发育过程中的动态表达与分布规律,进而探究其表达调控作用及所赋予的生物学功能,为进一步探索BOLL在绵羊乃至其他哺乳动物睾丸中的时空表达转录调控机制及在精子发生中的作用机理提供依据。
1 材料与方法
试验于2019年3月至2020年8月在甘肃农业大学进行。
1.1 试验动物与样品采集
以甘肃省夏河甘加藏羊养殖合作社提供的 24只健康雄性藏绵羊(同父异母)作为试验动物。试验羊来自相同父本,不同母本,分为3个发育阶段:性成熟前(3月龄,n=8)、性成熟(1岁龄,n=8)和成年(3 岁龄,n=8)。所有羊只在海拔 3 000—3 500 m的天然草场围栏放牧,昼夜自由采食,无补饲。屠宰后收集每只绵羊的右侧睾丸组织,一部分液氮速冻后存放于-80 ℃,另一部分置于4%多聚甲醛溶液中固定用于制作石蜡切片。
1.2 总RNA提取及质检
冷冻的各睾丸组织经液氮快速研磨后,使用Trizol试剂盒(全式金生物公司, 北京)进行总RNA提取;使用NanoDrop ND-2000分光光度计(德国)对提取的 RNA浓度和纯度进行检测。选取 OD260/OD280介于1.8—2.1,OD260/OD230大于2.0的RNA样品,并经 1.5%的琼脂糖凝胶电泳对其完整性进行检测。
1.3 第一链cDNA的合成
以质量和浓度检测合格的RNA样品作为模板,根据反转录试剂盒(Evo M-MLV RT Kit with gDNA Clean for qPCR,艾克瑞,湖南)的操作说明合成第一链cDNA。
1.4 引物设计与合成
从 NCBI数据库中获取绵羊BOLL(XM_004004798.4)和β-actin(NM_001009784.2)的核苷酸序列,随后利用Primer Premier 6.0软件设计引物并送至擎科生物公司(西安)进行合成。
1.5 BOLL的克隆
以合成的cDNA为模板,利用RT-PCR技术进行BOLLCDS区的扩增。扩增总反应体系为 50 μL:2×TransStart FastPfu Fly PCR SuperMix(全式金生物公司,北京)25 μL,PCR Stimulant 10 μL(全式金生物公司,北京),模板 1 μL,上下游引物(BOLL-F:5′-ATG GAGACCGAGTCCGGG-3′,BOLL-R:5′-TTAATAAT GAATGCTCCACAC-3′)各 2 μL, ddH2O 10 μL。PCR扩增程序:95 °C 5 min;95 °C 30 s,55 °C 30 s,72 °C 1 min,30 个循环;72 °C 5 min。对 PCR 产物进行 1%琼脂糖凝胶电泳分离后,用胶回收试剂盒对目的扩增产物进行纯化并回收,随后与pEASY-Blunt载体相连并转化Trans1-T1化学感受态细胞。随机挑取8个独立的阳性克隆通过菌落PCR法鉴定后,由擎科生物公司(西安)进行测序。
1.6 生物信息学分析
利用BLAST工具将获得的藏绵羊BOLL序列与NCBI中现有的核苷酸序列数据库进行比对,以确定最近的亲缘关系和序列相似度,并用 DNAMAN 8.0软件对不同物种 BOLL的氨基酸序列同源性进行比较。使用NCBI中的ORFfinder在线工具(https://www.ncbi.nlm.nih.gov/orffinder/)对藏绵羊CDS区所有开放阅读框(ORF)进行查找分析。分别利用 ProtParam(https://web.expasy.org/protparam/)、InterPro(http://www.ebi.ac.uk/interpro/)、SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和 Phyre 2.0(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)在线软件对藏绵羊BOLL蛋白的理化性质、功能结构域、二级结构和三级结构进行分析。用 WebLogo 在线工具(http://weblogo.berkeley.edu/logo.cgi)对结构域区段的核苷酸和氨基酸序列保守性进行分析。该蛋白的跨膜区和信号肽区分别通过 TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM- 2.0)和 SignalP 4.0(http://www.cbs.dtu.dk/ services/SignalP-4.0/)软件进行预测。采用邻接算法通过MEGA 7.0软件构建BOLL蛋白的系统发育树。采用 STRING 11.0数据库(https://stringdb.org)对与绵羊BOLL蛋白相互作用的潜在蛋白质进行分析,并使用Cytoscape 3.8.0软件进行可视化。
1.7 qRT-PCR 分析
采用qRT-PCR技术在罗氏LightCycler 96荧光定量PCR仪(瑞士)上对各年龄组睾丸中BOLLmRNA的表达量进行实时定量检测。以β-actin作为内参。采用两步法反应程序:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,共 40 个循环。反应体系如下:1 μL cDNA, 每 0.4 μL上下游引物(表 1),10 μL 2×SYBR GreenPro TaqHS Premix(艾克瑞,湖南),补充ddH2O至20 μL。每个样本设计3个重复。

表1 qRT-PCR引物信息Table 1 Primer information used for qRT-PCR
1.8 Western blot 检测
使用含PMSF的RIPA裂解液(索莱宝生物公司,北京)提取各睾丸组织总蛋白,BCA试剂盒(碧云天生物公司,上海)对其浓度进行测定,并经 5×SDS上样缓冲液(索莱宝生物公司,北京)变性后,用12%SDS-PAGE进行电泳分离。电泳结束后,转移蛋白至PVDF膜上,并经5%脱脂奶粉室温封闭2.5 h后,用一抗BOLL(稀释比1∶500;博奥森,北京)或β-actin(稀释比1∶1 500;博奥森,北京)4 ℃孵育过夜。0.1% PBST洗膜后,添加山羊抗兔二抗(稀释比1∶5 000;博奥森,北京)孵育2 h。使用ECL发光液进行化学发光、曝光和显影。用AlphaEaseFC软件对条带灰度值进行量化,并对BOLL蛋白的相对表达丰度进行计算。
1.9 免疫荧光染色
使用常规方法对石蜡切片进行脱蜡、梯度乙醇脱水后,用微波炉加热法加入EDTA抗原修复缓冲液对组织切片进行抗原修复。5%BSA室温封闭30 min后,添加一抗 BOLL(1∶200;博奥森,北京)4 ℃孵育过夜。同时用5%BSA代替一抗BOLL作为阴性对照。PBS清洗后,切片中滴加带有FITC绿色荧光标签的山羊抗兔二抗(稀释比1∶200;赛维尔,武汉),室温避光孵育1 h。PBS冲洗后,DAPI核染10 min,用含抗荧光猝灭剂的溶液封片,荧光显微镜(尼康,日本)观察并拍照。
1.10 绵羊BOLL的功能注释与ceRNA调控网络分析
利用非冗余蛋白质(NR)数据库和基因本体论(GO)数据库,对绵羊BOLL所参与的生物学过程或功能进行注释。基于课题组前期通过 RNA测序(RNA-seq)技术获得的藏绵羊睾丸组织全转录组测序数据,利用 RNAhybrid 2.1.2(https://bibiserv.cebitec.uni-bielefeld)+SVM_light 6.01(http://svmlight.joachims.org/),Miranda 3.3a(http://www.microrna.org/microrna/home)和 TargetScan 7.0(http://www.targetscan.org/)数据库对潜在的调控BOLL的长链非编码RNA(long noncoding RNA,lncRNA)/环状 RNA(circular RNA,circRNA)-微小 RNA(microRNA,miRNA)-BOLL作用网络进行预测分析,并对上述数据库的预测结果取交集作为 ceRNA整合分析结果,使用 Cytoscape 3.8.0软件对网络进行可视化。
1.11 潜在靶向调控BOLL的miRNAs及ceRNAs表达量验证
对通过上述 ceRNA网络分析获得的潜在调控BOLL的lncRNAs、circRNAs和miRNAs表达量进行qRT-PCR验证,并与RNA-seq结果进行比较。以1.2中提取的总RNA为模板,根据Evo M-MLV RT Kit with gDNA Clean for qPCR(艾克瑞,湖南)和Mir-XTM miRNA First-Strand Synthesis Kit(Takara,日本)反转录试剂盒操作说明分别合成用于 lncRNA/circRNA和miRNA qRT-PCR扩增的第一链cDNA。qRT-PCR操作方法同1.7所述。运用Primer Premier 6.0软件设计lncRNA引物和circRNA发散引物,并经Blast对其特异性检测;采用加尾法设计miRNA的上游引物。引物(表1)均由擎科生物公司(西安)进行合成。每个样本设计 3个重复。分别以β-actin和 U6作为内参对lncRNA/circRNA和miRNA的表达量进行校正。
1.12 荧光素酶报告基因载体构建及双荧光素酶活性检测
将分别含有 oar-miR-760-3p、oar-miR-127-5p和oar-miR-382-5p假定靶结合位点的绵羊源BOLL3′UTR区序列(XM_004004798.4),即BOLL-m760-3′UTR WT(核苷酸位点 1539-1843)、BOLL-m127-3′UTR WT(核苷酸位点 1599-1918)和 BOLL-m382-3′UTR WT(核苷酸位点 2430-2769);含有 oar-miR-760-3p假定靶结合位点的circ-SPHKAP、Circ-ECT2L、LOC105602204(XR_001020768,核苷酸位点 16-325)、LOC105603195(XR_001022217,核苷酸位点50-369)和 LOC105616228(XR_001043211,核苷酸位点190-494)野生型序列;以及它们分别对应的突变型(MUT)序列克隆至pmirGLO双荧光素酶miRNA靶向表达载体(Promega, 美国)的XhoI和SalI位点。上述序列均由金唯智生物科技有限公司(苏州)合成。miR-760-3p、miR-127-5p和 miR-382-5p的模拟物(mimic)及其阴性对照物(mimic NC)由吉玛制药技术有限公司(上海)合成。复苏购自北京北纳生物的 HEK-293T细胞,以 1.0×105/孔的密度接种于 24孔板中,使用含10% FBS的DMEM高糖培养基,在5% CO2、37℃恒温箱静置培养24 h后(汇合率为70%—80%),采用 LipofectamineTM 2000转染试剂盒(Invitrogen, 美国),按照操作说明将构建的野生型及突变型重组双荧光素酶报告载体分别与对应的mimics或 mimic NC 共转染 293T 细胞。48 h 后收集细胞,采用 E1910双荧光素酶报告基因检测试剂盒(Promega, 美国)分析相对荧光素酶活性,结果以萤火虫荧光素酶活性与海肾素荧光素酶活性的比值来表示。每个试验设置3个重复。
1.13 数据统计分析
采 用 2–ΔΔCt法 ( ΔCt=CtmRNAcircRNA/lncRNA/miRNACtβ-actin/U6)对 qRT-PCR 结果进行计算。用 SPSS21.0统计软件进行单因素方差分析,结果用均值±标准差(Mean±SD)表示。采用 Duncan多重检验比较各组间的差异,P<0.05和P<0.01分别表示组间差异显著和极显著。
2 结果
2.1 藏绵羊BOLL完整CDS区序列特征
绵羊BOLL位于2号常染色体上,总长为63 155 bp,由11个外显子和10个内含子组成(图1-A)。1%琼脂糖凝胶电泳显示,PCR扩增的cDNA片段大小位于预期位置(图1-B)。对该cDNA片段进行克隆并测序,获得了长度为888 bp的核苷酸序列,对应的序列信息已提交至 GenBank(Accession No. MT424763)。经与NCBI中的核苷酸序列数据库进行Blast检索比对发现,与设计克隆引物所用绵羊BOLLCDS区序列(XM_004004798.4)完全一致。该克隆序列存在14个ORF,其中全长开放阅读框ORF1(即完整CDS区)为888 bp,可编码295个氨基酸(图1-C、D)。藏绵羊BOLL编码蛋白的分子量、等电点(pI)和分子式分别为32.43 kD、6.07和C1459H2217N379O440S10。该蛋白序列不含信号肽和跨膜区域。
2.2 藏绵羊与其他哺乳动物BOLL的同源性比较和结构域分析
不同哺乳动物BOLL的氨基酸序列多重比较结果发现:藏绵羊与山羊的序列相似度最高(100%),其次为川南山地黄牛(99.66%)、牦牛(99.66%)、水牛(98.98%)、威德海豹(98.31%)、白尾鹿(97.97%)、非洲象(97.97%)、家猫(97.63%)、单峰驼(97.29%)、瘤牛(97.29%)、双峰驼(97.29%)、灰熊(96.95%)、北极熊(96.95%)、家马(96.95%)、野猪(96.95%)、北海狮(96.61%)、家犬(96.27%)和抹香鲸(95.93%),与兔的序列相似度最低(94.92%)(图2-A)。结构域分析发现,在藏绵羊 BOLL氨基酸序列靠近 N端43—123位点处(核苷酸序列位点127—369)和172—196处(核苷酸序列位点514—588)分别存在RNA识别基序(RNA recognition motif,RRM)结构域和DAZ重复基序,并且对应的核苷酸和氨基酸序列在不同哺乳动物间具有高度保守性(图2-B—E)。用系统进化树对来自20种哺乳动物的BOLL氨基酸序列进行分析发现,藏绵羊与山羊聚为一支,在进化上与山羊、水牛、牦牛和川南山地黄牛的亲缘关系较近,这与氨基酸同源性比较结果基本一致(图3)。

图1 藏绵羊BOLL CDS区克隆及序列分析Fig. 1 Cloning and sequence analysis of Tibetan sheep BOLL CDS region
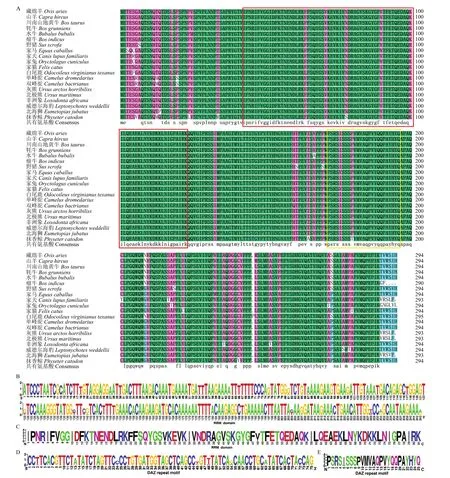
图2 不同哺乳动物BOLL CDS区序列特征比较分析Fig. 2 Comparative analysis of BOLL CDS sequence features among different mammals
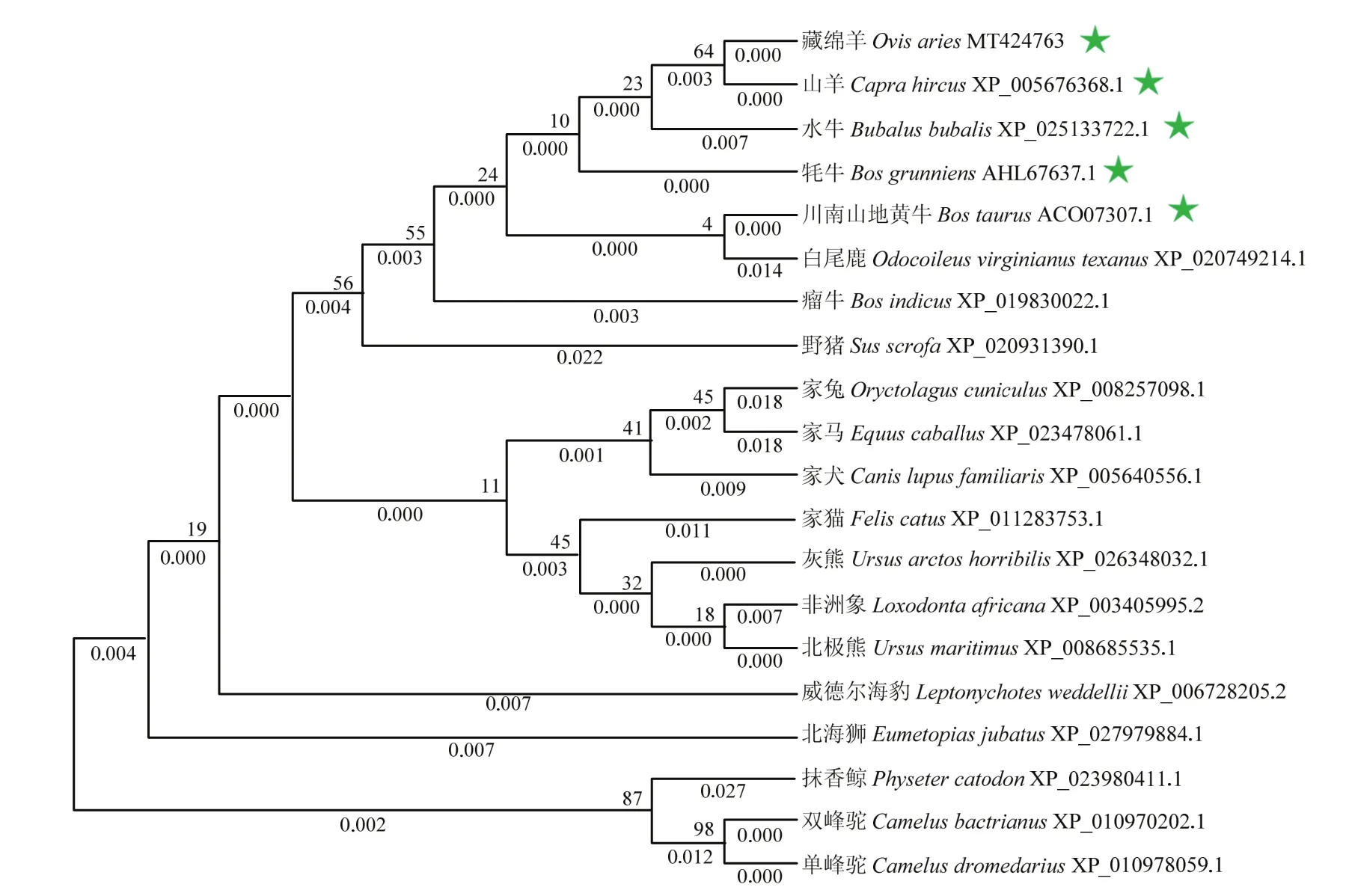
图3 基于邻接算法的BOLL蛋白氨基酸序列系统进化树Fig. 3 Phylogenetic tree for amino acid sequences of BOLL protein based on Neighbor-joining algorithm
2.3 藏绵羊BOLL蛋白空间结构分析
藏绵羊BOLL蛋白的二级结构由无规则卷曲、延伸链、α螺旋和β转角4种结构组成,分别占64.07%、18.64%、13.56%和3.73%(图 4-A)。BOLL蛋白的三级分子结构预测结果与其二级结构组成相似,主要含无规则卷曲、延伸链、α螺旋和β转角4种结构类型(图4-B)。
2.4 与绵羊BOLL蛋白互作蛋白分析
由蛋白相互作用网络分析结果(图 5)可知,绵羊BOLL编码蛋白与10个蛋白可能存在相互作用,包括细胞质多腺苷酸化元件结合蛋白 1(CPEB1)、细胞分裂周期蛋白 25A(CDC25A)、细胞分裂周期蛋白20B(CDC20B)、DEAD家族的RNA解螺旋酶4(DDX4,也称VASA)、生长抑制蛋白家族成员2(ING2)、RNA结合基序蛋白25(RBM25)、核转运蛋白2(KPNA2)、核转运蛋白7(KPNA7)、鸟氨酸脱羧酶抗酶 2(OAZ2)、泛素样修饰活化酶 5(UBA5)。BOLL蛋白处于网络核心位置,且与CPEB1、UBA5和CDC20B蛋白间的相互作用具有更高的可信度。
2.5 BOLL的表达模式
由图6可知:在mRNA和蛋白质水平,3月龄藏绵羊睾丸中BOLL表达量均较低;与3月龄相比,1岁龄和3岁龄睾丸中的BOLL mRNA和蛋白表达量极显著上调(P<0.01);相比1岁龄,3岁龄睾丸中BOLLmRNA表达呈上调趋势,而蛋白呈现一定的下调趋势。
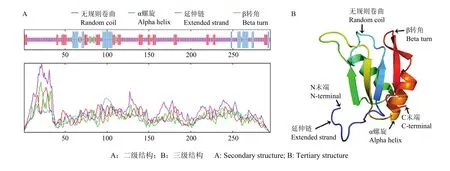
图4 藏绵羊BOLL蛋白的空间结构Fig. 4 Spatial structure of Tibetan sheep BOLL protein
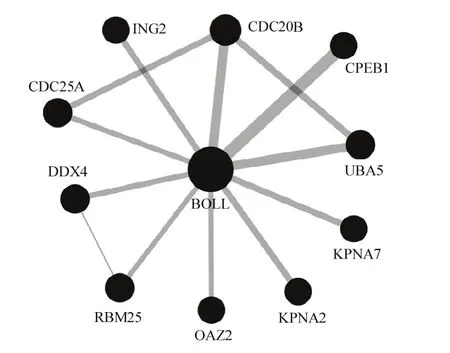
图5 与绵羊BOLL蛋白互作蛋白网络分析Fig. 5 Network analysis of the proteins interacting with ovine BOLL protein
2.6 BOLL蛋白的免疫荧光染色
由BOLL蛋白的阳性免疫染色结果(图7)可知:在3月龄藏绵羊睾丸中,较弱的BOLL蛋白阳性信号定位于部分精原细胞;在1岁龄和3岁龄睾丸中,强的BOLL蛋白阳性信号主要定位于长形精细胞中,其次为圆形精细胞,也有较弱的阳性信号被发现存在于精原细胞和精母细胞中,并且随着年龄的增加,在精原细胞和精母细胞中的阳性信号强度减弱。
2.7 绵羊BOLL潜在的ceRNA表达调控网络及功能注释
通过对BOLL的功能注释结果发现:在分子功能方面,其主要涉及结合、翻译调节活性等;在生物学过程方面,其主要参与组织细胞发育、生殖、配子生成、基因转录后及翻译起始调控等过程(图8)。ceRNA网络分析发现:绵羊BOLL受到3个潜在的miRNAs(oar-miR-127-5p、oar-miR-760-3p 和 oar-miR-382-5p)靶向调控,并且 3个 lncRNAs(LOC105602204、LOC105603195和 LOC105616228)和 2个 circRNAs(circ-ECT2L和circ-SPHKAP)分子可与BOLL共同竞争性结合oar-miR-760-3p(图8)。
2.8 潜在调控BOLL的非编码RNAs在睾丸中的表达特征
实时荧光定量PCR结果(图9-A)显示:随着年龄的增加,oar-miR-127-5p、oar-miR-382-5p和oar-miR-760-3p的表达量逐渐下降;LOC105616228和circ-ECT2L的表达量逐渐上调;LOC105602204、LOC105603195和circ-SPHKAP的表达量先上调而后呈现不同程度的下调趋势。由图9-A可知,qRT-PCR与RNA-seq获得的各非编码RNAs在睾丸中的表达模式基本一致:相比3月龄,在1岁龄和3岁龄睾丸中各 miRNAs的表达量极显著下调(P<0.01),而lncRNAs和circRNAs的表达量极显著上调(P<0.01)。
2.9 BOLL-miRNAs及miRNAs-circRNAs/lncRNAs靶向关系验证
由双荧光素酶报告基因检测结果(图9-B)可知,相比对照组,oar-miR-127-5p和oar-miR-760-3p模拟物均显著降低了293T细胞中的BOLL3′UTR野生型报告者的荧光素酶活性(P<0.01),并且oar-miR-760-3p模拟物显著降低了野生型Circ-ECT2L(P<0.05)以及LOC105616228(P<0.01)报告者的荧光素酶活性,但对相应的突变型报告者均无明显影响。oar-miR-382-5p模拟物对BOLL3′UTR野生型报告基因的荧光素酶活性无明显影响(P>0.05),oar-miR-760-3p模拟物对野生型 circ-SPHKAP、LOC105602204和LOC105603195报告基因的荧光素酶活性也无明显影响(P>0.05;结果未显示)。这些结果表明,绵羊BOLL是oar-miR-127-5p和oar-miR-760-3p共有的直接作用靶标,并且 circRNA Circ-ECT2L和 lncRNA LOC105616228作为ceRNAs, 可与BOLL共同竞争性靶向结合oar-miR-760-3p。
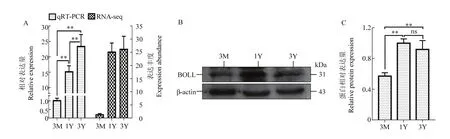
图6 发育的藏绵羊睾丸中BOLL mRNA和蛋白的表达规律Fig. 6 Expression patterns of BOLL mRNA and protein in developmental Tibetan sheep testes
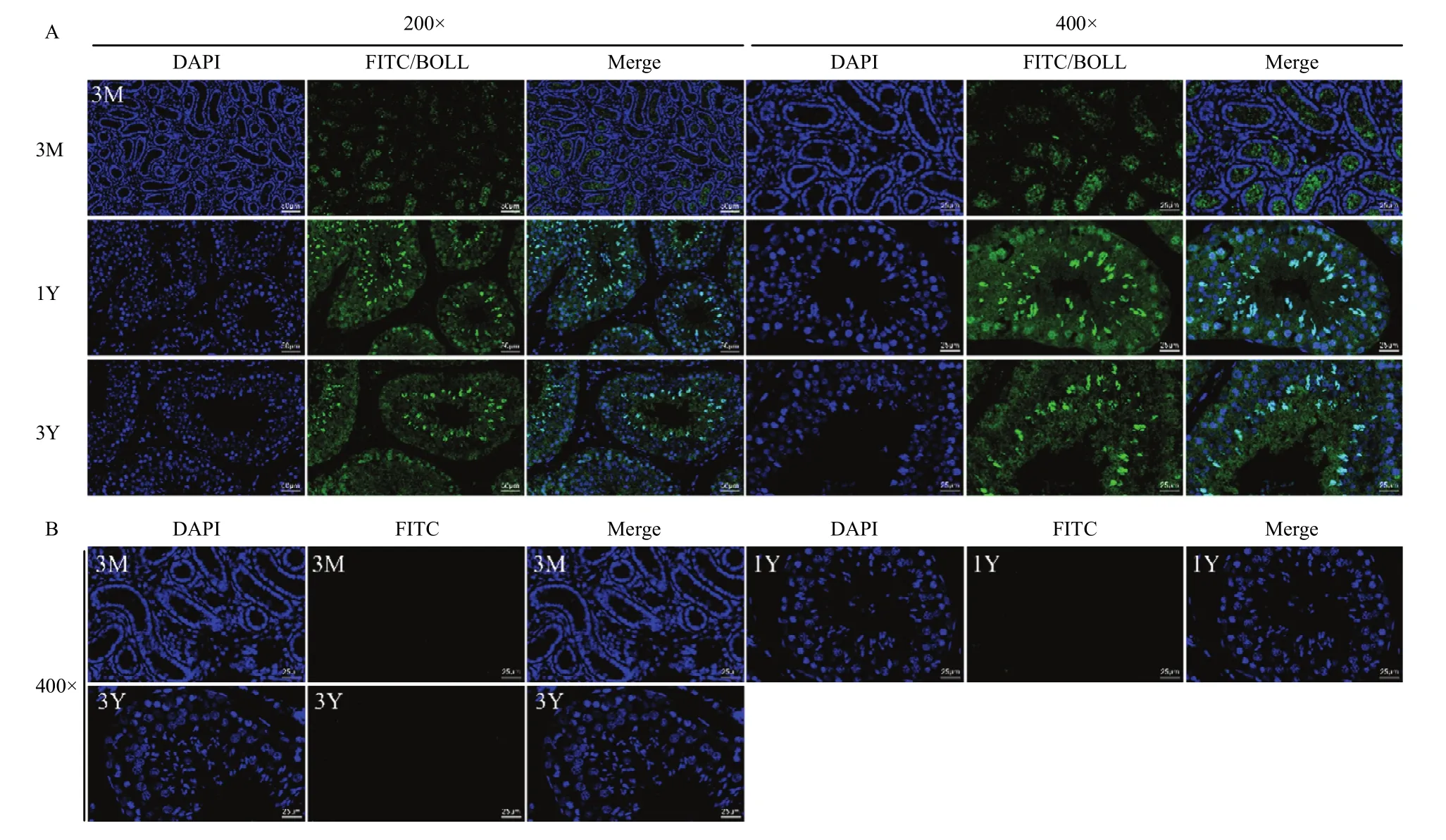
图7 藏绵羊睾丸中BOLL蛋白的免疫荧光染色Fig. 7 Immunofluorescence staining of BOLL protein in Tibetan sheep testis
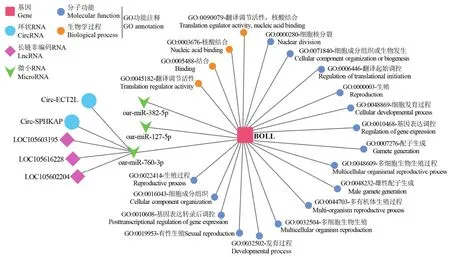
图8 绵羊BOLL的功能注释及潜在的ceRNA调控网络Fig. 8 The functional annotation and potential ceRNA regulatory network for ovine BOLL
3 讨论
首先通过克隆并测序获得了藏绵羊BOLL的完整 CDS区,由 888 bp的核苷酸组成,可编码 295个氨基酸,这与 NCBI中提供的绵羊预测序列(XM_004004798.4)完全一致,并且与在山羊[19]、川南山地黄牛[12]和牦牛[20]上报道的该基因 CDS区长度一致。通过氨基酸序列比对发现,藏绵羊BOLL编码区对应的氨基酸序列与山羊、川南山地黄牛和牦牛的相似度均在99%以上,与其他哺乳动物的序列相似度也高于95%(兔除外)。在进化上,其与山羊、牦牛、川南山地黄牛也具有比较近的亲缘关系。这些结果表明,藏绵羊BOLL同其他哺乳动物具有高度的序列同源性和进化保守性。
DAZ家族成员(包括BOLL)所编码蛋白中均含有一个 RRM 结构域和至少一段 DAZ重复基序[6]。RRM结构域作为真核生物细胞中最丰富的RNA结合元件,可通过与其他核酸或蛋白质相互作用,以调节mRNA的转录、翻译和衰变等环节而参与基因表达的精细调控(尤其转录后调控),进而发挥相应的生物学功能[21]。但对于DAZ重复基序,其在DAZ家族基因编码蛋白中所承担的具体功能目前仍不清楚。YEN等[22]依据自己的研究结果推测,DAZ重复基序可能参与RNA结合,也可能参与与其他蛋白质的相互作用。EWIS等[23]研究发现,DAZ重复基序的缺失可造成男性生精能力下降。综上所述,DAZ重复基序可能具有与 RRM结构域相似的功能,即通过与其他蛋白质分子结合从而贡献于DAZ家族各成员自身的功能维持。与其他哺乳动物一样,本研究通过氨基酸序列分析表明,藏绵羊BOLL蛋白也具有一个RRM结构域(靠近N端)以及一段由25个氨基酸残基组成的DAZ重复基序,并且二者对应的氨基酸序列在不同物种中高度相似。由此推测,藏绵羊BOLL蛋白所含的RRM结构域和 DAZ重复基序在进化和功能上是非常保守的,并且可能是其执行并维持正常的生物学功能所必需的。
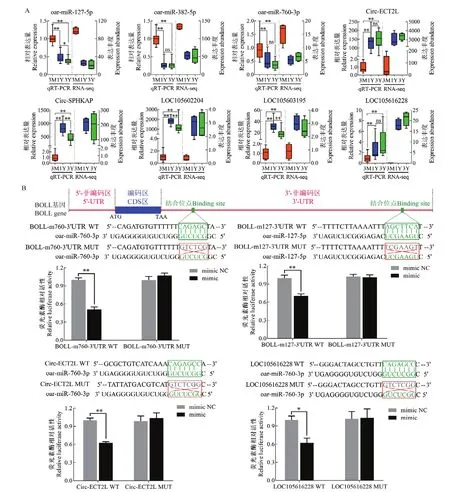
图9 潜在调控BOLL的非编码RNAs的时间表达模式及靶向关系验证Fig. 9 Temporal expression patterns of potential regulatory non-coding RNAs for BOLL gene and verification of the target relationship
鉴于BOLL蛋白可通过结合相应的蛋白质而发挥作用,本研究进一步借助STRING数据库对潜在的与绵羊BOLL蛋白互作的基因编码蛋白进行分析发现,CDC25A、CDC20B 等 10个蛋白质分子与 BOLL蛋白可能存在相互作用。其中,CDC25A和CDC20B作为细胞周期相关蛋白,被报道在哺乳动物精子发生中广泛参与生殖细胞的有丝分裂和减数分裂等过程[24-25]。在成年雄性小鼠睾丸中发现,BOLL可与CDC25AmRNA靶向结合以调节其翻译过程,从而对精母细胞的减数分裂进行调控[9]。LI等[11]在山羊上研究发现,过表达BOLL可通过上调减数分裂相关基因CDC25A的表达,进而启动山羊生殖干细胞的减数分裂过程。与细胞周期蛋白所发挥的功能类似,OAZ2和DDX4也被报道在雄性哺乳动物生殖细胞中表达并参与调控细胞的增殖与分化[26-27]。在女性原始生殖细胞中过表达BOLL可增加DDX4的表达,进而促进生殖细胞的减数分裂进程及单倍体卵子的形成[15]。同DAZ家族成员一样,CPEB1也是一种RNA结合蛋白,已被报道在小鼠卵母细胞成熟过程中,可靶向调控DAZ家族中另一成员DAZL的mRNA翻译和蛋白积累,并且CPEB1和DAZL可通过调节共同靶标mRNA的翻译表达从而对卵母细胞的减数分裂进行正向调控[28]。CPEB1的缺失不仅造成雌性小鼠不育,也可导致雄鼠精子发生停滞在减数分裂阶段而严重损害其生育力[29]。类似于CPEB1在睾丸中的调节功能,ING2可通过与相应的靶标结合参与精子发生过程,其缺失也可导致精子生成异常甚至雄性不育[30]。UBA5是泛素激活酶 E1的主要成员之一,而KPNA2和KPNA7均属于核转运蛋白。泛素化作为广泛存在于真核生物细胞内的一种蛋白质翻译后修饰,在精子发生过程中可将泛素分子结合到靶蛋白上,以参与调节蛋白质的稳定性和功能[31-32]。同样,在雄性生殖细胞发育的特定阶段,核转运蛋白通过与不同的转录因子或蛋白质结合以转运其穿过核孔参与调控生殖细胞分化和精子发生[33]。RBM25作为RNA结合基序蛋白家族中的一员,被报道在小鼠睾丸精子细胞中特异表达[34]。综上,这些潜在与绵羊BOLL互作的蛋白分子均在雄性动物精子发生过程中表达并扮演不同的角色,且大多已被报道具有结合并调节其他蛋白质分子的能力。这为深入理解BOLL在哺乳动物,尤其绵羊睾丸发育期间的分子作用机理提供了新的见解和思路,但这些蛋白是否与BOLL存在相互作用并且通过何种机制贡献于生精细胞的表型维持及精子生成,仍有待进一步探索。
本研究发现,在性成熟后藏绵羊睾丸中BOLL在转录和蛋白质水平的表达均显著上调,这与以前研究报道其在具有正常生精能力(性成熟后)的哺乳动物睾丸中高表达[35-36]的结果相符,表明BOLL可能主要在性成熟后藏绵羊睾丸中发挥作用。尽管BOLL是一个减数分裂相关基因,但在睾丸中的表达不仅仅局限于减数分裂阶段的精母细胞中。如在成年小鼠睾丸中,BOLL蛋白不仅存在于精母细胞中,也在圆形和长形精细胞、精子及部分精原细胞中分布[37]。在地鼠睾丸中,BOLL蛋白除了在精原细胞、精母细胞和圆形精细胞中表达外,在睾丸间质细胞中也有分布[10]。为了明确BOLL蛋白在发育的藏绵羊睾丸细胞中所呈现的表达特征及发挥的生物学功能,本研究进一步通过免疫荧光染色发现,BOLL蛋白在性成熟前睾丸中存在于部分精原细胞中,而在性成熟后主要分布于精细胞中,在精母细胞和精原细胞中也有较弱的阳性信号且随着年龄的增加信号强度更加弱化。这与在小鼠睾丸中[9],研究发现该基因在圆形精细胞中高表达并发挥作用的结果类似,提示BOLL可能主要在藏绵羊精子发生中参与调控减数分裂后的单倍体精子细胞进一步向成熟精子发育。这也与在小尾寒羊(低海拔、性成熟早、繁殖力高)[36]和地鼠[10]上报道BOLL蛋白在睾丸生殖细胞中的分布特征基本一致,但与在小尾寒羊上研究结果不同的是,BOLL蛋白在性成熟后小尾寒羊睾丸中主要表达于精母细胞和圆形精细胞中。此外,有趣的是,在藏绵羊睾丸中,随着年龄的增加,BOLL在转录水平的表达逐渐上调,而在翻译水平的表达先(性成熟)上调而后(成年)出现下调,这与在转录和翻译水平都发现BOLL在成年小尾寒羊睾丸中表达上调(相比性成熟阶段)[36]的结果不一致。这些差异可能是由于基因的表达调控机制和表观遗传机制因品种的繁殖力和生存环境不同而异所造成的。已有研究报道,在具有不同繁殖力的绵羊品种卵巢中许多基因的表达及调控作用存在差异[38]。正如在雄性不育的人[18]和犏牛[20]睾丸中发现的BOLL表达很微弱甚至不表达,在发育的绵羊睾丸中,相比同处于成年期(精子产生旺盛期)的具有高繁殖力的小尾寒羊[36],藏绵羊睾丸内BOLL蛋白的表达呈现一定的下降趋势,这可能与其繁殖力较低有关,但其具体作用机制的差异性还需进一步研究与证实。这为从细胞和分子水平探究不同繁殖力的种公羊品种在睾丸发育分子遗传机理方面的差异提供了思路。
在藏绵羊睾丸中BOLL mRNA和蛋白的表达趋势及表达改变倍数存在差异,这可能是由于基因的表达受到转录、转录后等水平的严格调控所造成的。非编码 RNAs(lncRNAs、miRNAs、circRNAs等)作为转录调节因子,在转录、转录后、表观遗传等层面参与调控基因的表达[39]。同样,哺乳动物精子发生过程也受到许多非编码RNAs的调控[40-41]。有研究发现,在斑马鱼原始生殖细胞中miR-430可靶向抑制DAZL(与BOLL属同一家族)的mRNA表达[42],但有关靶向调控BOLL的miRNAs研究仍处于空白阶段。本研究基于前期有关藏绵羊睾丸组织的全转录组测序数据并借助相关数据库对潜在调控BOLL的非编码RNAs,即ceRNAs作用网络进行了鉴定与分析,并通过qRT-PCR和双荧光素酶报告实验对其表达特征和靶向关系进行了验证。发现,oar-miR-127-5p和 oar-miR-760-3p可直接靶向调控藏绵羊BOLL的表达并且 circRNA Circ-ECT2L和lncRNA LOC105616228可通过竞争性结合 oar-miR-760-3p而调控BOLL的表达。为了进一步理解ceRNA介导调控绵羊BOLL所涉及的生物学功能,对BOLL参与的生物学过程和功能进行了详细注释,发现其主要参与配子生成、细胞分裂、基因转录后及翻译起始调控等生物学过程。以上结果表明,在藏绵羊睾丸中,BOLL的表达受miRNAs及ceRNAs调控,进而可能与可激活下游信号或事件的蛋白分子相互作用,以响应生精细胞的发育及精子生成。该研究填补了在绵羊睾丸中有关BOLL表达与调控作用认识的空白,丰富了对其在绵羊甚至其他哺乳动物精子发生中潜在的分子作用机制的理解,并为进一步的功能探究提供了新的理论视角。
4 结论
本研究成功克隆获得藏绵羊BOLL完整CDS区序列,其在哺乳动物间具有高度进化保守性,并发现该基因主要表达于性成熟后藏绵羊睾丸精子细胞中,且在精母细胞和整个发育阶段的精原细胞中也有表达。oar-miR-127-5p和oar-miR-760-3p直接靶向抑制绵羊BOLL的表达;circRNA Circ-ECT2L和 lncRNA LOC105616228作为ceRNAs,可与BOLL共同竞争性吸附oar-miR-760-3p而正向调控绵羊BOLL的表达,进而通过与下游分子相互作用以激活细胞增殖或分化相关信号事件来调控精子发生,尤其减数分裂后的单倍体精子细胞的进一步发育。

