lnc-MTBP-5 在结直肠癌中的表达及其对细胞侵袭能力的影响
严宇青,沈超琴,陈豪燕,洪 洁,王震华
上海交通大学医学院附属仁济医院消化科,上海市消化疾病研究所,上海 200001
研究[1-2]显示,结直肠癌(colorectal cancer,CRC)的发病率及病死率分别位居恶性肿瘤的第3、4 位,是常见的消化系统恶性肿瘤,也是导致癌症死亡的第二大病因。CRC 患者的死亡与病灶转移密切相关,据统计患者因转移而发生死亡的数量分别占初诊和确诊人数的25%和50%[3]。对于CRC 转移的患者,切除原发灶和转移灶是最佳的治疗方案,但由于癌症细胞的潜伏性、播散性和抗药性,患者于术后复发十分常见[4]。因此,对CRC 转移的具体机制及其诊治标志物的研究,已成为当前关注的焦点。
在人类基因组中,细胞仅利用其中的2%来产生具有蛋白质编码功能的转录本,其余大部分不编码蛋白质的基因则被转录为非编码RNA。其中,长链非编码RNA(long noncoding RNA,lncRNA)是长度大于200 个核苷酸的非编码转录本[5]。lncRNA 可定位在胞核内或胞质中。在分子水平上,lncRNA 通过不同机制调节染色质组成、基因转录和转录后修饰[6]。在许多肿瘤尤其是恶性肿瘤中,如胃癌、乳腺癌、前列腺癌、肾癌等[7-10],lncRNA 的表达可发生改变。
基于此,我们假设有特定lncRNA 参与CRC 的转移,通过筛选CRC 相关的lnc-MTBP-5,就该lncRNA 对人CRC 细胞侵袭能力的影响进行检测,以期为CRC 的早期诊断及治疗提供新的靶点。
1 资料与方法
1.1 数据收集
1.1.1 数据集获取 从SRA(Sequence ReadArchive)数据库中提取PRJNA218851 数据集和PRJNA376161 数据集。前者包括18 例CRC 患者的正常结肠黏膜、CRC 原发灶及肝转移灶的数据,后者包括10 对肿瘤组织和正常结肠组织、5 对异常隐窝灶和匹配普通隐窝灶的数据。SRA数据库信息均采用R3.5.2 软件提取,用以筛选与CRC 转移相关的lncRNA。
1.1.2 组织芯片数据获取 在癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中提取含有638 例CRC 组织芯片和51 例正常结直肠组织芯片的数据,分析lnc-MTBP-5在正常结直肠组织和CRC 组织中的表达差异。根据lnc-MTBP-5在CRC 患者癌组织表达量的中位数,将TCGA 数据库中的638 例患者分为lnc-MTBP-5高表达组和lnc-MTBP-5低表达组。从TCGA 数据库中提取CRC 患者的临床信息,包括初次病理诊断年龄、性别、美国癌症联合委员会(American Joint Committee on Cancer,AJCC)临床分级、淋巴结转移、远处转移、脉管侵及、结肠息肉病史和治疗后复发,分析lnc-MTBP-5与患者临床预后的相关性。TCGA 数据库CRC 组织芯片信息均采用R3.5.2软件提取。
1.2 标本收集
选择2016 年1 月—2017 年12 月于上海交通大学医学院附属仁济医院的53 例CRC 手术患者,从癌组织和癌旁组织获取标本,并于-80 ℃长期保存。本研究经上海交通大学医学院附属仁济医院伦理委员会批准(伦理审批号为[2015]097),所有参与者均签署了知情同意书。
1.3 细胞和主要试剂
人正常结直肠黏膜FHC 细胞以及人CRC 细胞HT29、SW480、DLD1、HCT116、SW1116、Lovo、RKO 和Caco2 均购自美国模式菌种保藏中心(American Type Culture Collection,ATCC)。
RPMI-1640 培养液、McCoy's 5A 培养液、OptiMEM培养液和胎牛血清均购自美国Gibco 公司,DharmaFECT 1 转染试剂购自美国GE Healthcare 公司,RNAiso Plus、反转录试剂和实时荧光定量PCR 试剂盒均购自日本TaKaRa公司,PCR 引物由生工生物工程(上海)股份有限公司合成,CCK-8 检测试剂盒购自日本同仁化学研究所,Transwell 小室购自美国Millipore 公司,Matrigel 胶购自美国BD Bioscience 公司。
1.4 CRC 细胞培养及转染实验
分别采用含5%胎牛血清的McCoy's 5A 完全培养基和RPMI-1640 完全培养基,于37 ℃、5%CO2的培养箱中培养HT29、SW480 细胞,于3 d 后传代。待细胞生长至指数生长期时,分别以3×105个/孔、2×105个/孔接种于6 孔板中,用于后续转染。
用Control siRNA、lnc-MTBP-5siRNA 分 别 转 染HT29 和SW480 细胞,并用正常表达lnc-MTBP-5的质粒(pcDNA3.1)、lnc-MTBP-5过表达质粒(pcDNA3.1-lnc-MTBP-5)分别转染SW480 细胞。于6 孔板中继续分别培养上述细胞24 h 后,将培养液更换为无血清培养基。于37 ℃培养6 h 后将培养基更换为完全培养基,继续培养细胞48 h 后提取RNA,用于后续研究。转染实验siRNA 序列见表1。

表1 siRNA 序列Tab 1 Sequence of siRNA
1.5 细胞和组织中lnc-MTBP-5 的表达量检测
采用实时荧光定量PCR(quantitative real-time PCR,qPCR)检测lnc-MTBP-5的表达量。采用磷酸盐缓冲液洗涤FHC 细胞、8 种CRC 细胞及转染的HT29、SW480 细胞各2 次,同时将53 例结直肠癌组织和癌旁组织标本研磨成粉末后,按照操作说明抽提总RNA。细胞质/细胞核RNA 抽提按照试剂盒说明操作。分别取1 μg 上述RNA,用PrimeScript RT Reagent Kit 试剂盒反转录成cDNA,并以此为模板用SYBR Premix Ex Taq Ⅱ试剂盒、StepOne real-time PCR 系统进行qPCR 检测。反应条件为:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,共40 个循环。以2-△△CT值评估基因的相对表达量,每组实验设置3 个复孔。细胞、组织中总表达量的检测以GAPDH为相对内参,细胞质和细胞核内表达量检测以U6为相对内参。引物序列见表2。
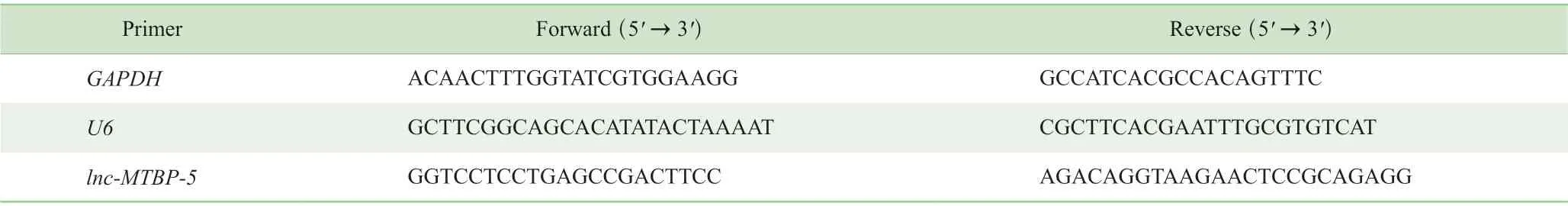
表2 PCR 引物序列Tab 2 Primer sequences for PCR
1.6 细胞增殖能力检测
采用CCK8 实验检测CRC 细胞的增殖能力。将HT29、SW480 细胞接种至96 孔板中,按1.4 方法分别转染Control siRNA 和lnc-MTBP-5siRNA。分别在转染后的第0、1、2、3、4 日,向每孔加入100 μL 含10% CCK8试剂的无血清培养基,于37 ℃孵育2 h 后,用酶标仪测定样品在450 nm 处的吸光度。
1.7 细胞克隆形成能力检测
采用克隆形成实验检测CRC 细胞的增殖能力。将HT29、SW480 细胞以800 个/孔接种至6 孔板中,按1.4方法分别转染Control siRNA 和lnc-MTBP-5siRNA,并静置于培养箱中。7 d 后弃去培养基,经固定染色后拍照,观察克隆形成的大小和数量。
1.8 细胞侵袭能力检测
采用侵袭实验检测SW480 细胞的侵袭能力。将SW480 细胞按2×105个/孔接种于6 孔培养板中,培养24 h 后向其中分别转染Control siRNA 和lnc-MTBP-5siRNA,以及对照质粒和lnc-MTBP-5过表达质粒。继续培养24 h 后进行消化,并用无血清RPMI-1640 培养液重悬细胞。在24 孔培养板中放入Transwell 小室,铺入40 μL Matrigel;4 ~6 h 后向小室下层加入600 μL 含20%胎牛血清的RPMI-1640 培养液,小室上层加入200 μL 上述4 组细胞悬液。继续培养48 h 后取出小室,固定染色并于光学显微镜下进行观察。随机选取4 个视野(放大倍数为40 倍),计数穿过Matrigel 的细胞数,取平均值。
1.9 统计学方法
采用SPSS 22.0 软件对研究数据进行统计分析,所有实验均重复3 次。组间比较采用配对t检验。P<0.05 表示差异具有统计学意义。
2 结果
2.1 SRA 数据库中与CRC 转移相关的lncRNA 分析
采用t检验对PRJNA218851 数据集进行分析,共获得105 条[假发现率(false discovery rate,FDR)<0.05)]在正常-原发灶-转移灶中依次表达上调的lncRNA;在PRJNA376161 数据集中,对10 对肿瘤组织和癌旁正常组织的数据分析得到990 条(FDR<0.05)差异表达lncRNA。对上述lncRNA 取交集,共得到10 条在2 个数据集中均有意义的lncRNA。通过在UCSC(http://genome.ucsc.edu/)中比对序列,我们将关注点放在ENSG00000254814、ENSG00000257453 和ENSG00000272502 这3 条lncRNA上,筛选流程如图1A。由于前2 条lncRNA 的引物未能成功合成,因此仅重点研究ENSG00000272502。通过在LNCipedia 数据库(https://lncipedia.org/)中对该lncRNA的编码潜能进行验证(排除假阳性),最终确认其(lnc-MTBP-5)是一种非编码RNA(图1B)。
在正常肠上皮细胞和8 个结直肠癌细胞系中对lnc-MTBP-5表达量进行检测,结果显示lnc-MTBP-5在5 个结直肠癌细胞中的表达均显著高于FHC 细胞(图1C)。通过对HT29、SW480 细胞的胞质和胞核RNA 进行分离发现,lnc-MTBP-5在胞质、胞核中均有分布(图1D、E)。
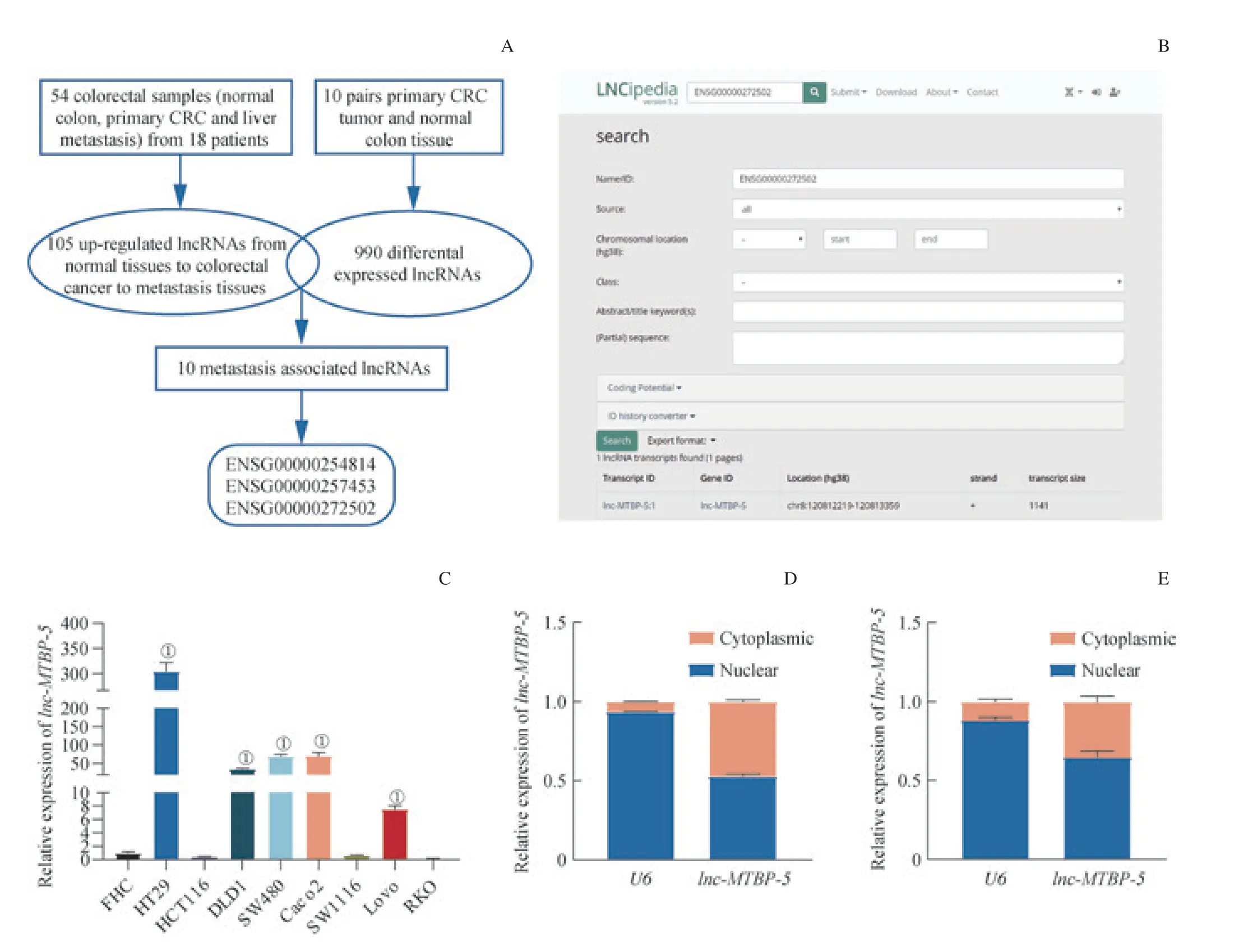
图1 SRA 数据库中与CRC 转移相关的lncRNA 分析Fig 1 Analysis of lncRNAs related to CRC metastasis in SRA database
2.2 lnc-MTBP-5 在CRC 患者癌组织和癌旁组织中的表达差异
为了探讨lnc-MTBP-5与CRC 的临床相关性,本研究对53 例结直肠癌组织和癌旁组织标本中lnc-MTBP-5的表达进行检测,结果(图2A)显示癌组织中lnc-MTBP-5的表达显著高于癌旁组织(P=0.000)。进一步针对TCGA数据库中638 例癌组织和51 例正常组织中的lnc-MTBP-5表达进行评估,结果(图2B)显示癌组织中其表达高于正常组织(P=0.000)。继而提示,lnc-MTBP-5的高表达可能参与了CRC 的发生和发展。
2.3 CRC 组织中lnc-MTBP-5 表达与患者临床预后的关系
为进一步验证lnc-MTBP-5与CRC 的临床相关性,本研究发现在TCGA 数据库的638 例患者中,lnc-MTBP-5表达的高低与患者的初次病理诊断年龄(P=0.020)、临床分期(P=0.006)、淋巴结转移情况(P=0.017)、远处转移情况(P=0.033)有关(表3);且与低表达组相比,lnc-MTBP-5高表达组中初次病理确诊年龄小于65 岁以及临床分期为Ⅲ、Ⅳ期的患者居多,且易发生淋巴结转移和远处转移。
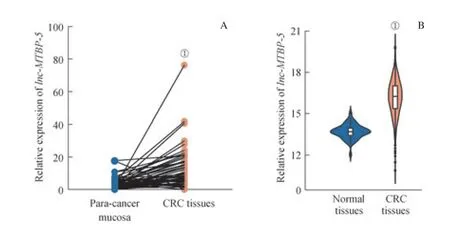
图 2 lnc-MTBP-5 在正常结直肠组织与CRC 组织中的表达Fig 2 Expressions of lnc-MTBP-5 in CRC tissues and normal colorectal tissues
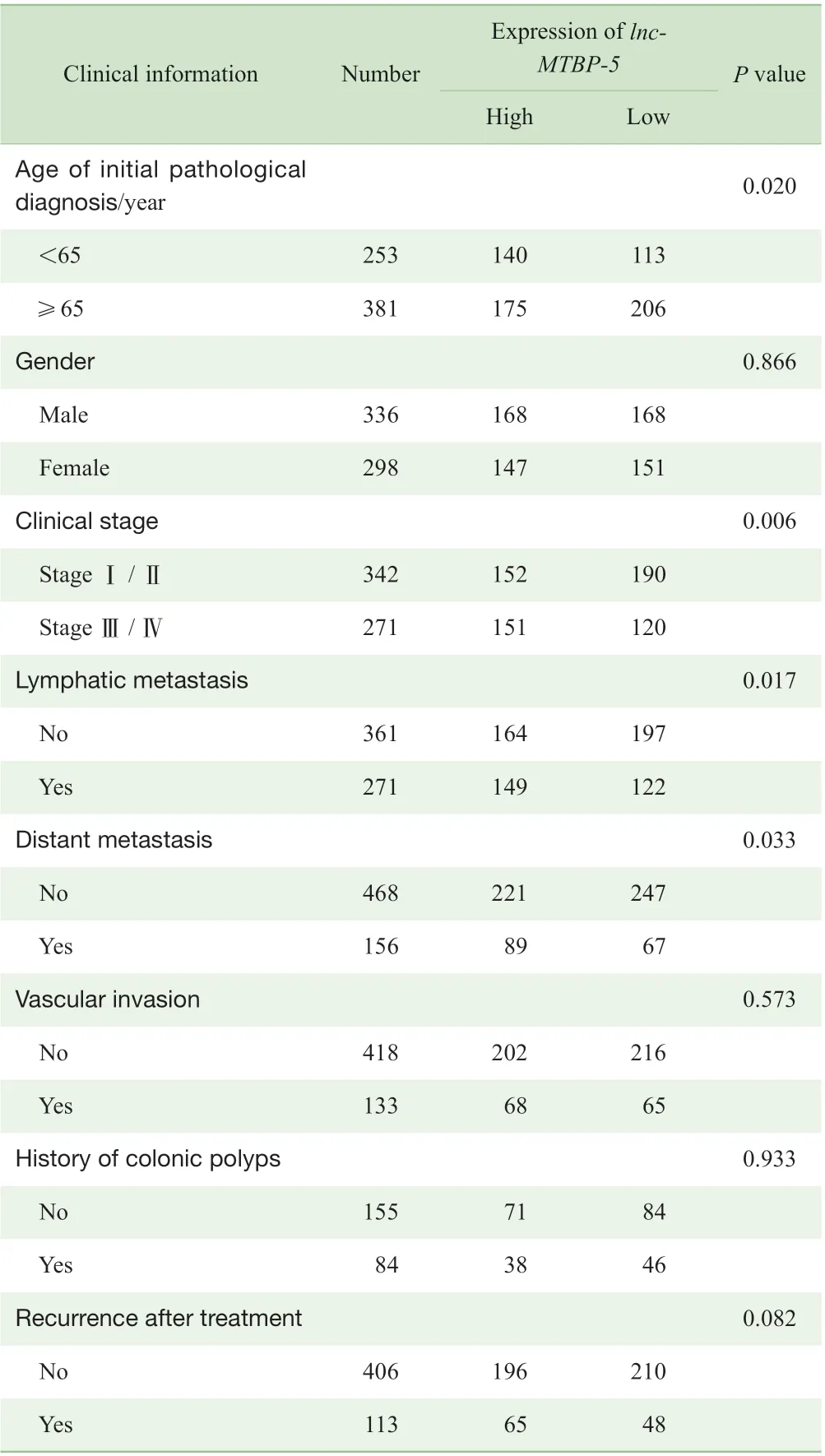
表3 CRC 患者中lnc-MTBP-5 表达与临床信息的关系Tab 3 Relationship between lnc-MTBP-5 expression and clinical information in patients with CRC
2.4 lnc-MTBP-5 对CRC 细胞侵袭能力的影响
为评估lnc-MTBP-5在CRC 发生与发展中的生物学功能,本研究通过向HT29、SW480 细胞中转染lnc-MTBP-5siRNA 发现,lnc-MTBP-5的表达量显著下降;而在SW480细胞中转染pcDNA3.1-lnc-MTBP-5后发现,lnc-MTBP-5的表达量显著升高(图3A ~C)。随后,进一步开展功能实验发现,在HT29 和SW480 细胞中转染lnc-MTBP-5siRNA后,与转染Control siRNA 的细胞相比,前者的增殖和克隆形成能力无明显差异(图3D、E)。因HT29 是高分化型CRC 细胞系,侵袭能力较弱,侵袭功能实验仅选择低分化的SW480 细胞系进行。侵袭实验结果表明,经lncRNA siRNA 转染的SW480 细胞穿过Transwell 小室的数量少于经Control siRNA 转染的细胞(图3F)。相反,与转染对照质粒的SW480 细胞相比,上调lnc-MTBP-5后,SW480 细胞侵袭能力增强(图 3G)。以上结果表明,lnc-MTBP-5可以增强SW480 细胞的侵袭能力,但对HT29 和SW480 细胞的增殖和克隆形成能力无明显影响。
2.5 lnc-MTBP-5 与转移起始基因表达的相关性
转移起始基因的缺失或过表达均能促使细胞外基质的降解、细胞黏附的减少、细胞侵袭能力的增强、血管生成以及肿瘤微环境的改变[11]。为进一步探索lnc-MTBP-5的作用机制,本研究对TCGA 数据库中lnc-MTBP-5的表达与转移起始基因的表达做相关性分析。结果(图4)显示,lnc-MTBP-5与结肠癌转移相关因子1(metastasis associated in colon cancer 1,MACC1)呈正相关;经进一步探索其下游机制发现,lnc-MTBP-5与间质上皮转换因子(mesenchymal to epithelial transition factor,MET)、钙黏着蛋白关联蛋白(cadherin-associated protein,CTNNB1)均呈正相关,而与肝细胞生长因子(hepatocyte growth factor,HGF)、蛋白激酶B(protein kinase B,PKB/AKT)均无明显正相关性。继而提示,lnc-MTBP-5促进CRC 的转移可能与MACC1 激活HGF/MET 通路及其下游AKT/CTNNB1 通路有关。
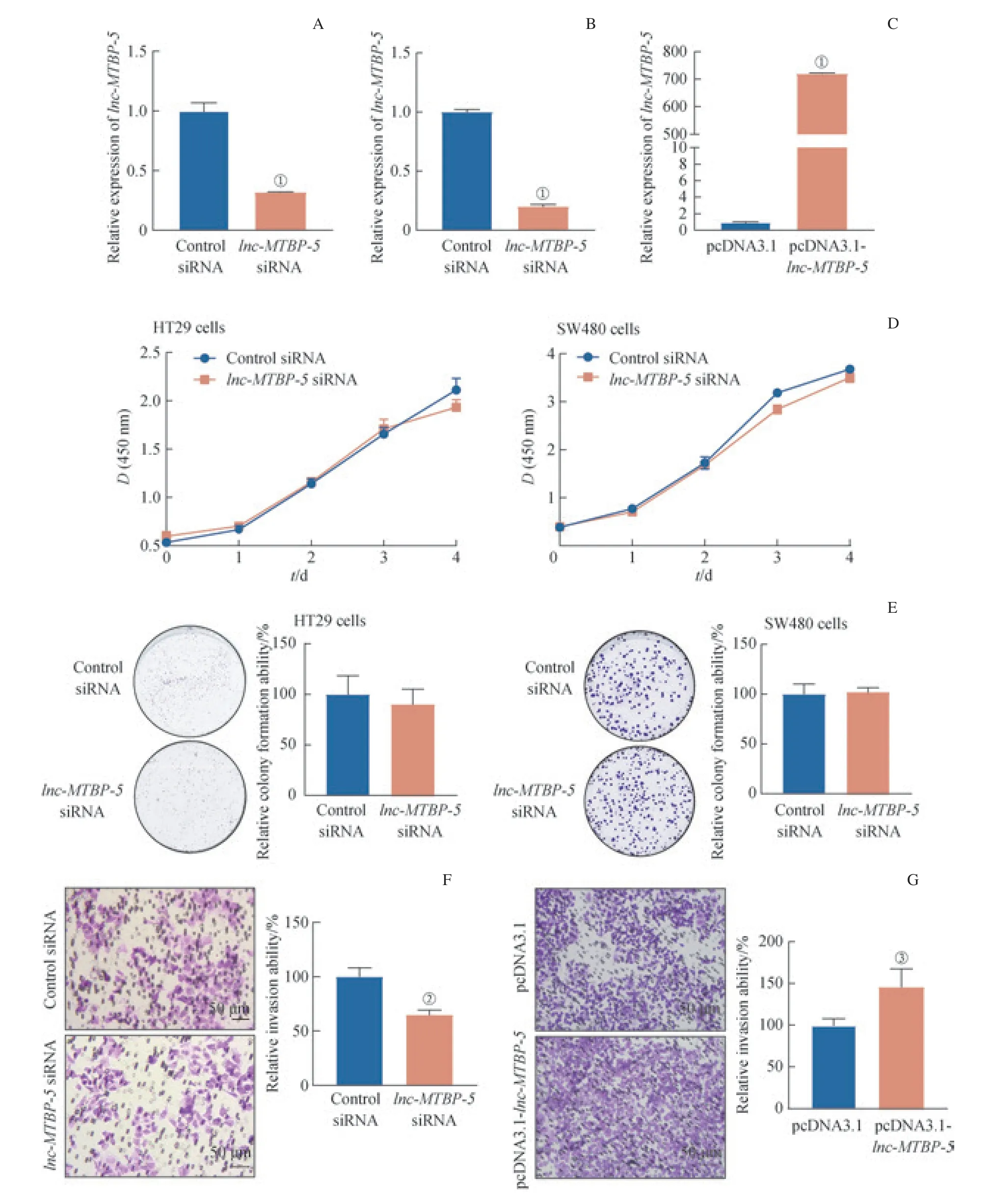
图3 lnc-MTBP-5 在CRC 发生与发展中的生物学功能Fig 3 Biological function of lnc-MTBP-5 in the occurrence and development of CRC
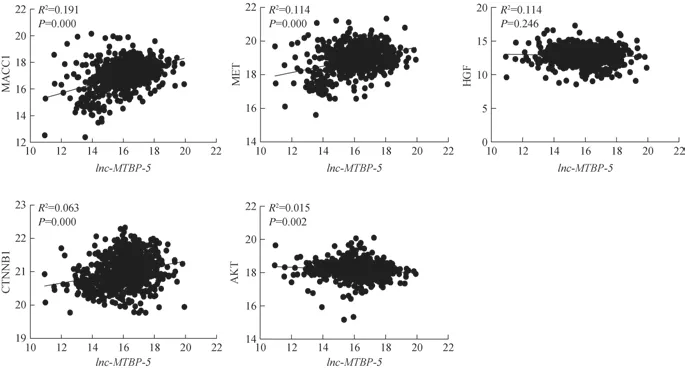
图4 lnc-MTBP-5 与转移相关基因表达的相关性分析Fig 4 Correlation analysis of lnc-MTBP-5 and the expression of metastasis associated genes
3 讨论
研究[12-13]显示,CRC 的发病率和死亡率呈逐年上升趋势,且其病灶转移是CRC 患者死亡的主要原因。因此,针对CRC 转移的早期检测,并寻找有效的分子标志物成为近年来预防和治疗CRC 的关键点。
已有研究报道,CRC 的转移与许多分子标志物相关,如JMJD2C、FOXC1、SGK1、RHBDD1[14-17]。同时,非编码RNA 也被广泛报道参与了CRC 转移的发生。依据RNA 长度的不同,非编码RNA 被分为两类;其中,小于200 个核苷酸的转录本被称为microRNA,通常在转录后调节基因的表达;而大于200 个核苷酸的转录本被称为lncRNA,该类RNA 具有较强的组织和细胞表达特异性,参与调节多种重要的细胞活动,如募集染色质修饰物(HOTAIR)[18]、调节X 染色体基因表达(XIST)[19]、调节基因组表达和蛋白质活性等。此外,lncRNA 还具有结构相对稳定,易于在血液和尿液中被检测,且定量方法灵敏、快速、成本低廉等优势[20],使得该类RNA 可作为癌症初筛、早期诊治的分子标志物,具有较强的应用价值。
在本研究中,通过整合分析2 个SRA 数据集的数据,筛选出在正常-原发灶-转移灶中表达依次上调的lncRNA,通过结合文献以及细胞实验、临床标本信息的验证,我们选择了lnc-MTBP-5继续研究;结果显示,该lncRNA 在多个CRC 细胞系和CRC 组织中均呈高表达。同时,本研究通过提取TCGA 数据库中CRC 患者的临床信息进行研究,结果显示lnc-MTBP-5的表达量与患者预后紧密相关。继而推测,lnc-MTBP-5或可作为早期预警和诊断CRC 的分子标志物;由于高表达lnc-MTBP-5的患者易发生CRC 转移,临床医师应在患者早期给予积极治疗,手术后密切随访。
CRC 的肝转移是影响患者预后的重要危险因素之一,因此揭示CRC 转移的发生机制亦是改善患者预后的关键点。本研究通过下调lnc-MTBP-5的表达发现,SW480 细胞的侵袭能力减弱,继而证实lnc-MTBP-5的高表达可提高CRC 的转移能力。
先前已有研究表明lncRNA 在CRC 中呈现异常表达,但有关其在CRC 转移中的作用及机制仍知之甚少。目前,基于lncRNA 在序列、结构以及生物功能上的高度异质性,存在有多种lncRNA 的分类方法,如根据亚细胞定位可将lncRNA 分为细胞核lncRNA 与细胞质lncRNA。lncRNA 的亚细胞定位与其功能密切相关,如在细胞质中lncRNA 可作为竞争性内源RNA(competing endogenous RNA,ceRNA)与microRNA 竞争性结合,调节miRNA抑制的靶基因表达[21]。已有研究[22]报道,lncRNAUICLM通过竞争性结合miR-215 来调节其下游ZEB2的表达,进而促进CRC 的肝转移。lncRNAXIST可直接结合miR-200b-3p,调节其下游ZEB1的表达,从而促进CRC 细胞的增殖、侵袭、上皮间充质转化及小鼠模型中肿瘤的生长和转移[23]。考虑到本研究中lnc-MTBP-5在胞质、胞核均有分布,我们推测lnc-MTBP-5可通过竞争性结合相关microRNA,调节下游靶基因的表达,促进CRC 的转移。
癌症的转移是一个复杂的过程,包括细胞外基质的降解、细胞黏附性的降低、癌细胞的黏附性增加和肿瘤微环境的改变等[24-28],是许多肿瘤发展、侵袭的重要过程[29-30]。根据在转移中所起的作用,与转移相关的基因可分为促癌基因、转移起始基因、转移进展基因和转移毒力基因[11]。转移起始基因MACC1已被证实在恶性肿瘤组织中有较高的表达,尤其在发生转移的恶性组织中。MACC1可通过结合Met的启动子区,激活HGF/MET 通路,进而促进肿瘤的增殖、上皮间质转化、血管生成、侵袭转移、调节细胞代谢和凋亡[31-32]。HGF/MET 通路的激活可使其下游的相应信号通路被激活,如通过TWIST/VEGF 通路促进血管生成,激活Nanog 分子增强细胞干性,激活STAT1/3、MCL1/FAS调节细胞凋亡和免疫重建。其中,AKT/CTNNB1 通路是HGF/MET 通路促进肿瘤转移的中间信号通路。本研究初步分析了TCGA 数据库中lnc-MTBP-5与转移相关基因表达的相关性,并发现lnc-MTBP-5促进CRC 的转移可能与MACC1 激活HGF/MET通路及其下游AKT/CTNNB1 通路有关。结合lnc-MTBP-5在胞质、胞核的分布,我们推测lnc-MTBP-5可能通过竞争性结合microRNA 提高MACC1 的表达,进而激活下游AKT/CTNNB1 通路。而其具体机制还有待实验进一步 探究。
综上所述,lnc-MTBP-5在CRC 中表达增加,且其高表达与患者预后呈负相关;同时,lnc-MTBP-5可促进CRC 细胞的侵袭。因此,lnc-MTBP-5或可作为临床评估CRC 发生与发展、远处转移的新型肿瘤标志物,其相关信号通路及分子机制亦将是我们下一步的研究重点。随着研究的不断深入,lnc-MTBP-5或可为临床预防、治疗CRC 及相关预后评估提供更加广阔的前景。
参·考·文·献
[1] Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65(2): 87-108.
[2] Jemal A, Ward EM, Johnson CJ, et al. Annual report to the nation on the status of cancer, 1975-2014, featuring survival[J]. J Natl Cancer Inst, 2017, 109(9). DOI: 10.1093/jnci/djx030.
[3] Tauriello DV, Calon A, Lonardo E, et al. Determinants of metastatic competency in colorectal cancer[J]. Mol Oncol, 2017, 11(1): 97-119.
[4] Benson AB, Venook AP, Al-Hawary MM, et al. NCCN guidelines insights: colon cancer, version 2.2018[J]. J Natl Compr Canc Netw, 2018, 16(4): 359-369.
[5] Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629-641.
[6] Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: insights into functions[J]. Nat Rev Genet, 2009, 10(3): 155-159.
[7] Sun TT, He J, Liang Q, et al. LncRNA GClnc1 promotes gastric carcinogenesis and may act as a modular scaffold of WDR5 and KAT2A complexes to specify the histone modification pattern[J]. Cancer Discov, 2016, 6(7): 784-801.
[8] Kim J, Piao HL, Kim BJ, et al. Long noncoding RNAMALAT1suppresses breast cancer metastasis[J]. Nat Genet, 2018, 50(12): 1705-1715.
[9] Hua JT, Ahmed M, Guo HY, et al. Risk SNP-mediated promoter-enhancer switching drives prostate cancer through lncRNAPCAT19[J]. Cell, 2018, 174(3): 564-575. E18.
[10] Zhang YJ, Pitchiaya S, Cieślik M, et al. Analysis of the androgen receptorregulated lncRNA landscape identifies a role for ARLNC1 in prostate cancer progression[J]. Nat Genet, 2018, 50(6): 814-824.
[11] Nguyen DX, Massagué J. Genetic determinants of cancer metastasis[J]. Nat Rev Genet, 2007, 8(5): 341-352.
[12] Nishida T, Egashira Y, Akutagawa H, et al. Predictors of lymph node metastasis in T1 colorectal carcinoma: an immunophenotypic analysis of 265 patients[J]. Dis Colon Rectum, 2014, 57(8): 905-915.
[13] Zhang L, Zhang SY, Yao J, et al. Microenvironment-induced PTEN loss by exosomal microRNA primes brain metastasis outgrowth[J]. Nature, 2015, 527(7576): 100-104.
[14] Wu XN, Li RX, Song Q, et al. JMJD2C promotes colorectal cancer metastasisviaregulating histone methylation of MALAT1 promoter and enhancing β-catenin signaling pathway[J]. J Exp Clin Cancer Res, 2019, 38(1): 435.
[15] Zhang Y, Liao Y, Chen CY, et al. P38-regulated FOXC1 stability is required for colorectal cancer metastasis[J]. J Pathol, 2020, 250(2): 217-230.
[16] Lee LYW, Woolley C, Starkey T, et al. Serum- and glucocorticoid-induced kinase Sgk1 directly promotes the differentiation of colorectal cancer cells and restrains metastasis[J]. Clin Cancer Res, 2019, 25(2): 629-640.
[17] Zhang MM, Miao F, Huang R, et al. RHBDD1 promotes colorectal cancer metastasis through the Wnt signaling pathway and its downstream target ZEB1[J]. J Exp Clin Cancer Res, 2018, 37(1): 22.
[18] Loewen G, Jayawickramarajah J, Zhuo Y, et al. Functions of lncRNAHOTAIRin lung cancer[J]. J Hematol Oncol, 2014, 7: 90.
[19] Sahakyan A, Yang YH, Plath K. The role ofXistin X-chromosome dosage compensation[J]. Trends Cell Biol, 2018, 28(12): 999-1013.
[20] Yan XH, Hu ZY, Feng Y, et al. Comprehensive genomic characterization of long non-coding RNAs across human cancers[J]. Cancer Cell, 2015, 28(4): 529-540.
[21] Salmena L, Poliseno L, Tay Y, et al. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?[J]. Cell, 2011, 146(3): 353-358.
[22] Chen DL, Lu YX, Zhang JX, et al. Long non-coding RNA UICLM promotes colorectal cancer liver metastasis by acting as a ceRNA for microRNA-215 to regulate ZEB2 expression[J]. Theranostics, 2017, 7(19): 4836-4849.
[23] Chen DL, Chen LZ, Lu YX, et al. Long noncoding RNAXISTexpedites metastasis and modulates epithelial-mesenchymal transition in colorectal cancer[J]. Cell Death Dis, 2017, 8(8): E3011.
[24] Said AH, Raufman JP, Xie GF. The role of matrix metalloproteinases in colorectal cancer[J]. Cancers (Basel), 2014, 6(1): 366-375.
[25] Marei H, Carpy A, Woroniuk A, et al. Differential Rac1 signalling by guanine nucleotide exchange factors implicates FL Ⅱ in regulating Rac1-driven cell migration[J]. Nat Commun, 2016, 7: 10664.
[26] Highfill SL, Cui YZ, Giles AJ, et al. Disruption of CXCR2-mediated MDSC tumor trafficking enhances anti-PD1 efficacy[J]. Sci Transl Med, 2014, 6(237): 237ra67.
[27] Gregorieff A, Pinto D, Begthel H, et al. Expression pattern of Wnt signaling components in the adult intestine[J]. Gastroenterology, 2005, 129(2): 626-638.
[28] Xie CQ, Ji N, Tang ZG, et al. The role of extracellular vesicles from different origin in the microenvironment of head and neck cancers[J]. Mol Cancer, 2019, 18(1): 83.
[29] Scully OJ, Bay BH, Yip G, et al. Breast cancer metastasis[J]. Cancer Genomics Proteomics, 2012, 9(5): 311-320.
[30] Boire A, Brastianos PK, Garzia L, et al. Brain metastasis[J]. Nat Rev Cancer, 2020, 20(1): 4-11.
[31] Birchmeier C, Birchmeier W, Gherardi E, et al. Met, metastasis, motility and more[J]. Nat Rev Mol Cell Biol, 2003, 4(12): 915-925.
[32] Radhakrishnan H, Walther W, Zincke F, et al. MACC1-the first decade of a key metastasis molecule from gene discovery to clinical translation[J]. Cancer Metastasis Rev, 2018, 37(4): 805-820.

