大豆脱落酸受体基因家族鉴定、系统发育进化及表达模式分析
张兆涵 张天旭 王万鹏 解莉楠


摘要:【目的】鑒定大豆脱落酸受体基因(GmPYLs)家族成员,并进行系统发育进化及表达模式分析,为深入研究该基因家族在大豆生长发育和非生物胁迫中的作用机制提供理论依据。【方法】以拟南芥PYLs基因(AtPYLs)家族成员序列为参考,从JGI和NCBI数据库鉴定出GmPYLs基因家族成员;利用生物信息学方法对该基因家族的组成、基因结构、保守性、系统进化、启动子区顺式作用元件及其编码蛋白理化性质和结构域等进行全面分析,并基于转录组测序数据,对GmPYLs基因家族成员的表达模式进行分析。【结果】从大豆全基因组序列中鉴定出21个GmPYLs基因,长度为500~4000 bp,分布在15条染色体上,编码蛋白的氨基酸数量为178~267个,分子量为19457.11~29105.43 Da,理论等电点(pI)为4.82~7.23,均为亲水蛋白,但稳定性存在明显差异,主要定位于细胞质中。GmPYLs蛋白的二级结构以α-螺旋和无规则卷曲为主。GmPYLs基因家族成员可分为四大组,同一组成员基因结构及其编码蛋白的氨基酸序列、三级结构、结构域和保守基序(motif)均相似。大豆、拟南芥、苜蓿和水稻的PYLs基因被分为五大类群(Ⅰ~Ⅴ),大豆、拟南芥、水稻和苜蓿的大多数PYLs基因归于Ⅰ、Ⅱ和Ⅲ类群;Ⅳ类群含少量的拟南芥和大豆PYLs基因,而Ⅴ类群仅含少部分苜蓿PYLs基因。拟南芥与大豆直系同源基因对数量多于拟南芥与苜蓿直系同源基因对数量。21个GmPYLs基因的启动子序列主要包含响应逆境胁迫、调节生长发育及响应植物激素的顺式作用元件,且所有GmPYLs基因的启动子区至少含有1个植物激素响应元件,其中,以含ABA响应元件的GmPYLs基因数量最多。GmPYLs基因具有组织表达特异性,且品种间的表达模式也存在差异。【结论】GmPYLs基因家族成员在系统发育进化上较保守,其基因组复制事件可能发生在豆科植物分化以后,且大部分GmPYLs基因被保留,在响应非生物胁迫中存在广泛的潜在机制,尤其与激素响应密切相关。
关键词: 大豆;GmPYLs;基因家族;鉴定;系统进化;非生物胁迫;表达分析
中图分类号: S565.1 文献标志码: A 文章编号:2095-1191(2020)08-1904-13
Identification,phylogenetic evolution and expression analysis of abscisic acid receptors gene family in Glycine max L. Merr
ZHANG Zhao-han, ZHANG Tian-xu, WANG Wan-peng, XIE Li-nan*
(College of Life Science, Northeast Forestry University/Key Laboratory of Saline-alkali Vegetation Ecology Restoration,Ministry of Education, Harbin 150040, China)
Abstract:【Objective】The abscisic acid receptor gene family in Glycine max L. Merr GmPYLs was identified by bioinformatics,and analyzed its evolution model and expression pattern. The purpose of this study was to provide a theoretical basis for in-depth study of the role of PYL gene in soybean development and abiotic stress. 【Method】The reported members of PYLs gene family in Arabidopsis thaliana(AtPYLs) were used as a reference,and the members of GmPYLs gene family were identified by JGI and NCBI databases. The gene family composition,gene structure,conservatism,evolutionary relationship,cis-acting element of promoter region, and its coding protein physicochemical properties and domain were analyzed by bioinformatics method. Based on high throughput transcriptome sequencing data,the expression patterns of GmPYLs gene family members were analyzed. 【Result】In this study,21 GmPYLs genes were identified from soybean whole genome sequence, which were distributed on 15 chromosomes with a sequence length of 500-4000 bp. The protein encoded was 19457.11-29105.43 Da in molecular weight, amino acids number was 178 to 267, theoretical isoelectric point(PI) was 4.82-7.23,which was hydrophilic protein mainly distributed in the cytoplasm. But the stability was different. The secondary structure of the protein was mainly α-helix and random coil. The GmPYLs gene family could be divided into four groups. In the same group,the gene structure,amino acid sequence of conded protein,protein tertiary structure,functional domain and conserved motif were very similar. The PYLs genes of G. max, Arabidopsis thaliana, Oryza sativa and Medicago truncatula were divided into five groups (Ⅰ-Ⅴ). Most of the PYLs genes in G. max, Arabidopsis thaliana, Oryza sativa and Medicago truncatula were classified into groups Ⅰ, Ⅱ and Ⅲ. Group IV contained a small amount of AtPYLs and GmPYLs genes, while group Ⅴ contained only a small part of MtrPYLs genes. The promoter sequences of 21 GmPYLs genes mainly contained cis-acting elements that responded to stress, regulate growth and respond to plant hormones. The promoter regions of all GmPYLs genes contained at least one hormone response element, of which the largest number of GmPYLs genes containing ABA response elements. The results of transcriptome sequencing analysis showed that GmPYLs gene had tissue-specific expression|, and there were differences in the expression patterns between different varieties. 【Conclusion】Members of the GmPYLs gene family are conservative in phylogenetic evolution, and their genome replication events may occur after legume differentiation, and most of the GmPYLs genes are retained. There are a wide range of potential mechanisms in response to abiotic stress, closely related to hormone response.
Key words: soybean; GmPYLs; gene family; identification; systematic evolution; abiotic stress; expression analysis
Foundation item: National Natural Science Foundation of China for Young Scholars(31801444)
0 引言
【研究意义】大豆(Glycine max L. Merr )是我国重要的经济作物,具有极高的营养价值,是人类获取植物蛋白的主要来源,在世界范围内广泛种植。干旱、盐碱和极端温度等非生物胁迫严重制约大豆的产量和品质。PYR(Pyrabactin resistance)/PYL(PYR1-like)/RCAR(Regulatory components of ABA receptor)(简称PYL)蛋白家族作为脱落酸(ABA)的直接受体,在响应ABA介导的非生物胁迫过程中发挥重要作用(Ma et al.,2009;Park et al.,2009;Rodriguez et al.,2014)。因此,鉴定大豆PYLs基因(GmPYLs)家族成员,分析其进化及表达模式,以期深入了解大豆响应非生物胁迫的分子机制,对提高大豆抗胁迫能力具有重要竟义。【前人研究进展】PYL蛋白位于ABA信号转导途径中的顶端,以抑制2C型蛋白磷酸酶(PP2C)活性作为主要调控方式(Park et al.,2009)。在缺乏ABA时,PYL蛋白不与PP2C结合,因此PP2C活性很高,从而阻止SNF1相关蛋白激酶2(SNF1-related protein kinases 2,SnRK2)及其下游因子的活化;当存在ABA时,PYL结合PP2C,抑制PP2C活性(Melcher et al.,2009;Miyazono et al.,2009),被抑制的PP2C释放SnRK2,随后SnRK2磷酸化,激活ABA响应元件结合因子(ABA-responsive elements bin-ding factors,ABFs),从而启动下游因子响应逆境(Soon et al.,2012)。因此,PYL蛋白在ABA信号转导途径发挥重要作用,间接调控植物响应非生物胁迫(Miao et al.,2006;Yu et al.,2015)。迄今,已有较多有关PYLs基因的研究报道,陆续从拟南芥(Ma et al.,2009)、葡萄(Boneh et al.,2012)、水稻(He et al.,2014)、番茄(González-Guzmán et al.,2014)、玉米(李鸿杰等,2015)、橡胶树(Guo et al.,2017)、棉花(Chen et al.,2017)和苜蓿(黄思源等,2019)中分别鉴定出14、8、13、14、21、27、13和14个PYL蛋白。其中,部分PYL蛋白的生物学功能已被验证。如ABA以不依赖于AtPYR1、AtPYL1、AtPYL2和AtPYL4的方式启动拟南芥保卫细胞中的MeJA信号(Ye et al.,2016);过表达AtPYL4、AtPYL8和AtPYL13基因可增强拟南芥的耐旱性(Santiago et al.,2009;Fuchs et al.,2014;Pizzio et al.,2017;Quan et al.,2018);过表达AtPYR1、AtPYL1、AtPYL2和AtPYL3基因可提高拟南芥的抗旱性和水分利用率(Li et al.,2018);过表达AtPYL5基因可降低干旱胁迫下拟南芥植株的蒸腾速率,减少水分损失,减轻氧化胁迫伤害,进而提高光合作用效率(Quan et al.,2018);AtPYR1和AtPYL2是脱落酸诱导气孔关闭的关键受体(Dittrich et al.,2019);AtPYL8和AtPYL9蛋白在擬南芥侧根生长中发挥重要作用(Zhao et al.,2014,2016)。此外,过表达玉米ZmPYL8、ZmPYL9和ZmPYL12基因可提高植株的耐寒性(He et al.,2018);敲除水稻OsPYL1、OsPYL4和OsPYL6基因可提高水稻的产量(Miao et al.,2018);过表达小麦TaPYL4基因可减少蒸腾作用并增强光合作用以提高水分利用效率和抗旱性(Mega et al.,2019);玉米ZmPYL9基因响应干旱胁迫和ABA诱导,导致该基因的表达量明显提高(王延召等,2020)。【本研究切入点】目前,针对PYLs基因家族的研究主要集中在拟南芥、棉花、番茄、水稻和苜蓿等模式植物,虽然GmPYLs基因家族已被鉴定(Bai et al.,2013),但目前在JGI数据库(https://phytozome.jgi.doe.gov/pz/portal.html)中尚未找到本研究鉴定得到的2个基因(GmPYL19和GmPYL22),也未进行全面的生物信息学分析。【拟解决的关键问题】以拟南芥PYLs基因(AtPYLs)家族成员序列为参考,利用JGI和NCBI数据库从大豆全基因组鉴定出GmPYLs基因家族成员;利用生物信息学方法对该基因家族的组成、基因结构、保守性、进化关系、启动子区顺式作用元件及其编码蛋白的理化性质和结构域等进行全面分析,并基于高通量转录组测序数据,对GmPYLs基因家族成员的表达模式进行分析,为探究该基因家族的生物学功能及其在大豆生长发育和响应非生物胁迫中的调控作用提供理论依据。
1 材料与方法
1. 1 试验材料
供试大豆品种为Williams 82和Jack,由东北林业大学生命科学学院提供。主要试剂:RNA提取试剂盒(北京百泰克生物技术有限公司)、无水乙醇(天津市永大化学试剂有限公司)。
1. 2 样品采集及RNA提取
将大豆种子种植于温室(26 ℃,光照14 h/黑暗10 h)中,待幼苗的第一片真叶充分展开后,选取长势一致的幼苗,采集其顶端分生组织、真叶、上胚轴、下胚轴和根。每个组织设3个独立的生物学重复,放入液氮速冻,提取总RNA。高通量转录组测序由上海凌恩生物科技有限公司完成。
1. 2 GmPYLs基因家族鉴定
从TAIR数据库(https://www.arabidopsis.org/)查找14个AtPYLs基因及其编码蛋白的相关信息,并在JGI数据库(https://phytozome.jgi.doe.gov/pz/portal.html)进行氨基酸序列同源比对,筛选出与AtPYLs蛋白同源的GmPYLs候选蛋白。通过NCBI数据库进行BLASTp比对分析,最终确定GmPYLs基因家族成员,并命名。
1. 3 系统进化分析
从NCBI数据库(http://plants.ensembl.org/index.html)中获取苜蓿PYLs基因(MtrPYLs)家族成员和水稻PYLs基因(OsPYLs)家族成员编码蛋白氨基酸序列。利用MEGA 10.0的邻接法(Neighbor-joining,NJ)构建系统发育进化树,并使用Bootstrap(自展法)进行3000次可信度检测,分析GmPYLs基因家族与AtPYLs、MtrPYLs和OsPYLs基因家族的系统进化关系。
1. 4 基因结构及保守性分析
通过NCBI数据库获得大豆全基因组序列信息,利用TBtools提取GmPYLs基因序列信息,采用GSDS(Gene structure display server)在线分析GmPYLs基因结构。使用DNAMAN 6.0对GmPYLs、AtPYLs、MtrPYLs和OsPYLs蛋白进行氨基酸序列比对;利用MEME和TBtools分析GmPYLs蛋白的保守motif;并以SMART在线分析GmPYLs蛋白的结构域。
1. 5 染色體定位与共线性分析
在NCBI数据库中获取GmPYLs基因的染色体定位信息,利用MapChart绘制染色体定位图。运用MCScanx和TBtools分析GmPYLs基因家族成员间及其与AtPYLs和MtrPYLs间的共线性关系,阐释不同物种PYLs基因家族间的复制事件,确定直系同源基因和旁系同源基因。
1. 6 启动子序列分析
在NCBI数据库中提取GmPYLs基因家族成员起始密码子上游2000 bp基因组序列,利用PlantCARE在线分析启动子区顺式作用元件。
1. 7 理化性质分析、亚细胞定位及结构预测
利用ProtParam在线分析GmPYLs和AtPYLs蛋白的分子量、等电点、稳定性、亲/疏水性等理化性质。使用PSORT II Prediction预测GmPYLs蛋白的亚细胞定位情况;采用SOPMA和SWISS-MODEL预测GmPYLs蛋白的二、三级结构。
1. 8 表达特性分析
表达量数据由高通量转录组测序获取,测序平台为Illumina HiseqTM 2500,每个样品产生6.0 Gb测序数据,测序方式为双末端(PE)测序,读长为150 bp,将转录组分析通用的FPKM(Fragments per kilobase of transcript per million fragments mapped)值作为基因的表达量,分析GmPYLs基因的组织表达特异性及品种间表达差异。
1. 9 统计分析
利用Excel 2016统计试验数据,并采用T-test进行显著性差异分析。
2 结果与分析
2. 1 GmPYLs基因家族鉴定及命名
根据氨基酸序列相似性比对及NCBI数据库BLASTp序列验证,最终获得21个GmPYLs基因,命名为GmPYL1~GmPYL21,其基因长度、染色体位置及编码氨基酸数量如表1所示。21个GmPYLs基因序列长度为570~3927 bp,编码蛋白的氨基酸数量为178~267个。
2. 2 系统发育进化分析结果
由图1可知,大豆、拟南芥、苜蓿和水稻的PYLs基因被分为五大类群(Ⅰ~Ⅴ),其中,大豆、拟南芥、水稻和苜蓿的大多数PYLs基因归属于Ⅰ、Ⅱ和Ⅲ类群,Ⅳ类群含少量的拟南芥和大豆PYLs基因,而Ⅴ类群只有少部分苜蓿PYLs基因,说明PYLs基因在植物进化及分化过程中相对保守,在不同物种间未发生明显变异;21个GmPYLs基因成员分布在Ⅰ、Ⅱ、Ⅲ和Ⅳ类群中,其中,Ⅰ类群有8个成员,Ⅱ类群有6个成员,Ⅲ类群有5个成员,Ⅳ类群有2个成员;在Ⅰ~Ⅲ类群中,GmPYLs基因数量明显多于AtPYLs基因数量,推测在十字花科和豆科植物分化后大豆全基因组发生复制事件并保留下较多基因。Ⅳ类群中GmPYLs基因数量少于AtPYLs基因数量,说明Ⅳ类群PYLs基因在大豆全基因组复制事件中可能发生丢失。故推测相同类群的基因结构及其编码蛋白的结构和功能域相似。
2. 3 GmPYLs基因家族的基因结构及其编码蛋白结构分析结果
由GmPYLs基因家族的系统发育进化树(图2-A)可知,21个GmPYLs基因可分为四大组(Ⅰ~Ⅳ),仅Ⅲ组中的5个GmPYLs基因存在内含子,其他16个GmPYLs基因均无内含子(图2-B)。蛋白保守基序(motif)分析结果显示,从GmPYLs和AtPYLs蛋白中共鉴定出10个保守motif(motif 1~motif 10)(图2-C),长度为8~50个氨基酸(表2),其蛋白保守结构域的氨基酸序列分布情况如图2-D所示。所有GmPYLs蛋白共有的motif为motif 1、motif 2和motif 3,与多序列比对分析结果(图3)一致。在同一组内系统发育进化树同一节支点的基因具有相似的保守motif结构(图2-A和图4)。结构域预测结果显示,21个GmPYLs蛋白均包含1个共有的聚酮环化酶结构域2(Pfam:Polyketide_cyc2),部分GmPYLs蛋白含有3个特有的结构域:聚酮环化酶结构域(Pfam:Polyketide_cyc)、桦树花粉蛋白结构域(Pfam:Bet_v_1)和过氧化物酶结构域(Pfam:peroxidase)(图4和表3)。其中,Pfam:peroxidase是GmPYL14蛋白所特有,说明其可能行使与其他GmPYLs基因不同的生物学功能,如负责清除叶绿体和细胞质中的过氧化氢及其他氧化有毒化合物等。
2. 4 GmPYLs基因家族的染色体定位及共线性分析结果
利用MapChart绘制GmPYLs基因家族成员在染色体上的位置,结果如图5所示。21个GmPYLs基因分别位于1、2、3、4、6、7、8、9、11、13、14、15、16、17和18号染色体上,其中1号染色体上分布的GmPYLs基因数量最多,为3个,其次是6、7、13和14号染色体上分布的GmPYLs基因数量,均为2个,其他染色体仅包含1个基因。共线性和复制基因分析结果(图6-A)显示,GmPYL1/2/3/4、GmPYL5/6/7/8、GmPYL9/10/11、GmPYL12/13/14、GmPYL15/16、GmPYL17/19和GmPYL20/21均发生片段复制,且片段复制使基因数目增多,尤其是Ⅰ、Ⅱ和Ⅲ类群的基因数目。在对拟南芥、大豆和苜蓿3个物种的直系同源基因进行对比分析,结果(图6-B)发现,拟南芥与大豆直系同源基因对数量多于拟南芥与苜蓿直系同源基因对数量,说明PYLs基因的基因组复制事件可能发生在豆科植物分化以后,且3个物种间直系同源基因对数量较多,也间接表明同源基因序列间高度的保守性和相似性。
2. 5 GmPYLs基因家族的启动子区序列分析结果
为了预测GmPYLs基因家族的功能,对其启动子区的顺式作用元件进行分析,结果(图7)显示,GmPYLs基因家族的启动子序列主要包含响应逆境胁迫、调节生长发育及响应植物激素的顺式作用元件。其中,响应逆境胁迫的顺式作用元件主要包括创伤响应元件、防御和压力响应元件、低温响应元件和干旱响应元件,说明GmPYLs基因家族在响应非生物胁迫方面存在广泛的潜在机制,如GmPYL15基因启动子区包含6个参与干旱诱导的MYB结合位点,说明GmPYL15蛋白可能调节某些MYB相關基因的转录,以响应干旱胁迫。调节生长发育的顺式作用元件包括种子特异性调控元件、顶端分生组织表达元件、昼夜节律调控元件及参与类黄酮生物合成基因调控的MYB结合位点,如GmPYL9和GmPYL12基因启动子区包含种子特异性调控元件,GmPYL17基因启动子区包含参与类黄酮生物合成基因调控的MYB结合位点。说明GmPYL9和GmPYL12基因可能在种子发育过程中发挥重要作用,而GmPYL17基因可能参与类黄酮合成。GmPYLs基因家族共含有5种植物激素响应元件,包括ABA响应元件、赤霉素(GAs)响应元件、生长素(IAA)响应元件、响应茉莉酸甲酯(MeJA)应答的元件和水杨酸(SA)响应元件。其中,以含ABA响应元件的GmPYLs基因数量最多,为15个,其次是含MeJA响应元件的GmPYLs基因数量,为14个;GmPYL2、GmPYL16和GmPYL20基因均包含4个激素响应元件,GmPYL13、GmPYL14、GmPYL17和GmPYL19基因均包含3个激素响应元件,说明这7个基因在激素响应方面可能发挥重要调控作用。
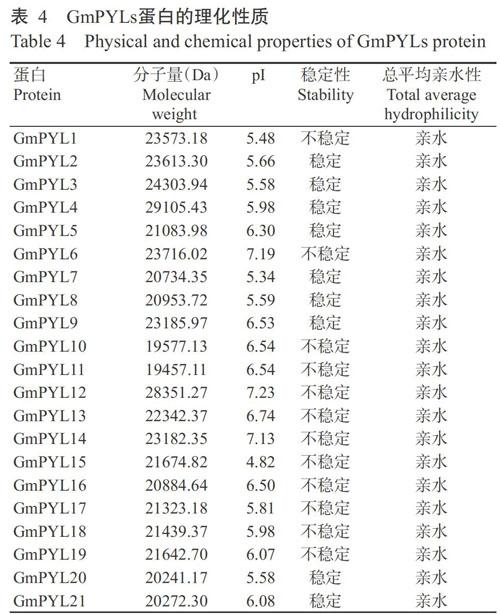
2. 6 GmPYLs蛋白理化性质分析、亚细胞定位及结构预测结果
GmPYLs蛋白理化性质如表4所示。GmPYLs蛋白的分子量为19457.11~29105.43 Da,理论等电点(pI)4.82~7.23,均为亲水蛋白,但稳定性存在明显差异,由此推测其在不同微环境中行使的功能。亚细胞定位结果显示,GmPYLs蛋白主要定位在细胞质(图8-A)。利用SOPMA预测GmPYLs蛋白二级结构,结果(图8-B)表明GmPYLs蛋白二级结构均以α-螺旋和无规则卷曲为主,其次是β-折叠,β-转角最少。GmPYLs蛋白三级结如图8-C所示,位于系统发育进化树节支点的GmPYLs蛋白具有类似三级结构,如GmPYL1~GmPYL4具有相似三级结构,GmPYL5~ GmPYL8也存在相似的三级结构,因此推测三级结构相似的GmPYLs蛋白具有相似的生物学功能。
2. 7 GmPYLs基因家族的表达特异性分析结果
鉴于幼苗子叶期是大豆受非生物胁迫最敏感的阶段,本研究选取2个代表性的大豆品种Williams 82和Jack分析其GmPYLs基因家族成员在子叶期幼苗的顶端分生组织、真叶、上胚轴、下胚轴和根中的表达模式。品种间的表达量差异分析结果(图9)显示,品种间基因表达量差异极显著(P<0.01)的基因包括顶端分生组织中的GmPYL1、GmPYL15和GmPYL19基因,真叶中的GmPYL4、GmPYL9、GmPYL10、GmPYL11、GmPYL16和GmPYL19基因,上胚轴中的GmPYL1、GmPYL2、GmPYL3和GmPYL9基因,下胚轴中的GmPYL1、GmPYL3、GmPYL4、GmPYL9、GmPYL10、GmPYL11和GmPYL15基因,以及根中的GmPYL1、GmPYL3、GmPYL4、GmPYL9、GmPYL10、GmPYL11、GmPYL13、GmPYL14、GmPYL15、GmPYL16和GmPYL19基因。组织特异性表达结果(图10)显示,GmPYLs基因具有组织表达特异性,Williams 82中的GmPYL1和GmPYL9基因分别在上胚轴和真叶表达,且其表达量均高于其他器官,Jack中的GmPYL10基因在根中表达量均高于其他器官,说明GmPYL9和GmPYL10基因不仅具有组织器官特异性,还在品种间差异表达;GmPYL19基因在5个组织中的表达量均表现为Jack高于Williams 82,说明GmPYL19基因可能在Jack品种响应非生物胁迫方面发挥重要调控作用。此外,Williams 82和Jack品种间在根中的差异表达基因居多,且大多数基因表达量表现为Jack高于Williams 82,说明这2个品种根中的工作基因存在明显差异;GmPYL5、GmPYL6、GmPYL7、GmPYL8、GmPYL20和GmPYL21在2个品种中的表达量非常低,说明这些基因功能可能已冗余,或不在子叶期表达。
3 讨论
本研究中GmPYLs基因家族的系统发育进化和序列保守性分析结果显示,21个GmPYLs基因分成四大组,同一组中的基因结构较相似,且系统发育进化树中同一节支点内保守motif和三级结构也高度相似,故推测同组基因具有类似的调控功能。前人的研究也发现,AtPYR1、AtPYL1、AtPYL2和AtPYL3基因均能促进ABA诱导的气孔关闭(Nishimura et al.,2010;Wang et al.,2013;Yin et al.,2013)。本研究的系统进化分析结果显示,GmPYL1、GmPYL2、GmPYL3、GmPYL4与AtPYR1、AtPYL1、AtPYL2、AtPYL3基因直系同源,暗示这些基因可能也具有促进ABA诱导的气孔关闭生物学功能。GmPYLs蛋白均包含Pfam:Bet_v_1、Pfam:Polyketide_cyc和Pfam:Polyketide_cyc2结构域,其中,Pfam:peroxidase是GmPYL14基因所特有,说明GmPYL14蛋白可能在GmPYLs基因家族中具有除响应ABA信号以外的其他功能。由于Pfam:peroxidase结构域在植物中主要负责清除叶绿体和细胞质中的过氧化氢及其他氧化有毒化合物,且参与细胞壁生物合成、伤害防御反应、IAA分解代谢及乙烯生物合成等(http://pfam.xfam.org/family/peroxidase),故推测GmPYL14蛋白也具有相似功能。Pfam:Polyketide_cyc和Pfam:Polyketide_cyc2是START结构域超家族成员,其蛋白折叠具有保守的配体结合模式,以适应各种催化活性和非催化调节功能;Pfam:Bet_v_1是桦树花粉蛋白结构域(Ma et al.,2009),与Pfam:Polyketide_cyc结构域同源(Iyer et al.,2001),且在位置上互相重叠。这些结构域是PYL蛋白作为ABA直接受体的关键,通过结合PP2C而负调控ABA核心信号通路(Fujii et al.,2009)。由于大豆是古四倍体生物,在基因家族中存在一定程度上的功能冗余,对同一组别的GmPYLs基因进行多基因编辑,有利于高效实现研究目标。
本研究的GmPYLs基因启动子顺式作用元件分析结果显示,Ⅲ组中的GmPYL17、GmPYL18和GmPYL19基因位于系统发育进化树同一节支点上,均包含IAA响应元件。而前人的研究结果表明,AtPYL8以不依赖于ABA核心信号通路(PYL-PP2C-SnRK2)的方式促进响应IAA的相关基因转录,从而解除ABA核心信号通路对侧根生长的抑制,促进侧根生长(Zhao et al.,2014)。本研究中不同物种的PYLs基因系统发育进化树分析结果显示,GmPYL17、GmPYL18和GmPYL19基因與AtPYL8基因归于Ⅲ类群,表明四者亲缘关系较近。由此推测,GmPYL17、GmPYL18和GmPYL19基因可能在大豆中也参与调控侧根生长。本研究还发现,所有GmPYLs基因至少包含1个植物激素响应元件,而植物激素信号是响应干旱或水分不足的关键,故推测GmPYLs基因家族在干旱响应中发挥至关重要的作用。
PYL作为ABA的直接受体,参与植物多种非生物胁迫响应。前人的研究结果表明,AtPYL5基因过表达可降低干旱胁迫下植株的蒸腾速率,减少水分损失,减轻氧化胁迫伤害,进而提高光合作用(Quan et al.,2018)。本研究发现,GmPYL9基因在叶中的表达量较高,在今后的研究中可深入探究其表达量与光合速率和蒸腾速率的相关性。此外,AtPYR1、AtPYL1、AtPYL2和AtPYL3基因过表达可提高拟南芥的抗旱性和水分利用率(Li et al.,2018)。在本研究构建的不同物种PYLs基因系统发育进化树中,GmPYL1~GmPYL8基因与上述基因同归于Ⅰ类群,故推测这些基因也具有类似的生物学功能,可提高大豆的抗旱性和水分利用率。本研究通过转录组数据分析发现,GmPYLs基因家族具有组织表达特异性,且品种间的表达模式也存在差异。与大豆不同器官转录组在线数据(http://bar.utoronto.ca/eplant/)(Libault et al.,2010)相比,本研究中GmPYLs基因家族成员具有相似的表达模式,证明本研究结果的可靠性。
PYLs是植物中复杂且庞大的基因家族。通过对模式植物拟南芥在逆境胁迫下的生理和分子水平进行分析,研究人员揭示了植物如何通过ABA应对干旱胁迫,包括对基因表达进行重新编程、关闭气孔及调节渗透等,最终实现植株适应性生长发育(Gong et al.,2020)。基于上述植物抗逆分子机理研究对重要经济作物进行遗传改良,从而获得高抗逆性植物材料。本研究利用大豆基因组和转录组数据,对GmPYLs基因家族进行全面分析,为研究GmPYLs基因响应非生物胁迫和生长发育的机制及GmPYLs基因的定向研究提供了理论依据。在后续研究中,应对GmPYLs基因家族成员进行多重基因编辑,分析其对大豆生长发育及应对非生物胁迫具体表型效应影响。
4 结论
GmPYLs基因家族成员在系统发育进化上较保守,其基因组复制事件可能发生在豆科植物分化以后,且大部分GmPYLs基因被保留,在响应非生物胁迫中存在广泛的潜在机制,尤其与激素响应密切相关。
参考文献:
黄思源,呼天明,杨培志. 2019. 蒺藜苜蓿PYL基因家族的全基因组鉴定、表达和功能分析[J]. 草业科学,36(2):422-433. [Huang S Y,Hu T M,Yang P Z. 2019. Identification and function analysis of the PYL gene family in Medicago truncatula[J]. Pratacultural Science,36(2):422-433.]
李鴻杰,王盈阁,张夏伟,付凤玲,李晚忱. 2015. 玉米脱落酸受体基因家族的生物信息学分析[J]. 核农学报,29(9):1657-1667. [Li H J,Wang Y G,Zhang X W,Fu F L,Li W C. 2015. Bioinformatic analysis for abscisic acid perceptor gene family in maize[J]. Journal of Nuclear Agricultural Sciences,29(9):1657-1667.]
王延召,鲁晓民,魏良明,周波,黄保. 2020. 玉米脱落酸受体ZmPYL9基因的克隆及功能探究[J]. 河南农业科学,49(1):18-23. [Wang Y Z,Lu X M,Wei L M,Zhou B,Huang B. 2020. Cloning and function analysis of maize abscisic acid receptor ZmPYL9 gene[J]. Journal of Henan Agricultural Sciences,49(1):18-23.]
Bai G,Yang D H,Zhao Y,Ha S,Yang F,Ma J,Gao X S,Wang Z M,Zhu J K. 2013. Interactions between soybean ABA receptors and type 2C protein phosphatases[J]. Plant Molecular Biology,83(6):651-664.
Boneh U,Biton I,Zheng C,Schwartz A,Ben-Ari G. 2012. Characterization of potential ABA receptors in Vitis vini-fera[J]. Plant Cell Reports,31(2):311-321.
Chen Y,Feng L,Wei N,Liu Z H,Hu S,Li X B. 2017. Overexpression of cotton PYL genes in Arabidopsis enhances the transgenic plant tolerance to drought stress[J]. Plant Physiology & Biochemistry,115:229-238. doi:10.1016/j.plaphy.2017.03.023.
Dittrich M,Mueller H M,Bauer H,Peirats-Llobet M,Rodriguez P L,Geilfus C M,Carpentier S C,Al-Rasheid K A S,Kollist H,Merilo E,Herrmann J,Müller T,Ache P,Hetherington A M,Hedrich R. 2019. The role of Arabidopsis ABA receptors from the PYR/PYL/RCAR family in stomatal acclimation and closure signal integration[J]. Nature Plants,5(9):1002-1011.
Fuchs S,Tischer S V,Wunschel C,Christmann A,Grill E. 2014. Abscisic acid sensor RCAR7/PYL13,specific regulator of protein phosphatase coreceptors[J]. Proceedings of the National Academy of Sciences of the United States of America,111(15):5741-5746.
Fujii H,Chinnusamy V,Rodrigues A,Rubio S,Antoni R,Park S Y,Cutler S R,Sheen J,Rodriguez P L,Zhu J K. 2009. In vitro reconstitution of an ABA signaling pathway[J]. Nature,462(7273):660-664.
Gong Z Z,Xiong L,Shi H Z,Yang S H,Herrera-Estrella L R,Xu G H,Chao D Y,Li J R,Wang P Y,Qin F,Li J J,Ding Y L,Shi Y T,Wang Y,Yang Y Q,Guo Y,Zhu J K. 2020. Plant abiotic stress response and nutrient use efficiency[J]. Science China Life Sciences,63(5):635-674.
González-Guzmán M,Rodríguez L,Lorenzo-Orts L,Pons C,Sarrión-Perdigones A,Fernández Ma,Peirats-Llobet M,Forment J,Moreno-Alvero M,Cutler S R,Albert A,Granell A,Rodríguez P I. 2014. Tomato PYR/PYL/RCAR abscisic acid receptors show high expression in root,differential sensitivity to the abscisic acid agonist quinabactin,and the capability to enhance plant drought resistance[J]. Journal of Experimental Botany,65(15):4451-4464.
Guo D,Zhou Y,Li H L,Zhu J H,Wang Y,Chen X T,Peng S Q. 2017. Identification and characterization of the abscisic acid(ABA) receptor gene family and its expression in response to hormones in the rubber tree[J]. Scientific Reports,7:45157. doi:10.1038/srep45157.
He Y,Hao Q,Li W Q,Yan C Y,Yan N,Yin P. 2014. Identification and characterization of ABA receptors in Oryza sativa[J]. PLoS One,9(4):e95246.
He Z H,Zhong J W,Sun X P,Wang B C,Terzaghi W,Dai M Q. 2018. The maize ABA receptors ZmPYL8,9,and 12 facilitate plant drought resistance[J]. Frontiers in Plant Science,9:422. doi:10.3389/fpls.2018.00422.
Iyer L M,Koonin E V,Aravind L. 2001. Adaptations of the helix-grip fold for ligand binding and catalysis in the START domain superfamily[J]. Proteins,43(2):134-144.
Libault M,Farmer A,Joshi T,Takahashi K,Langley R J,Franklin L D,He J,Xu D,May G,Stacey G. 2010. An integrated transcriptome atlas of the crop model Glycine max,and its use in comparative analyses in plants[J]. The Plant Journal,63(1):86-99.
Li X Y,Li G M,Li Y,Kong X G,Zhang L,Wang J M,Li X F,Yang L. 2018. ABA receptor subfamily Ⅲ enhances abscisicacid sensitivity and improves the drought toleran-ce of Arabidopsis[J]. International Journal of Molecular Sciences,19(7):1938.
Ma Y,Szostkiewicz I,Korte A,Moes D,Yang Y,Christmann A,Grill E. 2009. Regulators of PP2C phosphatase activity function as abscisic acid sensors[J]. Science,324(5930):1064-1068.
Mega R,Abe F,Kim J S,Yuuri T,Keisuke T,Hisato K,Yoichi S,Kousuke H,Hisashi T,Jun K,Sean R S,Masanori O. 2019. Tuning water-use efficiency and drought toleran-ce in wheat using abscisic acid receptors[J]. Nature Plants,5(2):153-159.
Melcher K,Ng L M,Zhou X E,Soon F F,Xu Y,Suino-Powell K M,Park S Y,Weiner J J,Fujii H,Chinnusamy V,Kovach A,Li J,Wang Y H,Li J Y,Peterson F C,Jensen D R,Yong E L,Volkman B F,Cutler S R,Zhu J K,Xu H E. 2009. A gate-latch-lock mechanism for hormone signalling by abscisic acid receptors[J]. Nature,462(7273):602-608.
Miao C B,Xiao L H,Hua K,Zou C S,Zhao Y,Bressan R A,Zhu J K. 2018. Mutations in a subfamily of abscisic acid receptor genes promote rice growth and productivity[J]. Proceedings of the National Academy of Sciences of the United States of America,115(23):6058-6063.
Miao Y,Lv D,Wang P,Wang X C,Chen J,Miao C,Song C P. 2006. An Arabidopsis glutathione peroxidasefunctions as both a redox transducer and a scavenger in abscisic acid and drought stress responses[J]. The Plant Cell,18(10):2749-2766.
Miyazono K I,Miyakawa T,Sawano Y,Kubota K,Kang H J,Asano A,Miyauchi Y,Takahashi M,Zhi Y,Fujita Y,Yoshida T,Kodaira K-S,Kazuko Y S,Tanokura M. 2009. Structural basis of abscisic acid signalling[J]. Nature,462(7273):609-614.
Nishimura N,Sarkeshik A,Nito K,Park S Y,Wang A,Carvalho P C,Lee S,Caddel D F,Cutler S R,Chory J,Yates J R,Schroeder J I. 2010. PYR/PYL/RCAR family members are major in-vivo ABI1 protein phosphatase 2C-interacting proteins in Arabidopsis[J]. The Plant Journal,61(2):290-299.
Park S Y,Fung P,Nishimura N,Jensen D R.,Fujii H,Zhao Y,Lumba S,Santiago J,Rodrigues A,Chow T F,Alfred S E,Bonetta D,Finkelstein R,Provart N J,Desveaux D,Rodriguez P L,McCourt P,Zhu J K,Schroeder J I,Volkman B F,Cutler S R. 2009. Abscisic acid inhibits type 2C protein phosphatases via the PYR/PYL family of START proteins[J]. Science,324(5930):1068-1071.
Pizzio G A,Rodriguez L,Antoni R,Gonzalez-Guzman M,Yunta C,Merilo E,Kollist H,Albert A,Rodriguez P L. 2017. The PYL4 A194T mutant uncovers a key role of PYR1-LIKE4/PROTEIN PHOSPHATASE 2CA interaction for abscisic acid signaling and plant drought resistance[J]. Plant Physiology Biochemistry,163(1):441-455.
Quan W L,Hu Y L,Mu Z X,Shi H T,Chan Z L. 2018. Overexpression of AtPYL5 under the control of guard cell specific promoter improves drought stress tolerance in Arabidopsis[J]. Plant Physiology & Biochemistry,129:150-157.
Rodriguez L,Gonzalez-Guzman M,Diaz M,Rodrigues A,Izquierdo-Garcia A C,Peirats-Llobet M,Fernandez M A,Antoni R,Fernandez D,Marquez J A,Mulet J M,Albert A,Rodriguez P L. 2014. C2-domain abscisic acid-related proteins mediate the interaction of PYR/PYL/RCAR abscisic acid receptors with the plasma membrane and regulate abscisic acid sensitivity in Arabidopsis[J]. The Plant Cell,26(12):4802-4820.
Santiago J,Rodrigues A,Saez A,Rubio S,Antoni R,Dupeux F,Park S Y,Márquez J A,Cutler S R,Rodriguez P L. 2009. Modulation of drought resistance by the abscisic acid receptor PYL5 through inhibition of clade A PP2Cs[J]. The Plant Journal,60(4):575-588.
Soon F F,Ng L M,Zhou X E,West G M,Kovach A,Tan M H E,Suino-Powell K M,He Y Z,Xu Y,Chalmers M J,Brunzelle J S,Zhang H M,Yang H Y,Jiang H L,Li J,Yong E L,Cutler S,Zhu J K,Griffin P R,Melcher K,Xu H E. 2012. Molecular mimicry regulates ABA signaling by SnRK2 kinases and PP2C phosphatases[J]. Science,335(6064):85-88.
Wang Y Z,Chen Z H,Zhang B,Hills A,Blatt M R. 2013. PYR/PYL/RCAR abscisic acid receptors regulate K+ and C1- channels through reactive oxygen species-mediated activation of Ca2+ channels at the plasma membrane of intact Arabidopsis guard cells[J]. Plant Physiology,163(2):566-577.
Ye Y,Yuji A,Yoshimasa N,Shintaro M,Izumi C M,Yoshiyuki M. 2016. Involvement of OST1 protein kinase and PYR/PYL/RCAR receptors in methyl jasmonate-induced stomatal closure in Arabidopsis guard cells[J]. Plant and Cell Physiology,57(8):1779-1790.
Yin Y,Adachi Y,Ye W,Hayashi M,Nakamura Y,Kinoshita T,Mori I C,Murata Y. 2013. Difference in abscisic acid perception mechanisms between closure induction and opening inhibition of stomata[J]. Plant Physiology,163(2):600-610.
Yu Y T,Wu Z,Lu K,Bi C,Liang S,Wang X F,Zhang D P. 2015. Overexpression of the MYB37 transcription factor enhances abscisic acid sensitivity,and improves both drought tolerance and seed productivity in Arabidopsis thaliana[J]. Plant Molecular Biology,90(3):267-279.
Zhao Y,Chan Z,Gao J,Xing L,Cao M,Yu C,Hu Y,You J,Shi H,Zhu Y,Gong Y,Mu Z,Wang H,Deng X,Wang P,Bressan R A,Zhu J K. 2016. ABA receptor PYL9 promotes drought resistance and leaf senescence[J]. Procee-dings of the National Academy of Sciences of the United States of America,113(7):1949-1954.
Zhao Y,Xing L,Wang X,Hou Y J,Gao J H,Wang P C,Duan C G,Zhu X H,Zhu J K. 2014. The ABA receptor PYL8 promotes lateral root growth by enhancing MY B77-dependent transcription of auxin-responsive genes[J]. Science Signaling,7(328):ra53.
(责任编辑 陈 燕)
收稿日期:2020-05-13
基金项目:国家自然科学基金项目(31801444)
作者简介:*为通讯作者,解莉楠(1977-),博士,副教授,主要從事植物抗逆生物学研究工作,E-mail:linanxie@nefu.edu.cn。张兆涵(1994-),研究方向为大豆抗逆生物学,E-mail:zhangzhaohan@nefu.edu.cn

