不同饲养模式下成年滩羊瘤胃真菌多样性及菌群结构的变化
付子琳,陈 林,李 娜,徐晓锋,张力莉
(宁夏大学农学院,宁夏银川 750021)
滩羊是我国西北地区独特的绵羊品种,以宁夏中、北部为中心产区,年存栏规模约300 万只。瘤胃是反刍动物最主要的消化器官,其菌群结构影响瘤胃的发酵功能[1]。Owens 等[2]研究发现,瘤胃微生物对摄入饲料进行代谢作用,其代谢产物和微生物蛋白将作为反刍动物的能量和蛋白质的最主要来源。与此同时,原虫吞噬行为、自溶现象以及高的脱氨基活性会造成氮的无效利用。因此,改善瘤胃菌群结构对促进反刍动物生长健康、提高其纤维降解和饲料转化效率具有重要意义。瘤胃微生物主要包括纤毛虫、真菌及细菌,其中,真菌占总量的5%~20%。瘤胃真菌含有消化植物组织所需要的酶,包括纤维素酶、木聚糖酶以及其他水解酶等,主要附着于纤维类植物饲料表面,参与复杂碳水化合物的初步降解。虽然真菌占瘤胃微生物的比例不多,但真菌个体较大,且其复杂性以及酶活性在消化纤维中起主要作用[3-5]。
目前能够检测并定义的瘤胃厌氧真菌主要包括新美鞭菌属、盲肠鞭菌、梨囊鞭菌属、Anaeromyces属、Orpinomyces属及Cyllamyces属6 个属[6]。不同种属微生物在瘤胃内占据不同生态位,其组成与种群密度随饲喂而改变[7]。近年来,滩羊的传统生产方式由放牧转为舍饲,由采食荒漠及干旱草原牧草为主转向以人工牧草和作物秸杆以及补充精料为主。为此本研究基于ITS 测序分析技术首次检测饲养方式对滩羊瘤胃真菌菌群结构与多样性的影响,为滩羊的营养调控提供理论基础。
1 材料与方法
1.1 试验设计 选择体重(30.12±3.23)kg、体况健康的9 月龄去势滩羊24 只,随机分为放牧组和舍饲组,放牧组滩羊于2017 年9 月份在盐池宁鑫生态牧场全天放牧,为期3 个月,可自由采食草场牧草(沙打旺、胡枝子、苦豆子、柠条、甘草等),每天放牧13 h(07:00—20:00),无补饲;舍饲组饲喂混合日粮,原料成分及营养成分见表1,每天早晚饲喂2 次,自由采食。
1.2 样品采集与处理 在试验滩羊12 月龄时,2 组随机各取5 只滩羊进行屠宰,采集瘤胃内容物于50 mL 离心管中,放置于-80℃冰箱(DW-HL528S,中科美菱)中冻存,用于瘤胃真菌菌群分析。共10 个样品,其中放牧组5 个(G12-1、G12-2、G12-3、G12-4、G12-5),舍饲组5个(R12-1、R12-2、R12-3、R12-4、R12-5)。

表1 舍饲组滩羊日粮组成及营养成分(干物质基础)
1.3 样品DNA 提取与测序 运用试剂盒法提取样本总DNA,试剂盒由南京建成生物研究所提供。样本总DNA 提取后,利用PCR 仪(ETC811,EASTWIN),用带有barcode 的特异引物扩增ITS rDNA 的ITS2 区域。引物序列为KYO2F:5´-GATGAAGAACGYAGYRAA-3´;ITS4R:5´-TCCTCCGCTTATTGATATGC-3´。PCR 反应体系为50 µL:DNA 1 µL,上、下游引物(10 μmol/L)各1.5 µL,2 mmol/L dNTPs 5µL,25 mmol/L MgSO43 µL,10×Buffer KOD 5 µL,KOD 酶1 µL,ddH2O(无酶水)32 µL。PCR 反应条件:94℃预变性2 min,98℃变性10 s,退火30 s,退火温度为62℃,68℃延伸30 min,共32个循环;最后68℃延伸5 min。使用AMPure XP Beads对扩增产物进行纯化,用ABI StepOnePlus Real-Time PCR System(Life Technologies)对其纯化产物进行定量,引物序列同上,根据illumina Hiseq2500 的PE250 模式pooling 上机测序。
1.4 统计分析 数据采用Excel 2016 进行初步处理,采用 SAS 8.2 统计软件中的完全随机设计方差分析进行组间差异显著性比较;菌群丰度采用Metastats 软件进行分析。结果表示为平均值±标准误,P<0.05 为差异显著,P<0.01 为差异极显著。
2 结果
2.1 样品测序深度分析 如图1 所示,当测序深度超过5 000 reads 时,曲线趋于平缓,各样品达到饱和状态,表明本试验的测序深度足以覆盖各样品中大多数微生物,测序深度增加已经不影响物种多样性,样品Shannon 曲线高度没有明显组间差异。
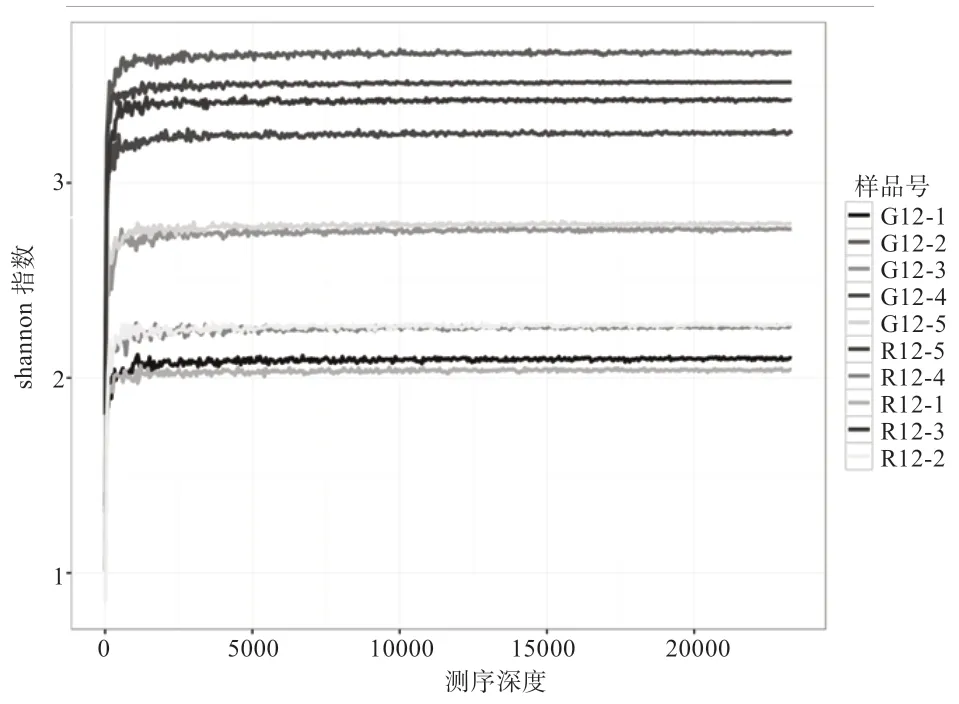
图1 Shannon 曲线
2.2 瘤胃真菌OTUs 比较 经Illumina Hiseq 测序结束后,除去低质序列,通过序列拼接,应用Mothur,根据97% 的序列相似度,对序列进行OTU 划分。根据OTU 聚类分析结果,分析不同样品的聚类信息,依照其共有、特有的OTU 信息绘制韦恩图(Venn graph)。如图2 所示,放牧组(G12)滩羊瘤胃真菌OTU 总数有347 个,舍饲组(R12)OTU 总数有322 个,2 组共有的OTU 数量为169。
2.3 PCoA 分析 基于样本间的加权(Weighted Unifrac)和非加权(Unweighted Unifrac)数据结果,绘制出PCoA(Principal Coordinates Analysis)图形,直观显示不同环境样品中微生物进化上的相似性及差异性。分析结果中,样品越相似,反映在PCoA 图中的距离越近,而且不同环境间的样品可能表现出各自聚集的分布情况。
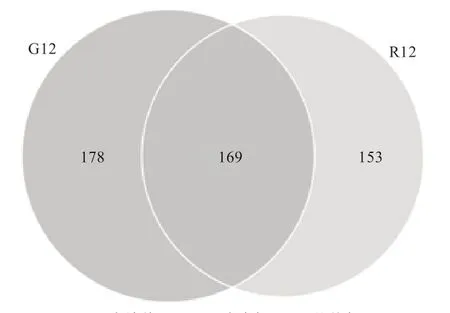
图2 试验滩羊瘤胃真菌菌群韦恩图
如图3-a 所示,未加权仅考虑物种有无变化时,放牧组(G12)5 个样本相对集中,舍饲组(R12)1、3、5 号样本相对集中,而2 号、4 号在第二主成分纵向上样本距离较大,与组内其他样本相似性较小;而加权后(图3-b)同时考虑了物种有无和丰度的变化,放牧组中1 号与其他4 个样本差异较大,舍饲组内样本差异性较大。
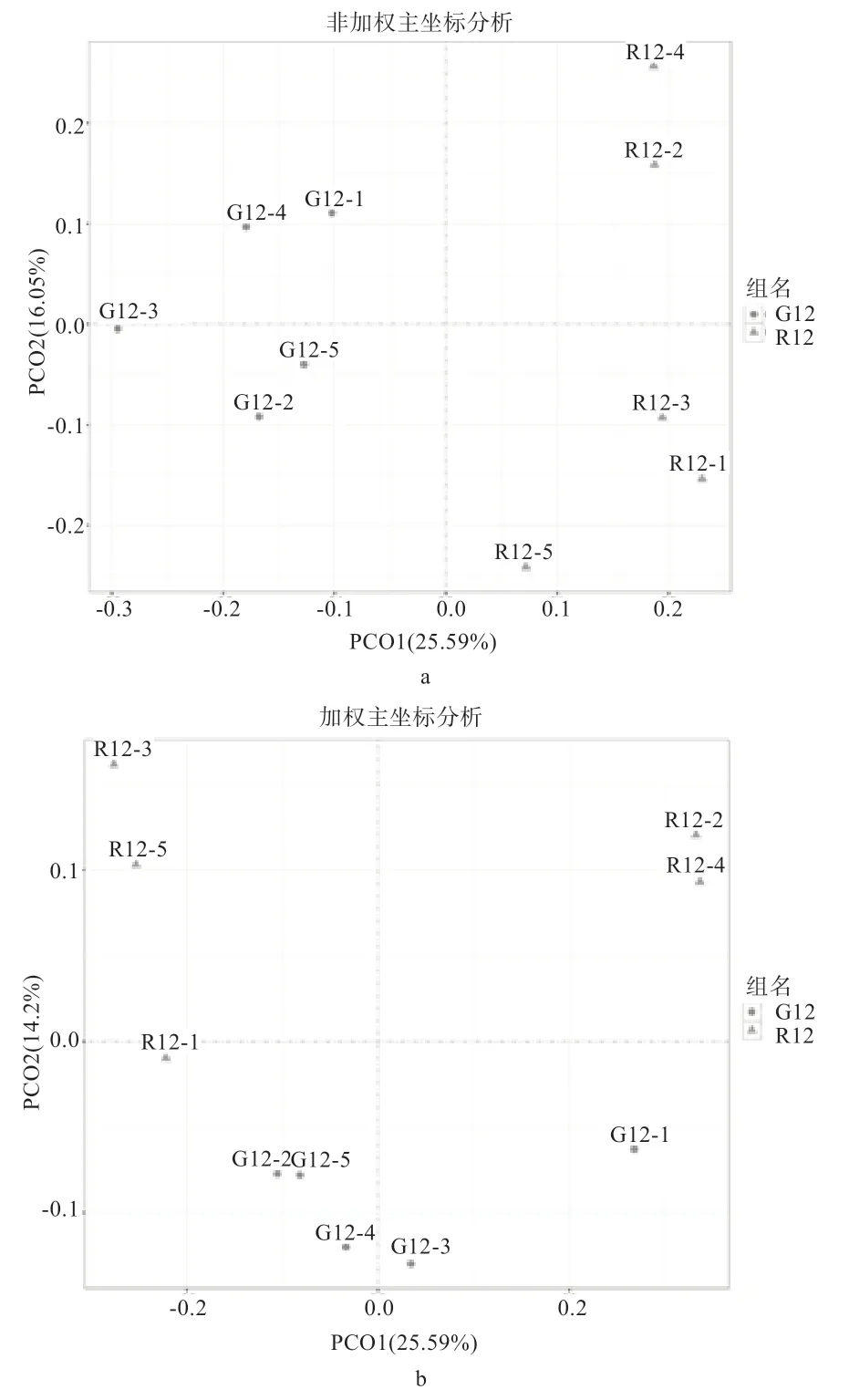
图3 PCoA 分析
2.4 瘤胃真菌Alpha 多样性分析 由表2 可知,放牧组的Chao1 和ACE 值均显著高于舍饲组,说明放牧组滩羊瘤胃真菌丰富度高于舍饲组;而Shannon 和Simpson指数组间差异不显著,表明当综合物种丰富度和均匀度比较时,不同饲养方式对成年滩羊瘤胃真菌多样性影响不显著。

表2 样品在0.03 距离下的Alpha 丰富程度
2.5 瘤胃真菌菌群结构
2.5.1 门水平 由表3 可知,放牧组中鉴定出6 个门,舍饲组中鉴定出4 个门,含量在5% 以上的优势菌门均为新丽鞭毛菌门(Neocallimastigomycota)和子囊菌门(Ascomycota),含量超过90%;含量在0.5%~5%的次优菌门均为担子菌门(Basidiomycota) 和1 种未注释菌门;此外,仅在放牧组中检测出微量的毛霉门(Mucoromycota)和被孢霉门(Mortierellomycota)。滩羊瘤胃真菌不同菌门在不同饲养方式下均无显著差异,但放牧组鉴定出的滩羊瘤胃菌门多于舍饲组。
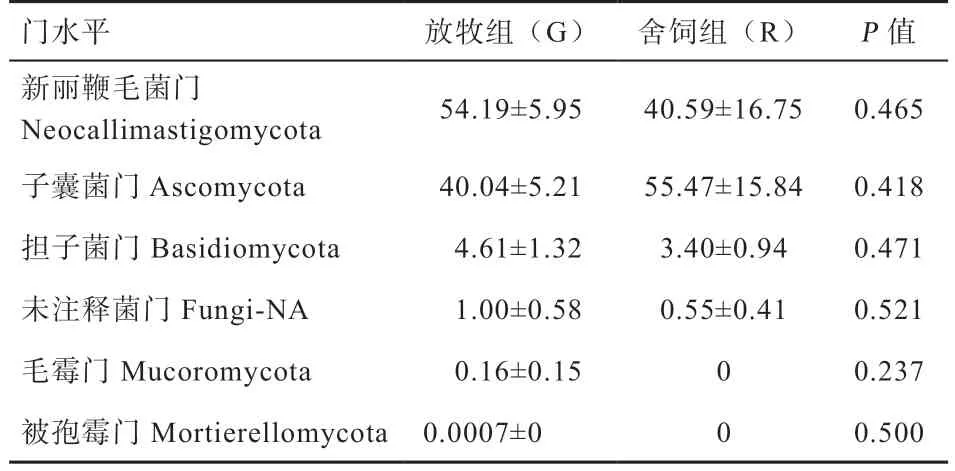
表3 滩羊瘤胃真菌结构门水平的变化
2.5.2 属水平 放牧组共鉴定出65 个属,舍饲组共鉴定出58 个属,放牧组多于舍饲组。2 组属水平上的优势菌(丰度在5% 以上)、次优菌属(丰度均在0.5%~5%)及差异菌属见表4。本试验中,差异菌属共有6 种,其中优势菌属均无显著性差异,次优菌属有4种。放牧组共鉴定出3 个优势菌属,分别为梨囊鞭菌属(Piromyces)、Kazachstania属及1 个未注释菌属;9个次优菌属分别为赤霉菌属(Gibberella)、附球菌属(Epicoccum)、Naganishia属、线黑粉菌属(Filobasidium)、酵母属(Saccharomyces)、链格孢属(Alternaria)、Meyerozyma属、Papiliotrema属、未注释新美鞭菌科(Neocallimastigaceae-NA)、新美鞭菌属(Neocallimastix)及Pleonectria属;其中放牧组Naganishia属、线黑粉菌属(Filobasidium)和酵母属(Saccharomyces)3 个菌属含量极显著高于舍饲组。舍饲组共鉴定出4 个优势菌群,分别为梨囊鞭菌属(Piromyces)、Kazachstania、1 个未注释菌属及附球菌属(Epicoccum);8 个次优菌属分别为链格孢属(Alternaria)、Meyerozyma属、未注释新美鞭菌科(Neocallimastigaceae-NA)、新美鞭菌属(Neocallimastix)、黄丝曲霉属(Talaromyces)、节担菌属(Wallemia)、双足囊菌属(Dipodascus)及Paraphaeosphaeria属;其中,节担菌属(Wallemia)含量极显著高于放牧组;Paraphaeosphaeria属含量较放牧组有升高趋势(P>0.05)。此外,放牧组中Pleonectria属含量显著高于舍饲组;而平脐蠕孢属(Bipolaris)和裂褶菌属(Schizophyllum)含量有升高趋势(P>0.05);香蘑属(Lepista)含量显著降低;而盲肠鞭菌属(Caeco myces)含量有降低趋势(P>0.05)。
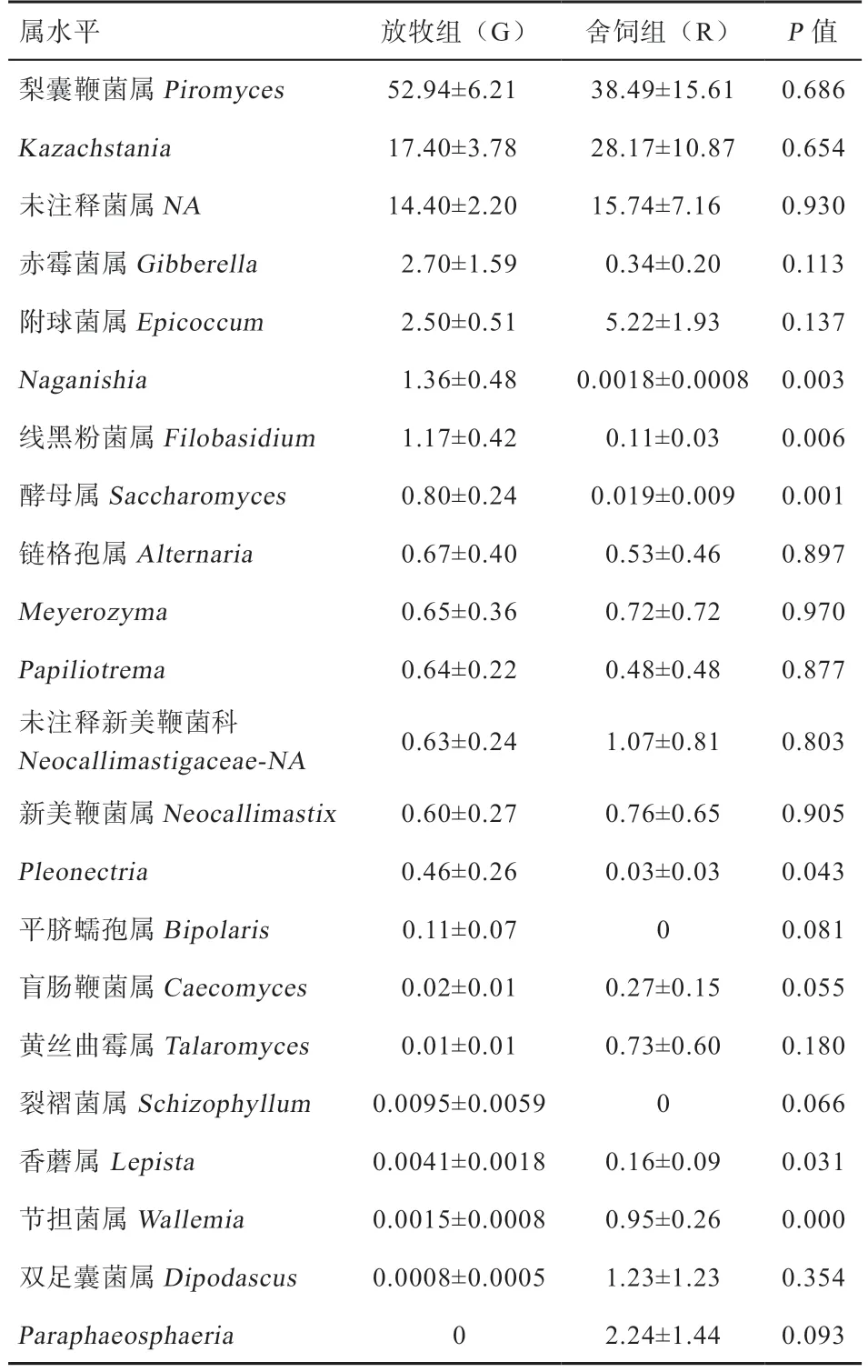
表4 滩羊瘤胃真菌结构属水平的变化
3 讨 论
3.1 饲养方式对滩羊瘤胃真菌菌群丰富度影响 厌氧真菌在瘤胃微生物占比较低且生长速度慢,与细菌相比,在瘤胃系统中处于劣势。但真菌能够与细菌竞争生长环境并存活,应当具备其特有的功能[8]。瘤胃真菌假根可深入刺穿到植物细胞壁角质层内,利用酶对植物细胞壁进行分解。虽然目前研究表明植物纤维主要由瘤胃细菌降解,但是真菌能够首先植入纤维组织内部进行反应[9-10]。本试验中,结合OTU 比较以及Alpha 多样性Chao1和ACE 指数的分析可以看出,放牧组瘤胃真菌丰富度显著高于舍饲组。Grenet 等[11]研究发现,与淀粉含量较高的饲料相比,木质素含量较高的饲料更有利于厌氧真菌生长,而且真菌会选择性地附着于纤维成分。孙云章等[12]采用体外厌氧共培技术发现,适当的精粗比有利于瘤胃真菌和纤维降解细菌在发酵前期建立起相对稳定的共培养体系;但随着精料比例的增大,真菌数量逐渐降低直到消失,细菌数量逐渐上升,同时系统的产气量显著增加。Belanche 等[13]研究表明,与高淀粉日粮相比,瘤胃细菌真菌的丰富度在奶牛采食高中性洗涤纤维日粮时丰富度更高,且纤维降解菌的浓度显著增加。本试验中,采食相对较高含量纤维的放牧组滩羊瘤胃真菌丰富度显著增加,与前人结果相一致。
3.2 饲养方式对瘤胃真菌菌群结构的影响 反刍动物所摄入的营养物质大部分是进入瘤胃后通过瘤胃微生物发酵进而吸收的[14],因此日粮的营养结构影响瘤胃的发酵功能,对瘤胃菌群结构有所影响。多数瘤胃厌氧真菌属都能分解纤维素,可产生纤维素酶、半纤维素酶和果胶酶等,用于降解植物细胞壁[15]。而瘤胃不同属厌氧真菌降解植物的能力有所不同,新美鞭菌属(Neocallimastix)和梨囊鞭菌属(Piromyces)降解秸秆的能力较强,而盲肠鞭菌属(Caecomyces)相对较弱[16]。本试验中,梨囊鞭菌属(Piromyces)在2 组中均是第一优势菌属,虽然含量均值相差约14%,但并没有显著的组间差异;而盲肠鞭菌属(Caecomyces)属含量在舍饲组中有升高趋势;其他纤维降解真菌,如未注释新美鞭菌科(Neocallimastigaceae-NA)和新美鞭菌属(Neocallimastix)组间差异也不显著。Kazachstania及1 个未注释菌属作为2 个优势菌属,其功能属性还有待更深的研究。
Naganishia、线黑粉菌属(Filobasidium)和酵母属(Saccharomyces)3 个菌属在放牧组中为次优菌属,且含量显著高于舍饲组。这3 个菌属均属于酵母菌[17-18]。饲粮中添加酵母菌制剂可以改善反刍动物能过改善瘤胃内环境,且酵母属(Saccharomyces)具有降解赭曲霉毒素的能力,有一些酵母菌还可以降解呕吐毒素、玉米赤霉烯酮、伏马毒素[19-20]。
舍饲组节担菌属(Wallemia)和香蘑属(Lepista)含量显著升高。有研究表明,香蘑属(Lepista)能够较好地利用二糖,可以在偏酸性(pH=4.5)环境中生存[21]且存在腐生菌种[22];节担菌属(Wallemia)通常生长在食物与室内环境中,在腐生植物上也可以提取到[23],节担菌属(Wallemia)在高温发酵和腐熟阶段均为优势菌群[24]。Paraphaeosphaeria属在舍饲组内有升高趋势,但其在瘤胃中的功能作用报道较少,有待更进一步研究。
4 结 论
综上所述,不同饲养方式对12 月龄滩羊瘤胃真菌菌群多样性并未产生显著影响,但放牧组滩羊瘤胃真菌丰富度显著高于舍饲组;不同饲养方式对瘤胃真菌菌群结构存在存在一定影响,其中差异菌属有6 种,其中包含4 种次优菌属。

