藜麦内参基因筛选及盐胁迫相关基因表达分析
贾冰晨,王 宇,张东亮,吴筱林,尹海波,陈世华,郭善利
(烟台大学生命科学学院,山东 烟台 264005)
实时荧光定量PCR(quantitative Real-Time PCR,qRT-PCR)是一种能够高效、定量分析基因表达量的技术手段,具有灵敏度高、特异性强、重复性好以及反应速度快等特点[1].但是有许多因素会对定量分析结果的准确性造成影响,如RNA提取的质量与产量、RNA反转录效率、qRT-PCR反应体系初始模板量、酶促反应效率等[2].因此,为了排除基因表达的时空间差异、减少试验材料间的样本差异、减小实验过程中的系统误差,获得精确度高、科学合理的数据,通常需要引入一个或多个表达较为稳定的基因作为内参基因,通过稳定表达的内参基因对检测基因的表达进行校正及标准化分析[3].筛选特定物种、或者在特定处理中稳定表达的内参基因是进行荧光定量实验的前提[4-5],目前基于常用的geNorm、NormFinder和Bestkeeper3种分析方法,可以筛选出特定实验条件下表达稳定的内参基因[6-7].
藜麦(Chenopodiumquinoawilld)是苋科、藜亚科、藜属的一年生自花授粉双子叶植物,具有耐寒、耐旱、耐瘠薄、耐盐碱等特性[8],其种植、推广和不断增长的消费需求也使得其遗传育种等基础生物学研究受到了科研工作者越来越多的关注.本研究利用筛选出的最佳内参基因对藜麦盐胁迫下小分子渗透调节物质合成相关基因脯氨酸合成酶基因(P5CS1)以及甜菜碱合成相关基因胆碱单加氧酶基因(CMO)的基因表达进行了分析[9].
1 材料与方法
1.1 材料与处理
用500 mmol NaCl溶液浇灌处理8片真叶期藜麦苗2 d,样品做3个生物学重复,剪取地上部分为试验材料.
1.2 RNA提取与反转录
使用Vazyme公司生产的Fast Pure Plant Total RNA Isolation Kit试剂盒提取处理后藜麦苗地上部分组织材料RNA,然后用1%琼脂糖凝胶电泳检测RNA的完整性,并且使用GE Healthcare公司生产的Nano VueTM紫外/可见光分光光度计检测RNA浓度和纯度.随后使用Vazyme公司生产的HiScript®Ⅱ Q Select RT SuperMix for qPCR反转录试剂盒合成cDNA第一链.
1.3 候选内参基因特异性引物设计
根据NCBI网站(https://www.ncbi.nlm.nih.gov/)发布的藜麦全基因组序列和在其他物种中常用的候选内参基因,选取了(以NCBI网站发布的Gene ID为准)LOC110715281 (ACT-1),LOC110686159 (TUB-1),LOC110711758 (TUB-6),LOC110711181 (eIF3),LOC110710371 (EF1-α),LOC110711214 (GAPDH)共6个基因作为候选内参基因,以及盐胁迫相关基因LOC110722294 (P5CS1)和LOC110710124 (CMO).使用BeaconDesign8软件设计特异性引物(表1)并且在Primer-BLAST网站(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)上限定物种为藜麦对特异性引物进行检测,并送至北京六合华大基因科技有限公司合成引物.
1.4 候选内参基因实时荧光定量PCR分析
qRT-PCR在QIAGEN®公司生产的Rotor-Gene Q实时荧光定量PCR仪中进行.使用Vazyme公司生产的Cham QTMUniversal SYBR®qPCR Master Mix试剂进行qRT-PCR反应.实时荧光定量PCR反应体系总体积为20 μL:2×Cham Q Universal SYBR qPCR Master Mix,10 μL;Forward Primer (10 μmol·L-1),0.4 μL;Reverse Primer (10 μmol·L-1),0.4 μL;10倍稀释cDNA模板,1 μL;ddH2O,8.2 μL.反应程序为:95 ℃,3 min;95 ℃,10 s,58 ℃,30 s,72 ℃,30 s,40个循环.进行72~95 ℃熔解曲线分析.
1.5 数据处理与分析
数据处理使用geNorm、NormFinder和BestKeeper 3个软件进行分析,并利用Excel 2016作图.
2 结果与分析
2.1 RNA检测
将提取的藜麦地上部分样品RNA用1%琼脂糖凝胶电泳进行检测.结果(图1)显示, 28S和18S RNA条带清晰,表明RNA样品没有降解.使用Nano VueTM紫外/可见光分光光度计检测的结果显示,各样品A260/A280在1.9~2.0之间,A260/A230在2.0~2.3之间,表明RNA的纯度高,能满足反转录实验的要求.
2.2 候选内参基因引物特异性PCR检测
使用反转录合成的cDNA作为模板,通过普通PCR验证内参引物扩增特异性(图2).结果显示,引物特异性较好,没有非特异性扩增及引物二聚体出现,并且确定6对候选内参基因引物Tm值为57 ℃.
2.3 候选内参基因表达丰度
对6个候选内参基因的循环阈值(Ct)进行整理分析,评估这些基因的平均表达丰度(图3)[10].结果显示,这6个候选内参基因的Ct值位于17~28之间,其中4个候选内参基因Ct值集中在20~26之间,说明表达丰度适中.其中GAPDH的Ct值最低,范围位于17~19之间,说明其表达丰度最高;而EF1-α的Ct值最高,范围位于于25~28之间,说明其表达丰度最低.
2.4 geNorm程序分析候选内参基因的稳定性
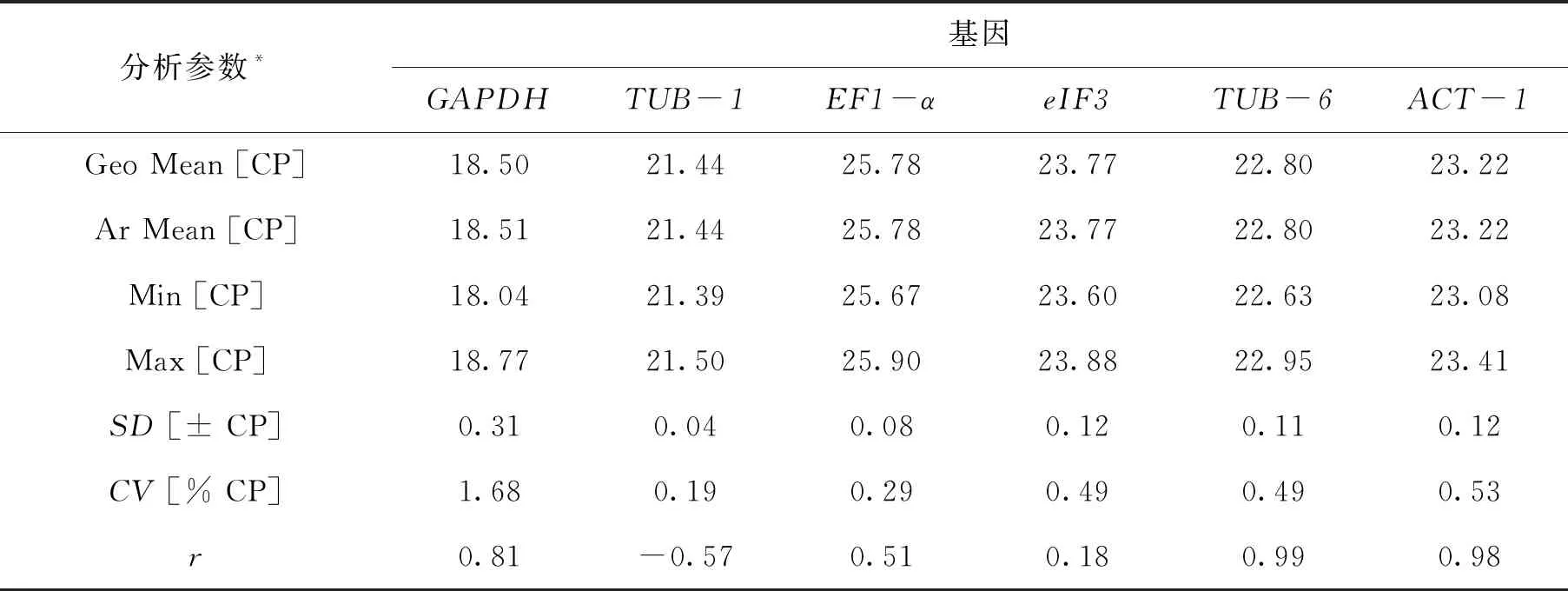
geNorm通过计算每个候选内参基因在特定实验条件下、特定组织材料中表达稳定值(M)来判定其表达稳定性[11].该程序以M=1.5为阈值,当候选内参基因的M值大于1.5则不适于作为候选内参基因.因此M值越小,候选内参基因稳定性越高,越适合特定条件下作为候选内参基因.由程序分析结果可知(图4(a)),6个候选内参基因的M值均小于1.5,其中ACT-1最小,为0.255;GAPDH最大,为0.378,表明这些基因都可作为候选内参基因,按照M值由低到高排序依次为:ACT-1 NormFinder与geNorm类似, 通过计算候选内参基因的表达稳定值M来判定表达稳定性的大小[11].M值越小,越适合作为候选内参基因;反之,则不适合.由程序分析结果可知(图5), 6个候选内参基因M值从低到高排列为ACT-1 BestKeeper程序根据不同候选内参基因在样品中平均Ct值分别计算相关系数(coefficient of correlation,r)、标准差(standard deviation,SD)和变异系数(coefficient of variation,CV)来判定候选内参基因的稳定性[11].判定原则是相关系数越大,标准差和变异系数越小,内标基因表达稳定性越好.由程序分析结果可知(表2),6个候选内参基因SD值均小于1,说明这些基因都适合作为内参基因.其中,ACT-1和TUB-6相关系数较大,并且标准差和变异系数比较小,故二者适合作为内参基因.这与geNorm和NormFinder程序分析结果相似. 表2 Bestkeeper分析候选内参基因表达稳定性 综合上述3种程序分析显示,在盐胁迫下藜麦ACT-1与TUB-6作为荧光定量PCR内参基因最为稳定.故对2条内参基因引物的熔解曲线和标准曲线进行分析[12],结果如图6、表3所示,ACT-1和TUB-6熔解曲线(n=3)出现明显单一峰,说明引物特异性较好,并且Tm值分别为80.5 ℃和83.5 ℃. 表3 实时荧光定量PCR中2条内参基因标准曲线参数 在盐胁迫下,由于外界渗透势较低,植物细胞会发生水分亏缺现象,即渗透胁迫.多数植物能够通过积累大量的代谢物质如糖类(果糖、蔗糖、海藻糖等)、氨基酸(脯氨酸)等来调节植物细胞内渗透压与外界平衡,降低体细胞水势,保持膨压.维持植物体内生理代谢平衡,保持内环境稳态.植物的脯氨酸和甜菜碱是公认的小分子渗透调节物质[13-14],两种物质的合成与积累是植物应答渗透胁迫、离子胁迫以及次生氧化胁迫的手段.分别使用筛选出的ACT-1和TUB-6作为内参基因通过qRT-PCR对脯氨酸合成相关基因P5CS1和甜菜碱合成相关基因CMO基因表达分析.结果显示(图7),P5CS1和CMO基因在以ACT-1和TUB-6为内参基因时,表达情况一致,由此可见ACT-1和TUB-6可作为藜麦盐胁迫时qRT-PCR分析时合适的内参基因.并且在盐胁迫下,藜麦P5CS1基因上调6倍左右,CMO基因上调30~40倍. 本研究分别使用geNorm、NormFinder和Bestkeeper内参基因分析程序对藜麦在NaCl胁迫下6个候选内参基因进行筛选,并利用实时荧光定量PCR技术对藜麦盐胁迫相关基因P5CS1和CMO基因相对表达量进行分析.综合上述3种程序的分析结果,在NaCl胁迫下藜麦实时荧光定量PCR内参基因最佳选择数目为2,最佳内参基因分别是ACT-1和TUB-6,并且2个内参基因表达丰度处于22~24之间,基因表达丰度适中,能够满足实验需求.利用筛选出的ACT-1和TUB-6作为内参基因,检测到在NaCl胁迫下,藜麦P5CS1基因表达上调6倍左右,CMO基因表达上调30~40倍.由于基因表达存在时空间差异,经验式的认为管家基因都可以作为内参基因是片面的[15],所以筛选特定物种、特定处理下实时荧光定量PCR内参基因是有必要的.在不同物种,在同一物种不同组织、不同生长阶段、不同胁迫条件下内参基因表达的稳定性不尽相同[16],所以筛选特定物种中、特定处理下表达稳定的内参基因变得尤为重要.随着藜麦在基因表达、抗逆分子生物学领域研究的深入,筛选出稳定表达的内参基因成为研究的前提.此实验结果为NaCl胁迫下藜麦实时荧光定量PCR筛选出了可靠、稳定的内参基因,并从基因表达角度验证了甜菜碱的积累是藜麦应答NaCl胁迫、调节渗透平衡的主要方式,这与其他藜科、苋科植物盐胁迫下物质积累相关报道一致[17-18].2.5 NormFinder程序分析候选内参基因稳定性
2.6 Bestkeeper程序分析候选内参基因稳定性
2.7 内参基因熔解曲线和标准曲线分析

2.8 盐胁迫相关基因P5CS1和CMO基因表达分析
3 讨 论

