基于全基因序列的中国北方3省(区)马铃薯Y病毒遗传多样性分析
马俊丰 李小宇 张春雨 周雪平 王永志

摘要 本研究以馬铃薯Y病毒(PVY)全基因组为基础,分析吉林、黑龙江和内蒙古3省(区)PVY群体遗传多样性和群体分化,并评估突变、重组、选择等遗传力所起的作用。根据已报道的PVY全基因序列保守区设计4对引物,采用片段重叠法对来自内蒙古和吉林的24个PVY分离物全基因序列进行测定,并联合NCBI中已登录的9个黑龙江分离物全基因组序列进行遗传多样性参数评估、群体分化检验和分子变异等分析。结果显示,我国北方3省(区)PVY群体遗传多样性高,其中内蒙古和黑龙江PVY群体遗传多样性高于吉林群体,并且3个群体之间呈现一定程度的遗传分化。分子变异分析发现在PVY基因组中存在1 786个变异位点,表明我国北方3省(区)PVY群体变异程度较高,并且这种高变异度有85.54%来自各个马铃薯种植区内PVY个体的遗传变异。重组分析和系统发育分析发现,我国北方3省(区)PVY群体中重组株系占比高达90.3%,并具有明显的株系多样性,表明PVY重组株系已成为我国北方3省(区)马铃薯种植区的流行株系。选择压力分析显示,使用FEL和IFEL法分别检测出501个和315个净化压力选择位点,这表明3省(区)PVY群体受净化选择压力为主。以上结果表明,中国北方3省(区)PVY群体遗传多样性高,突变、重组和自然选择都对遗传多样性和群体分化存在一定影响。
关键词 马铃薯Y病毒(PVY); 遗传多样性; 基因组序列
中图分类号:
S 432.41
文献标识码: A
DOI: 10.16688/j.zwbh.2019571
Genetic diversity analysis of Potato virus Y in three provinces of north China based on the full genomic sequences
MA Junfeng1, LI Xiaoyu1, ZHANG Chunyu1, ZHOU Xueping2, WANG Yongzhi1, 2*
(1. Institute of Plant Protection, Jilin Academy of Agricultural Sciences, Gongzhuling 136100, China;
2. Institute of Plant Protection, Chinese Academy of Agricultural Sciences, Beijing 100193, China)
Abstract
Complete genome sequences of Potato virus Y (PVY) from three provinces (Jilin, Heilongjiang and Inner Mongolia) of north China were used to investigate its genetic diversity and population differentiation. Meanwhile, the effects of mutations, recombination and selection on population genetic dynamics were evaluated. The complete genomes of 24 PVY isolates from Jilin and Inner Mongolia were amplified and sequenced using 4 pairs of primers designed according to the conserved regions of the reported PVY isolates, and were then combined with complete genome sequences of 9 Heilongjiang isolates for estimation of genetic variation, population differentiation, molecular variation, etc. The genetic diversity of the PVY populations in the three provinces was high. Furthermore, the genetic diversities of PVY isolates from Heilongjiang and Inner Mongolia were higher than that of PVY from Jilin and there was a certain degree of genetic differentiation among the three populations. Analysis of molecular variation revealed that there were 1 786 mutation sites in PVY genomes, which indicated that the degree of variation in PVY populations in the three provinces was high; 85.54% of the variation was from individuals of different potato planting areas. Recombination and phylogenetic analysis showed that 90.3% strains were recombinant and there was obvious strain diversity in the PVY populations, indicating that the recombinant strains had become the epidemic strain in the potato cropping regions of the three provinces. Totally 501 and 315 purifying selection sites were detected by using FEL and IFEL methods, respectively, suggesting that the PVY populations in the three provinces were mainly affected by purifying selection pressure. In summary, the PVY populations in the three provinces of northern China had high genetic diversity, and mutations, recombination and selection all contributed to the genetic diversity and population differentiation.
Key words
Potato virus Y(PVY); genetic diversity; genome sequence
马铃薯病毒是危害马铃薯的主要病原物之一,已有报道显示,世界范围内能够侵染马铃薯的病毒至少有53种,其中10种已在我国被发现[1]。在我国已经发现的10种病毒中,马铃薯Y病毒Potato virus Y(PVY)发生范围最广,造成经济损失最为严重。近些年在我国多个马铃薯种植区检测到PVY[2-7]。PVY侵染马铃薯可引起叶脉坏死、叶面黄化褪绿、块茎环斑坏死等症状,导致马铃薯种质退化,产量降低,给马铃薯的生产造成严重损失[8-10]。
PVY是马铃薯Y病毒科Potyviridae马铃薯Y病毒属Potyvirus代表成员,其病毒粒体呈弯曲线状,每个病毒粒体包含一套完整的基因组。该基因组由正义单链RNA构成,长约9.7 kb,包含一个大的开放阅读框(open reading frame, ORF),两端含有非编码区(untranslated regions,UTRs)。基因组翻译时,开放阅读框先编码一个多聚蛋白(polyprotein),随后再通过自身编码的3个蛋白酶将多聚蛋白切割加工为P1、HC-Pro、P3等10个成熟蛋白[11]。除此之外,PVY基因组内还有一个通过移码翻译产生的蛋白——PIPO,该蛋白与P3蛋白的N′端以P3N-PIPO融合形式存在[12]。最终,PVY基因组通过这两种方式生成11个成熟的多功能蛋白,使整个基因组呈现不同程度的遗传多样性。
近些年,PVY呈高变异、多株系发展趋势,因此,PVY研究已不局限在分子检测、株系鉴定、蛋白质功能验证等方面[13-17],遗传变异及分子进化已成为新的研究方向。已有工作主要是选择其中1~2个功能基因研究PVY的遗传多样性[18-20],不能全面地反映PVY进化和遗传机制。因此,本研究通过扩增、克隆等手段获得吉林(JL)和内蒙古(NMG)PVY分离物全基因组序列24条,并联合GenBank中已提交的9条黑龙江(HLJ)分离物的全基因组序列进行序列比对分析、遗传多样性参数评估及群体分化检验等一系列分析,旨在基于全基因组序列更为准确地探明我国北方3省(区)PVY群体遗传多样性及进化机制。该研究结果将有助于了解我国北方地区PVY的发生、流行及变异趋势,为其有效防控提供理论依据。
1 材料与方法
1.1 毒源
2017年—2018年从吉林、内蒙古马铃薯种植区,用随机抽样法采集带有花叶、褪绿和坏死等典型病毒症状的马铃薯叶片。先采用酶联免疫吸附法(enzyme linked immunosorbent assay, ELISA)检验病毒,之后对病毒CP基因进行扩增,将确定为PVY分离物的叶片密封保存在-80℃超低温冰箱中。
1.2 主要试剂
RNeasy Plant Mini Kit,QIAGEN公司;ReverAid First Strand cDNA Synthesis Kit,赛默飞世尔科技公司(Thermo Fisher Scientific);TaKaRa LA Taq、pMD18-T Vector Cloning Kit、DNA Marker,宝生物工程有限公司;AxyPrep DNA Gel Extraction Kit,爱思进生物技术公司(Axygen)。
1.3 试验方法
1.3.1 RNA提取及基因克隆
按照RNeasy Plant Mini Kit说明书,从感染PVY的马铃薯叶片中提取总RNA。以提取的叶片总RNA为模板,以OligodT为引物,按照反转录试剂盒说明书合成cDNA第一链。根据GenBank中已提交的PVY不同分离物基因组序列的保守区,设计用于扩增PVY全基因序列的简并引物和特异性引物(表1)。引物委托吉林省库美生物科技有限公司合成。
PCR扩增采用50 μL反应体系:TaKaRa LATaq (5 U/L)0.5 μL、10×LA PCR buffer Ⅱ(Mg2+Plus)5 μL、dNTP mixture 5 μL、cDNA 1 μL、正、反向引物(10 μmol/L)各 1 μL、超纯水36.5 μL。PCR反应条件:94℃预变性5 min;94℃变性30 s,退火30 s(退火温度视基因片段而定),72℃延伸若干分鐘(延伸时间按1 kb/min计算),共30个循环;最后72℃再延伸10 min。
取4 μL PCR产物用1.5%琼脂糖凝胶电泳进行检测。检测后切取目的条带,利用胶回收试剂盒进行纯化,纯化产物分别与pMD18-T载体连接,连接产物转化到大肠杆菌DH5α感受态细胞中。经菌液PCR验证后筛选出阳性克隆,随机选取3~5个阳性克隆,委托吉林省库美生物科技有限公司进行测序,并通过测序峰图及序列比对排除PCR过程中可能引起的突变。
1.3.2 序列处理
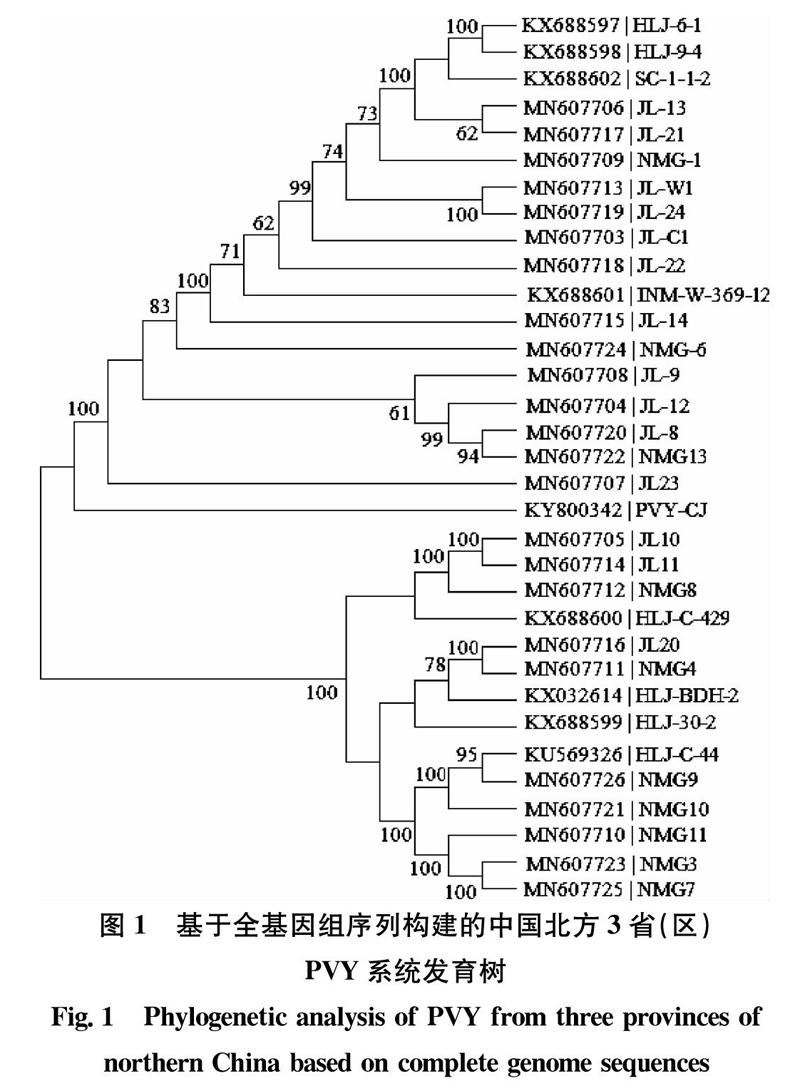
测序获得的吉林和内蒙古PVY分离物各序列片段使用DNAMAN 6.0软件进行拼接,并通过PVY参考序列(GenBank登录号为NC_001616)进行校正。校正时先使用MEGA 6.0软件中的Muscle程序对PVY全基因组序列进行多重比对,之后使用Muscle(codon)程序对PVY基因组编码区进行比对,最后将编码区翻译为相应的氨基酸序列,进行人工校正,以确保基因组序列的准确性。试验所用的黑龙江PVY分离物基因组序列均为NCBI中已登录的序列(序列名称及登录号见图1)。最终用于本试验研究的PVY全基因组序列共33条。
1.3.3 遗传多样性参数评估及群体分化检验
使用DnaSP 5.10软件计算遗传多样性参数:多态性位点数目(segregating sits, S)、单倍型数目(number of haplotypes, h)、单倍型多样性(haplotype diversity, Hd)、核苷酸差异平均数(average number of nucleotide differences, k)、核苷酸多样性(nucleotide diversity, Pi)。使用Arlequin 3.5软件计算群体分化系数(fixation index, Fst)并根据Fst值的大小判断群体间分化程度。当0
1.3.4 PVY基因组的分子变异
以PVY全基因组序列为分子标记,利用Arlequin 3.5软件进行不同群体的分子变异分析(analysis of molecular variance, AMOVA)。使用DnaSP 5.10软件进行变异位点分析。在网站http:∥blast.ncbi.nlm.nih.gov/上进行序列一致性分析。
1.3.5 PVY基因组的重组
以PVYO株系分离物Oz(EF026074)和PVYN株系分离物Mont(AY884983)作为亲本,使用SimPlot 3.5软件进行重组分析。同时在网站(http:∥www.datamonkey.org)使用遗传算法中的重组检测法(genetic algorithm recombination detection,GARD)[23]进行重组位点检测及可靠性评估。
1.3.6 压力分析
使用在线网站(http:∥www.datamonkey.org),基于密码子,通过IFEL(internal branches fixed-effects likelihood)、FEL(fixed effects likelihood)、MEME(mixed effects model of evolution)3种不同方法,对PVY基因组进行压力检测及统计检验。压力选择方向根据非同义核苷酸替换速率(dN)与同义核苷酸替换速率(dS)比值(ω=dN/dS)大小进行判断。当ω>1且群体差异显著时,说明基因组受到正向选择(positive selection)作用;当0<ω<1时,说明基因组受到净化選择(purifying selection)作用;当ω=1时,基因组不受压力选择[24]。
1.3.7 系统发育分析
使用最大似然法(maximum likelihood, ML)基于全基因组序列编码区(coding sequence, CDs)核苷酸序列构建PVY系统发育树。建树前,使用MAFFT软件对建树的33条PVY编码区核苷酸序列进行多重比对,并使用DAMBE软件检测核苷酸替换是否饱和,之后利用MEGA 6.0软件选择最优化的核苷酸替换模型,并参照BIC(Bayesian information criterion)标准设置相应参数,最后通过1 000次自举(bootstrap)抽样来对各分支节点的置信值进行评估。
2 结果与分析
2.1 全基因组序列特征
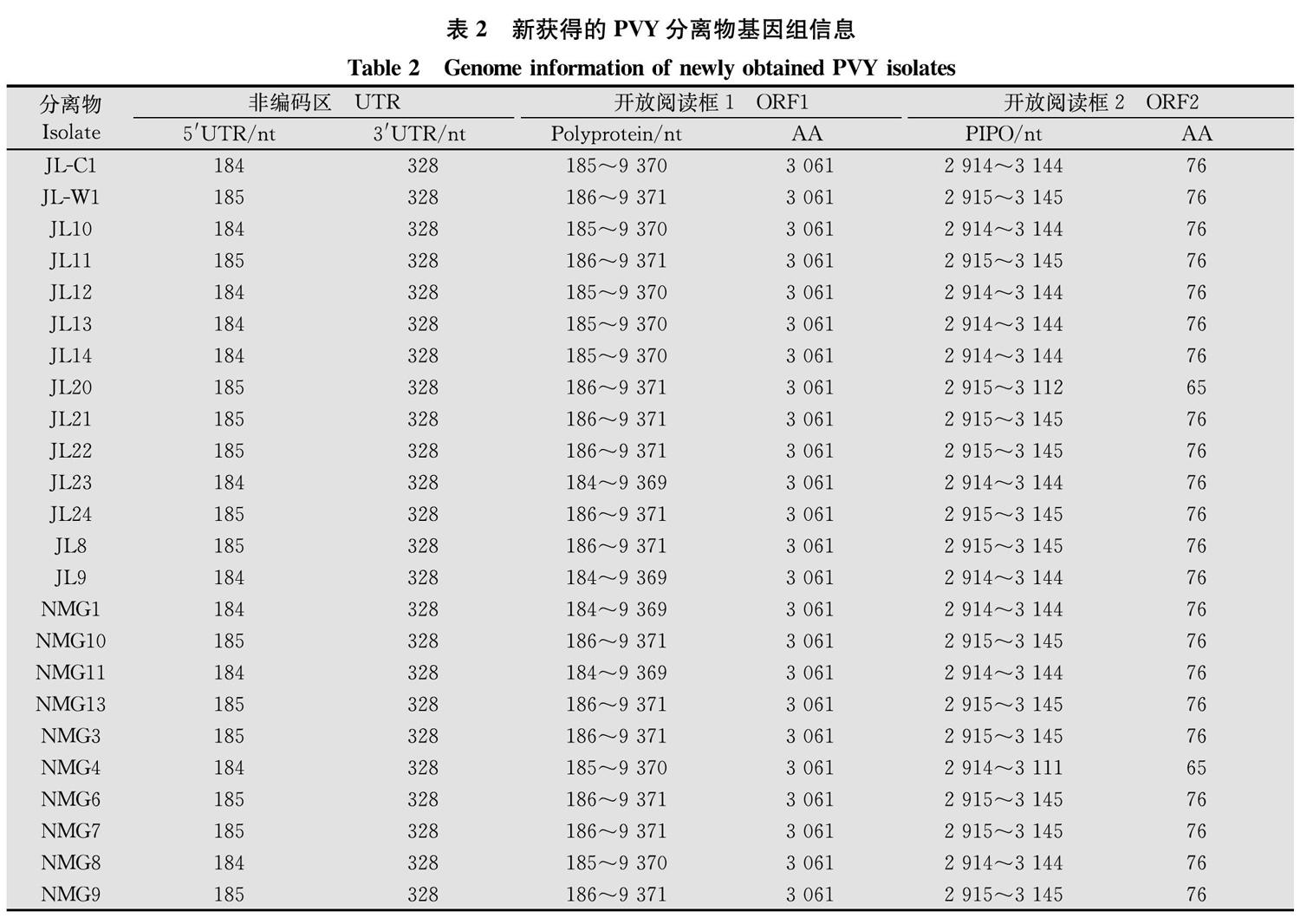
本研究共获得24个PVY分离物全基因组序列(9 698 nt或9 699 nt),5′非编码区长度为184 nt或185 nt,3′非编码区长度为328 nt,编码区长度为9 186 nt;其中在编码区部分,大开放阅读框对应的多聚蛋白编码序列长度为9 186 nt,编码一个包含3 061个氨基酸残基的多聚蛋白;小开放阅读框对应的蛋白PIPO编码序列长度为195 nt或228 nt,并通过移码翻译编码一个包含65或76个氨基酸残基的蛋白PIPO(表2)。
2.2 遗传多样性参数评估及群体分化检验
PVY基因组遗传多样性参数如表3所示:3个群体的多态性位点数目(S)表现出一定差异,其中黑龙江和内蒙古群体S值相对较大,多态性位点数目分别为1 232个和1 229个,吉林群体相对较小,多态性位点数目为807个;核苷酸差异平均数(k)方面,黑龙江和内蒙古群体k值较大,分别为491.361和526.933,吉林群体k值较小为249.659;单倍型多样性(Hd)方面,3个群体Hd值均为1,大于0.5,且33个分离物中共发现33种单倍型(h),未出现两条完全一致的序列,各个群体均表现出较高的单倍型多样性;核苷酸多样性方面(Pi),3个群体的Pi值分别为0.050 61、0.025 72、0.054 29,均大于0.005,表明3个群体均具有较高的核苷酸多样性。以上数据表明我国北方3省(区)PVY群体遗传多样性高,且黑龙江和内蒙古PVY群体遗传多样性高于吉林群体。
以PVY基因组为分子标记,黑龙江和吉林PVY群体之间Fst值为0.051 71,Z统计显著,Kst和Snn值统计不显著,表明在黑龙江和吉林种植区的PVY群体间呈现中度分化但差异不显著;黑龙江和内蒙古,群体之间Fst值为0.072 52,Z、Kst和Snn统计均不显著,表明黑龙江和内蒙古种植区的PVY群体呈现中度分化,但差异不显著;吉林和内蒙古群体之间Fst值为0.233 52,Kst统计显著,Z统计极显著,Snn统计不显著,表明吉林和内蒙古种植区的PVY群体呈现高度分化且差异显著(表4)。
2.3 分子变异分析
全基因组序列一致性比对分析结果显示,本研究获得的24个分离物之间核苷酸一致性为90.3%~99.0%,氨基酸一致性为99.0%~100%,进一步与NCBI中已登录的9个黑龙江PVY分离物比对发现,核苷酸一致性在89.3%~99.5%之间,氨基酸一致性在98.6%~100%之间。综上所述,本次研究3个群体之间核苷酸一致性为89.3%~99.5%,氨基酸一致性为98.6%~100%。
突变位点分析发现,3个群体PVY全基因组多态性位点(polymorphic sites)共1 786个,其中单一变异位点(singleton variable sites)450个,简约信息位点(parsimony informative sites)1 336个,表明PVY基因组存在较高程度的变异。
以PVY基因组为基础,AMOVA分析结果如表5所示,14.46%的变异来自群体间,85.54%的变异来自群体内,因此不同种植区PVY群体之间的遗传变异主要来自各个种植区内PVY个体的遗传变异。
2.4 重组分析
使用Simplot软件分析发现,33个PVY分离物中有30个检测到潜在重组信号,约占分离物总数的90.3%,并且所有检测到的重组分离物均为N×O重组类型。吉林和黑龙江PVY群体分离物均存在重组,而内蒙古PVY群体存在3个O株系非重组分离物。进一步通过GARD程序确认,3个群体存在4个或5个重组位点,每个重组位点都具有较高的模型支持率,且KH检验均达到显著水平(表6)。
2.5 选择压力分析
PVY全基因组选择压力分析显示(表7),使用FEL、IFEL、MEME 3种检测方法均检测到大量选择压力位点,在3个群体中利用FEL和IFEL法分别检测出501个和315个净化选择位点,但仅检测出9个和12个正向选择位点;使用MEME法检测出43个正向选择位点。而以上3种方法共同检测出6个正向选择位点,且这些位点均显著(表8)。以上结果表明尽管PVY基因组受净化选择压力为主,但基因组中也存在个别密码子位点受到强烈的正向选择压力。
2.6 系统发育分析
建树序列多重比对后长度为9 186 nt,根据MEGA 6.0软件的BIC标准,建树序列最合适的核苷酸替换模型为GTR+I+G。ML法构建的系统发育树如图1所示。从图中可以看出3省(区)PVY分离物并未以地区为标准相聚成簇,因此北方3省(区)PVY分离物在系统发育关系上与地理分布无显著联系。根据Chang等[25]的研究结果,以PVY基因组编码区构建系统发育树,相同株系的分离物会相聚成簇,表现出明显的株系特异性,因此,本研究中33个PVY分离物是以不同株系类型为标准相聚成簇,由此可以看出北方3省(区)的PVY群体具有较为明显的株系多样性。
3 讨论
遗传多样性是体现病原物群体对环境适应力的一项重要指标,是突变、基因重组、基因流(基因迁移)、随机漂变和自然选择5种遗传力长期相互作用的结果。遗传多样性直接影响着病原物群体的进化潜力。高遗传多样性的病原物具有更强的生存和进化优势,能更快适应新的寄主和生存环境[26]。开展病原物遗传多样性及群体分化研究有助于了解植物病原物的发生规律,流行机制,并为相关病害的监测及防控提供理论依据。
中国北方3省(区)PVY遗传多样性参数结果表明,黑龙江、吉林和内蒙古3省(区)PVY群体遗传多样性高,与前人研究结果一致,符合中国PVY群体遗传多样性高的特征[27-28]。3省(区)的马铃薯种植区存在一定程度的种薯调运,这会导致不同种植区间PVY群体的二次接触,从而造成高遗传多样性。此外,种群内这种高遗传多样性的形成是一个长期过程,本次研究的3个省(区)PVY群体基因组单倍型多样性(Hd)均大于0.5,核苷酸多样性(Pi)均大于0.005,且两两种植区之间PVY群体呈现一定程度的群体分化,表明3省(区)PVY群体是由一个相对较大且稳定的群体经过长期演化所形成的。
PVY具有高度变异性,而突变在PVY的遗传变异过程中起到重要作用。PVY是RNA病毒,其依赖于RNA的RNA聚合酶(RNA-dependent RNA polymerase, RdRp)缺乏校正功能,会在复制过程中产生大量的点突变[29],在所产生的突变中有利突变会被逐渐积累,而有害突变则在自然选择过程中被逐渐淘汰,由此突变常被视作推动PVY进化的原始动力。本试验基于PVY全基因序列发现了大量变异位点,表明我国北方3省(区)PVY群体存在较高程度的变异。AMOVA分析结果显示3省(区)PVY群体之间的遗传变异主要来自各个种植区内PVY个体的遗传变异。在序列一致性方面,3个PVY群体之间的核苷酸一致性为89.3%~99.5%,符合同一种病毒全基因序列一致性不低于85.0%的标准[11]。
基因重组同样是PVY遗传变异的主要驱动力,在PVY群体中发挥着重要的作用。已经确认的重组位点主要集中在P1、HC-Pro/P3、VPg、NIb和CP基因区域[30-32]。本试验3个PVY群体中均检测出大量的重组分离物,并且3个PVY群体中都存在着多个重组位点,表明相较于基因突变,基因重组可能是PVY进化过程中更为重要的机制,因为重组产生的遗传变异远比仅由突变造成的变异更快、更频繁[33]。常飞等在进行PVY群体遗传分析时并未检测到重组信号[28],原因是选用的P3和PIPO基因位于PVY基因组的非重组区域,因此基于全基因序列的重组分析可以更准确的了解PVY群体的重组状况。此外PVY N×O重组分离物的大量发现也表明此类型重组株系已成为北方3省(区)马铃薯种植区的主流株系。而PVY重组株系的频繁发生可以归纳于以下两点:一是重组分离物相较于非重组分离物有着更强的致病性;二是重组分离物兼具非重组分离物的适应性优势,可以快速适应环境变化,并发展为优势株系[10, 34]。因此,在生产上需密切关注重组株系的流行趋势。
选择压力分析中,中国北方3省(区)PVY群体基因组净化选择位点远高于正向选择位点,表明PVY基因组中多数位点的突变是有害的,在进化过程中这些有害突变会被剔除,从而保证PVY各个基因发挥正常功能。而采用3种方法共同检测到6个显著的正向选择压力位点,表明PVY基因组中存在个别位点的突变是有利于该病毒生存竞争的,在遗传进化过程中这些有利突变会被不断积累。
CP和PIPO基因由于高度保守的特点,在进行Potyvirus病毒系统发育分析时,常被用以构建系统发育树[12],但对于PVY等变异程度高,重组频繁的病毒來说,单基因建树不足以准确地反映同种病毒不同株系分离物之间的系统发育关系,因此,以全基因组构建系统发育树成为解决此类问题的有效方法[25,35]。本研究以我国北方3省(区)PVY分离物全基因序列进行系统发育分析,准确地反映出了PVY分离物的系统发育关系,为我国北方3省(区)PVY群体遗传多样性研究提供了准确数据。
參考文献
[1] 路秉翰, 汤亚飞, 于琳, 等. 基于小RNA深度测序和RT-PCR检测侵染广东省冬种马铃薯的病毒[J]. 植物保护学报, 2019, 46(3): 604-610.
[2] 刘洪义, 张洪祥, 李明福, 等. 黑龙江省马铃薯病毒病的普查及鉴定[J]. 东北农业大学学报, 2006, 37(3): 307-310.
[3] 钟婷婷, 蒲志刚, 何俊蓉, 等. 四川省马铃薯主产区最新病毒病普查及血清学鉴定[J]. 西南农业学报, 2008, 21(1): 96-99.
[4] 胡新喜, 雷艳, 何长征, 等. 湖南省马铃薯主产区马铃薯病毒种类及流行分析[J]. 中国马铃薯, 2012, 26(6): 358-361.
[5] 黄丹, 余琨, 陈建斌, 等. 马铃薯病毒PVY、PVS和PLRV多重RT-PCR检测[J]. 云南农业大学学报, 2015, 30(4): 535-540.
[6] 苑智华. 乌兰察布市马铃薯病毒病调查分析[J]. 江苏农业科学, 2015, 43(11): 189-191.
[7] 王炳森, 王丽, 陈惠兰. 山东省马铃薯病害调查[J]. 中国马铃薯, 2017, 31(2): 104-112.
[8] NOLTE P, WHITWORTH J L, THORNTON M K, et al. Effect of seedborne Potato virus Y on performance of Russet Burbank, Russet Norkotah, and Shepody potato [J]. Plant Disease, 2004, 88(3): 248-252.
[9] RAHMAN M S, AKANDA A K. Performance of seed potato produced from sprout cutting, stem cutting and conventional tuber against PVY and PLRV [J]. Bangladesh Journal of Agricultural Research, 2009, 34(4): 609-622.
[10]CHIKH A M, MAOKA T, NATSUAKI T, et al. PVYNTN-NW, a novel recombinant strain of Potato virus Y predominating in potato fields in Syria [J]. Plant Pathology, 2010, 59(1): 31-41.
[11]KING A M, LEFKOWITZ E, ADAMS M J, et al. Virus taxonomy, ninth report of the international committee on taxonomy of viruses [M]. Amsterdam: Elsevier Academic Press, 2012.
[12]高芳銮, 沈建国, 史凤阳, 等. 马铃薯Y病毒pipo基因的分子变异及结构特征分析[J]. 遗传, 2013, 35(9): 1125-1134.
[13]LORENZEN J H, MEACHAM T, BERGER P H, et al. Whole genome characterization of Potato virus Y isolates collected in the western USA and their comparison to isolates from Europe and Canada [J]. Archives of Virology, 2006, 151(6): 1055-1074.
[14]CHIKH A M, GRAY S M, KARASEV A V. An improved multiplex IC-RT-PCR assay distinguishes nine strains of Potato virus Y [J]. Plant Disease, 2013, 97(10): 1370-1374.
[15]GAO Fangluan, CHANG Fei, SHEN Jianguo, et al. Complete genome analysis of a novel recombinant isolate of Potato virus Y from China [J]. Archives of Virology, 2014, 159(12): 3439-3442.
[16]GLAIS L, FAUREZ F, TRIBODET M, et al. The amino acid 419 in HC-Pro is involved in the ability of PVY isolate N605 to induce necrotic symptoms on potato tubers [J]. Virus Research, 2015, 208: 110-119.
[17]KIM J, CHA D J, KWON M, et al. Potato virus Y (PVY) detection in a single aphid by one-step RT-PCR with boiling technique [J]. Entomological Research, 2016, 46(4): 278-285.
[18]TIAN Yanping, LIU Jinliang, ZHANG Chengling, et al. Genetic diversity of Potato virus Y infecting tobacco crops in China [J]. Phytopathology, 2011, 101(3): 377-387.
[19]GAO Fangluan, ZOU Wenchao, XIE Lianhui, et al. Adaptive evolution and demographic history contribute to the divergent population genetic structure of Potato virus Y between China and Japan [J]. Evolutionary Applications, 2017, 10(4): 379-390.
[20]MAO Yanzhi, SUN Xuhong, SHEN Jianguo, et al. Molecular evolutionary analysis of Potato Virus Y infecting potato based on the VPg gene [J/OL]. Frontiers in Microbiology, 2019, 10: 1708. DOI:10.3389/fmicb.2019.01708.
[21]HUDSON R R, BOOS D D, KAPLAN N L. A statistical test for detecting geographic subdivision [J]. Molecular Biology and Evolution, 1992, 9(1): 138-151.
[22]HUDSON R R, RICHARD R. A new statistic for detecting genetic differentiation [J]. Genetics, 2000, 155(4):2011-2014.
[23]KOSAKOVSKY P S L, POSADA D, GRAVENOR M B, et al. GARD: a genetic algorithm for recombination detection [J]. Bioinformatics, 2006, 22(24): 3096-3098.
[24]NIELSEN R. Molecular signatures of natural selection [J]. Annual Review of Genetics, 2005, 39(1): 197-218.
[25]CHANG Fei, GAO Fangluan, SHEN Jianguo, et al. Complete genome analysis of a PVYN-Wi recombinant isolate from Solanum tuberosum in China [J]. Potato Research, 2015, 58(4): 377-389.
[26]祝雯, 詹家绥. 植物病原物的群体遗传学[J]. 遗传, 2012, 34(2): 157-166.
[27]史凤阳. 中国4个省份马铃薯Y病毒(PVY)P1基因的群体遗传结构[D]. 福州: 福建农林大学, 2014.
[28]常飞. 马铃薯Y病毒pipo基因的遗传结构分析[D]. 福州: 福建农林大学, 2015.
[29]谢联辉, 林奇英. 植物病毒学[M]. 北京: 中国农业出版社, 2011.
[30]LORENZEN J H, PICHE L M, GUDMESTAD N C, et al. A multiplex PCR assay to characterize Potato virus Y isolates and identify strain mixtures [J]. Plant Disease, 2006, 90(7): 935-940.
[31]VISSER J C, BELLSTEDT D U, PIRIE M D. The recent recombinant evolution of a major crop pathogen, Potato virus Y [J/OL]. PLoS ONE, 2012, 7(11): 50631. DOI: 10.1371/journal. pone. 0050631.
[32]QUENOUILLE J, VASSILAKOS N, MOURY B. Potato virus Y: a major crop pathogen that has provided major insights into the evolution of viral pathogenicity [J]. Molecular Plant Pathology, 2013, 14(5): 439-452.
[33]CHAO Lin. Fitness of RNA virus decreased by Mullers ratchet [J]. Nature, 1990, 348(6300): 454-455.
[34]STEINHAUER D A, HOLLAND J J. Rapid evolution of RNA viruses [J]. Annual Review of Microbiology, 1987, 41(1): 409-433.
[35]ADAMS M J, ANTONIW J F, FAUQUET C M. Molecular criteria for genus and species discrimination within the family Potyviridae [J]. Archives of Virology, 2005, 150(3): 459-479.
(責任编辑:田 喆)

