辣椒FAR1基因家族的全基因组分析
夏承东 方大伟


摘要 FAR-RED IMPAIRED RESPONSE 1(FAR1) 基因在植物生长发育中起着重要的调控作用。该研究采用生物信息学的方法,从辣椒基因组中鉴定得到20个CaFAR1转录因子家族成员,系统进化分析表明,20个 CaFAR1基 因分为6个亚家族,其中18个 CaFAR1 基因分布在8条染色体上,8 号染色体上有3个,2号、6号、7号、12号染色体各分布1个,6号和10号染色体上各分布2个,3号染色体上有6个。GO功能注释表明,20个CaFAR1蛋白序列基于氨基酸的相似性被分为三大类:生物过程(BP)、细胞成分(CC)和分子功能(MF)。
关键词 辣椒; FAR 基因家族;生物信息学;全基因组分析
中图分类号 Q943.2 文献标识码 A
文章编号 0517-6611(2020)14-0097-06
doi:10.3969/j.issn.0517-6611.2020.14.027
Abstract FAR-RED IMPAIRED RESPONSE 1 (FAR1) gene plays an important regulatory role in plant growth and development.In this study,bioinformatics was used to identify 20 CaFAR1 transcription factor family members from the pepper genome.Phylogenetic analysis showed that 20 CaFAR1 genes were divided into 6 subfamilies.Of all 20 CaFAR1 genes,18 genes were mapped to the 8 chromosomes.Three genes were mapped to chromosome 3,four genes were mapped to chromosome 2,6,7 and 12 with one for each.Two genes were mapped to the chromosome 6 and 10.There are 6 genes were mapped to the chromosome 3.GO analysis showed that the functions of 20 CaFAR1 genes were enriched in biological process,cell component and molecular function.
Key words Pepper; FAR gene family;Bioinformatics;Whole genome analysis
FAR-RED IMPAIRED RESPONSE 1(FAR1)是转座酶衍生的一类转录因子,是能够在远红光下直接激活FHY1/FHL的基因表达,其在植物淀粉合成代谢和碳饥饿引发的能量匮乏过程中起着重要调控作用[1-2]。目前,已在拟南芥中分离得到了14个FAR1家族成员被鉴定,编码的蛋白长度为531~851个氨基酸[3]。而在棉花中鉴定出88个 FAR1 基因,其中亚洲棉27个、雷蒙氏棉35个、陆地棉26个,编码的蛋白长度为158~1 134個氨基酸[4]。辣椒是我国一种重要的蔬菜作物,光照时间、光照强度以及不同光质对辣椒产量和品质都有很大的影响。然而关于辣椒光敏色素调控相关基因的报道鲜见。该研究利用生物信息学方法,筛选、鉴定辣椒 FAR1 基因家族成员,通过分析其系统进化关系、基因结构,以及染色体定位,探索辣 椒FAR1 基因功能及其应用。
1 材料与方法
1.1 植物序列来源
辣椒 (Capsicum annuum L.)FAR1转录因子家族数据来源于NCBI(https:∥www.ncbi.nlm.nih.gov/genome/?term=Capsicum+annuum)与PlantTFDB(http:∥planttfdb.cbi.pku.edu.cn)等数据库。拟南芥 (Arabidopsis thaliana)与番茄(Solanum lycopersicum) FAR1转录因子氨基酸序列来源于PlantTFDB数据库。
1.2 辣椒FAR1转录因子家族基因的筛选
从TAIR(https:∥www.arabidopsis.org)数据库中下载拟南芥FAR1转录因子序列,利用BlastP筛选出辣椒的FAR1转录因子,得到候选CaFAR1蛋白序列。将这些序列导入CD-HIT Suite(http:∥weizhongli-lab.org/cdhit_suite/cgi-bin/index.cgi)在线网站中去除冗余序列,再经过SMART(http:∥smart.embl-heidelberg.de/opennewwindow)在线预测,得到具有FAR1保守结构域的蛋白序列。参考Lin等[5]的分类方法,将辣椒FAR1蛋白序列归为6类。
1.3 辣椒FAR1转录因子家族理化分析与进化树构建
利用在线ProtParam网站(https:∥web.expasy.org/protparam/)对鉴定出的辣椒FAR1蛋白进行理化性质分析。 使用MEGA 7.0软件内置的 Clustal W程序对鉴定出的FAR1类蛋白序列和拟南芥FAR1家族、番茄FAR1家族蛋白序列进行序列比对,采用邻接(neighbor-joining,N)法构建系统发育树,选用Poisson模型并设置Bootstrap value为1 000,缺失值处理方式为配对删除(pairwise deletion),其他参数使用默认值[5]。
1.4 辣椒FAR1转录因子家族氨基酸序列比对、保守结构域分析 将辣椒FAR1转录因子家族的氨基酸序列上传至DNAMAN8.0中进行比对,得到氨基酸序列比对结果。利用MEME在线软件(http:∥meme suit.org)对辣椒FAR1转录因子保守元件进行预测分析,将motif数量设为10。对辣椒FAR1转录因子保守元件进行图形化显示。
1.5 辣椒FAR1转录因子家族基因结构与染色体定位分析
利用本地软件UltraEdit得到辣椒FAR1转录因子家族基因结构信息与染色体信息,将辣椒FAR1转录因子家族基因结构信息上传至在线软件GSDS(http:∥gsds.cbi.pku.edu.cn/)中得到基因结构示意图。用在线软件MG2C(http:∥mg2c.iask.in/mg2c_v2.0/)对辣椒FAR1转录因子家族的染色体信息进行图形化显示。
1.6 辣椒FAR1转录因子家族基因GO注释分析
使用Blast2GO的非冗余蛋白质数据库软件与默认参数,首先将CaFAR1氨基酸序列与NCBI进行BlastP比对。然后对每一个CaFAR1都获取GO注释,得到GO注释的ID,导出数据之后使用GraphPad作图。
2 结果与分析
2.1 CaFAR1转录因子家族的鉴定及特征 利用本地 Blast 软件对辣椒全基因组蛋白序列进行检索,同时手动剔除重复序列,只保留一个FAR1结构域的蛋白序列。最后得到20个只具有一个FAR1结构域的转录因子家族成员[6],利用ExPASy(https:∥web.expasy.org/cgi-bin/protparam/protparam)在线软件对CaFAR1转录因子家族进行分析,得到CaFAR1转录因子的氨基酸大小、分子量和等电点等信息(表1)。辣椒FAR1转录因子家族成员蛋白序列含有氨基酸数目为220(XP_016567405.1)~877(XP_016580413.1),利用 Expasy 软件预测CaFAR1分子量大小为 25 546.68(XP_016567405.1)~100 799.3 Da (XP_016580413.1),等电点为 5.12(XP_016553207.1)~9.39 (XP_016567405.1),除CaFAR1-14之外,其余CaFAR1蛋白都不稳定。从以上理化性质来分析,辣椒FAR1转录因子家族成员之间的各项指标跨度较大,具有差异性,在筛选出的20个转录因子家族成员中,有13个转录因子的等电点小于7,占 65%,呈弱酸性。
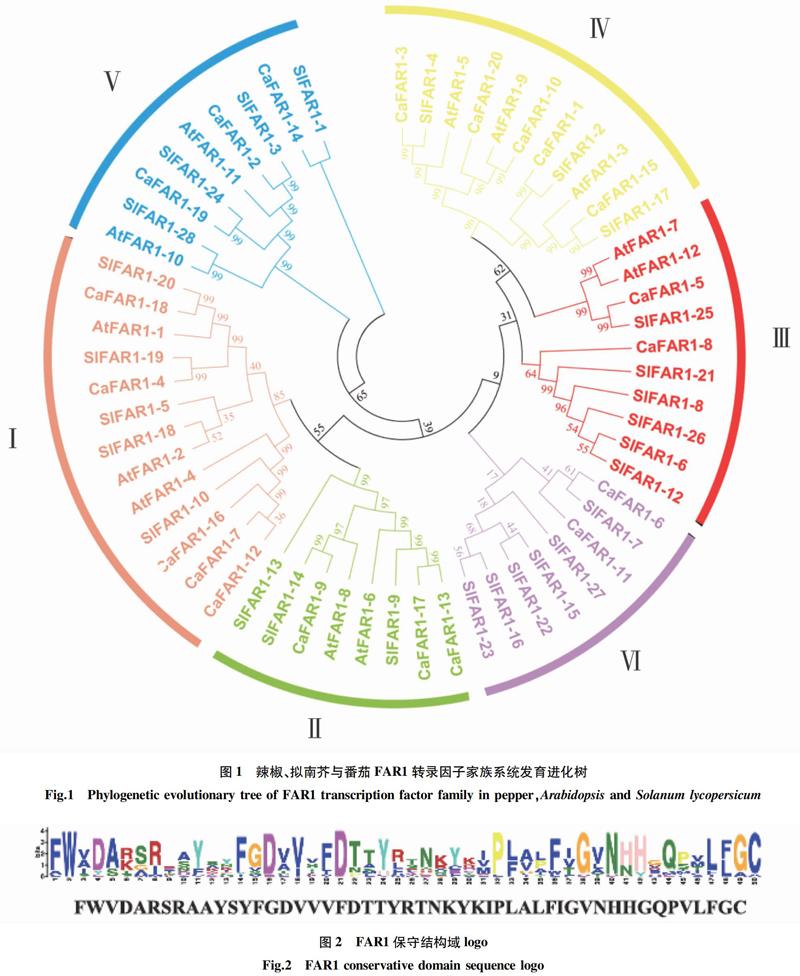
2.2 CaFAR1转录因子家族系统进化树 PlantTFDB v5.0(http:∥planttfdb.cbi.pku.edu.cn)在线软件中下载拟南芥与番茄的FAR1转录因子家族,利用MEGA7系统进化树构建软件构建拟南芥、番茄与辣椒的系统进化树。系统进化树的构建使用最大近似然法,MEGA7参数采用系统默认值,最终得到图1所示的进化树。根据Lin等[5]对拟南芥的分类方法对得到的系统进化树进行亚家族分类,共分为6个亚家族,命名为I~VI。
2.3 氨基酸序列比对
使用DANMAN8.0 序列分析软件对辣椒 FAR1 基因保守结构域进行分析,结果表明,20个蛋白序列均含有FAR1 保守结构域(图2、3)。所有FRS亚组的大多数成员都具有N末端C2H2锌指结构域,N末端FAR1 DNA结合结构域是WRKY-神经胶质细胞缺失(WRKY-GCM1)超家族中的一种C2H2锌指结构域,它与多种靶标的启动子区域中的特定顺式元件结合。
2.4 CaFAR1保守結构域与基因结构分析
将CaFAR1氨基酸序列上传至在线软件MEME(http:∥meme suit.org)对辣椒FAR1序列的保守元件进行预测分析,将motif数量设为10,分析结果如图4B所示。发现,IV亚家族中除CaFAR1-10与CaFAR1-20外均含有10个motif;亚家族Ⅰ中所有CaFAR1都含有10个motif,亚家族Ⅲ中缺少motif 9;亚家族V中均含有motif 3;亚家族VI中均含有motif 1与motif 8。
通过辣椒GFF注释文件中获得CaFAR1的内含子外显子的数据,上传至在线软件GSDS(http:∥gsds.cbi.pku.edu.cn/)对辣椒 FAR1 基因家族的基因结构进行分析。由图4C可知,不同亚组成员的内含子数量存在差异。亚家族I中基因内含子较为相似,内含子数量较多,CaFAR1-16内含子有10个,在所有CaFAR1中数量最多;亚家族Ⅱ的成员含有1~3个内含子;亚家族Ⅲ的CaFAR1-5内含子有1个,CaFAR1-8有2个内含子;亚家族Ⅳ成员含有2~5个内含子;亚家族Ⅴ成员含有2~5个内含子;亚家族Ⅵ成员含有2~6个内含子。
2.5 染色体定位
通过辣椒GFF注释文件中获得 CaFAR1的 位置信息,上传至在线软件mg2c(http:∥mg2c.iask.in/mg2c_v2.0/)中,将18 个 CaFAR1 基因定位到辣椒染色体上,有2 个CaFAR1基 因未被定位到染色体上。 如图5所示,辣椒的18个 CaFAR1 基因分布在8条染色体上,8 号染色体上有3个,2号、6号、7号、12号染色体各分布1个,6号和10号染色体上各分布2个,3号染色体上有6个。
2.6 GO分析 使用Blast2GO的非冗余蛋白质数据库软件与默认参数,首先将CaFAR1氨基酸序列与NCBI进行BlastP比对。然后对每一个CaFAR1都获取GO注释,得到GO注释的ID,导出数据之后使用GraphPad作图(图6)。可以发现,20个CaFAR1蛋白序列基于氨基酸的相似性被分为三大类:生物过程(BP)、细胞成分(CC)和分子功能(MF),大部分CaFAR1与离子结合相关,显然与CaFAR1的锌指结构域功能吻合。CaFAR1在细胞成分(CC)和分子功能(MF)这两方面发挥了很大的作用,由此可以推断CaFAR1在辣椒的生长发育与信号传递中发挥了很大的作用。
3 讨论与小结
光是植物生长和发育的能量来源,光敏色素(phytochromes)是高等植物光受体网络中不可或缺的一个,其通过感受红光和远红光,直接或间接调控下游的转录因子,从而控制下游基因表达,影响植物的种子萌发、幼苗去黄化、叶绿体运动等生长发育进程[4,7-10]。FAR1是phyA信号通路中重要的正向调控因子之一。已在拟南芥、棉花中分离得到了多个FAR1家族成员[3-4]。该研究通过对辣椒全基因组扫描,从中鉴定得到了20个CaFAR1转录因子家族成员,结合基因结构分析与系统进化分析,20个 CaFAR1基 因分为6个亚家族。其中,18个 CaFAR1基 因分布在8条染色体上。而对其功能注释表明,20个CaFAR1蛋白序列基于氨基酸的相似性被分为三大类:生物过程(BP)、细胞成分(CC)和分子功能(MF)。
参考文献
[1]陈芳,邓兴旺.远红光受体伴侣蛋白FHY1在介导基因表达和植物生长发育过程中的独特作用[J].遗传,2014,36(9):958.
[2]CASAL J J.Phytochromes,cryptochromes,phototropin:Photoreceptor interactions in plants[J].Photochemistry & photobiology,2000,71(1):1-11.
[3]LIN R C,WANG H Y.Arabidopsis FHY3/FAR1 gene family and distinct roles of its members in light control of Arabidopsis development[J].Plant physiology,2004,136(4):4010-4022.
[4]袁娜,王彤,劉廷利,等.棉花FAR1/FHY3基因家族的全基因组分析[J].棉花学报,2018,30(1):1-11.
[5]LIN M ,GANG L.FAR1-RELATED SEQUENCE (FRS) and FRS-RELATED FACTOR (FRF) family proteins in arabidopsis growth and development[J].Frontiers in plant science,2018, 9:692.
[6]惠甜,沈兵琪,王连春,等.桑树bHLH转录因子家族全基因组鉴定与分析[J].分子植物育种,2019,17(17):5624-5637.
[7]HEIJDE M,ULM R.UV-B photoreceptor-mediated signalling in plants[J].Trends in plant science,2012,17(4):230-237.
[8]刘圈炜,何云,齐胜利,等.光敏色素研究进展[J].中国农学通报,2005,21(5):237-241.
[9]FURUYA M.Phytochromes:Their molecular species,gene families,and functions[J].Annual review of plant physiology and plant molecular biology,1993,44(1):617-645.
[10]TEPPERMAN J M,ZHU T,CHANG H S,et al.Multiple transcription-factor genes are early targets of phytochrome A signaling[J].Proceedings of the national academy of sciences of the United States of America,2001,98(16):9437-9442.

