环境DNA技术在生态保护和监测中的应用
张 辉 , 线薇薇
(1. 中国科学院海洋研究所 中国科学院海洋生态与环境科学重点实验室, 山东 青岛 266071; 2. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室, 山东 青岛 266237; 3. 中国科学院海洋大科学研究中心, 山东 青岛 266071)
生物多样性是全人类最终所依赖的所有尺度上生态系统服务的最初支撑者, 也是人类生存和发展的基础, 它与“全球变迁”和“持续发展”同被列为当代生态学和环境科学的三大前瞻性领域[1]。近一个世纪以来, 随着人类活动的不断加剧, 生物多样性和生态系统遭到了严重的破坏, 物种灭绝的速度不断加快, 人类赖以生存的生态系统中 60%已处于持续退化状态, 自然资源的三分之二已被损耗, 生物多样性减少、生态系统功能退化、全球气候变化等早已成为全球性的重大问题[2]。
基于此背景, 在避免对当前生态环境再破坏的前提下, 开展全面准确的生态监测来查清和明确生物多样性, 对生态保护和可持续利用具有极其重要的意义。环境DNA技术(eDNA)的发展就提供了一种生态和生物多样性监测监测的新手段。该技术样品采集简单易行, 是一种便捷地、环境友好型地大范围生态监测技术。
eDNA, 是指环境中存在的 DNA, 在大型生物研究中主要指生物脱落在环境中的组织或者细胞内含有的 DNA, 例如皮毛、黏膜、粪便等[3]。通过采集 eDNA样品, 基于测序技术(现在通常使用第二代测序技术, 即高通量测序)监测调查区域的生物种类,称为环境DNA宏条形码技术(图1)。

图1 环境DNA宏条形码技术——以海洋鱼类为例Fig. 1 Workflow of eDNA metabarcoding in the research on marine fishes
1 环境DNA技术的发展
eDNA技术在水生生态系统中应用的灵感, 来自于 20世纪 90年代利用潜水采集到的粪便来识别座头鲸(Megaptera novaeangliae), 抹香鲸(Physeter macrocephalus)及露脊鲸(Eubalaena glacialis)等的相关研究中[3]。而世界上首例证明eDNA适用于水生生态系统的研究是2008年Ficetola 等对美国牛蛙入侵的报道[4]。该研究表明即使目标水域内的牛蛙数量很少(例如每平方公里水域仅有1~2只牛蛙), 亦可通过eDNA将该物种检测出来。
此后, 一些对不同水环境、不同生物门类的研究均验证了eDNA在物种检测中的有效性和灵敏性。例如, 美国水产生物保护中心进行的一项大型研究开发了在大湖系统的运河中检测白鲢(Hypophthalmichthys molitrix)和鳙(H.nobilis)等物种的 eDNA 技术[5]。Goldberg等通过测试多种DNA提取和PCR方法完善了eDNA技术, 以检测源头水流中的蝾螈(Dicamptodon aterrimus)和尾蟾落基山亚种(Ascaphus montanus)[6]。Thomsen等[7]表明基于eDNA技术对于甲壳动物、水生昆虫、水生哺乳动物以及鸟类和陆生哺乳动物等的监测是有效的。2012年以来, 利用eDNA检测脊椎动物的论文大量增加, 其中包括综述论文, 如“环境DNA生态学研究及其在保护遗传学中的应用”[8], “基于eDNA的野生生物及生物多样性监测”[9]和“基于eDNA对水生动物的监测——生态学研究中 eDNA作为调查工具的综述”[10]以及生物保护集刊《环境DNA: 生物保护的高效新工具》中的12篇研究论文对eDNA在生物保护学领域的应用进行了报道等等[11]。这些研究为 eDNA技术在水生生态系统, 尤其是海洋生态系统监测中的应用奠定了必要的基础(图2)。
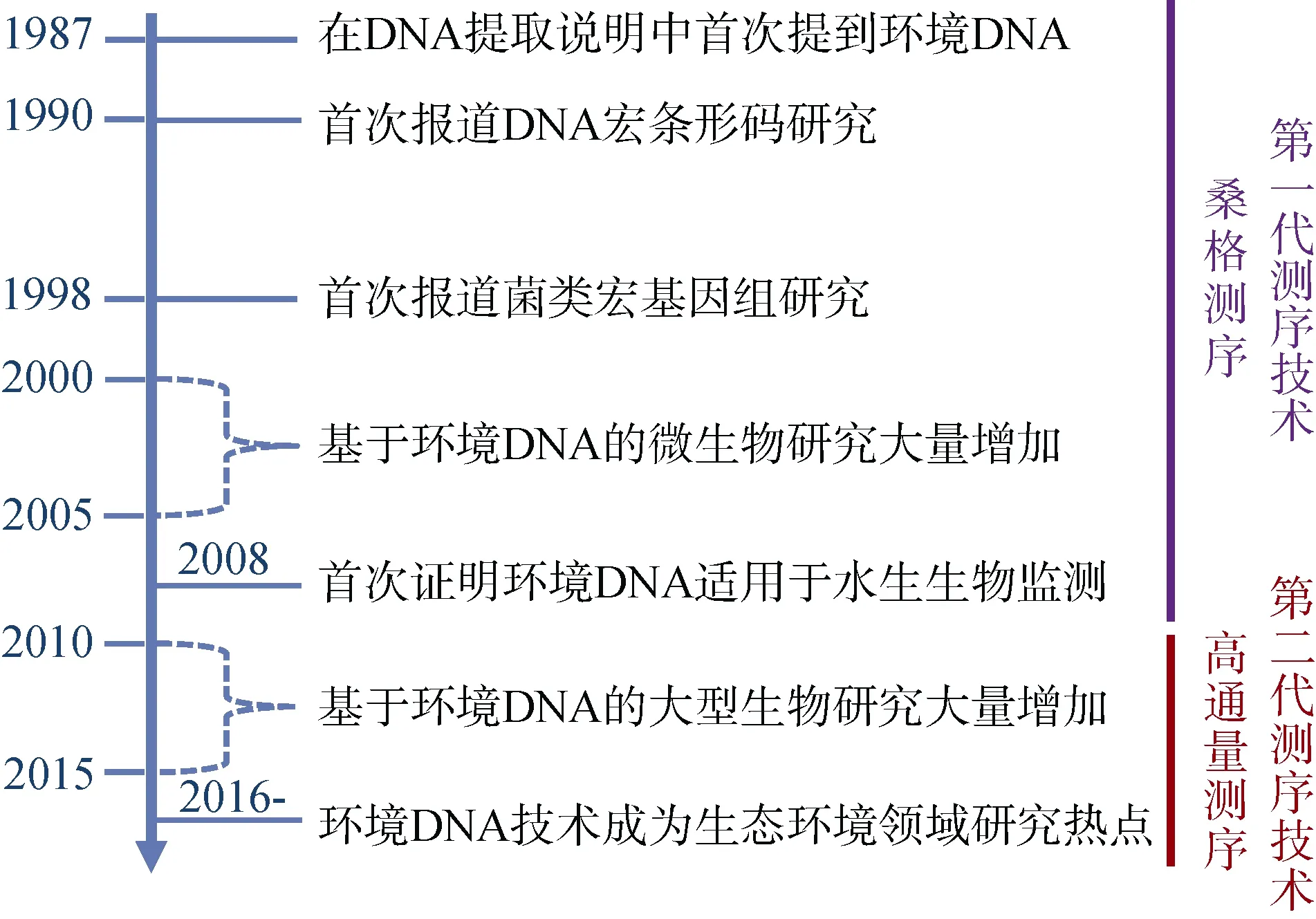
图 2 环境DNA研究发展概略图Fig. 2 Overview of the research history of eDNA
2 国际上环境DNA技术在生态监测中的应用
国际上相关研究验证了 eDNA技术在生态监测中的有效性和灵敏性。Bálint 等[12]综述了最近的研究成果, 强调 eDNA为生态研究提供了强有力的新时态数据来源, 并讨论了基于 eDNA技术的潜在研究方向, 概述了相关挑战。《2018研究前沿》[13]显示在生态与环境科学领域, eDNA技术监测生物多样性连续第2年入选热点研究前沿(图3)。本研究主要对eDNA技术在生态监测中的几个主要方向进行介绍。

图3 生态与环境科学领域Top10 热点前沿的施引论文Fig. 3 Top 10 research topics in the fields of ecology and environment science注: 改编自《2018研究前沿》[13]
2.1 环境 DNA技术与传统监测方法的互补性
野生生物资源调查通常使用网具进行, 这在监测高丰度物种时较为可靠, 而对低丰度、珍惜、濒危物种的捕获概率较低, 随着野生生物资源不断减少,传统资源调查得到的结果会存在较大误差[14]。研究表明, eDNA方法比传统的调查方法更为灵敏、有效。Dejean等[15]在外来物种入侵研究中, 以两种美国牛蛙为研究对象, 将传统调查方法与 eDNA技术进行了比较, 结果表明 eDNA技术更加便捷有效。Davy等[16]利用 eDNA方法对 8种淡水龟进行了监测, 结果表明 eDNA方法在龟类资源调查和监测中具有适用性, 可以推广至濒危龟类物种的监测等领域。Sigsgaard等[17]在丹麦水域同时使用传统调查方法和eDNA技术对泥鳅进行了监测, 结果表明eDNA方法结果可靠, 且所需成本较小。Smart等[18]认为eDNA方法比传统方法在水生生物分类领域更高效, 并就优化eDNA的成本进行了探索。Evans等[19]以美洲红点鲑(Salvelinus fontinalis)为研究对象比较了电击法和 eDNA方法的优劣, 表明 eDNA方法更加省时省力, 约能节省67%的成本, 因此eDNA可以成为电击法调查的有力补充。
2.2 基于环境 DNA技术监测物种生活史过程
由于 eDNA技术对目标物种的干扰和生态系统的破坏性较小, 因此可用于监测不同门类的物种,如日本大鲵(Andrias japonicus)[20]、泥鳅(Misgurnus fossilis)[21]、阿拉巴马鲟(Scaphirhynchus suttkusi)[22]等。相关研究进一步表明, 通过监测eDNA浓度的变化, 可以实现对目标物种在特定区域的产卵场、索饵场及迁移路径等生活史过程的监测和预测, 显著提高了生态监测的效率, 同时降低了大量人工成本和科研成本。Spear等[23]利用 eDNA对隐鳃鲵(Cryptobranchus alleganiensis alleganiensis)进行资源调查发现在隐鳃鲵的繁殖期, 其 eDNA浓度处于最高值。Erickson等[24]基于 eDNA方法研究了鳙鱼入侵的迁移路径、产卵场位置, 发现了eDNA浓度与其迁移路径的相关性。Buxton等[25]研究发现在疣螈(Triturus cristatus)繁殖期(6月开始)及稚鱼期(8月中旬开始)水域中的eDNA浓度最高。Bylemans等[26]证明eDNA浓度变化可以作为监测濒危物种澳洲麦氏鲈(Macquaria australasica)产卵行为的重要手段, 并有望在其他门类物种产卵过程中推广使用。
2.3 基于环境DNA技术推算物种丰度
研究表明, 基于 eDNA浓度与物种生物量之间的正相关关系可以确定物种的丰度和区系分布。Pilliod等[27]在美国爱达荷州的13条河流中利用传统野外调查方法和 eDNA方法采集了样品进行比较研究, 结果表明 eDNA浓度与野外调查得到的生物密度、生物量等结果呈显著正相关关系。Evans等[28]测定了9种动物(包括8种淡水鱼类和1种两栖动物)的 6个线粒体基因片段序列, 发现序列拷贝数与上述9种生物的丰度存在正相关关系, 提示eDNA具有生物丰度评价指标的潜力。Lacoursiere-Roussel等[29]认为关于 eDNA浓度与物种丰度之间关系的大量例证使得eDNA在渔业评估管理中具有广泛应用前景。Doi等[30]基于日本佐波河水域浮潜调查研究, 证明了香鱼(Plecoglossus altivelis)eDNA浓度与其丰度及生物量的关系, 结果表明 eDNA分析是估算鱼类丰度/生物量以及它们在河流中的空间分布的有效工具。Salter等[31]比较了基于eDNA技术与传统拖网调查结果, 发现大西洋鳕鱼(Gadus morhua)的捕获量与定量PCR结果呈显著正相关关系, 提出eDNA技术可以用于重要海洋经济鱼类的区域性资源量评估。
2.4 基于环境DNA技术研究生态系统结构变化
生态系统结构变化是生态监测的重要方面, 通过 eDNA分析, 可以同时监测生物在多个营养级和群落中的动态变化, 从而提供与生态系统变化相关的生物间作用的关键信息。Djurhuus等[32]利用eDNA高通量测序技术, 基于加利福尼亚蒙特雷湾18个月(2015—2016年)的海水样品, 研究了该生态系统中的生物多样性, 推断了群落结构变化及其与环境的关系, 提供了预期的捕食者与被捕食者之间的关系、营养级关系及季节性变化的证据, 认为基于 eDNA分析可以掌握海洋生态系统变化并为保护敏感生物提供依据。Holman等[33]基于eDNA调查了英国沿海4个码头的沉积物和水样, 并与历史结果进行了比较。结果发现了含多个新引进种在内的多个非本地种, 凸显了 eDNA宏条形码技术在非本地种早期检测和时空监测中的实用性, 认为在是评估群落结构变化相关研究时需选择不同类型的环境样本。Sigsgaard等[34]在丹麦沿海进行了为期一年的水样采集和浮潜观察, 基于eDNA技术分析了水样品, 并与已有的 7年历史数据进行了比较。结果证明 eDNA鉴定的OTUs随鱼类群落结构的季节性变化而变化。尽管浮潜观察结果与 eDNA结果存在差异, 但是通过浮潜观察到的的绝大多数鱼类都可以在 eDNA结果中获得, 该研究证明了 eDNA在重建海水鱼类群落结构季节变化中至关重要。
3 我国环境DNA技术在生态监测中的应用
国内学者近年来亦开展了 eDNA相关研究, 但总体仍处于起步状态, 且大部分聚焦于淡水生态系统, 对海洋生态系统的研究报道较少。
徐浩等[35]、陈炼等[36]、赵明等[37]通过综述介绍了环境DNA技术在生物多样性和生态学研究领域中的应用。姜维等[38]以川陕哲罗鲑为目标物种, 对环境DNA 分析流程进行了设计优化; 刘军等[39]对鱼类环境DNA 研究中的通用引物进行了筛选验证。徐念和常剑波[40]基于长江中下游干流环境 DNA样本, 开展了鱼类物种检测的相关工作, 该研究从来源于 17 个采样点的115条匹配成功的序列中检测出了15 种鱼类。孙晶莹等[41]研究表明 eDNA 宏条形码技术可实现对浮游动物物种的半定量检测, 在生物多样性监测和生物完整性评价有显著的应用价值。李苗等[42]建立了一套中国对虾 eDNA 技术的操作流程, 提高了中国对虾的检出率。Zhang等[43]基于eDNA技术研究了长江口及其邻近水域鱼类群落结构的季节变化特征。结果显示: eDNA技术适用于长江口及其邻近水域的资源监测, 与传统方法相比更加灵敏和有效。张辉和线薇薇开发了一种收集海水中环境DNA的装置[44]。陈治等[45]以曼氏无针乌贼(Sepiella japonica)为研究对象, 通过绝对定量技术建立和优化了舟山近海高浊度水样 eDNA的获取方法, 建立了舟山近海水样大生物 eDNA最适获取方法, 为相似水域的水样采集及eDNA提取提供了借鉴参考。
4 结论与展望
eDNA技术的优势使其能够作为传统资源调查的有力补充, 在生态保护和监测中能够发挥重要作用, 已在美国、日本等发达国家广泛应用。我国的相关研究, 尤其是在海洋领域的研究十分匮乏, 亦应当稳步开展、不断加强, 充分发挥eDNA技术的优势服务于生态保护。
需要指出的是, 任何技术的发展都会存在一系列挑战。在eDNA技术研究中, 亦存在一些问题。例如, 在海洋监测中, 海水样品高质量保存与提取难度大; 野外现场水样品容易被污染; 不同门类选择的通用引物尚没有统一认识; 在鉴定OUTs时往往依赖于相关数据库中的已有序列, 而当数据库中不存在该物种的相关信息或者信息存在错误时, 物种鉴定结果就不准确; 虽然有大量研究尝试了基于qPCR产物浓度或者 PCR序列数量来推测生物量, 但基于eDNA技术计算生物量不仅流程复杂, 而且其结果与实际情况通常存在较大差距等等, 这些都是亟需科学工作者努力解决的问题。

