基于广西普通野生稻染色体片段代换系的落粒性QTL鉴定及相关主效QTL定位
袁睿智 黄泽键 罗亮 赵能 陈媛 梁燕青 万瑶 刘芳 李容柏


摘要:【目的】挖掘廣西普通野生稻(Oryza rufipogon Griff.)蕴藏的落粒性基因,为广西普通野生稻进化及栽培稻起源等研究提供参考依据。【方法】通过杂交、回交和分子标记辅助选择(MAS)等方法构建广西普通野生稻DP30和DP15的染色体单片段代换系(CSSLs)群体(DP30-CSSLs和DP15-CSSLs),对CSSLs群体进行落粒性鉴定和数量性状位点(QTL)分析;并以筛选出的强落粒性代换系Y99(CSSL-Y99)与受体亲本93-11杂交构建次级F2群体,对落粒性相关QTL进行定位。【结果】构建出由144个CSSLs组成的DP30-CSSLs群体和由59个CSSLs组成的DP15-CSSLs群体。DP30-CSSLs群体代换片段累计全长737.5 Mb,对DP30全基因组的覆盖率约为94.71%;DP15-CSSLs群体代换片段累计全长337.36 Mb,对DP15全基因组的覆盖率约为73.11%。从2个广西普通野生稻CSSLs群体中鉴定出12个落粒性CSSLs(CSSL-Y104、CSSL-Y68、CSSL-Y83、CSSL-Y328、CSSL-Y235、CSSL-Y64、CSSL-Y63-2、CSSL-Y303、CSSL-Y99、CSSL-Y106-3、CSSL-Z37和CSSL-Z38),检测出6个落粒性QTLs(qSH2.1、qSH4.1、qSH5.1、qSH9.1、qSH11.1和qSH11.2)。其中,qSH11.1的加性效应(-37.5)和表型贡献率(-23.4%)最高。利用在qSH11.1所在区间内开发的6对InDel分子标记(M1、M2、M3、M4、M5和M6)对从次级F2群体进行基因型分析,筛选出4个重组单株(43、167、128和136),并将落粒性主效QTL qSH11.1定位于第11染色体M5~M6间约1.5 Mb的范围内。【结论】从构建的广西普通野生稻核心种质资源(DP30和DP15)CSSLs群体中检测出6个落粒性QTLs,主效QTL qSH11.1定位于第11染色体M5~M6间约1.5 Mb的范围内,是新发现的落粒性QTL。
关键词: 普通野生稻;落粒性;染色体片段代换系(CSSLs);数量性状位点(QTL);广西
中图分类号: S511.9 文献标志码: A 文章编号:2095-1191(2020)05-1004-09
Abstract:【Objective】Based on exploitation of the shattering genes from Guangxi common wild rice(Oryza rufipogon Griff.), in order to provide a reference for the study of the evolution of Guangxi common wild rice and the origin of cultivated rice (O. sativa L.). 【Method】DP30-CSSLs and DP15-CSSLs, two chromosome segment substitution lines (CSSLs) populations of Guangxi common wild rice were constructed respectively by hybridization, backcrossing and molecular marker-assisted selection(MAS), which were used for shattering identification and quantitative trait loci(QTL) analysis. The secondary F2 population was constructed by crossing the strong shattering substitution line Y99 (CSSL-Y99) with the recipient parent 93-11 to map the shattering-related QTL. 【Result】DP30-CSSLs population consisted of 144 DP30-CSSLs and DP15-CSSLs population consisted of 59 DP15-CSSLs were obtained. The total length of DP30-CSSLs population was 737.5 Mb, covering 94.71% of DP30 genome; while the total length of DP15-CSSLs population was 337.36 Mb, covering 73.11%. of DP15 genome. Twelve CSSLs(CSSL-Y104,CSSL-Y68,CSSL-Y83,CSSL-Y328, CSSL-Y235,CSSL-Y64,CSSL-Y63-2,CSSL-Y303,CSSL-Y99,CSSL-Y106-3,CSSL-Z37 and CSSL-Z38) showed shattering trait and six grain-shattering QTLs(qSH2.1, qSH4.1, qSH5.1, qSH9.1, qSH11.1 and qSH11.2) were identified from the two Guangxi common wild rice CSSLs populations. Among them, qSH11.1 had the highest additive effect(-37.5) and phenotypic contribution rate(-23.4%). Six pairs of InDel molecular markers(M1, M2, M3, M4, M5 and M6) were developed in the interval of qSH11.1 to analyze the genotypes from the secondary F2 population, and four recombinant plants(43,167,128 and 136) were selected. A major grain-shattering QTL qSH11.1 from CSSL Y99 was mapped to the approximately 1.5 Mb region between M5-M6 on chromosome 11. 【Conclusion】Six shattering QTLs are detected from DP30-CSSLs and DP15-CSSLs. Among them, the major QTL qSH11.1 is located in the range of about 1.5 Mb between the M5-M6 on chromosome 11, which is a newly discovered shattering QTL.
Key words:common wild rice(Oryza rufipogon Griff.); grain shattering; chromosome segment substitution lines (CSSLs); quantitative trait loci (QTL); Guangxi
Foundation item: Guangxi Innovation Driven Development Special Project (Guike AA17204070)
0 引言
【研究意義】普通野生稻(Oryza rufipogon Griff.)是水稻育种的种质资源宝库,具有许多与栽培稻(O. sativa L.)存在显著差异的优良性状(Huang et al.,2012;Stein et al.,2018)。栽培稻在长期的人工驯化过程中,丢失了许多优良性状且遗传基础变窄(Doebley et al.,2006;江川等,2018),优良数量性状位点(QTL)的丢失极大限制了栽培稻产量和品质的进一步提高(Wu et al.,2020)。因此,加强普通野生稻农艺及抗逆等优良性状的研究与利用,对揭示水稻的驯化起源及提高栽培稻的产量和品质均具有重要意义。【前人研究进展】经人工驯化后,许多不同物种间的相同性状被固定下来,如分蘖角度、株型及种子落粒性变化等(Yu et al.,2008;Zhang et al.,2009;Wu et al.,2013)。落粒性对水稻种子传播有积极作用,但不利于人们收获。相对于野生稻而言,栽培稻种子成熟时仍保留在穗部(Zhang et al.,2009)。因此,不落粒性作为水稻驯化过程中一个人为选择的重要性状,有效保证了水稻的收获产量,而开展落粒性研究对了解水稻起源演化具有重要意义(Zhao et al.,2015)。水稻落粒性是一个复杂的生物学性状,其中离层(Abscission zone,AZ)形成和断裂是控制落粒的主要原因(Zhou et al.,2012)。AZ是小枝梗与种子间的连接部位,由一层或数层形态相似的致密细胞组成(Yan et al.,2015)。在前人的相关研究中,已有多个控制水稻落粒性的QTLs被检测出来,并克隆获得SH1、SHAT1和GL4等落粒性基因(Lin et al.,2007;Zhang et al.,2009;Huang et al.,2012;Zhou et al.,2012;Wu et al.,2017)。其中,SH1基因位于第1染色体上,cDNA序列全长2450 bp,包含4个外显子,编码612个氨基酸残基。qSH1基因开放阅读框12 kb的5'端调节区存在一个单核苷酸多态性(SNP)位点,在Kasalath中为G,在日本晴中则为T(Zhang et al.,2009)。qSH1基因编码一个BEL1类型的同源异型蛋白,主要影响水稻AZ的发育。qSH1基因在花器官分化期及花序轴和枝梗快速伸长期的花序原基表达,还在花药和小穗基部将要形成脱离层的区域表达;花序轴形成期在不落粒品种日本晴的花序原基也有表达,但在花器官分化期及花序轴和枝梗快速伸长期的小穗基部将要形成脱离层的区域并未表达(Huang et al.,2012)。SH4基因是一个影响水稻落粒性的主效QTL,且编码一个未知功能的转录因子,而影响水稻AZ形成(Xiong et al.,1999)。Lin等(2007)从栽培稻克隆获得的SHAT1基因与SH4基因是等位基因,二者的不同之处在于SHAT1基因并不影响AZ形成。SHAT1基因编码的APETALA2转录因子是落粒性形成的必需条件,并参与AZ发育(Lin et al.,2007)。SHAT1基因在亚洲栽培稻中广泛存在,对其进行研究有助于探索水稻的籼粳分化机理(Zhou et al.,2012)。在水稻的驯化过程中,SH4非落粒等位基因被人为选择保留下来(Zhang et al.,2009)。除了SH1和SHAT1基因外,GL4基因通过调控外颖和内颖纵向细胞的伸长,而控制非洲栽培稻的粒长,同时能调控种子落粒性,主要在幼穗中表达,其次是茎部,在叶片和根部不表达;GL4基因发生单核苷酸突变时提前产生终止密码子,致使种子粒长宽变小、落粒性丧失,进而影响水稻产量(Wu et al.,2017)。【本研究切入点】染色体单片段代换系(CSSLs)是基因挖掘的理想群体,可从普通野生稻CSSLs群体中挖掘出更多新的落粒性基因。广西野生稻遗传多样性丰富,但至今鲜见有关广西普通野生稻落粒性基因挖掘的研究报道。【拟解决的关键问题】通过构建CSSLs群体发掘广西普通野生稻中与落粒性相关QTL,为落粒性基因精细定位和克隆打下基础,同时为广西普通野生稻进化及栽培稻起源等研究提供参考依据。
1 材料与方法
1. 1 试验材料
试验材料包括普通野生稻DP30和DP15及栽培稻品种93-11。其中,DP30和DP15是广西普通野生稻核心种质,由亚热带农业生物资源保护与利用国家重点实验室提供,易落粒;93-11为测序品种,是优良栽培籼稻中的常用恢复系,不易落粒。
1. 2 普通野生稻CSSLs群体构建
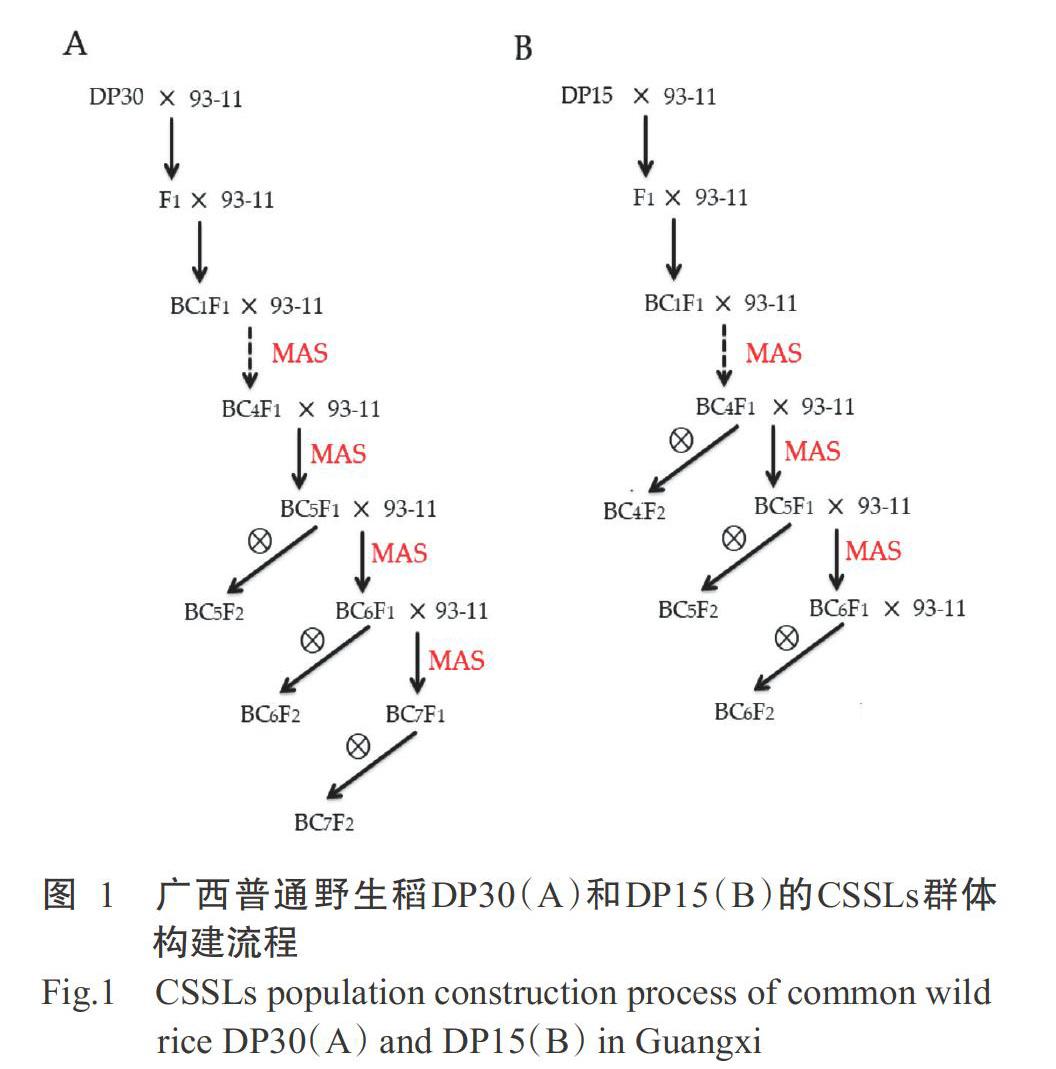
参考T?rjék等(2008)的研究方法,通过杂交、回交和分子标记辅助选择(MAS)等方法构建广西普通野生稻DP30和DP15的CSSLs群体(DP30-CSSLs和DP15-CSSLs)。93-11与DP30和DP15分别杂交获得F1。93-11作轮回亲本,与杂交后代连续回交,从BC1F1开始每个世代均通过MAS跟踪目标DNA片段,选择含有目标DNA片段植株再继续回交;直至获得高世代回交群体BC4F2、BC5F2、BC6F2和BC7F2,从BC4F1开始通过MAS检测目标片段植株的遗传背景,筛选出携带少数几个或1个供体亲本片段的CSSLs(图1)。由此构建获得DP30-CSSLs群体和DP15-CSSLs群体。
1. 3 落粒性表型调查
在2018年晚稻(7—11月)栽培条件下,采用随机区组设计(RCBD)每3 d在广西大学试验田(东经108°13′,北纬22°38′)记录1次表型数据。具体方法:所有表型数据记录重复3次,取平均值,每次重复选取15株种植材料进行表型调查。参考Zhou等(2012)关于落粒/非落粒的判定方法,即种子完熟后,自然条件下每穗脱落种子数量占每穗种子总数的百分比,80%以上记为落粒,10%以下为不落粒。参考吕树伟(2018)的方法判断落粒性强弱,即种子开花至种子完熟间各阶段采用永红牌拉力仪测量拉力值。具体步骤:夹子挂在拉力仪挂钩上,用夹子固定颖壳中部,在水平方向上对其进行拉拽,使种子与小枝梗分离,记录测得的最大拉力值,重复3次,记录平均值。表型值(TV)选取拉力值大幅变化时,即第6 d的拉力值(Zhao et al.,2019)。
1. 4 基因组测序及分子标记开发
采用Illumina HiSeq 2500?提取试剂盒制备DP30和DP15基因组样品,并委托北京诺禾致源生物信息科技有限公司进行测序。样品制备和测序均遵循标准的Illumine方案,参考已公布的93-11基因组序列(Kechin et al.,2017)。使用FastQC和Adapt进行测序结果质量检测(Pared质量评分),参数为-O 5和-m 32(Zhao et al.,2016);以Burrow-Wheeler Aligner(BWA)进行基因组比对分析(McKenna et al.,2010)。通过GATK检测1~50 bp的插入或缺失情况,并提取测序深度高(DP,≥50倍)的InDel区大片段(≥2 bp)设计InDel分子标记,所得候选分子标记经数据库(https://www.ncbi.nlm.nih.gov/)搜索,筛选出最终的分子标记(Wu et al.,2017)。
1. 5 基因组DNA提取及PCR
采用CTAB法提取基因组DNA,PCR扩增产物经7%聚丙烯酰胺变性凝胶分离后,用银染法显色(Zhou et al.,2012);参照Yoon等(2015)的方法确认PCR扩增產物的电泳条带和基因分型。
1. 6 QTL定位与数据分析
以Graphical Geno-Types 2(GGT 2.0)绘制CSSLs群体基因型图谱(van Berloo,2008)。参考Brondani等(2002)的方法分析CSSLs群体中普通野生稻DNA代换片段长度。采用重叠群QTL分析法检测CSSLs群体的QTL,具体方法:若具有重叠代换片段的几个CSSLs表型相同,相关QTL可能位于其重叠代换片段上(Furuta et al.,2014)。利用QTL IciMapping 4.1.0的复合区间作图法(CIM)进行QTL定位,根据排列(组合)进行检验,排列=1000,P=0.001,若LOD值≥2.5,则在P≤0.001水平上认定一个可能的QTL。参考Zhao等(2019)的方法判定QTL加性效应(A)和表型贡献率(AP),具体计算公式如下:
2 结果与分析
2. 1 全基因组测序及CSSLs群体构建情况
利用Illumina高通量测序技术对广西普通野生稻DP30和DP15进行测序,结果(图2)表明,DP30和DP15与93-11间的SNPs分别为1900132和1894103 bp。针对DP30-CSSLs群体和DP15-CSSLs群体分别开发285和254对亲本间具有多态性的分子标记。相邻分子标记的平均间距分别为4.92和5.62 cM,经数据库(https://www.ncbi.nlm.nih.gov/)搜索筛选,最终构建出由144个CSSLs组成的DP30-CSSLs群体和由59个CSSLs组成的DP15-CSSLs群体。DP30-CSSLs群体代换片段累计全长737.5 Mb,对DP30全基因组的覆盖率约为94.71%;DP15-CSSLs群体代换片段累计全长337.36 Mb,对DP15全基因组的覆盖率约为73.11%。
2. 2 DP30-CSSLs群体和DP15-CSSLs群体的落粒性鉴定结果
分别调查CSSLs群体和亲本的落粒性,从开花当天(第0 d)到种子成熟(第21 d)期间每隔3 d对CSSLs群体和亲本的种子拉力值进行测量,结果表明种子拉力值与其落粒性呈负相关(表1)。93-11种子未脱落,不具落粒性,在整个观察期间的落粒率为0,拉力值维持在155~165 g;而DP30、DP15及12个CSSLs群体(CSSL-Y104、CSSL-Y68、CSSL-Y83、CSSL-Y328、CSSL-Y235、CSSL-Y64、CSSL-Y63-2、CSSL-Y303、CSSL-Y99、CSSL-Y106-3、CSSL-Z37和CSSL-Z38)除空瘪粒外,其余种子均已完全脱落,具有落粒性。从第6 d起DP30、DP15及12个CSSLs群体的拉力值开始下降,第9 d的拉力值持续下降,至第12 d各拉力值均降为0 g。其中,CSSL-Y99(图3)在第0~3 d不落粒,其拉力值维持在200 g左右;从第6 d起开始出现脱落,落粒率约30%,拉力值下降至80 g;至第9 d种子大量脱落,落粒率约75%,拉力值下降至0 g;第9 d除空瘪粒外种子几乎完全脱落,落粒率约94%。可见,相对于其余11个CSSLs群体,CSSL-Y99的落粒率最高,拉力值下降最快。
2. 3 次级F2群体表型鉴定及遗传分析结果
CSSL-Y99是CSSLs群体中表型最强的CSSLs,为进一步明确其种子落粒性,故选取CSSL-Y99和93-11构建次级F2群体。遗传分析结果表明,杂交后代F1均具有落粒性,且相关分子标记显示供试F1的基因型均为杂合型。经自交一代后,共获得F2群体188株,其中落粒141株、不落粒47株,分离比为3∶1[χ2=1.630<χ2(0.05,1)=3.84],符合一对基因分离规律,说明CSSL-Y99与93-11的落粒性差异是受单个显性基因控制。
2. 4 落粒性QTL检测结果
由表2和图4可知,在2个CSSLs群体中共发现12个落粒性CSSLs,依据重叠群QTL分析检测出6个不同的落粒性QTLs(qSH2.1、qSH4.1、qSH5.1、qSH9.1、qSH11.1和qSH11.2),分别分布在第2、4、5、9和11染色体上。其中,qSH2.1位于第2染色体的C2-21~ C2-22,3.28 Mb区间,表型贡献率为-7.8%;qSH4.1位于第4染色体的C4-22~C4-23,2.01 Mb区间,表型贡献率为-10.9%,通过与前人的相关研究结果比较,发现该区间含有已克隆的SH4基因(Li et al.,2006);qSH5.1位于第5染色体的RM3227~5M13153,4.41 Mb区间,表型贡献率为-15.6%,通过与前人的相关研究结果比较,发现该区间含有已克隆的SH5基因(Yoon et al.,2015);qSH9.1位于第9染色体的C9-8~C9-9,1.25 Mb区间,贡献率为-10.9%;qSH11.1和qSH11.2分别位于第11染色体的C11-5~C11-8和C11-16~C11-17,4.5 Mb和0.5 Mb区间,贡献率分别为-23.4%和-15.6%。其中,qSH11.1的加性效应最强(-37.5),因而用于进行连锁分析。
2. 5 落粒性QTL qSH11.1连锁分析结果
CSSL-Y99与93-11的落粒性存在明显差异,是一个完全落粒性的CSSLs,所携带的qSH11.1表型贡献率(-23.4%)在6个落粒性QTLs中最高。CSSL-Y99的代换片段位于第11染色体的C11-4~C11-11区间,通过与CSSL-Y303、CSSL-Y106-3和CSSL-Y274的代换片段重叠而将qSH11.1缩小至CSSL-Y99、CSSL-Y303和CSSL-Y106-3的重叠片段上,即第11染色体的C11-5~C11-8区间。参考测序结果,本研究在qSH11.1所在区间内开发6对InDel分子标记(M1、M2、M3、M4、M5和M6)(表3),从构建的次级F2群体分析中筛选出4个重组单株(43、167、128和136),基于重组单株的基因型和表型,可将qSH11.1定位于InDel分子标记M5和M6间约1.5 Mb的范围内(图5),LOD值为25.0。
3 讨论
至今,针对广西普通野生稻各类性状的QTL定位已有较多研究报道(桑洪玉,2014;潘英华等,2017;刘剑镔,2018),但落粒性状QTL定位相对滞后。本研究从广西普通野生稻DP30-CSSLs群体和DP15-CSSLs群体中筛选出12个具有落粒性的CSSLs,检测出6个落粒性QTLs(qSH2.1、qSH4.1、qSH5.1、qSH9.1、qSH11.1和qSH11.2),且均呈负效应,对应的表型贡献率分别为-7.8%、-10.9%、-15.6%、 -10.9%、-23.4%和-15.6%。其中,qSH11.1的效应最强,qSH4.1与SH4基因(34231186~34233373 bp)在同一染色体区间内(Li et al.,2006),qSH5.1与SH5基因(22353997~22358568 bp)在染色体相同位置(Yoon et al.,2015)。在利用CSSLs构建次级F2群体定位落粒性QTL方面,郑丽媛(2017)以日本晴为受体亲本、优良恢复系R225为供体亲本,通过回交并结合MAS,鉴定了一个携带主效易落粒基因的水稻CSSL(Z481),并将SH6(t)定位于RM253~ZLY722,其物理距离在69 kb区间内;宗玉龙(2015)从236个植株中筛选出6个交换株,并将qSH4初步定位于RM6441~ RM1113,其物理距离为377 kb。本研究选取落粒性强、表型贡献率大的CSSL-Y99与93-11进行杂交,构建次级F2群体,从181个植株中筛选出4个交换株,将qSH11.1初步定位在第11染色体M5~M6间约1.5 Mb的区间内。由于该区间内未见已报道的落粒性基因,故推测其是一个新的落粒性基因。
本研究在DP30-CSSLs群体和DP15-CSSLs群体中新发现的QTL,可為后续精准定位新基因打下基础。除了精细定位外,CSSLs/SSSLs在QTL的聚合上也有广泛应用。谭全亚(2016)利用66个SSSLs对水稻柱头外露率QTL进行鉴定和聚合。徐建军和梁国华(2011)研究证实,通过遗传背景相同的CSSLs杂交可将不同代换片段上的多个优良基因聚合在一起,而培育出具有更多优良性状的新品种。本研究结果表明,12个携带落粒性QTL的CSSLs群体遗传背景中轮回亲本的回复率较高,且其他性状与93-11相似。由于93-11作为我国广泛栽培的优良品种,故这些CSSLs可直接搭配杂交组合。
数量性状遗传复杂,通过CSSLs群体进行QTL定位可消除遗传背景干扰,将复杂性状分解为单个孟德尔因子进行研究,避免不同QTL间的干扰(Brondani et al.,2002;Zhao et al.,2015),使基于CSSLs群体的QTL定位结果相对于利用F2初级作图群体的QTL定位结果更可靠(张向阳等,2014;汪欲鹏等,2016)。CSSLs群体是将亲本间的差异性状分离到各CSSLs中,降低各性状间的相互干扰,使得表型鉴定更直观。本研究对2个普通野生稻CSSLs群体进行全基因组深度测序,全面掌握其代换片段和遗传背景的信息,为落粒性QTL鉴定和基因克隆、QTL互作分析及杂种优势机理研究等提供了重要参考依据。
4 结论
从构建的广西普通野生稻核心种质资源(DP30和DP15)CSSLs群体中检测出6个落粒性QTLs,其中主效QTL qSH11.1定位于第11染色体M5~M6间约1.5 Mb的范围内,是新发现的落粒性QTL。
参考文献:
江川,朱业宝,张丹,郑苹立,王金英. 2018. 稻种资源收集、保存和更新中存在的问题及对策[J]. 江西农业学报,30(9):16-20. [Jiang C,Zhu Y B,Zhang D,Zheng P L,Wang J Y. 2018. Existent problems in collection,preservation and regeneration of rice germplasm resources and their countermeasure[J]. Acta Agriculturae Jiangxi,30(9):16-20.]
刘剑镔. 2018. 广西普通野生稻苗期耐冷QTL qCTS12精细定位及其调控模式研究[D]. 南宁:广西大学. [Liu J B. 2018. The fine mapping and regulation model research of common wild rice in Guangxi QTL qCTS12,a locus positively responses chill in rice seedling stage[D]. Nanning:Guangxi University.]
吕树伟. 2018. 非洲野生稻落粒基因SH3的克隆及其分子演化[D]. 北京:中国农业大学. [Lü S W. 2018. Cloning and molecular evolution of ObSH3 controlling seed shattering in African wild rice(Oryza barthii A. Chev.)[D]. Beijing:China Agricultural University.]
潘英华,温国泉,徐志健,梁云涛. 2017. 利用野栽杂交分离群体定位水稻结实率QTLs[J]. 西南农业学报,30(7):1473-1478. [Pan Y H,Wen G Q,Xu Z J,Liang Y T. 2017. Mapping of QTLs for seed setting rate using hybrid popu-lation of Oryza rufipogon Griff. ×cultivated rice[J]. Southwest Journal of Agriculture Sciences,30(7):1473-1478.]
桑洪玉. 2014. 广西普通野生稻育性恢复基因Rf3、Rf4的恢复效应及Rf3基因的定位[D]. 南宁:广西大学. [Sang H Y. 2014. Research on effects of restorer genes Rf3 and Rf4 from Guangxi common wild rice(Oryza rufipogon Griff.) and mapping of Rf3[D]. Nanning:Guangxi University.]
谭全亚. 2016. 基于SSSL水稻柱头外露率QTL的鉴定和聚合效应分析[D]. 广州:华南农业大学. [Tan Q Y. 2016. Identification and pyramiding of QTL related to stigma exsertion rate based on SSSL in rice[D]. Guangzhou:South China Agricultural University.]
汪欲鹏,段里成,龙启樟,徐林典,武志峰,万建林,石庆华,潘晓华,吴自明. 2016. 大穗型水稻穗部性状的QTL定位[J]. 南方农业学报,47(9):1445-1449. [Wang Y P,Duan L C,Long Q Z,Xu L D,Wu Z F,Wan J L,Shi Q H,Pan X H,Wu Z M. 2016. QTL analysis of panicle traits in large panicle rice[J]. Journal of Southern Agriculture,47(9):1445-1449.]
徐建军,梁国华. 2011. 水稻染色体片段代换系群体的构建及应用研究進展[J]. 安徽农业科学,39(4):1935-1938. [Xu J J,Liang G H. 2011. Research progress of construction and application of rice(Oryza sativa L.) chromosome segment substitution lines[J]. Journal of Anhui Agricultural Sciences,39(4):1935-1938.]
张向阳,张红宇,徐培洲,陈晓琼,田永航,王志,黄廷友,吴先军. 2014. 重穗型水稻穗部性状及剑叶宽的QTL定位[J]. 杂交水稻,29(6):56-61. [Zhang X Y,Zhang H Y,Xu P Z,Chen X Q,Tian Y H,Wang Z,Huang T Y,Wu X J. 2014. QTL mapping of flag leaf width and panicle traits of heavy-panicle rice[J]. Hybrid Rice,29(6):56-61.]
郑丽媛. 2017. 水稻落粒染色体片段代换系Z481的鉴定及SH6(t)定位[D]. 重庆:西南大学. [Zheng L Y. 2017. Identification of rice chromosome segment substitution line Z481 with seed shattering and fine mapping of the SH6(t)[D]. Chongqing:Southwest University.]
宗玉龍. 2015. 水稻易落粒突变体的鉴定与基因定位[D]. 南京:南京农业大学. [Zong Y L. 2015. Evaluation and gene mapping of a grain shatting mtuttant in rice(Oryza sativa L.)[D]. Nanjing:Nanjing Agricultural University.]
Brondani C,Rangel P,Brondani R,Ferreira M. 2002. QTL mapping and introgression of yield-related traits from Oryza glumaepatula to cultivated rice(Oryza sativa) using microsatellite markers[J]. Theoretical and Applied Gene-tics,104(6-7):1192-1203.
Doebley J F,Gaut B S,Smith B D. 2006. The molecular genetics of crop domestication[J]. Cell,127(7):1309-1321.
Furuta T,Uehara K,Angeles-Shim R B,Shim J,Ashikari M,Takashi T. 2014. Development and evaluation of chromosome segment substitution lines(CSSLs) carrying chromosome segments derived from Oryza rufipogon in the genetic background of Oryza sativa L[J]. Breeding Scien-ce,63(5):468-475.
Huang X H,Kurata N,Wei X H,Wang Z X,Wang A H,Zhao Q,Zhao Y,Liu K Y,Lu H Y,Li W J,Guo Y L,Lu Y Q,Zhou C C,Fan D L,Weng Q J,Zhu C R,Huang T,Zhang L,Wang Y C,Feng L,Furuumi H,Kubo T,Miyabayashi T,Yuan X P,Xu Q,Dong G J,Zhan Q L,Li C Y,Fujiyama A,Toyoda A,Lu T T,Feng Q,Qian Q,Li J Y,Han B. 2012. A map of rice genome variation reveals the origin of cultivated rice[J]. Nature,490(7421):497-501.
Kechin A,Boyarskikh U,Kel A,Filipenko M. 2017. CutPri-mers:A new tool for accurate cutting of primers from reads of targeted next generation sequencing[J]. Journal of Computational Biology,24(11):1138-1143.
Li C B,Zhou A L,Sang T. 2006. Rice domestication by reducing shattering[J]. Science,311(5769):1936-1939.
Lin Z W,Griffith M E,Li X R,Zhu Z F,Tan L B,Fu Y C,Zhang W X,Wang X K,Xie D X,Sun C Q. 2007. Origin of seed shattering in rice(Oryza sativa L.)[J]. Planta,226(1):11-20.
McKenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A,Garimella K,Altshuler D,Gabriel S,Daly M,DePristo M A. 2010. The genome analysis toolkit:A mapreduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research,20(9):1297-1303.
Stein J C,Yu Y,Copetti D,Zwickl D J,Zhang L,Zhang C J,Chougule K,Gao D Y,Iwata A,Goicoechea J L,Wei S,Wang J,Liao Y,Wang M H,Jacquemin J,Becker C,Kudrna D,Zhang J W,Londono C E M,Song X,Lee S,Sanchez P,Zuccolo A,Ammiraju J S S,Talag J,Dano-witz A,Rivera L F,Gschwend A R,Noutsos C,Wu C C,Kao S M,Zeng J W,Wei F J,Zhao Q,Feng Q,Baidouri M E l,Carpentier M C,Lasserre E,Cooke R,da Rosa Farias D,da Maia L C,Dos Santos R S,Nyberg K G,McNally K L,Mauleon R,Alexandrov N,Schmutz J,Flowers D,Fan C,Weigel D,Jena K K,Wicker T,Chen M S,Han B,Henry R,Hsing Y I C,Kurata N,de Oliveira A C,Panaud O,Jackson S A,Machado C A,Sanderson M J,Long M Y,Ware D,Wing R A. 2018. Genomes of 13 domesticated and wild rice relatives highlight genetic conservation,turnover and innovation across the genus Oryza[J]. Nature Genetics,50(2):285-296.
T?rjék O,Meyer R C,Zehnsdorf M,Teltow M,Strompen G,Witucka-Wall H,Blacha A,Altmann T. 2008. Construction and analysis of 2 reciprocal arabidopsis introgression line populations[J]. The Journal of Heredity,99(4):396-406.
van Berloo R. 2008. GGT 2.0:Versatile software for visualization and analysis of genetic data[J]. The Journal of Heredity,99(2):232-236.
Wu W G,Liu X Y,Wang M H,Meyer R S,Luo X J,Ndjion-djop M N,Tan L B,Zhang J W,Wu J Z,Cai H W,Sun C Q,Wang X K,Wing R A,Zhu Z F. 2017. A single-nucleo-tide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication[J]. Nature Plants,3:17064. doi:/10.1038/nplants.2017.64.
Wu X R,Tang D,Li M,Wang K J,Cheng Z K. 2013. Loose plant architecture1,an indeterminate domain protein involved in shoot gravitropism,regulates plant architecture in rice[J]. Plant Physiology,161(1):317-329.
Wu Y B,Li G,Zhu Y J,Cheng Y C,Yang J Y,Chen H Z,Song X J,Ying J Z. 2020. Genome-wide identification of QTLs for grain protein content based on genotyping-by-resequencing and verification of qGPC1-1 in rice[J]. International Journal of Molecular Sciences,21(2):408. doi:10.3390/ijms21020408.
Xiong L Z,Liu K D,Dai X K,Xu C G,Zhang Q F. 1999. Identification of genetic factors controlling domestication-related traits of rice using an F2 population of a cross between Oryza sativa and O. rufipogon[J]. Theoretical and Applied Genetics,98(2):243-251.
Yan H X,Ma L,Wang Z,Lin Z M,Su J,Lu B R. 2015. Multiple tissue-specific expression of rice seed-shattering gene SH4 regulated by its promoter pSH4[J]. Rice,8:12. doi:10.1186/s12284-015-0047-4.
Yoon J M,Cho L H,Kim S L,Choi H,Koh H J,An G. 2015. The BEL1-type homeobox gene SH5 induces seed shatte-ring by enhancing abscission-zone development and inhi-biting lignin biosynthesis[J]. The Plant Journal,79(5):717-728.
Yu B S,Lin Z W,Li H X,Li X J,Li J Y,Wang Y H,Zhang X,Zhu Z F,Zhai W X,Wang X K,Xie D X,Sun C Q. 2008. TAC1,a major quantitative trait locus controlling tiller angle in rice[J]. The Plant Journal,52(5):891-898.
Zhang L B,Zhu Q H,Wu Z Q,Ross I J,Gaut B S,Ge S,Sang T.2009. Selection on grain shattering genes and rates of rice domestication[J]. The New Phytologist,184(3):708-720.
Zhao L,Tan L B,Zhu Z F,Xiao L T,Xie D X,Sun C Q. 2015. PAY1 improves plant architecture and enhances grain yield in rice[J]. The Plant Journal,83(3):528-536.
Zhao H W,Sun L L,Xiong T Y,Wang Z Q,Liao Y,Zou T,Zheng M M,Zhang Z,Pan X P,He N,Zhang G Q,Zhu H T,Liu Z Q,He P,Fu X L. 2019. Genetic characterization of the chromosome single-segment substitution lines of O. glumaepatula and O. barthii and identification of QTLs for yield-related traits[J]. Molecular Breeding,39:51. doi:10.1007/s11032-019-0960-0.
Zhao S S,Wang C H,Ma J,Wang S,Tian P,Wang J L,Cheng Z J,Zhang X,Guo X P,Lei C L. 2016. Map-based cloning and functional analysis of the chromosome gene C in rice(Oryza sativa L.)[J]. Journal of Plant Bio-logy,59(5):496-505.
Zhou Y,Lu D F,Li C Y,Luo J H,Zhu B F,Zhu J J,Shangguan Y Y,Wang Z X,Sang T,Zhou B,Han B. 2012. Genetic control of seed shattering in rice by the APETALA2 transcription factor shattering abortion 1[J]. The Plant Cell,24(3):1034-1048.
(責任编辑 兰宗宝)

