白念珠菌CaPDE2基因的敲除与功能研究
毛红辰,张 幸,许海涛,李 峰,徐大勇,2①
(1. 淮北师范大学 生命科学学院,安徽 淮北235000;2. 淮北师范大学 信息学院,安徽 淮北235000)
0 引言
白念珠菌(Candida albicans)是临床上最常见的人体条件致病真菌,存在于健康人体的口腔、上呼吸道、消化道、生殖道等黏膜表面. 白念珠菌通常不会引起人体疾病,但是当人体免疫系统受到损害或体内正常微生物菌群失调时,该菌过度生长引起浅部感染(如鹅口疮、阴道炎等);在免疫能力严重低下的人群中(如艾滋病患者、器官移植病人、经过化疗和理疗的癌症病人等),白念珠菌过度繁殖引起严重的系统性感染[1-2]. 据报道,在世界范围内,白念珠菌感染每年造成25万到40万患者死亡,以及大约1亿次的复发性阴道炎的发病率[3-4]. 由于病人治疗过程中大量使用抗真菌药物,以及白念珠菌在病人人体组织和植入人体的导管等医疗器具表面形成生物膜,导致白念珠菌的耐药现象日益严重[5-8]. 因此,研究揭示白念珠菌的致病、耐药机制,发现新型抗真菌药物的基因靶点,对预防和治疗白念珠菌感染具有重要意义.
白念珠菌SC5314基因组测序的完成[9],为其基因功能的研究提供了极大的便利. 通过基因敲除并测试基因缺失株的表型是研究基因功能最常用的方法之一,由于白念珠菌一般情况下是二倍体细胞,基因敲除有一定的难度. 目前白念珠菌的基因敲除技术一般是基于营养缺陷型或抗药性标记. 例如:URA3-blaster(尿苷合成基因标记)[10]、SAT1-flipper(诺尔斯菌素抗性基因标记)[11]、从非白念珠菌PCR 扩增HIS1/LEU2/ARG4营养标记敲除盒等[12]. 但是单一使用一种基因标记敲除方法存在敲除效率低的问题.真核生物细胞中,cAMP信号通路调控多种细胞过程,其过程是胞外信号与细胞表面相应受体结合,从而导致细胞内第二信使cAMP 的水平变化而引起细胞各种反应[13]. 环核苷酸磷酸二酯酶(Cyclic nucleo⁃tide phosphodiesterase,PDE)是催化细胞内环核苷酸水解的一类磷酸酯酶,对细胞内cAMP/AMP 或者cGMP/GMP水平进行控制,从而调控的细胞生理代谢反应[14]. 在酿酒酵母中,ScPDE2编码对cAMP具有高亲和力的环核苷酸磷酸二酯酶2(ScPde2),其在控制细胞内cAMP/AMP浓度及调节多个生理过程起到重要作用[15]. 本研究以人体条件致病真菌白念珠菌CaPDE2为例,综合应用尿苷合成基因标记(URA3)和诺尔斯菌素抗性基因标记(SAT1)高效敲除CaPDE2基因,并测试CaPDE2基因缺失株的表型,对CaPDE2基因在白念珠菌细胞中的功能进行探究.
1 材料与方法
1.1 材料
1.1.1 菌株、质粒和引物:本研究使用的菌株和质粒见表1,引物见表2.

表1 本研究使用的菌株和质粒

表2 本研究使用的引物
1.1.2 主要药品及试剂
限制性内切酶、DNA 聚合酶、氨苄青霉素钠等购自宝生物工程有限公司;Ezmax One-Step 克隆试剂盒购自吐露港生物科技有限公司;诺尔斯菌素(ClonNAT)购自Jena Bioscience 公司;所有引物及5-氟乳清酸(5-FOA)购自生工生物工程(上海)股份有限公司;鲑鱼精DNA、醋酸锂、聚乙二醇3350、酮康唑、氟康唑等购自Sigma公司.
1.1.3 培养基
LB+Amp培养基:NaCl 10 g/L,酵母提取物5 g/L,蛋白胨10 g/L,pH 7.0,灭菌后加氨苄青霉素钠浓度至100 μg/mL;YPD培养基:酵母浸出粉10 g/L,蛋白胨20 g/L,葡萄糖20 g/L;YPD+200 μg/mL ClonNAT培养基:YPD 培养基灭菌后添加终浓度为200 μg/mL 的ClonNAT;YPD+25 μg/mL ClonNAT 培养基:YPD 培养基灭菌后添加终浓度为25 μg/mL的ClonNAT;YPD+0.1% 5-FOA培养基:YPD培养基灭菌后添加终浓度为0.1%的5-氟乳清酸(5-FOA). SD-URA 培养基:无氨基酸酵母氮源6.7 g/L,省却尿苷酸氨基酸混合物粉末0.71 g/L,葡萄糖20;SD-URA+200 μg/mL ClonNAT 培养基:SD-URA培养基灭菌后添加终浓度为200 μg/mL的ClonNAT.
1.2 方法
1.2.1 CaPDE2基因的SAT1-flipper敲除盒的构建
以质粒pSFS2为模板,用引物PDE2-F、PDE2-R,以白念珠菌SC5314基因组作为模板,通过PCR方法扩增敲除CaPDE2的SAT1-flipper敲除盒.
1.2.2 CaPDE2基因的URA3-blaster敲除质粒的构建
用引物PDE2-L-UP、PDE2-L-DOWN,以白念珠菌SC5314 基因组作为模板,通过PCR 扩增出CaP⁃DE2 开放阅读框上游序列965 bp 作为基因敲除上游同源臂,通过无缝克隆试剂盒克隆到质粒p5921 的SacI和KpnI位点;然后,用引物PDE2-R-UP、PDE2-R-DOWN,以白念珠菌SC5314基因组作为模板,通过PCR扩增出CaPDE2开放阅读框下游序列713 bp作为基因敲除下游同源臂,克隆到质粒p5921的BamHI和HindⅢ位点;至此获得CaPDE2基因的URA3-blaster 敲除质粒,然后用SacI和HindⅢ双酶切此敲除质粒,即获得CaPDE2基因的URA3-blaster敲除盒.
1.2.3 CaPDE2双等位基因的敲除
用方法1.2.1 获得的CaPDE2 基因的SAT1-flipper 敲除盒,采用醋酸锂方法[11]转化到野生型菌株CAI4,在YPD+200 μg/mL ClonNAT平板上筛选获得敲除CaPDE2第一等位基因的菌株;用方法1.2.2获得的CaPDE2基因的URA3-blaster敲除盒,采用醋酸锂方法转化到敲除CaPDE2第一等位基因的菌株中,在SD-URA+200 μg/mL ClonNAT 平板上筛选获得敲除CaPDE2第二等位基因的菌株. 将敲除CaPDE2双等位基因的菌株依次培养在YPD+25 μg/mL ClonNAT、YPD+0.1% 5-FOA平板上,以去除诺尔斯菌素抗性基因标记SAT1和尿苷合成基因标记URA3.
1.2.4 CaPDE2缺失株的表型试验
挑取CAI4、CaPDE2缺失株菌株的单菌落接种到YPD液体培养基中过夜培养,之后将CAI4、CaPDE2缺失株菌株的细胞培养液调成5×107个细胞/mL. 按照文献[16-17]表型试验操作方法将菌液被稀释至10-4. 然后从每个离心管中吸取2 μL稀释菌液点接至YPD及含有相应药物的YPD培养基上. 30 ℃倒置培养,拍照观察.
2 结果与分析
2.1 CaPDE2基因的URA3-blaster敲除质粒的构建
以野生型菌株CAI4 基因组DNA 为模板,PDE2-L-UP、PDE2-L-DOWN为引物进行PCR 反应,扩增出CaPDE2 开放阅读框上游的同源片段965 bp,扩增产物纯化后连接到用SacI、KpnI处理的p5921 载体中,获得质粒pDY1. 接下来,以白念珠菌SC5314 基因组DNA 为模板,PDE2-R-UP、PDE2-R-DOWN 为引物进行PCR反应,扩增出CaPDE2 开放阅读框下游的同源片段713 bp,扩增产物纯化后连接到用Bam⁃HI、HindⅢ处理的pXD1载体中,获得CaPDE2基因的URA3-blaster敲除质粒pDY2,如图1所示. 将pDY2用SacI、HindⅢ双酶切,获得CaPDE2基因的URA3-blaster敲除盒(5.7 kb),如图2所示.

图1 CaPDE2基因的URA3-blaster敲除质粒的构建示意图

图2 CaPDE2基因的URA3-blaster敲除质粒pDY2的SacI、HindⅢ双酶切验证
2.2 联合应用尿苷合成基因标记URA3和诺尔斯菌素抗性基因标记SAT1敲除CaPDE2双等位基因
白念珠菌是二倍体,对白念珠菌基因的敲除必须敲除目的基因的两个等位基因. 本研究出发菌株CAI4是尿苷营养缺陷型菌株[10],而且对诺尔斯菌素敏感[11]. 白念珠菌CaPDE2编码的磷酸二酯酶2蛋白,是Ras/cAMP/PKA 通路中的重要成员,该酶催化cAMP 水解成5′-AMP 而降低胞内cAMP 浓度,以维持胞内适当的cAMP浓度. 应用诺尔斯菌素抗性基因标记SAT1敲除CaPDE2第一等位基因. 以质粒pSFS2为模板,用引物PDE2-F、PDE2-R,以CAI4 基因组作为模板,PCR 扩增出大小约为4.3 kb 的CaPDE2 的SAT1-flipper 敲除盒(图3). 将该敲除盒转化至CAI4 中,涂布于200 μg×mL-1ClonNAT 的YPD 平板,30 ℃培养48 h,挑取ClonNAT 抗性菌株纯化,获得敲除CaPDE2 第一等位基因的菌株,其基因型是Cap⁃de2::SAT1-flipper/CaPDE2. 接下来,将构建好的含有CaPDE2 的URA3-blaster 敲除盒的质粒pDY2,用SacI和HindⅢ双酶切,获得5.7 kb带有URA3筛选标记的CaPDE2敲除盒(图2). 将此敲除盒转化至菌株Capde2::SAT1-flipper/CaPDE2中,涂布在SD-URA+200 μg/mL ClonNAT平板上,30 ℃培养48~72 h,挑取阳性转化子并验证,获得敲除CaPDE2第二等位基因菌株. 之后用YPD+25 μg/mL ClonNAT平板挤掉SAT1 抗性基因,进一步用YPD+0.1% 5-FOA 平板去除URA3 基因,最终获得CaPDE2 基因缺失株DY⁃CA1,其基因型为Capde2::FRT/Capde2::hisG(图4,5). 如图5 所示,用引物PDE2-UP-CHECK/DOWCHECK:以CAI4基因组为模板,扩增出2.2 kb的片段(包含CaPDE2基因全长ORF序列及其一部分启动子和终止子序列);以CaPDE2 基因缺失株的基因组为模板,扩增出0.85 kb 和1.7 kb(包含hisG 序列及CaPDE2基因的一部分启动子和终止子序列)的2个片段. 用引物PDE2-ORF-UP/DOWN:以CAI4基因组为模板,扩增出0.55 kb的片段(CaPDE2基因ORF的一部分序列);以CaPDE2缺失株的基因组为模板,未扩增出目标条带,表明CaPDE2的2个等位基因都已被敲除.

图3 PCR扩增CaPDE2的诺尔斯菌素抗性SAT1-flipper敲除盒

图4 联合应用尿苷合成基因标记URA3和诺尔斯菌素 抗性基因标记SAT1敲除CaPDE2双等位基因示意图
2.3 CaPDE2基因缺失株对氟康唑、酮康唑、SDS及42 ℃的敏感
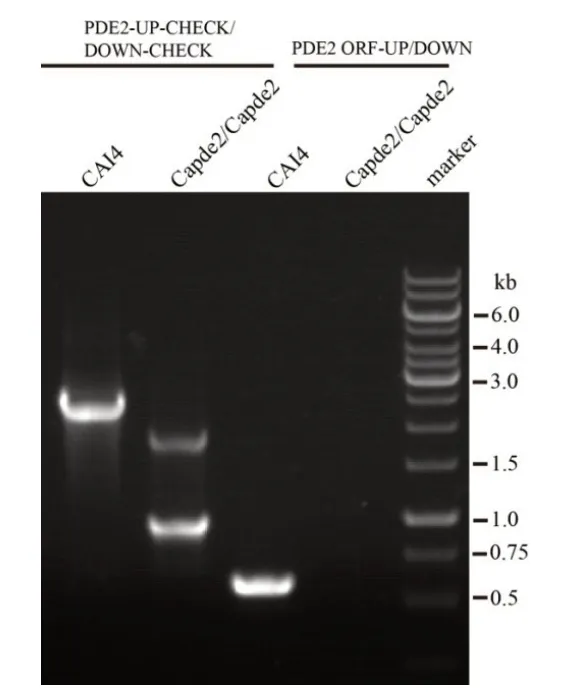
图5 CaPDE2基因缺失株的PCR验证

图6 白念珠菌CaPDE2基因缺失株对氟康唑、酮康唑、SDS及42 ℃的敏感性
通过倍比稀释表型试验发现(图6),与野生型菌株CAI4 相比,CaPDE2 基因缺失株对4 μg/mL KCZ(Ketoconazole,酮康唑)、5 μg/mL FLU(Fluconazole,酮康唑)、细胞膜干扰剂0.04% SDS(Sodium dodecyl sulfate,十二烷基硫酸钠)以及42 ℃均敏感,表明白念珠菌CaPDE2基因在耐受抗真菌药物、细胞膜压力及高温胁迫方面起作用.
3 结论与讨论
在本研究中,联合采用URA3 和SAT1 筛选标记完成了CaPDE2 双等位基因的敲除,并获得了去掉SAT1和URA3基因标记的CaPDE2基因缺失株,其基因型为Capde2::FRT/Capde2::hisG. 白念珠菌基因敲除的难度在于目的基因第二等位基因的敲除,本研究联合应用URA3和SAT1筛选标记进行基因敲除的优势:一是可以高效敲除目的基因的第二等位基因;二是去掉SAT1和URA3基因标记的缺失株可以用于构建多个基因的缺失株,研究多个基因(2个以上基因)的缺失对于白念珠菌的影响或用于研究多个基因之间的相互关系.
表型试验结果表明,CaPDE2基因缺失株对细胞膜干扰剂SDS敏感,与Jung等[18]报道的结果一致,表明敲除CaPDE2 基因很可能导致白念珠菌的细胞膜组分发生变化,从而导致CaPDE2 基因缺失株对SDS敏感. 研究还发现,CaPDE2基因缺失株对抗真菌药物氟康唑和酮康唑敏感,表明CaPDE2基因在耐受唑类抗真菌药物起作用. 唑类药物是目前临床常用的抗真菌药物,唑类药物发挥抗真菌作用是通过干扰羊毛甾醇14-α-去甲基化酶活性使得麦角甾醇的合成受阻,导致有毒的甾醇14-α-methyl-3,6-diol 的积累,该有毒甾醇的积累会使真菌细胞膜形成严重的压力,从而起到抗真菌作用[19]. 但是由于目前唑类抗真菌药物的大量使用,使得白念珠菌对唑类药物产生了严重的抗药性[20]. 白念珠菌缺失CaPDE2基因导致细胞对唑类药物敏感,因此,CaPDE2可以作为开发新的抗真菌药物的潜在靶点. 本研究还表明,CaP⁃DE2基因缺失株对42 ℃敏感,其具体耐热机制有待进一步研究.

