极地微生物基因组挖掘现状与展望
廖丽 陈波
研究综述
极地微生物基因组挖掘现状与展望
廖丽 陈波
(中国极地研究中心, 上海 200136)
测序技术的快速发展极大地促进了微生物基因组相关研究。极地微生物基因组的测序与研究虽然启动略晚, 但迅速成为极地微生物研究的一个重要内容。极地微生物基因资源具有重要的战略意义与社会效益, 但针对极地微生物基因组的深入研究以及基因资源的深度挖掘仍然非常有限, 没有真正发挥基因组测序的作用与价值。本文总结了国际上极地微生物基因组相关研究现状与主要的研究方向, 分析了极地微生物基因组挖掘潜力与目前研究的不足之处, 并在此基础上提出了重点需要突破的方向。极地微生物基因资源潜力巨大, 加强对极地微生物基因资源的挖掘将为未来生物技术的发展提供更多可能性。
极地微生物 基因组 基因资源 基因组挖掘
0 引言
极地具备最极端的自然环境与气候特征, 例如长期低温、常年冰雪覆盖、高辐射等极端条件对生命提出了诸多挑战。然而即便如此, 在极地各种生境中仍然存在丰富多样的微生物, 在极地生态系统中占据举足轻重的地位, 并在长期极端环境压力的选择下, 进化出适应极端环境的生理、遗传与代谢特征, 因而具有重要的基础研究价值与巨大的应用潜力。同时, 在《南极条约》的限制下, 微生物是目前可以开发利用的自然资源, 因此具有重要的战略意义, 也成为各国生物勘探的重点对象。
基因组测序技术的发展为极地微生物基因组及其基因资源的研究提供了新的机遇, 增加了我们对于极地微生物基因组特征与环境适应机制的认识, 也揭示了极地微生物基因资源的新颖性与潜在的应用价值。相比于模式物种或者常规环境中微生物的基因组测序, 极地微生物基因组测序研究历史并不长, 但完成基因组测序的极地微生物数量每年都在增加, 表明极地微生物基因组的研究越来越受到关注。但总体而言, 极地微生物基因组相关研究与基因资源的挖掘仍处于初级阶段。了解极地微生物基因组测序与研究的现状, 能更好地把握研究与发展趋势, 为开展极地微生物基因组相关研究与基因资源的挖掘提供指导。因此本文总结了极地微生物基因组研究现状、趋势与不足, 并提出了相应的建议, 以期为相关研究与政策制定提供参考。
1 极地微生物基因组测序现状及发展趋势
2003年首次报道了两个极地微生物基因组[1], 即来自南极埃斯湖(Ace Lake)的产甲烷古菌()和甲烷球菌()。在随后的数年里, 极地微生物基因组的研究与报道并没有大量增加, 2008年前总共才发表了7个极地微生物基因组, 以嗜冷或耐冷细菌为主, 例如比较经典的TAC 125基因组[2]和34H[3]。随着二代高通量测序技术的飞速发展, 测序成本能被更多的实验室接受, 极地微生物基因组报道在2011年之后迅速增加, 且呈现每年持续增加的趋势(图1)。据不完全统计, 截止2019年6月底共公布了168个极地微生物基因组, 其中2017、2018年最多, 分别发表了27、29个极地微生物基因组的测序与分析(图1)。2019年截至目前也已报道了8个极地微生物基因组, 并还会继续增加。根据美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)和美国能源部联合基因组研究所(Department of Energy-Joint Genome Institute, DOE-JGI)的数据库分析, 还可能有大量的基因组提交了数据库但尚未公布, 以及有一些基因组正在测序中。目前在专业公司或者高校、院所公用平台完成一个细菌的基因组草图测序价格低至1000元左右, 基因组测序成为了一种常规手段, 而且随着极地微生物菌株资源收集的增加, 将会有更多的极地微生物基因组完成测序, 进一步充实极地微生物基因组数据库。
从公布的极地微生物基因组种属分析, 绝大部分是细菌基因组, 仅有1个酵母基因组、2个病毒基因组、3个真菌基因组、5个古菌基因组以及6个藻类基因组。从属水平分析细菌基因组(图2), 共涵盖了68个属, 其中属共有20个基因组完成序列测定, 为目前基因组测序数量最多的属, 其次为和两个属, 各有10个基因组完成测序。余下大部分属类别仅有少数几个基因组甚至仅有1个基因组公布。可见, 完成基因组测序的极地微生物种属分布较广, 但集中研究的种属仅有少数几个。在测序的极地微生物基因组中, 也包括多个极地新物种, 例如南极新属[4]、南极新种[5]、北极新种[5]、北极新种[6]、南极新种[7]等。

图1 极地微生物基因组发表数量年度统计
Fig.1. Number of genomes of polar microorganisms announced annually

图2 极地细菌基因组在属水平的分类分布. 其他指的是仅有1个基因组测序的属类别总数
Fig.2. Distribution of bacterial genomes at the genus level. Others include genera containing only one genome
从已完成基因组测序的极地微生物来源环境分析, 所覆盖的范围很广, 包括来自南北极海水、沉积物、冻土、南极发草根际与叶际、冰川、南极湖泊、南极海绵、南极干谷土壤、南极“血瀑布”等环境样品。
2 极地微生物基因组资源挖掘现状
极地微生物基因组的研究目前还处于比较初级的阶段, 多数报道仅为基因组测序、注释与简单的分析, 仅有少数研究对基因组进行了较为深入的分析与挖掘。涉及的研究内容最常见的是跟极地低温等极端环境适应性相关的基因组特征认识, 仅有少数基因组研究提及功能基因及其产物资源, 但也未进行深入挖掘。以下总结分析了目前极地微生物基因组研究与资源挖掘的现状。
2.1 从基因组水平分析适应极端环境的机制及其抗逆功能基因
绝大部分极地微生物基因组的研究是围绕极端环境适应机制开展的基础研究, 并不涉及具体的基因资源认识, 但间接为耐逆基因等功能基因的发现与认识提供了信息。代表性研究包括基因组分析南极甲烷代谢古菌的低温适应机制[1]、从基因组与蛋白质组水平解析北极34H低温适应机制[3]、从基因组水平揭示北极TAC 125的适冷机制[2]、从基因组与转录组水平揭示北极BSW20308的温度适应机制[8]。这些研究从基因组层面分析了极地微生物适应低温相关的基因组特征, 发现极地微生物在蛋白质组成、基因组GC含量等方面存在适应特征。
同时, 从基因组分析了跟抗逆或环境适应相关的功能基因。例如抗冻蛋白/抑制重结晶蛋白编码基因、冷激蛋白编码基因、维持细胞膜在低温下流动性相关的基因、活性氧自由基抗性基因等[2,8-9]。这些功能基因在极地微生物的生存与环境适应中起到关键作用, 同时也可能在抗氧化、耐低温等方面具有实际的应用价值。代表性研究如南极发草叶际与根际的植物促生长菌株基因组的研究, 测序了5个属的细菌和1个15PA的基因组, 从基因组中发现了多个具有抑制重结晶功能的蛋白质编码基因, 可能在极地冰冻环境的适应中具有重要意义, 同时也可能应用于抗冻相关的生物技术与生物医药领域[9]。
2.2 从基因组挖掘天然产物生物合成相关基因
微生物, 尤其真菌和放线菌, 是天然产物的重要合成者。天然产物既是微生物防御、竞争与交流的化学工具, 也是人类寻找药物先导化合物的重要来源。极地微生物在适应极端环境的长时间尺度进化过程中, 可能产生了合成新颖天然产物的功能基因。传统的化学分离与鉴定手段受到诸多因素的限制, 例如多数基因或基因簇在一般条件下处于沉默状态, 得到的化合物仅是其合成天然产物潜力的极少部分。而从基因组测序入手, 通过生物信息学分析能全面了解和挖掘微生物的天然产物合成能力, 极大地促进了天然产物及其生物合成的研究。例如上面提到的南极发草菌株基因组中检测到编码合成生物活性物质(例如吲哚、氢氰酸、pyoverdine载铁蛋白)的基因[9]; 对产橘黄色色素的南极新属标准菌株ZS314T的基因组进行研究, 挖掘了5个次级代谢产物合成基因簇, 并对其中合成类胡萝卜素的基因簇进行了预测与产物验证(图3); 对极地海洋放线菌sp P6-10-X1[10]、strain NOCA502F[11]与sp. A1–2[12]的基因组测序, 分析了次级代谢产物合成相关基因簇, 并发现多个基因簇尚未研究, 预示着合成新化合物的可能性; 针对产抗菌活性物质的南极菌株sp. CAL575开展基因组研究, 发现跟次级代谢产物合成相关的基因资源[13]; 对南极海绵来源的菌株进行基因组测序, 发现有抑菌作用的挥发性有机物合成基因[14]; 以及南极3个具有抗菌活性物质合成能力的基因组测序研究[15]。

图3 南极新属标准菌株Marisediminicola antarctica ZS314T基因组中5个次级代谢产物合成基因簇以及基因簇2合成的类胡萝卜素分析. 该图修改自文献[4], 红色基因为关键合成基因, 黑色基因为合成辅助基因, 绿色基因为调控基因, 蓝色基因为转运基因, 白色为无关或者未知基因. 发酵液与菌体沉淀呈现典型的类胡萝卜色素特征
Fig. 3. The five secondary metabolite biosynthetic gene clusters predicted in the genome of the Antarctic type strainZS314Tand the analysis of carotenoids produced by cluster 2. The figure is modified from Liao et al[4], with red for core biosynthetic genes, black for additional biosynthetic genes, green for regulatory genes, blue for transporter genes and white for unrelated or unknown genes. The broth and the cell pellets are reddish orange due to the presence of carotenoids
这些研究极大地促进了对极地微生物天然产物合成潜力的快速认识, 为寻找新的天然产物及其合成基因提供了便利。随着遗传操作工具的创新与升级, 相信今后极地微生物天然产物的发现与生物合成将取得更多进展。
2.3 从基因组解析极地微生物代谢能力, 寻找生物材料、生物修复、生物质降解等方面的基因资源
微生物具有强大的代谢能力和丰富的代谢多样性, 在降解难以利用污染物的环境修复、合成新的生物材料以及生物能源利用等方面具有潜在的应用价值。随着人类活动在极地的增加, 人类对极地环境的扰动和破坏发生的概率也会增加, 因此利用极地来源的微生物进行极地环境的修复, 不仅能适应并提高低温下修复效率, 也能避免外来物种对极地环境与生态带来的不利影响。前期已经开展了极地微生物污染物降解与生物修复相关的基础研究, 例如从南极石油降解菌的基因组中测序发现了3个烷烃单加氧酶, 对该菌株及其基因资源应用于石油降解进一步研究, 发现其在寒冷海域的溢油污染生物修复方面有很好的应用前景[16-17]; 来自北极的R6-15基因组测序分析了该专性除烷烃菌的基因组特征与烷烃降解基因, 为将来实现低温环境烷烃生物修复提供材料[18]。此外, 通过基因组测序分析南极深海稀有放线菌P3-3-X1, 发现该菌株基因组编码一条完整的苯酚类污染物代谢降解途径(图4)[19]。苯酚为有毒有害致癌物质, 能降解并利用苯酚类化合物的菌株并不常见, 可见极地微生物具有很好的代谢能力与代谢多样性。此外, 通过对分离自北极地衣的木聚糖降解菌株sp. PAMC26640基因组进行测序研究, 发现了22个与木聚糖利用有关的基因, 因而在生物质降解与生物能源利用等方面具有潜在的价值[20]。在生物材料方面, 代表性研究为南极307与北极238菌株基因组分析, 发现具有新的细菌光驱动质子泵xanthorhodopsin合成基因, 能将太阳能转化为质子的电化学势能, 可能在光电器件、光探测器等方面成为潜在的信息材料, 以及在人造视网膜等方面有潜在应用[5]。
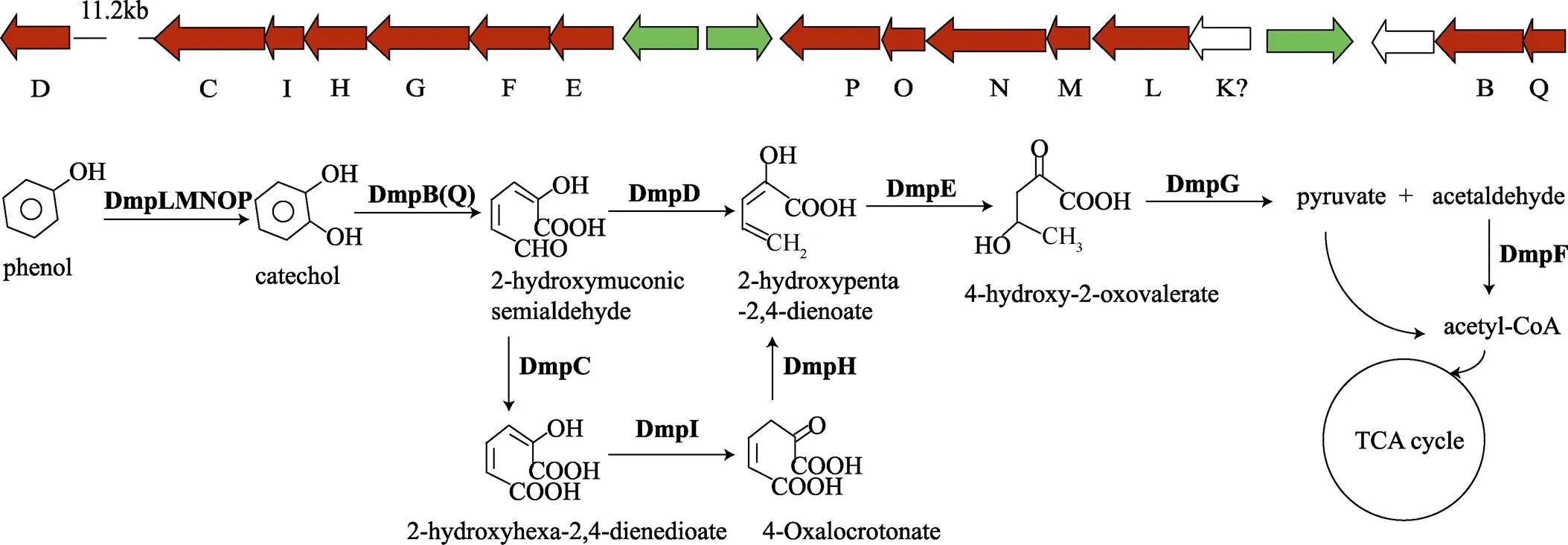
图4 南极深海稀有放线菌Janibacter limosus P3-3-X1基因组编码的苯酚降解基因簇及其降解途径示意图. 该图来自文献[19]
Fig. 4. The gene cluster and the schematic diagram for phenol degradation of rare actinobacteriumP3-3-X1 isolated from the Antarctic deep sea. The figure is adapted from Su et al[19]
3 极地微生物基因组挖掘潜力展望
3.1 极地微生物基因组具有巨大的基因资源挖掘潜力
通过上述举例分析, 可见极地微生物基因资源具有重大的研究与应用价值。前期的研究与探索表明了极地微生物在抗逆基因与产物、污染物的生物修复、惰性生物质的降解、生物新材料、天然产物合成等多方面具有重要的应用潜力。基因组测序与传统的基于单个基因或者活性开展的基因资源挖掘相比, 具有明显的优势。
优势1: 基因组测序能快速、全面地揭示整个基因组的基因资源。高通量测序技术的发展, 给极地微生物基因组研究带来了更多可能性。一旦基因组获得解析, 将全面、快速了解整个基因组的基因编码情况, 通过注释后将迅速获得已知功能基因的信息, 为其基因资源的深入挖掘提供序列信息。通过宏基因组技术, 则大大增加了对未培养微生物基因组的发现。因此极地微生物基因组蕴藏的基因资源将得到前所未有的认识与发现。
优势2: 尤其能高效发现需要多个基因参与完成的代谢与合成途径, 例如多种污染物的降解、天然产物合成等途径。尤其天然产物合成相关的基因资源往往成簇聚集在基因组上, 覆盖的基因片段长度很大, 一般在20—100 kb的范围, 只有在获得基因组的情况下才能对天然产物合成基因簇进行全面的认识与挖掘。
由于极地微生物在适应极端环境的过程中进化出了独特的基因组特征与基因资源, 而我们对于极地微生物基因组的研究与认识还相当有限, 在技术进步的推动下, 极地微生物基因组的研究与挖掘将为未来的生物技术与生物医药带来新的希望。
3.2 极地微生物基因组挖掘需要重点突破的方向
目前针对极地微生物基因组的研究大部分停留在测序与初步注释的层面, 即便开展了部分基因资源的认识, 也仅仅停留在预测阶段, 并没有充分把握基因组研究带来的机会。从资源挖掘的角度分析, 极地微生物基因组的研究与挖掘还存在不足, 需要重点突破的方向列举如下。
1. 极地微生物放线菌与真菌等资源价值明显的菌株测序研究不够, 需要有针对性地增加相应菌株基因组测序与研究。放线菌与真菌作为天然产物的主要合成者, 具有明显的资源挖掘价值。从已经发表的极地细菌基因组分析, 仅有少数放线菌基因组得到测序, 涉及的种类也非常有限, 而真菌基因组测序更少。因此增加对极地放线菌与真菌为主的基因组研究, 将有利于极地微生物天然产物及其合成基因的发掘。
2. 缺乏专门的极地微生物基因组数据库, 建立极地微生物基因组数据库将为基因资源的挖掘与研究提供便利。目前所有完成测序的基因组数据基本都被提交到以NCBI为主的综合型数据库中, 并且提交的数据存在关键信息缺乏的现象, 例如没有注明采集地点、样品环境、时间等关键信息, 导致数据的后续分析利用受阻, 或者被遗漏。建立一个极地微生物基因组数据库, 将有利于统一管理与分析相关数据, 为极地微生物基因资源的挖掘提供便利。
3. 极地微生物基因组的基因功能认识有待增加。由于极地微生物在亲缘关系、物种特异性等方面可能跟其他环境来源的微生物存在较大差异, 例如极地新发现的微生物种属在基因组完成测序后会得到更多的未知基因, 无法从已有的数据库中得到注释, 虽然这是一个较为普遍的问题, 但在极地新物种的注释时问题更突出。多组学研究以及生化功能验证将推动极地微生物新基因或者新功能的发现, 从而更好地挖掘极地微生物基因资源。
4. 极地低温菌株遗传操作体系缺乏, 研发合适的遗传操作体系将促进极地微生物基因功能研究。目前应用最多、研究最多的都是常温下的模式微生物遗传操作体系, 在研究极地微生物基因资源的过程中, 可能已有的遗传操作体系不具有普适性, 因此研发针对极地低温菌株的遗传操作体系将十分必要。
5. 利用多学科技术深入开展极地微生物基因组挖掘。由于目前大多数基因组研究仅仅报道了基本的基因组特征与序列信息, 并没有深入认识蕴藏其中的海量基因资源, 因此基因组的研究显得过于肤浅, 也无法发挥其在基因资源挖掘上的优势。深入开展极地微生物基因组的研究与挖掘, 需要生物信息学技术、遗传操作技术、代谢工程技术、酶学、化学与多组学等多学科技术的协同发展与有机结合, 需要构建多学科领域的研究团队持续开展工作。
1 SAUNDERS N F, THOMAS T, CURMI P M, et al. Mechanisms of thermal adaptation revealed from the genomes of the Antarctic Archaeaand[J]. Genome Research, 2003, 13(7): 1580-1588.
2 MÉDIGUE C, KRIN E, PASCAL G, et al. Coping with cold: The genome of the versatile marine Antarctica bacteriumTAC125[J]. Genome Research, 2005, 15(10): 1325-1335.
3 METHÉ B A, NELSON K E, DEMING J W, et al. The psychrophilic lifestyle as revealed by the genome sequence of34H through genomic and proteomic analyses[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(31): 10913-10918.
4 LIAO L, SU S, ZHAO B, et al. Biosynthetic potential of a novel Antarctic actinobacteriumZS314T revealed by genomic data mining and pigment characterization[J]. Marine Drugs, 2019, 17(7): 388-402.
5 Vollmers J, Voget S, Dietrich S, et al. Poles apart: Arctic and Antarcticstrains share high genome plasticity and a new type of xanthorhodopsin[J]. Plos One, 2013, 8(5): e63422.
6 Yin Y, Yue G, Gao Q, et al. Genome sequence ofsp. nov., a sea ice bacterium isolated from tundra soil[J]. Journal of Bacteriology, 2012, 194(23): 6688-6688.
7 Baker J, Riester C, Skinner B, et al. Genome sequence ofANT.BRT; a psychrophilic purple nonsulfur bacterium from an Antarctic microbial mat[J]. Microorganisms, 2017, 5(1): 1-16.
8 Liao L, Liu C, Zeng Y, et al. Multipartite genomes and the sRNome in response to temperature stress of an ArcticBSW20308[J]. Environmental Microbiology, 2019, 21(1): 272-285.
9 Cid F P, Maruyama F, Murase K, et al. Draft genome sequences of bacteria isolated from thephyllosphere[J]. Extremophiles, 2018, 22(3):537-552.
10 Zhao B, Liao L, Yu Y, et al. Complete genome ofsp. P6-10-X1 isolated from deep-sea sediments of the Southern Ocean[J]. Marine Genomics, 2017, 35: 27-29.
11 Yu Y, Zhao B, Wang L, et al. Complete genome of, strain NOCA502F isolated from sediment of the Arctic Ocean[J]. Marine Genomics, 2017, 34: 27-29.
12 Liao L, Su S, Yu Y, et al. Complete genome and data mining ofsp. A1–2 isolated from the Southern Ocean[J]. Marine Genomics, 2019, 45: 5-7.
13 Maida I, Fondi M, Papaleo M C, et al. Phenotypic and genomic characterization of the Antarctic bacteriumsp. CAL575, a producer of antimicrobial compounds[J]. Extremophiles, 2014, 18(1): 35-49.
14 Fondi M, Orlandini V, Maida I, et al. Draft genome sequence of the volatile organic compound-producing Antarctic bacteriumsp. strain TB23, able to inhibit cystic fibrosis pathogens belonging to thecomplex[J]. Journal of Bacteriology, 2012, 194(22): 6334-6335.
15 Fondi M, Orlandini V, Perrin E, et al. Draft genomes of three Antarcticstrains producing antimicrobial compounds againstcomplex, opportunistic human pathogens[J]. Marine Genomics, 2014, 13: 37-38.
16 Kube M, Chernikova T N, Alramahi Y, et al. Genome sequence and functional genomic analysis of the oil-degrading bacterium[J]. Nature Communications, 2013, 4(2): 2156.
17 Gentile G, Bonsignore M, Santisi S, et al. Biodegradation potentiality of psychrophilic bacterial strainRB-8T[J]. Marine Pollution Bulletin, 2016, 105(1): 125-130.
18 Dong C, Chen X, Xie Y, et al. Complete genome sequence ofR6-15, an obligate hydrocarbonoclastic marine bacterium from the Arctic Ocean[J]. Standards in Genomic Sciences, 2014, 9(3): 893-901.
19 Su S, Liao L, YU Y, et al. Genomic data mining of an Antarctic deep-sea actinobacterium,P3-3-X1[J]. Marine Genomics, 2019, DOI:10.1016/j.margen.2019.04.009
20 OH T J, HAN S R, KANG S, et al. Complete genome sequence of the xylan-degradingsp. strain PAMC26640 isolated from an Arctic lichen[J]. Journal of Biotechnology, 2016, 227: 23-24.
CURRENT STATUS AND PERSPECTIVES ON GENOME MINING OF POLAR MICROORGANISMS
Liao Li, Chen Bo
(Polar Research Institute of China, Shanghai 200136, China)
The rapid development of sequencing technology has greatly advanced research on microbial genomics. Although the initiation of polar microorganism genome sequencing was relatively late, it has become an important research area in the field of polar microbiology. The genetic resources of polar microorganisms hold strategic significance and social benefits. However, in-depth investigations of polar microbial genomes and genetic resources are quite limited, and the real power of genome sequencing has not been fully utilized. In this review, we summarize the current progress and research directions of polar microbial genomics, and analyze the shortcomings of current research and potential of genome mining. Accordingly, we propose key research directions requiring particular attention and breakthroughs. Overall, enhancing the mining of genetic resources from polar microorganisms provides more possibilities for future biotechnology.
polar microorganisms, genome, genetic resources, genomic mining
2019年7月收到来稿, 2019年8月收到修改稿
中国工程院战略研究项目(2018-ZD-08)资助
廖丽, 女, 1984年生。副研究员, 主要从事极地微生物研究。E-mail: liaoli@pric.org.cn
10. 13679/j.jdyj.20190034

