达氏鲟自噬基因MAP1LC3B克隆及其组织表达分析
胡伟 许巧情 郭慧芝 李由申 韩盼盼 袁汉文 张书环 陈敦学


摘要:【目的】掌握达氏鲟微管相关蛋白1A/1B轻链3B基因(MAP1LC3B)的表达模式,为后续研究MAP1LC3B基因功能及揭示达氏鲟的抗病分子机制提供理论依据。【方法】采用RACE技术从达氏鲟组织中克隆MAP1LC3B基因,通过ProtParam、TMpred、Phyre2及Singal 4.1等在线软件进行生物信息学分析,同时借助实时荧光定量PCR检测MAP1LC3B基因在达氏鲟不同组织中的表达情况。【结果】达氏鲟MAP1LC3B基因cDNA序列全长2208 bp,包含378 bp的开放阅读框(ORF)、202 bp的5'端非编码区(5'-UTR)和1628 bp的3'端非编码区(3'-UTR),编码125个氨基酸。MAP1LC3B氨基酸序列含有GABARAP泛素结构域、Agt7结合位点、脂化位点和维管束蛋白结合位点。达氏鲟MAP1LC3B蛋白由18种氨基酸组成,其中谷氨酸(Glu)含量最高(占9.6%)、丙氨酸(Ala)含量最低(占1.6%);蛋白分子量为14743.01 Da,理论等电点(pI)为8.92,不稳定系数为65.12,总平均亲水性为-0.484,是一种不稳定的疏水性蛋白,且不存在跨膜结构,也无信号肽序列。MAP1LC3B基因在达氏鲟鳃、脑、皮肤、头肾、前肾、脾脏、中肾、心脏和肠道等组织中均有表达,以在鳃和肠道组织中的相对表达量较高,显著高于在其他组织中的相对表达量(P<0.05)。【结论】达氏鲟MAP1LC3B基因具有较高的保守性,在鳃和肠道黏膜免疫组织中高表达,表明自噬参与达氏鲟黏膜免疫过程,调节机体免疫反应。
关键词: 达氏鲟;自噬;微管相关蛋白1A/1B轻链3B(MAP1LC3B);GABARAP泛素结构域;抗病机制
中图分类号: S965.215 文獻标志码: A 文章编号:2095-1191(2020)02-0445-08
Cloning and expression of autophagy-related gene MAP1LC3B in Dabrys sturgeon(Acipenser dabryanus)
HU Wei1,2, XU Qiao-qing2, GUO Hui-zhi2, LI You-shen2, HAN Pan-pan2,
YUAN Han-wen2*, ZHANG Shu-huan1*, CHEN Dun-xue3
(1Yangtze River Fisheries Research Institute, Chinese Academy of Fishery Sciences/Key Laboratory of Freshwater Biodiversity Conservation, Ministry of Agriculture and Rural Affairs, Wuhan 430223, China; 2School of Animal Science, Yangtze University, Jingzhou, Hubei 434020, China; 3School of Animal Science,
Guizhou University, Guiyang 550025, China)
Abstract:【Objective】The purpose of present study was to clone microtubule-associated protein 1-light chain 3B gene(MAP1LC3B) from Dabrys sturgeon(Acipenser dabryanus) and analyze its tissue expression, and lay the foundation forstudying the function of MAP1LC3B gene and revealing the molecular mechanism of disease resistance of Dabrys sturgeon. 【Method】The MAP1LC3B gene from A. dabryanus tissue was cloned by RACE technique and analyzed by online bioinformatics softwares ProtParam,Tmpred,Phyre2, Singal 4.1. The expression of MAP1LC3B gene in A. dabryanus tissues was analyzed by real-time fluorescent quantitative PCR(RT-PCR) technique. 【Result】The full-length ofMAP1LC3B gene of A. dabryanus was 2208 bp. Its open reading frame(ORF) region was 378 bp, in addition, the 5' noncoding region(UTR) and 3' UTR were 202 bp and 1628 bp respectively, encoded 125 amino acids. MAP1LC3B amino acid sequence contained GABARAP domain, Agt7 binding sites, lipidation sites and tubulin binding sites. MAP1LC3B protein consisted of 18 amino acids with highest proportion of glutamic acid(Glu)(9.6%) and lowest proportion of alanine(Ala)(1.6%). Its molecular mass(MM)was 14743.01 Da and the the oretical isoelectric point(pI) was 8.92. Its instability coefficient was 65.12, and the total average hydrophilicity was -0.484, which was an unstable hydrophobic protein. In addition, there was no transmembrane structure and signal peptide sequence. The expression of MAP1LC3B gene was detected in the tissues including gill, brain, skin, head kidney, forekidney, spleen, kidney, heart and intestinal tissues, and the expression of MAP1LC3B was high in the gill and intestinal tissues, and was significantly higher than in other tissues(P<0.05). 【Conclusion】MAP1LC3B gene is conservative, its high expression is found in gill and intestinal mucosal immune tissues, suggesting that MAP1LC3B gene may be related to immunity, and participate in the mucosal immunity to regulate the immune response of A. dabryanu.
Key words: Acipenser dabryanus; autophagy; microtubule-associated protein 1-light chain 3B(MAP1LC3B); GABARAP domain; resistance mechanism
Foundation item: National Natural Science Foundation of China(31460689); Open Project of Key Lab of Freshwater Biodiversity Conservation, Ministry of Agriculture and Rural Affairs(LFBC0905); Joint Funds of Science and Technology Department of Guizhou Province(Qiankehe LH〔2016〕7455)
0 引言
【研究意义】自噬(Autophagy)是生物在进化过程中形成的一种保守机制,能通过自噬体双层膜将胞内降解的蛋白质及各种细胞器物质包裹,随后运输至溶酶体进行分解并再利用(Baek and Kim,2017;李建华等,2018)。自噬不仅能维持细胞和生物机体的稳态平衡,还发挥着抵抗病原体感染及保护细胞的作用,其作用机制可能是通过抑制病原体的复制和分泌,激活先天性免疫及获得性免疫而降解病原体蛋白(Richetta and Faure,2013)。微管相关蛋白1A/1B轻链3B(Microtubule-associated protein 1-light chain 3B,MAP1LC3B,也称LC3B)是检测自噬诱导发生较可靠的标志性分子(Mizushima et al.,2010;Ktistakis,2015;孙大伟等,2018),为哺乳类动物同源酵母Atg8,具有两种亚型:一种是存在于细胞质中其分子量为16~18 kD的LC3B-I,另一种是与自噬体膜结合其分子量为14~16 kD的LC3B-II(Wang et al.,2016)。LC3B-II是通过多步翻译后修饰过程所产生,其过程包括新合成的MAP1LC3蛋白C末端残基(120Gly)进行蛋白水解裂解(Yabu et al.,2012)。自噬在鱼体抗细菌和病毒感染的过程中发挥重要作用(Wang et al.,2016),诱导自噬能增强机体抵抗病原的能力。因此,明确MAP1LC3B基因在鱼体内的表达情况,有助于揭示鱼类的抗病分子机制。【前人研究进展】在哺乳类动物中,LC3基因首先从小鼠脑组织中克隆获得(Wu et al.,2006),在人类中具有3种亚型(MAP1LC3A、MAP1LC3B和MAP1LC3C),在小鼠中只有MAP1LC3A和MAP1LC3B。MAP1-LC3A和MAP1LC3B参与自噬过程,而MAP1LC3C的功能尚不清楚(Dhingra et al.,2018b)。Kim等(2013)从猪的颗粒细胞中克隆出MAP1LC3A基因,并证实掌握其表达规律有助于揭示小型猪囊状滤泡发育的独特性。此外,有研究报道MAP1LC3B能维持视网膜色素上皮细胞(RPE)内脂质稳态,敲除该基因可致使RPE中吞噬体聚集及脂肪酸氧化功能降低(Dhingra et al.,2018a)。LC3除了指引干扰素诱导的GTP酶靶向抑制鼠诺如病毒复制(Biering et al.,2017)外,其过表达还能通过增加小鼠α7型神经烟碱胆碱能受体(α7nAchR)表达和自噬活性而降低Aβ神经毒素(Hung et al.,2015)。目前,在鱼类中已发现两种类型LC3基因(LC3A和LC3B),但相对于其他高等脊椎动物而言其研究较滞后。Seiliez等(2010)从虹鳟(Oncorhynchus mykiss)肌肉中克隆获得LC3B基因,并证实饥饿14 d能诱导LC3B基因上调表达。Ganesan等(2014)从斑马鱼(Danio rerio)中分别克隆获得LC3A基因和LC3B基因,并分析其在胚胎发育过程中的表达情况,为阿尔茨海默氏病(Alzheimer?s disease)的检测提供了基础数据。此后,相继有学者从其他鱼类中克隆出LC3基因。Wei等(2017)成功克隆出黄颡鱼(Pelteobagrus fulvidraco)的LC3A基因和LC3B基因,并研究LC3在锌胁迫后的响应情况;Xu等(2018)从团头鲂(Megalobrama amblycephala)中克隆获得LC3B基因,并探讨高糖胁迫后对其表达的影响。【本研究切入点】达氏鲟(Acipenser dabryanus)是我国特有的淡水性鱼类,主要分布在长江上游和部分支流水域(岳华梅等,2018),但由于人类活动(水电工程建设、航运、工业水体污染及人为过度捕捞等)频繁加剧,导致该物种已处于极度濒危状态,于1989年被列入一级保护动物白皮书,同时被列入世界自然保护联盟(IUCN)世界濒危动物红色名录(Zhang et al.,2013)。近年来发现达氏鲟除了易感白鲟虹彩病毒(WSIV)、白鲟疱疹病毒-1,2(WSHV-1,2)及擬铲鲟虹彩病毒(SSIV)等病毒外(Xu et al.,2019),还受迟缓爱德华氏菌(Edwardsiella tarda)和维氏气单胞菌(Aeromonas hydrophil)等致病菌的威胁(Yang et al.,2017;刘亚等,2018)。自噬在抗病原体感染过程中发挥积极作用,但目前有关自噬诱导发生标志性分子MAP1LC3B基因的研究主要集中在人类等高等脊椎动物和部分鱼类上,在达氏鲟上尚无报道。【拟解决的关键问题】从达氏鲟组织中克隆MAP1LC3B基因,并对其进行生物信息学分析,通过实时荧光定量PCR检测MAP1LC3B基因在不同组织/器官中的表达情况,掌握其表达变化规律,为后续研究MAP1LC3B基因功能及揭示达氏鲟的抗病分子机制提供理论依据。
1 材料与方法
1. 1 试验材料
供试达氏鲟为人工繁殖子代(2龄),其体重1.0~1.5 kg/尾,取自中国水产科学研究院长江水产研究所太湖试验场(中国荆州),试验前在实验室循环养殖系统中暂养2周。实验室循环养殖系统水温(20±3)℃,投喂频率2次/天,且饱食投喂。暂养结束后立即取样,取样前饥饿24 h,以MS-222(10 mg/L)麻醉后在冰上迅速取出其鳃、脑、皮肤、头肾、脾脏和中肾等组织样品,液氮速冻后,-80 ℃保存备用。TRIzol? Reagent购自Invitrogen公司,反转录试剂盒(Revert-AidTM First Stand cDNA)购自Fermentas公司,大肠杆菌(Escherichia coli)DH5α感受态细胞购自北京全式金生物技术有限公司,pMD18-T载体和实时荧光定量PCR相关试剂盒购自TaKaRa公司。
1. 2 达氏鲟MAP1LC3B基因克隆
1. 2. 1 总RNA提取及cDNA合成 达氏鲟组织用液氮研磨成粉末状后加入1 mL TRIzol进行处理,充分溶解提取达氏鲟组织总RNA,提取步骤参照Hu等(2017)的研究方法。cDNA合成严格按照Revert-AidTM First Stand cDNA试剂盒说明进行操作,-20 ℃保存备用。
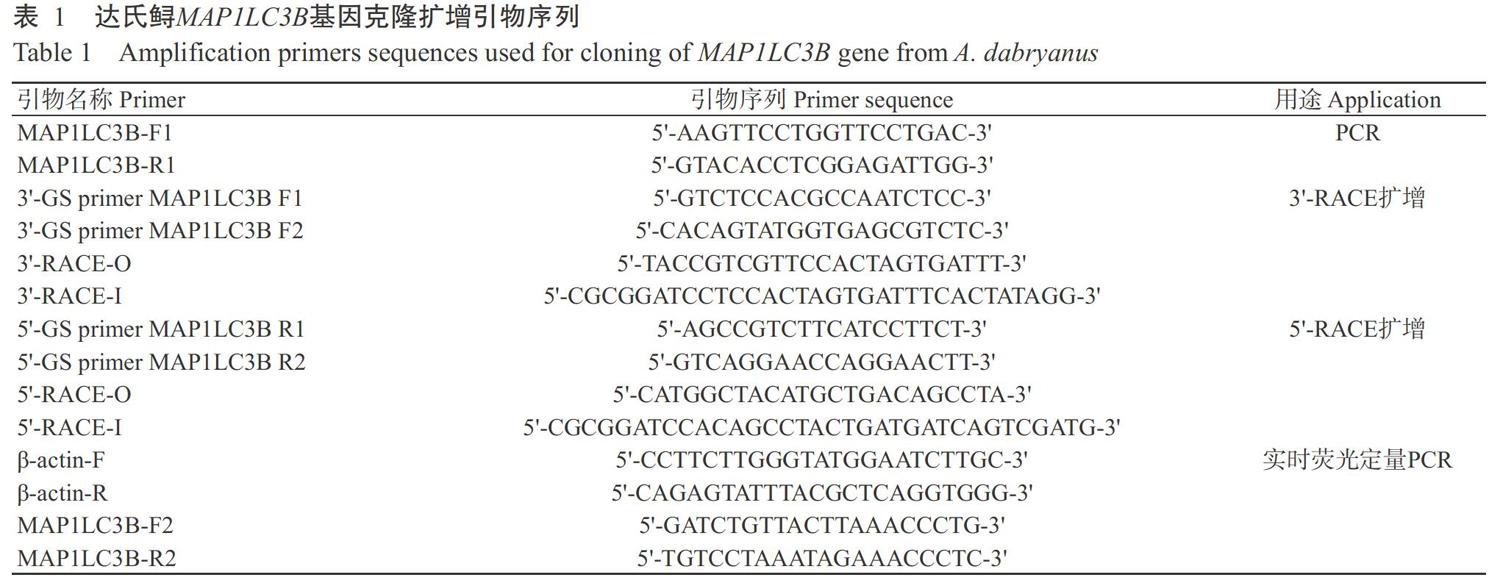
1. 2. 2 达氏鲟MAP1LC3B基因全长cDNA克隆及序列分析 搜索达氏鲟脾脏转录组数据得到MAP1-LC3B基因的部分序列信息,通过NCBI在线进行BLAST比对分析后,设计引物进行扩增验证。基于获得的cDNA保守序列,按照RACE试剂盒说明对MAP1LC3B基因的5'和3'端进行扩增,扩增引物见表1。
采用DNASTAR将RACE扩增获得的序列片段进行拼接,以获得MAP1LC3B基因全长cDNA序列,并通过NCBI中的BLAST(http://blast.ncbi.nlm.nih.gov/)确定拼接序列的正确性。利用NCBI网站的ORF Finder(https://www. ncbi.nlm.nih.gov/orffin-der/)查找MAP1LC3B基因的开放阅读框(ORF);使用MEGA 5.2的邻接法(Neighbor-joining,NJ)构建系统发育进化树,设定Bootstrap为1000。利用ProtParam对MAP1LC3B蛋白基本理化特性进行预测(https://web.expasy.org/protparam/),通过TMpred对MAP1LC3B蛋白跨膜结构域进行预测(http://www.cbs.dtu.dk/services/TMHMM/#opennewwindow),借助Phyre2对MAP1LC3B蛋白三级结构进行预测(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi? id=index),并以Singal 4.1预测其信号肽,最后運用ClustalW进行序列比对和同源性分析。
1. 2. 3 达氏鲟MAP1LC3B基因组织表达分析 选取4尾规格一致的达氏鲟(4个生物学重复),每个生物学重复设定3个技术重复。达氏鲟MAP1LC3B基因组织表达测定参考Hu等(2017)的研究方法,具体操作步骤:根据获得的达氏鲟MAP1LC3B基因序列,利用Primer 5.0设计实时荧光定量PCR扩增引物MAP1LC3B-F2和MAP1LC3B-R2(表1),以β-actin基因为内参基因,检测MAP1LC3B基因在达氏鲟不同组织(鳃、脑、皮肤、头肾、前肾、脾脏、中肾、心脏和肠道)中的相对表达量。反应体系25.0 μL:SYBR? Fast qPCR MasterMix II 20.0 μL,cDNA模板4.0 μL,上、下游引物各0.5 μL。扩增程序:95 ℃预变性5 min;95 ℃ 15 s,60 ℃ 30 s,72 ℃ 20 s,进行40个循环。使用2-△△Ct法换算MAP1LC3B基因在不同组织中的相对表达量,再以SPSS 17.0进行差异显著性分析。
2 结果与分析
2.1 达氏鲟MAP1LC3B基因全长cDNA序列及其推导氨基酸结构
RACE扩增获得的序列片段采用DNASTAR进行拼接,获得达氏鲟MAP1LC3B基因全长cDNA序列,提交至GenBank其序列号为MK749408。达氏鲟MAP1LC3B基因cDNA序列全长2208 bp,包含378 bp的ORF、202 bp的5'端非编码区(5'-UTR)和1628 bp的3'端非编码区(3'-UTR)。利用SMART和NCBI在线工具预测MAP1LC3B氨基酸结构域,发现其具有GABA-A受体关联蛋白样蛋白(GABARAP)泛素结构域、Agt7结合位点、脂化位点和维管束蛋白结合位点(图1)。通过ProtParam预测发现,达氏鲟MAP1LC3B基因编码125个氨基酸,由18种氨基酸组成,其中谷氨酸(Glu)含量最高(占9.6%),丙氨酸(Ala)含量最低(占1.6%)。带正电氨基酸残基数为19种,带负电氨基酸残基数为17种。编码蛋白的分子量为14743.01 Da,理论等电点(pI)为8.92,原子组成为C660H1058N182O192S4。蛋白不稳定系数为65.12,是一种不稳定蛋白。蛋白氨基酸残基总平均亲水性为-0.484,即为疏水性蛋白。
采用TMpred对达氏鲟MAP1LC3B蛋白跨膜结构域进行预测,结果表明其不存在跨膜结构(图2),即为胞内蛋白。通过SignalP 4.1预测分析达氏鲟MAP1LC3B蛋白氨基酸序列,未发现信号肽序列(图3),说明该蛋白为非分泌型蛋白。采用Phyre2对MAP1LC3B蛋白三级结构进行预测,结果(图4)显示达氏鲟MAP1LC3B蛋白三级结构与人类MAP1LC3B蛋白三级结构相似,而与斑马鱼MAP1LC3B蛋白三级结构的差异较明显。
2. 2 达氏鲟MAP1LC3B氨基酸序列比对及系统发育进化分析
利用ClustalW对达氏鲟MAP1LC3B氨基酸序列与GenBank已公布参考物种MAP1LC3B氨基酸序列进行同源性比对分析,得知达氏鲟与各参考物种的MAP1LC3B氨基酸序列同源性分别为:斑马鱼(D. rerio)96.7%,非洲爪蛙(Xenopus tropicalis)95.2%,人类(Homo sapiens)94.4%,草鱼(Ctenopharyngodon idella)93.6%,鲤鱼(Cyprinus carpio)92.8%,褐家鼠(Rattus norvegicus)92.0%,虹鳟(Oncorhynchus mykiss)91.2%,青鳉(Oryzias latipes)91.2%(表2)。MAP1LC3B氨基酸序列的多重比对分析结果表明,达氏鲟MAP1LC3B氨基酸序列中含有一个GABARAP泛素结构域和C端甘氨酸残基,同时具有维管束蛋白结合位点、Atg4识别位点、Atg7结合位点及脂化位点(图5)。基于MAP1LC3B氨基酸序列同源性,采用MEGA 5.2构建系统发育进化树(图6),发现哺乳类动物聚为一支,而达氏鲟与其他鱼类聚为另一支,表明其与鱼类的亲缘关系较近。
2. 3 MAP1LC3B基因在达氏鲟不同组织中的表达特征
采用实时荧光定量PCR检测MAP1LC3B基因在达氏鲟鳃、脑、皮肤、头肾、前肾、脾脏、中肾、心脏和肠道等组织中的表达情况,结果表明,MAP1LC3B基因在各检测组织中均有表达(图7),其中,以在鳃和肠道组织中的相对表达量较高,显著高于在其他组织中的相对表达量(P<0.05),而在脑组织中的相对表达量最低。
3 讨论
自噬在生理学上是一个动态过程,包括自噬体形成、自噬体与溶酶体融合及自噬体内包含物降解(Galluzzi et al.,2014)。MAP1LC3B作为自噬体延伸和成熟所必需的核心共轭系统,被Atg4蛋白酶水解暴露C端甘氨酸残基,并与磷脂酰乙醇胺结合形成定位于双膜胞质和腔面的LC3-II,待自噬体形成后LC3-II被水解(Belghit et al.,2013;Lee and Lee,2016)。LC3-I/LC3-II比值还能反映自噬体的数量(Lee and Lee,2016)。因此,MAP1LC3B在自噬过程中具有标志物作用(Behrends et al.,2010;刘晓健等,2018)。本研究通过RACE技术从达氏鲟组织中扩增获得的MAP1LC3B基因cDNA序列全长2208 bp,编码125个氨基酸,氨基酸序列中包括GABARAP泛素结构域、Agt7结合位点和C端区域甘氨酸残基,其中GABARAP泛素结构域由115个氨基酸组成,与在其他鱼类中的研究报道(Ganesan et al.,2014;Wei et al.,2017;Xu et al.,2018)一致,即GABARAP泛素结构域一般由114~119个氨基酸组成。此外,达氏鲟MAP1LC3B序列中含有结合纯化微管蛋白和微管的维管束蛋白结合位点,其在脊椎动物包括鱼类中也有发现(Yabu et al.,2012;Wei et al.,2017)。已有研究表明,MAP1LC3B氨基酸序列中80Phe和82Leu是Atg4的识别位点,49Tyr和50Leu则作为脂化的位点(Wei et al.,2017)。本研究也得出相似的结论,进一步印证MAP1LC3B基因具有较高的保守性。达氏鲟MAP1LC3B氨基酸序列与斑马鱼MAP1LC3B氨基酸序列的相似度最高(96.7%),说明达氏鲟与斑马鱼的亲缘关系较近。
MAP1LC3B基因在达氏鲟的鳃、脑、皮肤、头肾、前肾、脾脏、中肾、心脏和肠道等9个组织中均有表达,提示自噬可能发生在不同的组织中且参与代谢调控(Mizushima and Komatsu,2011;Xu et al.,2019)。MAP1LC3B基因在达氏鲟组织中的表达模式与其在其他物种的表达模式(He et al.,2003;Wu et al.,2006;Ganesan et al.,2014;Wei et al.,2017;Xu et al.,2018)相似。Wei等(2017)研究发现,MAP1LC3B基因在黄颡鱼的心脏、肾脏、鳃、脾脏、卵巢、肠道、肠系膜脂肪和肝臟等组织中均有表达,且以在脑组织中的相对表达量最高,暗示其可能主要参与神经细胞的形成和发育(Halpain and Dehmelt,2006)。Xu等(2018)研究报道,在团头鲂的肝胰脏、脂肪、脑、肌肉、心脏、皮肤、肠道和鳃等组织中均能检出MAP1LC3B基因,以肝胰脏组织中的表达水平最高,可能与肝脏作为重要的代谢器官参与糖、脂等代谢过程有关(Liu et al.,2017)。本研究发现,MAP1LC3B基因在达氏鲟鳃和肠道组织中的相对表达量较高,而在脑组织中的相对表达量最低,与Wei等(2017)、Xu等(2018)的研究结果存在差异,究其原因可能是MAP1LC3B基因表达存在物种特异性。
4 结论
达氏鲟MAP1LC3B基因具有较高的保守性,在鳃和肠道黏膜免疫组织中高表达,表明自噬参与达氏鲟黏膜免疫过程,调节机体免疫反应。
参考文献:
李建华,王润泽,李伟民,徐冶. 2018. 微管相关蛋白与自噬的研究进展[J]. 吉林医药学院学报,39(4):285-290. [Li J H,Wang R Z,Li W M,Xu Z. 2018. Advances in microtubule-associated proteins and autophagy[J]. Journal of Jilin Medical College,39(4):285-290.]
刘晓健,陈睿琦,胡峰,张秀冰,曾庆红,王艺,崔秀玲. 2018. 糖尿病大鼠心肌组织中自噬基因微管相关蛋白1轻链3的表达[J]. 中国老年学杂志,38(14):3464-3466. [Liu X J,Chen R Q,Hu F,Zhang X B,Zeng Q H,Wang Y,Cui X L. 2018. Expression of microtubule-associated protein 1 light chain 3 in myocardium of diabetic rats[J]. Chinese Journal of Gerontology,38(14):3464-3466.]
刘亚,杨锐,陈叶雨,宋明江,颜其贵,龚全,李强,杨焕超,赖见生. 2018. 达氏鲟维氏气单胞菌的分离鉴定及病理组织学观察[J]. 南方农业学报,49(6):1235-1241. [Liu Y,Yang R,Chen Y Y,Song M J,Yan Q G,Gong Q,Li Q,Yang H C, Lai J S. 2018. Isolation,identification of Aero-monas veronii in Acipenser dabryanus and its histological observations[J]. Journal of Southern Agriculture,49(6):1235-1241.]
孙大伟,冯丽莎,高青,索艳荣. 2018. 基于自噬基因Beclin-1与LC3探讨丹参素对H/R大鼠心肌细胞ATP5G1表达的影响[J]. 世界中西医结合杂志,13(11):1493-1497. [Sun D W,Feng L S,Gao Q,Suo Y R. 2018. Explore the protective effect of the tanshinol on cardiomyoytes ATP5G1 in the rats with H/R based on autophagy gene Beclin-1 and LC3[J]. World Journal of Integrated Traditional and Wes-tern Medicine,13(11):1493-1497.]
岳華梅,叶欢,阮瑞,刘志刚,李创举. 2018. 达氏鲟gpr54基因的克隆及Kisspeptin注射对其表达的影响[J]. 水生生物学报,42(5):896-902. [Yue H M,Ye H,Ruan R,Liu Z G,Li C J. 2018. Molecular cloning of gpr54 genes and effects of kisspeptin intraperitoneal injection on its expression in Acipenser dabryanus[J]. Acta Hydrobiologica Sinica,42(5):896-902.]
Baek S H,Kim K I. 2017. Epigenetic control of autophagy:Nuclear events gain more attention[J]. Molecular Cell,65(5):781-785.
Behrends C,Sowa M E,Gygi S P,Harper J W. 2010. Network organization of the human autophagy system[J]. Nature,466(7302):68-76.
Belghit I,Panserat S,Sadoul B,Dias K,Skiba-Cassy S,Seiliez I. 2013. Macronutrient composition of the diet affects the feeding-mediated down regulation of autophagy in muscle of rainbow trout(O. mykiss)[J]. PLoS One,8(9):e74308.
Biering S B,Choi J,Halstrom R A,Brown H M,Beatty W L,Lee S,McCune B T,Dominici E,Williams L E,Orchard R C,Wilen C B,Yamamoto M,Coers J,Taylor G A,Hwang S. 2017. Viral replication complexes are targeted by LC3-guided interferon-inducible GTPases[J]. Cell Host & Microbe,22(1):74-85.
Dhingra A,Alexander D,Reyes-Reveles J,Sharp R,Boesze-Battaglia K. 2018a. Microtubule-associated protein 1 light chain 3(LC3) isoforms in RPE and retina[J]. Advances in Experimental Medicine and Biology,1074:609-616.
Dhingra A,Bell B A,Peachey N S,Daniele L L,Reyes-Re-veles J,Sharp R C,Jun B,Bazan N G,Sparrow J R,Kim H J,Philp N J,Boesze-Battaglia K. 2018b. Microtubule-associated protein 1 light chain 3B,(LC3B) is necessary to maintain lipid-mediated homeostasis in the retinal pigment epithelium[J]. Frontiers in Cellular Neuroscience,12:351. doi: 10.3389/fncel.2018.00351.
Galluzzi L,Pietrocola F,Levine B,Kroemer G. 2014. Metabolic control of autophagy[J]. Cell,159(6):1263-1276.
Ganesan S,Nik S H M,Newman M,Lardelli M. 2014. Identification and expression analysis of the zebrafish orthologues of the mammalian MAP1LC3 gene family[J]. Experimental Cell Research,328(1):228-237.
Halpain S,Dehmelt L. 2006. The MAP1 family of microtubule-associated proteins[J]. Genome Biology,7(6):224.
He H,Dang Y,Dai F,Guo Z,Wu J,She X,Pei Y,Chen Y,Ling W,Wu C,Zhao S,Liu J Q,Yu L. 2003. Post-translational modifications of three members of the human MAP1LC3 family and detection of a novel type of modification for MAP1LC3B[J]. The Journal of Biological Chemistry,278(31):29278-29287.
Hu W,Mai K S,Luo Z,Zheng J L,Chen Q L,Pan Y X. 2017. Effect of waterborne copper on lipid metabolism in hepatopancreas and muscle of grass carp Ctenopharyngodon idella[J]. Aquaculture Research,48(4):1458-1468.
Hung S Y,Huang W P,Liou H C,Fu W M. 2015. LC3 overexpression reduces Aβ neurotoxicity through increasing α7nAchR expression and autophagic activity in neurons and mice[J]. Neuropharmacology,93:243-251.
Kim S H,Hwang S Y,Min K S,J T Yoon. 2013. Molecular cloning and expression analyses of porcine MAP1LC3A in the granulosa cells of normal and miniature pig[J]. Reproductive Biology and Endocrinology,11:8.
Ktistakis N T. 2015. Monitoring the localization of MAP1LC3B by indirect immunofluorescence[J]. Cold Spring Harbor Protocols,(8):751-755.
Lee Y K,Lee J A. 2016. Role of the mammalian ATG8/LC3 family in autophagy:Differential and compensatory roles in the spatiotemporal regulation of autophagy[J]. BMB Reports,49(8):424-430.
Liu Q N,Xin Z Z,Zhang D Z,Jiang S H,Chai X Y,Wang Z F,Li C F,Zhou C L,Tang B P. 2017. cDNA cloning and expression analysis of a hepcidin gene from yellow catfish Pelteobagrus fulvidraco(Siluriformes:Bagridae)[J]. Fish & Shellfish Immunology,60:247-254.
Mizushima N,Komatsu M. 2011. Autophagy:Renovation of cells and tissues[J]. Cell,147(4):728-741.
Mizushima N,Yoshimori T,Levine B. 2010. Methods in mammalian autophagy research[J]. Cell,140(3):313-326.
Richetta C,Faure M. 2013. Autophagy in antiviral innate immunity[J]. Cellular Microbiology,15(3):368-376.
Seiliez I,Gutierrez J,Salmerón C,Skiba-Cassy S,Chauvin C,Dias K,Kaushik S,Tesseraud S,Panserat S. 2010. An in vivo and in vitro assessment of autophagy-related gene expression in muscle of rainbow trout(Oncorhynchus mykiss)[J]. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,157(3):258-266.
Wang Y,Chen N,Hegazy A M,Liu X,Wu Z,Liu X,Zhao L,Qin Q,Lan J,Lin L. 2016. Autophagy induced by snakehead fish vesiculovirus inhibited its replication in SSN-1 cell line[J]. Fish & Shellfish Immunology,55:415-422.
Wei C C,Luo Z,Song Y F,Pan Y X,Wu K,You W J. 2017. Identification of autophagy related genes LC3 and ATG4 from yellow catfish Pelteobagrus fulvidraco and their transcriptional responses to waterborne and dietborne zinc exposure[J]. Chemosphere,175:228-238.
Wu J,Dang Y,Su W,Liu C,Ma H,Shan Y,Pei Y,Wan B,Guo J,Yu L. 2006. Molecular cloning and characterization of rat LC3A and LC3B—Two novel markers of autophagosome[J]. Biochemical and Biophysical Research Communications,339(1):437-442.
Xu Q Q,Luo K,Zhang S H,Gao W H,Zhang W B,Wei Q W. 2019. Sequence analysis and characterization of type I interferon and type II interferon from the critically endangered sturgeon species,A. dabryanus and A. sinensis[J]. Fish & Shellfish Immunology,84:390-403.
Xu W N,Chen D H,Liu W B,Xu J X,Yang S S. 2018. Molecular characterization of microtubule-associated protein 1-light chain 3B in Megalobrama amblycephala fed with high fat/berberine diets[J]. Journal of Applied Genetics,59(3):345-355.
Yabu T,Imamura S,Mizusawa N,Touhata K,Yamashita M. 2012. Induction of autophagy by amino acid starvation in fish cells[J]. Marine Biotechnology,14:491-501.
Yang R,Liu Y,Wang Y,Lei M K,Pan G Y,Wen J F,Gong Q,Ren L,Huang J,Wen X T,Cao S J,Huang Y,Huang X B,Ma X P,Han X F,Zhao Q,Wen Y P,Wu R,LinJ C,Yan Q G. 2017. Pathogenesis and pathological analysis of Edwardsiella tarda from Dabrys sturgeon (Acipen-ser dabryanus) in China[J]. Aquaculture,495:637-642.
Zhang S H,Luo H,Du H,Wang D Q,Wei Q W. 2013. Isolation and characterization of twenty-six microsatellite loci for the tetraploid fish Dabrys sturgeon(Acipenser dabryanus)[J]. Conservation Genetics Resources,5(2):409-412.

