多组学大数据在精准医学中的地位及应用
Zhifu SUN
(1 青岛大学医学部,山东 青岛 266071;2 Mayo Clinic,Rochester,Minnesota,U.S.A)
通过对基因突变、基因表达以及DNA修饰等信息的分析,临床医生能够对疾病进行更为精准的诊断和有效的防治。广义上,精准医学所应用的数据已扩展到像电子病历、医学影像资料和实验室检查结果等其他非基因组学数据,并同基因组学数据一起构成了精准医学的大数据[1]。目前临床最常用到的基因组学数据为胚系突变数据和肿瘤的体细胞突变数据,如携带BRAC1或BRAC2基因突变的健康人群可进行预防性乳房切除术以预防乳腺癌的发生,有EGFR突变的肺癌病人可进行酪氨酸激酶抑制剂(TKI)治疗等。尽管目前基因组学数据正在迅速发展,但是由于每种肿瘤中发生变异的基因数量庞大,许多疾病相关变异的临床意义也还不能进行明确的解释,只是被笼统地归为意义不明变异位点(VUSs),这极大地限制了基因组学信息的应用。更为复杂的是,DNA数据只是细胞内众多分子数据中的一种,还有DNA修饰(CpG甲基化)、组蛋白修饰、染色质开放区、基因转录(RNA或者miRNA表达)和蛋白质表达等多种分子数据,这些数据对于精准医学同样重要,统称为多组学数据。尽管很多数据还没有在临床上得到应用,但它们在解释意义不明位点、研究用于诊断的检测套餐、开发新的诊断和预后标记物、发现新治疗靶点等方面有着重要作用。在大数据时代,掌握如何使用这些资源对于现代医学实践和转化医学研究至关重要。
1 多组学数据的价值
人类生物学系统高度复杂,大量分子参与其中,且相互之间发生着多种多样的关系,因此仅研究其中一种分子会有很大的局限性,多组学数据的应用将会很大程度上解决这一问题。近年来,多个国内或国际大型合作项目产生了大量的多组学数据,研究取得的数据资源已作为公开资源向公众开放,这一举措改变了单分子研究的局限性。使用开放数据资源进行研究的首要优势在于,开放数据资源通常包含上千甚至上万个研究样本,可以确保能够检测到小到中等的基因效应,而如此大的样本量是任何一个实验室或研究机构无法达到的。即便是单一的肿瘤病种的研究,异质性也是研究者面临的巨大挑战。多而广的样本谱系将帮助研究者克服这一困难,进而进行更加全面和深入的研究,如在一组肿瘤病人中发现一种新突变,那么这一突变将有望成为新的治疗靶点。第二点优势是,许多开放数据库资源来源于多组学数据,包含像基因组学及表观基因组学数据等。通过数据库,研究者们可以对数据内部及数据间的关系进行整合分析。数据的整合分析虽然极具挑战性,但同时也会带来高回报。开放数据资源的第三点优势是数据库内资源可即时获取,因此可以快速生成假设并对其进行检验。基因组学数据的获取过程花费高、时间长,但研究人员通过挖掘开放数据资源中的多组学数据,在短时间内即可得到问题的答案。第四点优势是,小型实验的研究结果很可能存在潜在偏倚,因此有必要在开放数据资源中对结果进行验证,进而证实结果的有效性和通用性。第五点,近几年对于深层机器学习以及人工智能领域的关注日益增加,这也离不开开放的多组学数据资源的发展,因此开放数据资源不可或缺。
2 多组学数据库资源及其获取方法
开放的多组学数据库资源庞大,且数据量日渐增长。本文只能简单介绍一些研究人员常用或者容易获取的数据库,尤其是对临床实践指导作用比较大的数据库,主要有多组学数据库、罕见病或遗传性疾病相关突变数据库、群体变异数据库以及肿瘤体细胞突变数据库(表1)。这些数据库的资源并不是孤立的,而是互相关联的,或者一个数据库的数据来源于另外一个数据库。如肿瘤基因图谱(TCGA)数据库是目前最大的肿瘤多组学数据库,其中的突变数据是肿瘤体细胞突变图谱(COSMIC)和肿瘤突变位点临床解读数据库(CIVic)的重要组成部分。这些数据均来自于大样本量的临床研究并且可以作为更深层次的数据挖掘的基础。
多组学数据庞杂,包括原始数据、初步处理数据、高级筛选数据等多种不同处理水平数据。这些数据还与特定的参考基因组的版本有关(hg37版本或hg38版本)。根据用户的计算机及生物信息学技能以及使用目的不同,获取和使用数据的方法也有所不同。但几乎所有的基因组学数据都有其原始格式,这些原始数据可能来自于微阵列扫描仪或者下一代测序仪等。原始数据需要经过预处理才能够成为对终端用户有用的数据。由于在数据处理过程中存在多种选择,因此选择不同的分析路径可能会产生不同的结果。应用原始数据有助于不同来源数据的标准化或使其与内部生成的数据具有可比性(如对外来数据与内部产生的数据采用统一的序列匹配、采用序列比较工具来检测序列变异或突变,或者采用统一工具对基因表达进行定量和标准化处理)。然而原始数据数量庞大,往往需要专业生物信息人员先进行预处理。应用原始数据另一复杂之处在于它们包含可链接到研究入组人员的可识别的基因组信息。因此,大多数DNA变异原始数据,如单核苷酸多肽(SNP)阵列、全基因组数据或外显子测序数据是有使用权限的,需要得到特殊批准才可以访问和下载。如基因型和表型数据库(dbGAP,https://www.ncbi.nlm.nih.gov/gap/)以及欧洲基因表型数据库(EGA,https://ega-archive.org/)是最常使用的数据库。
对于数据分析师以及有经验的数据分析人员来说下载预处理数据是最为灵活的选择,比如DNA测序数据预处理产生的变异数据、RNA测序数据预处理产生的RNA表达数据、DNA甲基化数据预处理产生的GpG岛甲基化百分比数据等,使用者可以应用这些数据进行任何他们想做的分析。理解原始数据的处理过程、处理中使用了哪种标准化方法,并且找到与之匹配的临床数据以及样本数据是实现这种灵活选择的关键。这些数据均可以在相关资源网站上以文本文件的格式直接下载或者通过R语言或Python语言通过应用程序下载(表2)。R语言和Python语言是在生物信息学和机器学习方面最常用到的编程语言。对于没有编程技能的实验室人员来说,可以使用已经开发的基于网络的数据访问工具或者可视化的分析工具对原始数据进行分析。cBioportal和GEPIA2就是最常用、最方便的两个工具,用户只需点击鼠标就可进行各种分析。
3 多组学数据在精准医学中的应用
多组学数据在精准医学中的应用十分广泛,可用于任何临床学科及预防医学领域。以下用一些实例来说明基因组学的数据资源以及这些资源在医学领域中的应用。
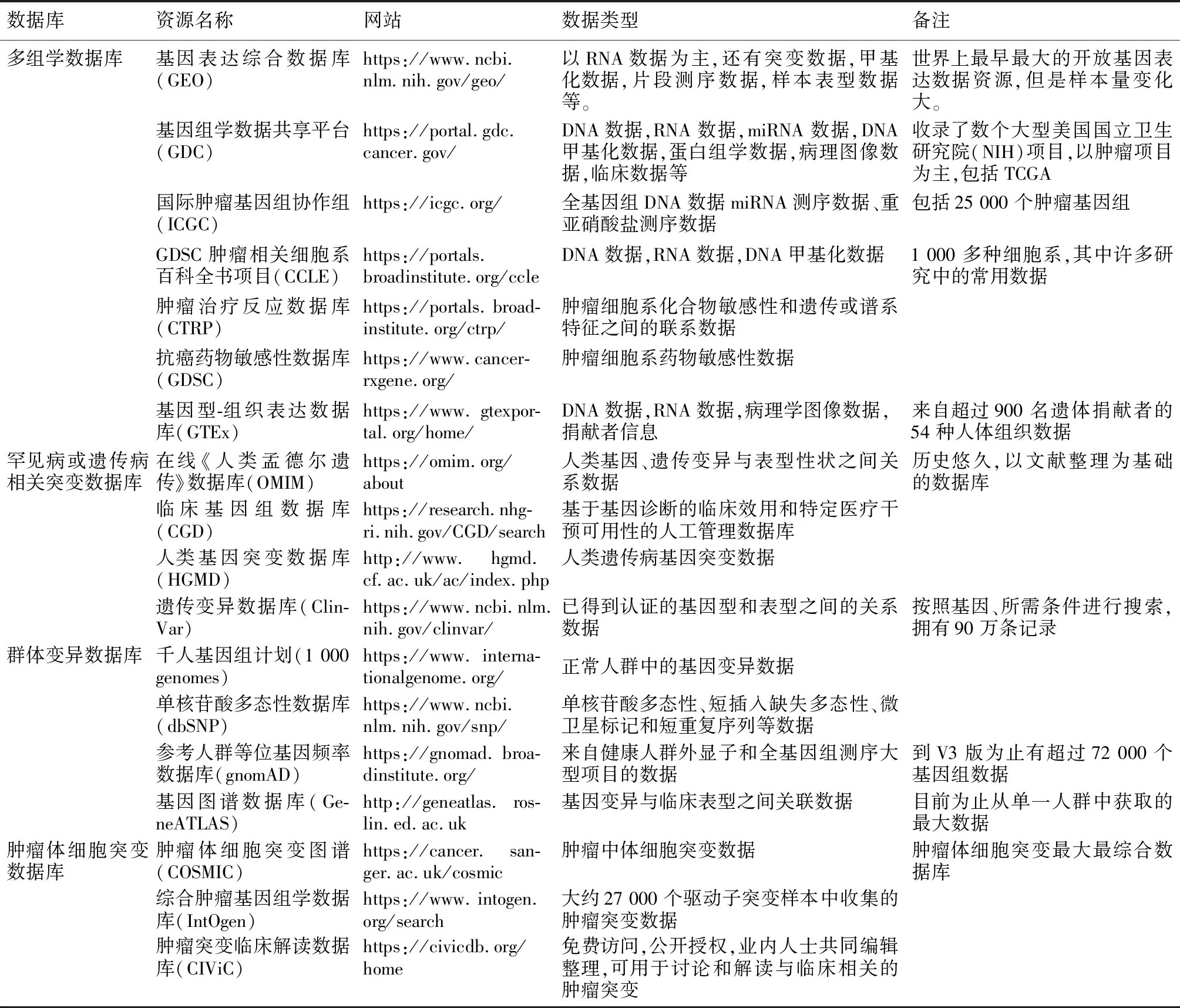
表1 主要基因组学数据库资源
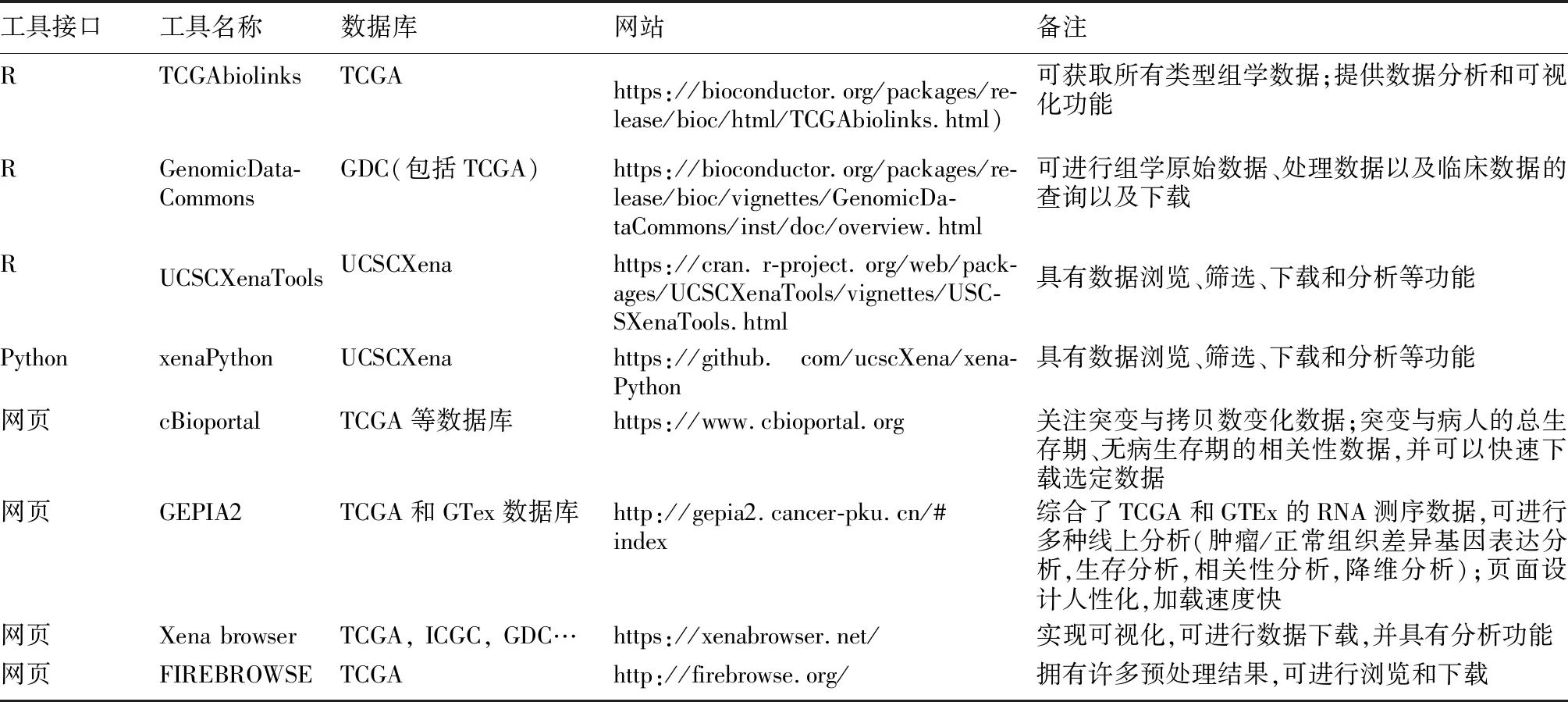
表2 访问开放数据库的常用工具
3.1 罕见遗传病相关突变在精准医学中的应用
据估计,从遗传角度讲,人类有超过10 000种疾病是单基因疾病,也就是说这些疾病是由于人类DNA的单个基因的单一错误引起的[2]。现已明确的与罕见遗传病相关的突变有6 528种(于2019年11月1日检索《人类孟德尔遗传》数据库(OMIM)的结果),但仍有很多突变没有被发现。对于这些致病原因不明的疾病,进行外显子或者全基因组测序是临床上常规的处理方法。进行大范围基因组测序通常会发现非常多的突变,但从中找出真正的致病突变并不容易,需要进行文献检索、罕见病数据库挖掘以及将病人的突变与数据库中报道的突变比对等一系列过程。通过基因组测序,许多疾病可以得到诊断。但如果检测发现该突变以往未见报道,则意味着该病人可能患有一种新疾病或者发现了另一个与某种疾病相关的致病突变。但是由于目前许多临床实验室的数据没有上传,所以罕见病数据库里数据并不集中。因此建立资源集中和数据共享的机制,将会有助于对未曾报道过的罕见病的诊断[3]。
3.2 多组学数据在预测人群风险变异中的应用
人群风险变异是指在人群中次等位基因频率(MAF)>1%的基因变异,它们是常见病或者复杂多基因病的主要病因,如2型糖尿病、高血压以及一些肿瘤等疾病。这些疾病并不是由单基因突变造成的,而是多基因突变以及与环境因素共同影响导致的。在过去的几十年里,已完成超过5 000例的全基因组关联分析(GWAS,https://www.ebi.ac.uk/gwas)。截至2018年,在GWAS目录中已经报道了71 673例突变-性状关联分析[4-5]。用户可以通过疾病名称、基因突变以及单核苷酸多肽编码(SNP ID)来进行搜索。但由于有些GWAS数据是来自于数年的多个研究,而且许多研究并没有足够的样本量,因此得出的关联关系可能不全面,从而导致GWAS搜索结果有时会差别较大。但令人高兴的是,一些大型生物标本库研究项目拥有了几十万甚至上百万样本量,并将研究所得的基因型-表型关系公之于众。例如有史以来最大的英国生物样本库项目(the UK biobank project)收集了452 264份志愿者的DNA样本,对778个性状和3 000万个基因组变异的关联性进行了研究。只需搜索疾病名称、基因名称、基因组变异或者位点等信息就可以在英国生物标本库项目(GeneATLAS,http://geneatlas.roslin.ed.ac.uk)上获取相应数据[6]。
但是GWAS结果在预测人群风险变异的应用中尚存在许多问题。①只有极少数有关联的变异或突变可以应用于临床,绝大多数关联没有发现临床应用价值。②GWAS研究中的大多数变异关联性是通过SNP阵列技术得到的,但是其所用SNP大部分是标签SNP,并不是与疾病或者性状相关的致病SNP。随着越来越多的GWAS研究方法转向外显子测序或者全基因组测序,这个问题可能会被解决;然而新平台同样会产生海量的SNP或者变异数据,从中寻找有意义的变异如“大海捞针”一般困难。超过95%的变异来自于基因组的非编码区,如何解释这些变异也是一项非常困难的工作。现在已经开发了一些软件工具对这些变异进行解释并同时进行数据优化。GWAVA是利用DNA元素百科全书计划(ENCODE)和GENCODE计划的非编码元素以及全基因组序列特性,如通过进化保守性以及GC含量,预测非编码区变异对基因功能的影响[7];结合注释依赖消耗评分系统(CADD)是另一个更全面的工具,其通过将自然选择中保存的变体与给定变异进行对比来进行评分。这个评分系统评价人类基因组中DNA变异和插入/缺失突变的有害程度[8]。尽管这些工具非常有用,但是GWAS结果应用于临床还有很长的路要走。
3.3 多组学数据在精准肿瘤学中的应用
应用基因组学信息指导肿瘤治疗是精准医学的核心,这一理念已被广泛认可并成功应用于临床。肿瘤的发生伴随着各种基因组畸变,产生肿瘤特异性靶向分子,在这些肿瘤治疗中,肿瘤标志成为其诊疗的主要靶点,此外肿瘤发生、发展、生长、转移过程中的众多靶点、信号传导通路以及异常的细胞周期、肿瘤微环境都是靶向药物的潜在治疗靶点。目前为止基因组学信息的临床应用仍仅局限于肿瘤DNA检测,如DNA突变、易位、插入/缺失或者肿瘤突变负荷(TMB)等的检测。肿瘤DNA检测常用于特定的基因和突变来增加选定序列的基因检测覆盖率,以避免许多可以解读的突变的漏检。其中很多检测的靶点是从TCGA这样的大型基因图谱计划中获得的。分析不同类型肿瘤的突变分布有助于鉴别出携带同种突变的其他肿瘤,携带同种突变的肿瘤患者可能会受益于相同的靶向治疗方案。
目前对于检测到的肿瘤突变如何解读以及临床应用的适应证还缺乏统一的标准。每个机构都有自己的测序平台及生物信息分析团队,对变异的解释也不尽相同。为了解决这一问题,专家们共同创建了肿瘤突变数据库(CIViC,https://civicdb.org/home)来帮助研究人员解读检测到的基因突变与肿瘤发生发展的相关性,以及对病人诊断、治疗、预后评估方面的应用价值[9]。
尽管精准医学强调的是个体化的医疗,但是知识的获取以及有意义突变的解读仍需要对大规模的人群数据进行分析,这就使得组学大数据显得尤为重要[10]。
3.4 多组学数据的转化医学研究
目前,对于基因变异以及变异对临床的影响还知之甚少,有很多的问题仍需进一步探索,因此基因组学大数据对开发诊断/预后标志物和发现新治疗靶点极为重要。开放数据库为我们提供了实现这些目标的资源。
利用数据库中的多组学数据在一系列肿瘤中进行单基因分析经常会出现这种问题:实验中发现了一个在某特定肿瘤中可能有临床意义的突变,但无法确定该突变在大数据中或者其他肿瘤中的临床意义如何。值得庆幸的是,现有的大型数据库以及一些在线资源可以很容易地解决这些问题。使用者无需掌握编程技巧,仅需一些基本的数据分析知识就可以利用这些开放资源进行数据分析,如通过检测发现,EGFR突变的肺癌病人可以使用酪氨酸激酶抑制剂进行治疗,那么携带EGFR突变的其他肿瘤病人会从酪氨酸激酶抑制剂靶向治疗中获益么?如果不能获益,是否可以找到其他的基因组变异来进行靶向治疗?通过对TCGA中11 000多种肿瘤进行cBioportal和GEPIA搜索,发现EGFR突变在其他肿瘤中是非常常见的,但突变功能域却各有不同,在脑肿瘤和其他器官的鳞状细胞癌中,EGFR扩增或表达增加提示病人预后差,这就需要使用不同的治疗策略来降低致癌基因表达。
肿瘤能否早期诊断决定着肿瘤病人的生存期长短以及能否制定合理治疗方案。大多数肿瘤还没有早期筛查手段,但一些肿瘤,如胰腺癌、卵巢癌和肺癌,如果不能早期诊断,后果将会是致命的。近几年来,以液体活检、无创血浆游离细胞、循环肿瘤细胞为基础的肿瘤检测技术已经取得了长足进展。几乎所有的基因学和表观基因学信息都可以作为这些检测的靶点,目前最常用的方法是体细胞突变的检测。研究发现,某些肿瘤携带许多特异性的突变,检测到一个基因突变或多个基因突变组合可以辅助这些肿瘤的确诊[11-13]。由于游离DNA中肿瘤DNA比例很低,通常选择感兴趣的基因区域(突变)进行测序来达到超高覆盖率,以避免目标基因突变的漏检。大型基因组数据库在帮助选择临床进行检测的突变位点上有重要作用。除此之外,突变检测还可用于治疗方案选择、治疗效果评估以及疾病预后评估等方面[14-15]。但由于肿瘤病人血液中肿瘤DNA量微,而且突变频率很低,这些检测方法的灵敏度仍需进一步提高。与基因突变不同的是肿瘤中广泛存在着DNA甲基化现象,常常是肿瘤特异性的表现[16]。因此检测DNA甲基化能够更好进地行肿瘤早期检测和分类[17-18]。寻找最有价值的通用的标记物以及肿瘤特异性标记物(或基因区域)通常需要大型泛癌或者组织基因组学数据。到目前为止,已有多项研究是通过TCGA数据资源发现肿瘤DNA甲基化标记物[19-23]。另外由于健康人群血浆中游离DNA片段的长度分布非常规律,而同源序列的循环肿瘤细胞DNA片段长度杂乱无章,所以血浆中游离DNA片段的长度及其分布模式,也可以提示病人是否患有肿瘤以及肿瘤的原发灶部位[24]。来自于细胞游离DNA中的microRNA或者RNA等其他表观基因组学数据也正逐步应用到临床试验中来[25]。从基因组学大数据中挖掘信息并应用于临床实践的潜力是无限的。
开放组学数据资源正在持续增长,不可能在文中一一列举,本文筛选出一些较为重要的数据库,并对其用法和获取数据方法进行简短的介绍。了解并正确如何使用这些资源将会极大地促进科学研究和临床实践的发展,这些开放资源会对项目申请、数据解读等给予很大的帮助。除此之外对于很多没有编程能力的研究者来说,利用在线的工具进行数据分析和数据挖掘会更加便捷和简单。高效使用这些数据首先要了解数据来源、处理方法并且学会解释这些数据的结果。随着时间的进展、资料不断完善,这些资源会变得越来越有条理,越来越容易获得。大数据正在转变传统医疗模式和生物医学研究方法,多组学大数据在其中起到重要作用。与电子病历、影像学资料、病理学图片等其他大数据以及精准医疗设备一样,大数据分析也正在逐步丰富精准医学研究的内涵。

