基于18S rDNA条形码技术的珊瑚礁区塔形马蹄螺(Tectus pyramis)食性分析
周天成, 胡思敏, 林先智, 刘 胜, 黄 晖, 3
基于18S rDNA条形码技术的珊瑚礁区塔形马蹄螺()食性分析
周天成1, 2, 胡思敏1, 林先智1, 2, 刘 胜1, 黄 晖1, 3
(1. 中国科学院南海海洋研究所 中国科学院热带海洋生物资源与生态重点实验室, 广东省应用海洋生物学重点实验室, 广东 广州 510301; 2. 中国科学院大学, 北京 100049; 3. 中国科学院海南热带海洋生物实验站, 海南 三亚 572000)
塔形马蹄螺()是一种暖水性较强的海洋贝类, 也是一种重要的礁栖生物, 研究其自然环境中的食物组成对于认识其生态功能具有重要意义, 但由于缺少直接的食物组成信息, 对其食性和生态功能定位尚不明确。本研究于2017年春季在南沙珊瑚礁区采集了塔形马蹄螺样品, 以18S rDNA可变区(V4)序列为靶标, 用高通量测序技术分析了其现场食物组成。共测得41个OTU, 分属11个门类, 包括节肢动物门(Arthropoda)、子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、丝足虫门(Cercozoa)、刺胞动物门(Cnidaria)、Stramenopiles(不等鞭毛类)、网粘菌门(Labyrinthulomycota)、软体动物门(Mollusca)、多孔动物门(Porifera)、甲藻门(Pyrrophyta)、捕虫霉亚门(Zoopagomycota)。与以往研究不同的是, 本研究发现塔形马蹄螺消化道中存在大量沉积物碎屑, 其中有孔虫、真菌、后生动物是最重要的类群, 占食物序列组成的99.76%, 它们主要存在于海洋沉积物、有机碎屑和礁石表生藻类基质(Epilithic algal matrix, EAM)中。研究结果揭示了塔形马蹄螺的食物主要来源于EAM中的小型生物和碎屑以及珊瑚礁石上的有害生物, 推测塔形马蹄螺属于沉积物碎屑食性生物, 可能在清除珊瑚表面藻类基质、促进珊瑚幼体附着过程中发挥一定的作用, 对于维护珊瑚生态系统的健康和稳定具有积极意义。
塔形马蹄螺(); 食性; DNA条形码; 生态功能; 珊瑚礁
珊瑚礁生态系统具有极高的生物多样性和生产力, 礁栖生物之间复杂的营养关系是保证物质和能量高效传递的基础, 其中底栖生物在珊瑚礁生态系统物质迁移与转化、能量流动中发挥重要作用[1]。底栖生物的摄食活动可在一定程度上维持底栖群落的稳定, 对生态系统的正常运转起到一定的调控作用[2]。因此, 研究底栖生物的现场食物组成有助于认识其在珊瑚礁生态系统稳定运转和自我调控过程中的生态功能[3]。
软体动物是珊瑚礁生态系统中底栖生物的重要优势类群, 其物种数可占底栖生物群落的38.85%[4], 生物量可占50.74%[5]。腹足纲(螺类)是软体动物门中最大的一纲, 珊瑚礁生态系统中的螺类主要栖息于珊瑚礁石或沉积物表面, 不同种的螺食性差别较大, 食物种类包括珊瑚组织、藻类和小型底栖动物[6-8]等, 其摄食特征将直接影响珊瑚礁的健康状况。但这些观点都是基于室内模拟或野外观察得出的推测, 缺少直接的胃容物证据, 因而容易误判其自然状况下的真实食性, 如Marshell等通过野外实验发现栉齿刺尾鱼()可以减少礁石表生藻类基质EAM(Epilithic algal matrix)的生物量而认为其属于植食性鱼类[9],但Tebbett通过显微镜检测消化道内容物发现栉齿刺尾鱼主要摄食碎屑和沉积物(占比99.6%~100%), 极少摄食EAM中的藻类, 从而确定其属于碎屑食性鱼类[10]。
对于类似植食性或碎屑食性底栖生物的摄食研究, 消化道内容物分析是目前比较常用的方法, 但是这种基于形态学鉴定的传统方法仍具有一定的局限性, 无法获取准确的食物种类组成信息[10], 从而难以准确分析其在珊瑚礁食物网中的具体位置和功能作用。基于高通量测序技术(High-throughput sequencing technology)的分子标记方法在摄食生态学研究中应用前景巨大, 解决了形态学鉴定中食物碎片难以分辨的问题, 可以更为全面的覆盖研究对象的食物谱, 往往能发现之前容易忽视的食物来源。此项技术已经被应用于多种海洋生物的摄食研究, 如桡足类、方格星虫、刺参、金钱鱼稚鱼、龙虾、樽海鞘等[11]。Zhang使用高通量测序技术分析了刺参()的食物组成, 共获取823个有效食物序列分属于 40个食物门类, 包括硅藻、节肢动物、有孔虫和软体动物等, 结果显示海参的食物来源并非只有沉积物中的有机碎屑, 也包括水体中的藻类和有机物[11], 而传统研究无法确定这些碎屑的具体组成和来源, 体现了高通量测序技术在底栖生物食性分析方面的有效性和独特优势。
南沙珊瑚礁区常夏无冬, 表层海水温度较高,而年温差较小, 平均水温27.9℃[12], 适合珊瑚生长, 其生物物种十分丰富, 是世界上热带生物种类最丰富的区域之一。南沙群岛已报道的底栖生物种类有309科837属1 444种[4], 但以往对南沙珊瑚礁区多关注造礁石珊瑚和礁栖鱼类的群落结构和生态分布, 对底栖生物生态功能的认识尚不明确。马蹄螺科暖水性较强, 生活于热带珊瑚礁和亚热带岩礁海域, 是南沙珊瑚礁区常见的底栖生物[13]。塔形马蹄螺()是其中重要的代表种, 也是南方沿海的重要经济贝类[14]。本文通过特异性引物扩增塔形马蹄螺消化道样本中真核生物18S rDNA 的可变区片段, 构建高通量测序文库并对18S rDNA可变区序列进行分析, 从而准确鉴定塔形马蹄螺的食物组成, 推测其食物来源以及在珊瑚礁生态系统中的生态功能。
1 材料与方法
1.1 样品的采集与处理
2017年春季在南沙群岛珊瑚礁水域进行了现场调查和采样, 调查区域(8°51′—9°54′N, 112°50′—115°35′E)见图1。本次调查区域礁石上的EAM覆盖面积大, 采集到的塔形马蹄螺大多生活在EAM中。在采样区域内徒手采集塔形马蹄螺样品装入网兜, 带回船上立即用封口袋包装放入–20℃冰箱保存待用。

图1 调查区域地理位置(红色方框, 审图号: 0010287159)及塔形马蹄螺自然生境情况
1.2 消化道内容物的处理及DNA提取
共采集到大小相近的塔形马蹄螺16个,重量95.14±16.66 g, 螺壳宽56.44±4.85 mm。用无菌解剖刀迅速解剖马蹄螺, 从消化道中小心提取其内容物, 使用MP粪便DNA快速提取试剂盒(FastDNA® Spin Kit for Feces, MP Biomedicals, Santa Ana, USA)提取消化道内容物DNA, 溶于30 μL Tris-HCl(10 mmol·L–1, pH 8.0), DNA纯度和含量使用分光光度法检测(Thermo NanoDrop ND-2000; Gene Company Limited, Waltham, Massachusetts, USA), 提取的DNA置于–20℃保存备用。
1.3 高通量测序
使用扩增片段约为380 bp的通用引物TAReuk454FWD1-TAReukREV3 (TAReuk454FWD1: 5′-CCAGCASCYGCGGTAATTCC-3′; TAReukREV3: 5′-ACTTTCGTTCTTGATYRA-3′)[15], 对塔形马蹄螺消化道内容物的18S rDNA V4区域进行扩增, 该区域是真核生物18S rDNA可变区域之一, 使用最多、数据库信息最全、分类效果最好, 是18S rDNA基因分析注释的最佳选择[16]。扩增在20 μL体系中进行: 0.4 μL FastPfu聚合酶, 2 μL 2.5 mmol·L–1dNTPs, 4 μL 5×FastPfu缓冲液, 5 μmol·L–1的正反引物各0.8 μL, 10 ng DNA模板。PCR扩增程序设定为: 95℃预变性 5 min; 95℃变性30 s, 55℃退火30 s, 72℃延伸45 s, 进行27个循环; 最后72℃延伸10 min。
PCR产物经2%琼脂糖电泳检测后, 在Illumina HiSeq测序平台(Illumina, San Diego, CA, USA)进行高通量测序。得到的原始下机数据(Raw Data)经过数据拆分、去引物序列、PE Reads拼接、Tags质量及长度过滤和截取, 然后去除嵌合体后得到最终的有效序列(Effective Sequence)。然后基于有效序列进行OUT (operational taxonomic units)聚类和物种分类分析。
1.4 数据分析
为了得到OTU对应的物种分类信息, 使用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析, 并在各个水平统计每个样本的群落组成, 比对数据库为Silva_128 18S rRNA database(http: //www.arb-silva.de/)。使用BLAST(basic local alignment search tool)与GenBank数据库进行核对(http: //blast.ncbi.nlm.nih.gov/Blast.cgi), 下载相似度最高的同源序列, 与测得的OTU序列进行排列(CLUSTAL W 1.8), 剔除掉塔形马蹄螺自身序列后, 使用MEGA7.0 构建系统进化树, 以大鼠(sp.)为外群, 算法选择最大似然法(Maximum Likelihood)。
2 结果与分析
2.1 测序数据评估
对塔形马蹄螺消化道内容物样品进行高通量测序, 共获得有效序列64 199条, 序列平均长度为363.12 bp, 去除塔形马蹄螺自身序列30 698条后, 有效食物序列共32 982条, 占总有效序列的51.37%。若序列相似性≥97%, 则认为可以鉴定到种水平[17]; 若序列相似性为90%~97%, 则认为可以鉴定到属水平[18]。根据比对结果, 本次鉴定到种的OTU占63.41%, 鉴定到属的OTU占31.71%, 鉴定到其他分类等级的OTU仅占4.88%(图2)。

图2 食物序列在不同分类水平上的丰度分布
2.2 塔形马蹄螺的食物组成
去除塔形马蹄螺自身序列、无法鉴定且数量较少的序列后, 共获得41个OTU, 分属于11个门类(图3): 节肢动物门(Arthropoda)、子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、丝足虫门(Cercozoa)、刺胞动物门(Cnidaria)、Stramenopiles(不等鞭毛类)、网粘菌门(Labyrinthulomycota)、软体动物门(Mollusca)、多孔动物门(Porifera)、甲藻门(Pyrrophyta)、捕虫霉亚门(Zoopagomycota)。有孔虫是食物序列中最丰富的类群, 占总食物序列的89.967%, 包含8个OTU, 均属于丝足虫门。后生动物占据食物序列组成的比例虽然不高, 仅为2.795%, 但种类丰富, 包括9个OTU, 属于石珊瑚、水螅虫、介形虫、海绵、螺等生物类群, 其中珊瑚2种(指状蔷薇珊瑚和细柱滨珊瑚), 水螅虫2种(植丛管螅和), 介形虫2种(陆奥弯角介和), 海绵动物2种(山海绵和穿贝海绵), 螺类1种(玉轮螺属)。此外, 还有少量的藻类, 均为甲藻(卵甲藻属2种, 费氏藻属1种), 占食物序列的0.236%。
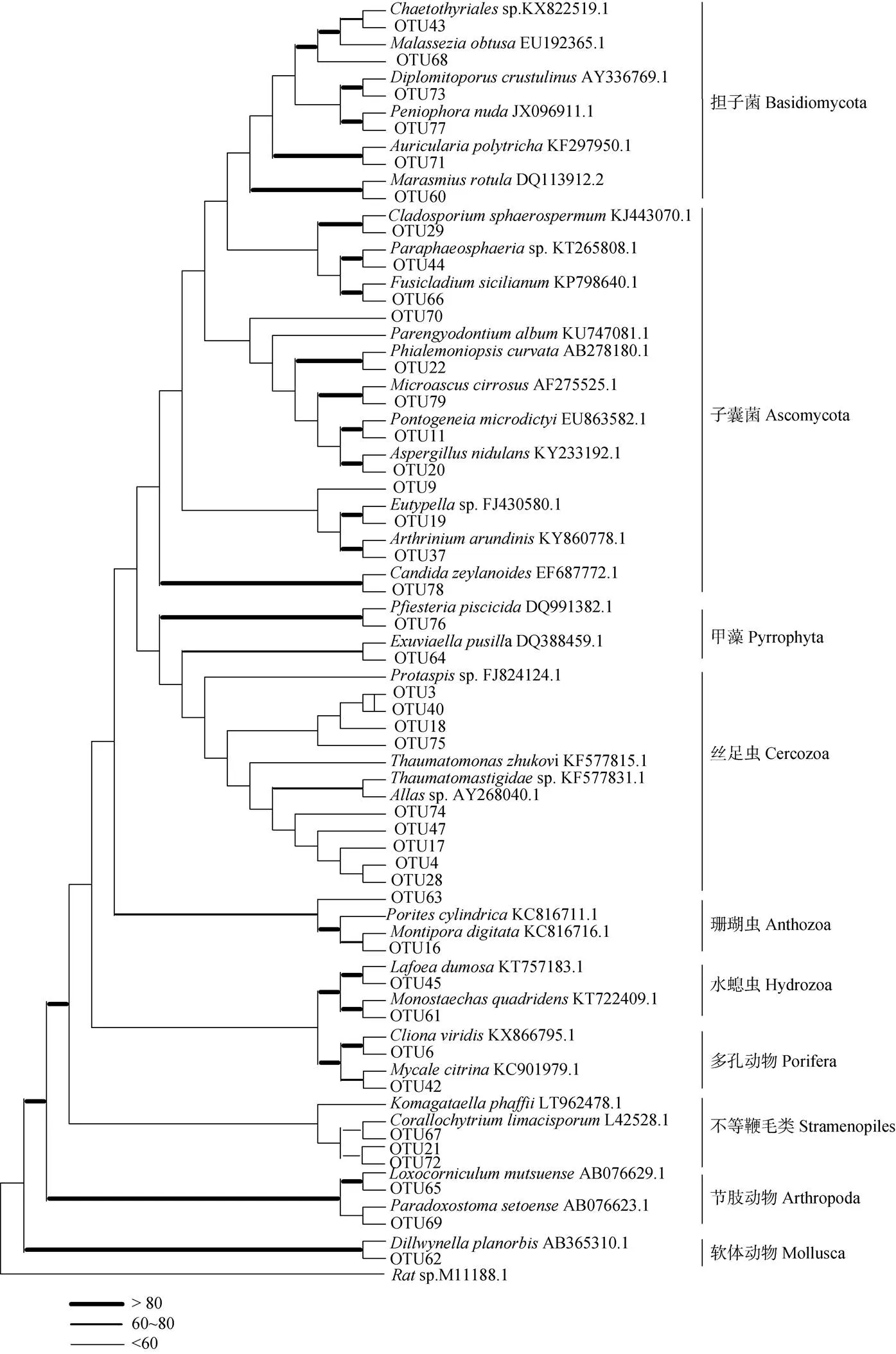
图3 基于OTU构建的 ML 系统进化树
食物序列中另一个重要类群是海洋真菌, 占食物序列的5.979%, 包含18个OTU, 主要为沉积物来源的子囊菌和担子菌, 如sp.、、、、, 也包括一些碎屑表生真菌, 如、、、以及少量藻类共附生真菌, 如、、。
此外还有一些分类地位尚不明确或无法鉴定到种属的OTU, 占食物序列的1.082%, 包括不等鞭毛类(sp., 相似性82%)、接合菌类(, 相似性85%)等。
综上, 塔形马蹄螺的食物序列种类十分多样(图4), 在门水平(phylum)上: 有孔虫占据了可识别食物序列总数的绝大部分(89.967%), 此外, 真菌占7.061%, 其余生物类群的序列所占比例较少: 珊瑚占0.894%、海绵占0.810%、介形虫占0.491%、水螅占0.409%、藻类仅占0.236%。根据食物序列的门类及其比例可以判断, 本次在南海海域采集到的塔形马蹄螺的主要食物类群是有孔虫、真菌等, 食物序列中有97.028%可能来源于碎屑和沉积物。
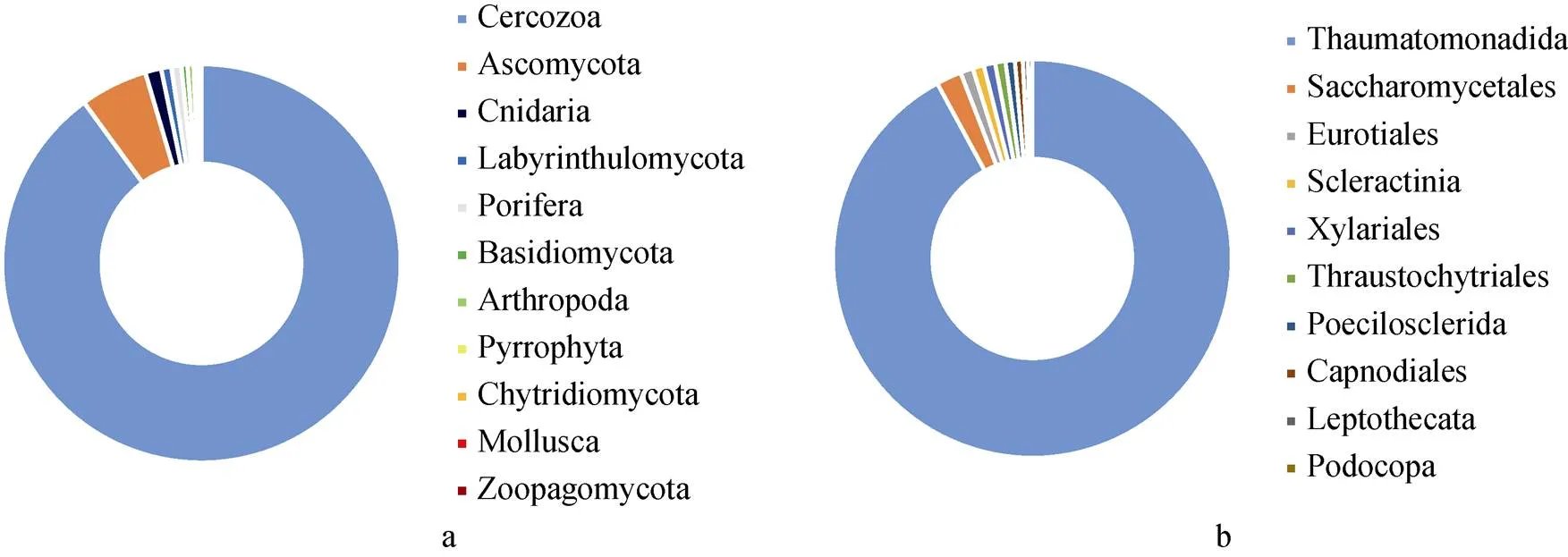
图4 塔形马蹄螺消化道内食物组成比例(扇形大小代表序列数目)
注: a: 门水平(Phylum) b: 目水平(Order, 选取序列相对含量前10的目)
3 讨论
自然海域中, 底栖生物食性与所处环境有一定的关系, 其食物来源广泛且食物种类多样, 利用光学显微镜鉴定与计数的传统食性分析方法难以准确获取食物信息[10-11], 传统测序方法(Sanger测序)可以比较准确地鉴定饵料生物的种类, 但是其工作量较大, 成本较高, 进而限制了对食物种类的覆盖范围[19]。用于胃肠内容物分析的DNA条形码有很多种类, 包括COI(线粒体细胞色素氧化酶I基因)、ITS(核糖体rRNA基因内转录间隔区)和18S rDNA(真核生物的核糖体小亚基RNA的编码DNA)等, 而18S rDNA是目前使用最多、数据库信息最全、分类效果最好的DNA条形码[16]。而且食物 DNA 往往会随消化过程逐步降解、片段化, 高通量测序技术恰好能有效利用长度很小的模版, 发掘更多种类信息, 适合于降解 DNA[20], 因此本研究选择了18S rDNA V4位点作为DNA条形码来进行塔形马蹄螺消化道内容物的分析。此前的研究认为塔形马蹄螺是植食性螺类, 本研究发现其消化道中存在大量沉积物碎屑, 使用基于18S rDNA条形码的高通量测序技术在其消化道内容物中检测到41个生物种类, 其中沉积物来源的有孔虫、真菌和后生动物是最丰富的食物类群。研究结果揭示了塔形马蹄螺食物组成的复杂性, 沉积物中丰富的生物碎屑和小型生物是其主要的食物来源。本研究也检测到了一些传统技术难以辨认或易忽略的类群, 如海绵、水螅、珊瑚等, 表明高通量测序技术比显微镜检技术更有优势, 因其提供了更准确、更全面的摄食数据, 有利于重新认识生物的食物来源和食性。因此, 推测珊瑚礁区塔形马蹄螺可能是碎屑食性而非植食性, 在清除有害真菌、霉菌、海绵等对珊瑚礁有潜在威胁的生物, 促进沉积物流出和珊瑚幼体附着, 维持珊瑚礁生态系统稳定等方面具有一定作用, 可能是一种重要的护礁生物。
3.1 塔形马蹄螺的食性及食物来源
一般认为塔形马蹄螺是植食性螺类[14], 主要摄食大型藻, 但这并非来源于直接的消化道内容物证据, 而是根据其栖息环境的推测。本研究中, 并未在塔形马蹄螺消化道内容物中检测到大型藻类的序列信息, 考虑到高通量测序的灵敏度及食物中其他微藻种类的存在, 这种结果的出现不太可能是由于对大型藻类的消化吸收速率更快导致无法被检测; 且许多大型藻类组织中会产生一些次级代谢产物或碳酸钙等, 来避免被动物摄食[21], 如网地藻(spp.)、葡扇藻(spp)和仙掌藻(spp.)等。因此, 塔形马蹄螺可能并非植食性生物。本研究从塔形马蹄螺消化道中仅鉴定出少量甲藻, 包括2种原甲藻和1种费氏藻。原甲藻在海洋中广泛分布, 通常附生于珊瑚礁区大型藻类或藻坪藻类表面[22], 推测其主要来源于珊瑚礁区藻坪EAM表面, 可能由塔形马蹄螺摄食其他有机成分时一并摄入。
本研究中检测到了丰富的原生动物和后生动物来源的序列, 在以往研究中未有报道, 这些生物可能被消化破碎以碎屑的形式存在, 从而未被传统方法检出, 也可能由于体积太小而被忽视[11], 这些生物和有机碎屑主要来源于海洋沉积物和EAM, 证明了塔形马蹄螺是沉积物碎屑食性。丝足虫在塔形马蹄螺食物序列中的占绝大部分且均属于Thaumatomastigidae科, 这是一类生活于海洋沉积物碎屑中的微型生物, 体型在10 μm左右, 在沉积物食性的刺参消化道中也检测到了大量丝足虫, 最高达到36.35%[11],一些丝足虫种类在珊瑚礁EAM中也有发现[23]。检测到的介形虫和小型腹足动物在海洋沉积物环境中分布广泛, 近期在大堡礁的EAM中也发现了多种类似的介形虫和腹足动物[24]。所以推测这些食物类群主要来源于EAM中的沉积物碎屑, 是塔形马蹄螺最主要的食物来源。
真菌在塔形马蹄螺的食物序列中比重很低但多样性很高, 其中大部分种类存在于沉积物和有机碎屑中, 如sp.、、、sp. 、, 这些真菌可以产生一些生物碱或次级代谢产物来抑制其他致病菌的生长繁殖, 一些种类具有分解植物纤维或蛋白质的能力[25], 同样生活于沉积物中的方格星虫和的肠道中也检测到了类似的真菌类群[25], 推测这些菌群可能是塔形马蹄螺等沉积物碎屑食性生物消化道中的共生真菌, 可以更好地促进底栖生物分解、利用沉积物中的各类有机碎屑。此外还检测到3种藻类附生真菌: 分别为、、, 这些真菌可能来源于EAM中的藻类表面, 塔形马蹄螺在这些藻类表面摄食其他有机成分时打包摄入。
礁石表生藻类基质即EAM是珊瑚礁区普遍存在的底质类型, 可占据珊瑚礁基底面积的 30%~80%[26], 其成分主要包括丝状藻类、微生物、沉积物、有机碎屑及小型无脊椎动物等[27]。本次研究发现调查区域礁石上的EAM覆盖面积很大, 且采集到的塔形马蹄螺大多生活在EAM中(图1), 因此推测EAM中的微小生物和碎屑是塔形马蹄螺的主要食物来源。EAM中菌类、微藻以及各类小型生物的存在改善了沉积物碎屑的营养价值, 使得碎屑的蛋白质/能量比率、C/N比率和总可水解氨基酸含量等营养指标均高于藻类[28-29], 这可能是塔形马蹄螺偏向于摄食EAM中有机碎屑的主要原因。
本研究也从塔形马蹄螺消化道中检测到了造礁石珊瑚来源的序列(指状蔷薇珊瑚和细柱滨珊瑚), 这两种珊瑚是潮间带或潮下带礁坪的优势种[30], 在采样区域也广泛存在。检测到的不等鞭毛类生物多生活于珊瑚表面的黏液中, 其中可能导致珊瑚患病[31], 塔形马蹄螺对这类生物的摄食对于维持珊瑚的健康具有重要的意义; 此外, 也检测到了一些常见的珊瑚礁固着生物[32], 包括海绵、水螅虫等, 其中由穿贝海绵引起的生物侵蚀会造成严重的珊瑚礁退化[33], 可见, 马蹄螺的摄食对于维持底栖群落稳定具有一定的积极作用。但是这些珊瑚或珊瑚共生/共存生物的序列相对丰度较低, 推测塔形马蹄螺主要还是以摄食EAM为主, 只是偶尔在珊瑚主导区域摄食海绵、水螅虫等固着生物及珊瑚粘液有机物。
3.2 塔形马蹄螺生态功能探讨
底栖生物是珊瑚礁生态系统内碎屑食物链的重要一环, 可摄食上层各营养级生物产生的有机颗粒, 避免颗粒态有机物POM(particulate organic matter)因埋藏而离开生物地球化学循环[34], 其自身又被其他高营养级的动物摄食, 从而将沉积物-水界面的物质和能量进行再加工和分配, 一定程度上驱动了珊瑚礁区有机碳的高效循环; 更重要的是, 底栖生物的摄食对于维持珊瑚礁区底栖生物群落的平衡和稳定具有重要意义。
研究发现EAM对沉积物的富集会对整个珊瑚礁生态系统造成许多潜在的生态威胁: EAM可能因为沉积物增加而转变为多沉积物藻类基质(long sediment-laden algal turfs, LSATs)状态, 这种转变会影响珊瑚幼体附着而减少珊瑚的补充量[35]; 此外, EAM中沉积物碎屑的增加也会减少植食性鱼类对大型藻的摄食[36], 导致珊瑚礁覆盖率降低和珊瑚礁生态系统退化, 这个过程将破坏珊瑚礁特有的结构复杂性和生物多样性, 导致生物多样性和生产力大大降低[37]。而塔形马蹄螺的摄食活动清除了EAM和珊瑚礁石表面的沉积物碎屑, 从而避免沉积物增加对珊瑚礁生态系统健康的影响, 如塔形马蹄螺可以通过摄食活动对沉积物进行再加工, 其消化过程会减小沉积物颗粒, 从而使沉积物更容易被水流从EAM中带离[38]。通过清除因水流和动物排泄滞留在礁石EAM中的沉积物和有机颗粒, 保持了珊瑚生活环境的洁净, 一定程度上可以减少大型藻的附着生存[38], 也有利于植食性鱼类对大型藻类的摄食[36]; 而且无沉积物的礁石表面更有利于珊瑚幼虫在礁石上的附着与成活[39], Omori(2005)通过野外培养证实了塔形马蹄螺的近源种—大马蹄螺()确实可以促进柔枝鹿角珊瑚幼体的附着与成活[40]。
此外, 人类活动增加了真菌、霉菌等病害生物的数量, 破坏了珊瑚礁正常的菌群组成, 从而降低了珊瑚的抵抗力和竞争力[41], 珊瑚礁生态系统中珊瑚的主导地位可能被其他竞争力更强的生物类群(如海绵)取代[42], 导致珊瑚礁生态系统结构和功能的破坏。本研究发现塔形马蹄螺的食物中包含许多种类的有害真菌、霉菌、海绵等对珊瑚礁有潜在威胁的生物, 塔形马蹄螺有可能控制或减缓这些有害生物形成优势群落, 从而推测塔形马蹄螺在维护珊瑚礁生态系统的健康和稳定中具有积极作用, 是一种典型的护礁生物, 但仍需进一步的实验验证其护礁能力, 以期将塔形马蹄螺作为人工修复珊瑚礁生态系统中引入的先锋种类, 在珊瑚礁生态系统早期的构建和稳定中发挥相应的生态功能。
4 结论
塔形马蹄螺的食物包括节肢动物、丝足虫、刺胞动物、不等鞭毛类、软体动物、多孔动物、甲藻、担子菌、网粘菌、子囊菌、捕虫霉等不同门类, 其中有孔虫、真菌和后生动物是最丰富的食物类群, 占99.76%, 这些种类多为典型的沉积物分布的小型底栖类群, 可能来源于珊瑚礁石表面的海洋沉积物和EAM, 推测南沙珊瑚礁区塔形马蹄螺属于沉积物碎屑食性生物, 而非传统认为的植食性生物, 可能在清除珊瑚表面藻类基质、促进珊瑚幼体附着过程中发挥作用。
[1] 严宏强, 余克服, 谭烨辉. 珊瑚礁区碳循环研究进展[J].生态学报, 2009, 29(11): 6207-6215. Yan Hongqiang, Yu Kefu, Tan Yehui. Recent development in the research of carbon cycle in coral reef ecosystem[J]. Acta Ecologica Sinica, 2009, 29(11): 6207-6215.
[2] Nystr M. Redundancy and response diversity of functional groups: implications for the resilience of coral reefs[J]. Ambio, 2006, 35(1): 30-35.
[3] Yachi S, Loreau M. Biodiversity and ecosystem productivity in a fluctuating environment: the insurance hypothesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(4): 1463-1468.
[4] 李新正, 王永强. 南沙群岛与西沙群岛及其邻近海域海洋底栖生物种类对比[J]. 海洋科学集刊, 2002: 74- 79. Li Xinzheng, Wang Yongqiang. Comparison between benthos of Nansha islands and Xisha islands, South China Sea[J]. Studia Marina Sinica, 2002: 74-79.
[5] 董栋, 李新正, 王洪法, 等. 海南岛三亚珊瑚礁区大型底栖动物群落特征[J]. 海洋科学, 2015, 39(3): 83-91. Dong Dong, Li Xinzheng, Wang Hongfa, et al. Macrobenthic community characters of coral reef at Sanya, Hainan[J]. Marine Sciences, 2015, 39(3): 83-91.
[6] Bessey C, Babcock R C, Thomson D P, et al. Outbreak densities of the coral predatorin relation to in situgrowth rates on Ningaloo Reef, Western Australia[J]. Coral Reefs, 2018, 37(4): 985-993.
[7] Altman-Kurosaki N T, Priest M A, Golbuu Y, et al. Microherbivores are significant grazers on Palau’s forereefs[J]. Marine Biology, 2018, 165(4): 1-11.
[8] Sharp W C, Delgado G A. Predator-prey interactions between the corallivorous snailand the carnivorous deltoid rock snail[J]. The Biological Bulletin, 2015, 229(2): 129-133.
[9] Marshell A, Mumby P J. The role of surgeonfish (Acanthuridae) in maintaining algal turf biomass on coral reefs[J]. Journal of Experimental Marine Biology and Ecology, 2015, 473: 152-160.
[10] Tebbett S B, Goatley C H R, Bellwood D R. Clarifying functional roles: algal removal by the surgeonfishesand[J]. Coral Reefs, 2017, 36(3): 1-11.
[11] Zhang H, Xu Q, Zhao Y, et al. Sea cucumber () eukaryotic food source composition determined by 18S rDNA barcoding[J]. Marine Biology, 2016, 163(7): 1-11.
[12] 赵焕庭, 王丽荣, 袁家义. 南海诸岛的自然环境、资源与开发——纪念中国政府收复南海诸岛70周年(3)[J].热带地理, 2017, 37(5): 659-680, 693. Zhao Huanting, Wang Lirong, Yuan Jiayi. Natural environment, resources and development of the South China Sea Islands: the 70th anniversary of recovery of the South China Sea Islands(3)[J]. Tropical Geography, 2017, 37(5): 659-680, 693.
[13] 王桢瑞. 中国动物志[M]. 北京: 科学出版社, 1997: 214-237. Wang Zhenrui. Fauna Sinica[M]. Beijing. Science Press, 1997: 214-237.
[14] 齐钟彦. 中国经济软体动物[M]. 北京: 中国农业出版社, 1998: 41-42. Qi Zhongyan. Economic Mollusca of China[M]. Beijing. China Agriculture Press, 1998: 41-42.
[15] Stoeck T, Bass D, Nebel M, et al. Multiple marker parallel tag environmental DNA sequencing reveals a highly complex eukaryotic community in marine anoxic water[J]. Molecular Ecology, 2010, 19(1): 21-31.
[16] Hadziavdic K, Lekang K, Lanzen A, et al. Characterization of the 18S rRNA gene for designing universal eukaryote specific primers[J]. Plos One, 2014, 9(2): 1-10.
[17] Peay K G, Garbelotto M, Bruns T D. Spore heat resistance plays an important role in disturbance-mediated assemblage shift of ectomycorrhizal fungi colonizingseedlings[J]. Journal of Ecology, 2009, 97(3): 537-547.
[18] Nieto M P, Carbone S S. Characterization of juvenile maritime pine (Ait.) ectomycorrhizal fungal community using morphotyping, direct sequencing and fruitbodies sampling[J]. Mycorrhiza, 2009, 19(2): 91-98.
[19] Jo H, Gim J A, Jeong K S, et al. Application of DNA barcoding for identification of freshwater carnivorous fish diets: Is number of prey items dependent on size class for?[J]. Ecology and Evolution, 2014, 4(2): 219-229.
[20] 张军毅, 朱冰川, 徐超, 等. 基于分子标记的宏基因组16S rRNA基因高变区选择策略[J]. 应用生态学报, 2015, 26(11): 3545-3553. Zhang Junyi, Zhu Bingchuan, Xu Chao, et al. Strategy of selecting 16S rRNA hypervariable regions for matagenome-phylogenetic marker genes based analysis[J]. Chinese Journal of Applied Ecology, 2015, 26(11): 3545-3553.
[21] Hanmer J, White J W, Pawlik J R. Application of diet theory reveals context-dependent foraging preferences in an herbivorous coral reef fish[J]. Oecologia, 2017, 184(1): 127-137.
[22] Faust M A, Morton S L, Quod J P. Further SEM study of marine dinoflagellates: The genus(Dinophyceae)[J]. Journal of Phycology, 1996, 32(6): 1053- 1065.
[23] Ekebom J, Patterson D J, Vørs N. Heterotrophic flagellates from coral reef sediments (Great Barrier Reef, Australia)[J]. Archivfür Protistenkunde, 1996, 146(3): 251-272.
[24] Kramer M J, Bellwood D R, Bellwood O. Large-scale spatial variation in epilithic algal matrix cryptofaunal assemblages on the Great Barrier Reef[J]. Marine Biology, 2014, 161(9): 2183-2190.
[25] Wang Y, Shi T, Huang G, et al., 2018. Molecular detection of eukaryotic diets and gut mycobiomes in two marine sediment-dwelling worms,and[J]. Microbes and Environments, 2018, 33(3): 1-11.
[26] Goatley C H R, Bellwood D R. The roles of dimensionality, canopies and complexity in ecosystem monitoring[J]. Plos One, 2011, 6(11): 1-8.
[27] Kramer M J, Bellwood O. Cryptofauna of the epilithic algal matrix on an inshore coral reef, Great Barrier Reef[J]. Coral Reefs, 2012, 31(4): 1007-1015.
[28] Crossman D J, Mcconochie J. Detritus as food for grazing fishes on coral reefs[J]. Limnology and Oceanography, 2001, 46(7): 1596-1605.
[29] Wilson S. Nutritional value of detritus and algae in blenny territories on the Great Barrier Reef[J]. Journal of Experimental Marine Biology and Ecology, 2002, 271(2): 155-169.
[30] 黄晖, 张成龙, 杨剑辉, 等. 南沙群岛渚碧礁海域造礁石珊瑚群落特征[J]. 应用海洋学学报, 2012, 31(1): 79-84. Huang Hui, Zhang Chenglong, Yang Jianhui, et al. Scleractinian coral community characteristics in Zhubi reef sea area of Nansha Islands[J]. Journal of Applied Oceanography, 2012, 31(1): 79-84.
[31] Sumathi J, Raghukumar S, Kasbekar D, et al. Molecular evidence of fungal signatures in the marine protistand its implications in the evolution of animals and fungi[J]. Protist, 2006, 157(4): 363-376.
[32] Redmond N E, Morrow C C, Thacker R W, et al. Phylogeny and systematics of demospongiae in light of new small-subunit ribosomal DNA (18S) sequences[J]. Integrative and Comparative Biology, 2013, 53(3): 388-415.
[33] Ashok A M, Schonberg C H L, Raj K D, et al. A sponge of thecomplex invades and excavates corals of the Gulf of Mannar, south-eastern India[J]. Marine and Freshwater Research, 2018, 69(6): 874-882.
[34] 宋金明, 李学刚. 海洋沉积物/颗粒物在生源要素循环中的作用及生态学功能[J]. 海洋学报, 2018, 40(10): 1-13. Song Jinming, Li Xuegang. Ecological functions and biogenic element cycling roles of marine sediment/ particles[J]. Haiyang Xuebao, 2018, 40(10): 1-13.
[35] Goatley C H R, Bonaldo R M, Fox R J, et al. Sediments and herbivory as sensitive indicators of coral reef degradation[J]. Ecology and Society, 2016, 21(1): 1-17.
[36] Goatley C H R, Bellwood D R. Sediment suppresses herbivory across a coral reef depth gradient[J]. Biology Letters, 2012, 8(6): 1016-1018.
[37] Graham N J. Habitat complexity: coral structural loss leads to fisheries declines[J]. Current Biology, 2014, 24(9): R359-R361.
[38] Tebbett S B, Goatley C H, Bellwood D R. Algal turf sediments and sediment production by parrotfishes across the continental shelf of the northern Great Barrier Reef[J]. Plos One, 2017, 12(1): 1-17.
[39] Quintana C O, Shimabukuro M, Pereira C O, et al. Carbon mineralization pathways and bioturbation in coastal Brazilian sediments[J]. Scientific Reports, 2015, 5(1): 1-13.
[40] Omori M. Success of mass culture ofcorals from egg to colony in open water[J]. Coral Reefs, 2005, 24(4): 563-563.
[41] Zaneveld J R, Burkepile D E, Shantz A A, et al. Overfishing and nutrient pollution interact with temperature to disrupt coral reefs down to microbial scales[J]. Nature Communications, 2016, 7: 1-12.
[42] Sisney M A, Cummins R H, Wolfe C R. Incidence of black band disease, brown band disease, and white syndrome in branching corals on the Great Barrier Reef[J]. Estuarine Coastal and Shelf Science, 2018, 214: 1-9.
Study on the feeding habits ofin the coral reef ecosystem based on 18S rDNA barcoding
ZHOU Tian-cheng1, 2, HU Si-min1, LIN Xian-zhi1, 2, LIU Sheng1, HUANG Hui1, 3
(1. Key Laboratory of Tropical Marine Bio-resources and Ecology, Guangdong Provincial Key Laboratory of Applied Marine Biology, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China; 3. Tropical Marine Biological Research Station in Hainan, Chinese Academy of Sciences, Sanya 572000, China)
primarily lives in warm seas and is widely distributed in coral reefs. However, there is a scarcity of knowledge about its ecological role in the coral reef ecosystem due to the lack of clear information regarding its feeding habits. This study was conducted to investigate the in situ diet ofcollected from the coral reefs in Nansha Islands in May 2017 by high-throughput sequencing (HTS) methods targeting the 18S rDNA gene. A total of 41 different operational taxonomic units (OTUs) at 97% similarity level were identified. Diverse prey species detected in this study demonstrated a wide and complicated food spectrum of, including Arthropoda, Ascomycota, Basidiomycota, Cercozoa, Cnidaria, Labyrinthulomycota, Mollusca, Porifera, Stramenopiles, Pyrrophyta, and Zoopagomycota. This finding was different from traditional study that reportedas herbivores, we found thatingests abundant sediments and sequence analysis revealed that foraminifera, fungi, fungus and metazoa were the most abundant food groups, constituting approximately 99.76% of the total sequences. Based on the results of previous studies, these abundant food resources might be derived from the benthic community consisting of the epilithic algal matrix (EAM) in coral reefs. Altogether, these findings indicate thatis a type of detritivore feeding primarily on the EAM in coral reefs and play an important role in cleaning the epilithic algal and promoting the attachment of coral larvae. Therefore, it can help in maintaining the stability and health of the coral reef ecosystem.
; feeding habits; 18S rDNA; ecological functions; coral reef
Jan. 17, 2019
[National Key Research and Development Project of China, No. 2016YFC0502800; Strategic Priority Research Program of Chinese Academy of Sciences, No. XDA13020100; Natural Science Foundation of China, No. 41806188]
P735
A
1000-3096(2020)02-0099-09
10.11759/hykx20190117002
2019-01-17;
2019-04-02
国家重点研发计划课题(2016YFC0502800); 中国科学院战略性先导科技专项(XDA13020100); 国家自然科学基金项目(41806188)
周天成(1995-), 男, 湖南省汨罗人, 硕士研究生, 主要从事珊瑚礁栖生物摄食生态学研究, 电话: 020-89023196, E-mail: zhoutiancheng17@mails.ucas.ac.cn; 刘胜,通信作者, 研究员, E-mail: shliu@scsio.ac.cn
(本文编辑: 杨 悦 李晓燕)

