33种鱼类微型反向重复转座元件鉴定
胡竞文 邵 峰 赵连鹏 韩民锦 彭作刚
(1. 西南大学生命科学学院, 淡水鱼类资源与生殖发育教育部重点实验室, 重庆 400715;2. 西南大学家蚕基因组生物学国家重点实验室, 重庆 400715)
转座元件(Transposable elements, TEs)是广泛存在于基因组中的重复序列, 也称为转座子(Transposons), 它可以在基因组中移动并插入到新的位点[1]。转座子由美国遗传学家Barbara McClintock在玉米中发现[2,3], 根据转座子不同的转座机制, 将转座子分为两大类[4]。第一类是RNA类转座子, 也被称为逆转录转座子(Retrotransposons), 这类转座子通过“复制-粘贴”机制, 以RNA为媒介进行转座; 第二类是DNA类转座子(DNA transposons), 这类转座子通过“剪切-粘贴”机制, 以DNA为媒介进行转座。
根据能否实现自主转座, 转座子可以分为自主型转座子和非自主型转座子。非自主型转座子最早在玉米(Zea mays)中被发现[5], 如今较受关注的非自主型转座子是微型反向重复转座元件(Miniature inverted-repeat transposable elements, MITEs), 它们由于缺少编码转座酶的序列而不能进行自主转座。这类转座子有一个明显的结构特征, 序列的两端存在反向重复序列(Terminal inverted repeat, TIR)和靶位点重复序列(Target site duplication, TSD)[5]。MITEs的序列长度通常小于800 bp, 序列中AT含量较高, 在基因组有较多的拷贝数, 可以形成稳定的二级结构, 并且能够插入到基因内部或附近的位置[6],对基因的结构[7]、基因组的大小[8,9]和基因的表达调控[10]都有影响。目前, 植物[8,9,11]、昆虫[12]、病毒[13,14]等基因组的MITEs预测和数据库构建已有相关文章发表, 例如在41种植物中鉴定出3527个MITEs家族, 大多数高等植物比低等植物含有更多的MITEs,植物基因组中MITEs序列的数量与基因组大小呈正相关[10]。在98种昆虫基因组中鉴定出6012个MITEs家族, 其中有601个新MITEs, TSD为TA的MITEs序列在昆虫基因组中大量存在, 并且昆虫基因组大小也与MITEs含量呈正相关[12]。
鱼类是最古老的脊椎动物, 在脊椎动物进化过程中具有独特的地位。鱼类的物种数量占脊椎动物物种总量的一半以上, FishBase数据库中记录的鱼类已达到34200种(2019年3月), 并且数量还在不断增加。根据形态特征和栖息环境等差异, 鱼类被分成多个类群[15—18], 如无颌类、软骨鱼纲、肉鳍鱼纲和辐鳍鱼纲。对于鱼类转座子的研究工作也在开展, 例如发现古代鱼类中的DNA转座子含量低于逆转录转座子含量[19], 较大基因组鱼类的转座子多样性更高[20], 鱼类基因组中转座子含量可能与物种栖息地等因素有关[21]等。目前, 更多研究关注转座子对“宿主”的作用, 比如转座子对鱼类性别分化有一定影响[22], 鱼类基因组加倍与转座子扩增有关[23]。虽然鱼类转座子的研究工作不断完善, 但是, 鱼类基因组中MITEs的系统研究尚未开展。
目前, 很多鱼类物种的基因组测序工作已经完成, 鱼类的基因组数据、蛋白质序列可以从NCBI(National Center of Biotechnology Information)中下载, 这为鱼类基因组中的MITEs鉴定提供了良好的契机, 使我们能够运用生物信息学方法对MITEs进行鉴定, 丰富脊椎动物的转座子数据。本研究从NCBI数据库搜集了具有代表性的33种鱼类基因组数据(包括无颌类3种、软骨鱼纲2种、肉鳍鱼纲1种、辐鳍鱼纲27种)并对它们进行鉴定和比较学分析以揭示MITEs在鱼类基因组中的分布规律、扩增模式以及对基因组进化的影响。
1 材料与方法
1.1 鱼类基因组数据的搜集
如表1所示, 从NCBI数据库中下载33种鱼类的基因组相关数据信息, 涵盖无颌类、软骨鱼纲、肉鳍鱼纲和辐鳍鱼纲, 包括小丑鱼(Amphiprion ocellaris)、欧洲鳗鲡(Anguilla anguilla)、日本鳗鲡(Anguilla japonica)、美洲鳗鲡(Anguilla rostrate)、墨西哥丽脂鲤(Astyanax mexicanus)、象鲨(Callorhinchus milii)、大西洋鲱(Clupea harengus)、半滑舌鳎(Cynoglossus semilaevis)、斑马鱼(Danio rerio)、蒲氏盲鳗(Eptatretus burger)、伯氏朴丽鱼(Haplochromis burtoni)、贝氏隆头鱼(Labrus bergylta)、大黄鱼(Larimichthys crocea)、矛尾鱼(Latimeria chalumnae)、猬鳐(Leucoraja erinacea)、斑点雀鳝(Lepisosteus oculatus)、日本七鳃鳗(Lethenteron camtschaticum)、斑点叉尾鮰(Ictalurus punctatus)、斑马宫丽鱼(Maylandia zebra)、黄鳝(Monopterus albus)、布氏新亮丽鲷(Neolamprologus brichardi)、弗氏假鳃鳉(Nothobranchius furzeri)、罗非鱼(Oreochromis niloticus)、日本青鳉(Oryzias latipes)、象鼻鱼(Paramormyrops kingsleyae)、海七鳃鳗(Petromyzon marinus)、红丽鱼(Pundamilia nyererei)、大西洋鲑鱼(Salmo salar)、亚洲龙鱼(Scleropages formosus)、红缚平鲉(Sebastes rubrivinctus)、犀角金线鲃(Sinocyclocheilus rhinocerous)、红鳍东方鲀(Takifugu rubripes)以及花斑剑尾鱼(Xiphophorus maculatus)。
1.2 基因组中MITEs的预测
使用MITE-Hunter[24](设定默认参数)对33种鱼类基因组的MITEs进行预测, 将得到的MITEs作为问询序列与基因组数据进行blastn比对(identity>80%、coverage>80%、e值<1e-5、长度<800 bp),保留在基因组中拷贝数多于3条的MITEs一致序列。然后提取MITEs在基因组的拷贝, 使用MUSCLE进行序列比对[25], Bioedit确定MITEs拷贝序列的TSD和TIR[26], 并在DAMBE中生成校正边界后的MITEs一致序列[27]。在同一个基因组中的MITEs遵从“80-80-80”规则去冗余[4], 记录每条MITEs的序列长度。再次使用blastn将得到的一致序列结果作为问询序列与基因组进行比对, 利用perl脚本统计MITEs在基因组中的拷贝数和序列长度等信息。若与MITEs一致序列有相同TIR和TSD, 称之为全长拷贝(Full-length copies); 若没有完整TIR的拷贝序列, 称为片段拷贝(Fragment copies)。得到MITEs在各个基因组中的含量信息后, 在SPSS中进行MITEs含量和基因组大小的Pearson相关性分析。
1.3 MITEs在鱼类基因组中的扩增和分布
MITEs可以在基因组中通过转座进行扩增, 因此我们分析了MITEs的插入时间和分布情况。通过K2P模型(Kimura 2 parameter distances)[28]计算全长拷贝序列在基因组中的插入时间, 首先使用DAMBE得到全长拷贝和MITEs一致序列之间的分化度, 进而利用公式T=K/2r[29]计算插入时间(取核苷酸位点替换速率r= 2.5×10-8[30,31],K值为分化度)。为了解MITEs在基因组中的分布, 下载所选鱼类物种的基因组注释文件, 其中的8个物种(蒲氏盲鳗、日本七鳃鳗、海七鳃鳗、亚洲龙鱼、日本鳗鲡、美洲鳗鲡、欧洲鳗鲡和红缚平鲉)没有内含子、外显子信息注释文件, 不能计算插入位置, 猬鳐的注释文件信息很少, 不能得到有效位置信息。对剩余的24种鱼类, 根据MITEs全长拷贝在基因组上的位置, 使用perl脚本统计全长拷贝插入到基因内、基因外显子和内含子中的数量。
2 结果
2.1 鱼类基因组中MITEs的含量
通过MITE-Hunter对33个鱼类基因组MITEs进行初步预测, 后续校准、去冗余后确定了所选物种基因组中MITEs一致序列和拷贝数, 共得到2433个MITEs一致序列, 487594个全长拷贝。不同鱼类基因组的MITEs拷贝数和含量差异较大(表1)。MITEs(全长拷贝和片段拷贝)含量最高的5个物种分别是
斑马鱼(237.76 Mb, 16.66%)、海七鳃鳗(213.53 Mb,21.18%)、大西洋鲑鱼(189.29 Mb, 6.38%)、墨西哥丽脂鲤(99.05 Mb, 7.42%)和日本七鳃鳗(83.17 Mb,8.07%)。也有部分物种的MITEs含量较低, 例如大西洋鲱MITEs含量为3.94 Mb(0.49%), 象鲨为3.83 Mb(0.39%), 欧洲鳗鲡为2.70 Mb(0.27%), 猬鳐的含量为1.73 Mb(0.11%), 红鳍东方鲀基因组中MITEs含量最低, 仅为0.61 Mb(0.16%)。从分类阶元来看, 无颌类的MITEs含量较高, 如海七鳃鳗; 软骨鱼纲和肉鳍鱼纲鱼类的MITEs含量较低, 如猬鳐和矛尾鱼; 辐鳍鱼纲的鱼类MITEs含量最为丰富,如斑马鱼。

表1 33种鱼类基因组MITEs存在情况Tab. 1 The presence of MITEs in 33 fish genomes
相关性分析发现基因组大小和MITEs含量之间呈正相关(r=0.124,P=0.045, 图1), 比如海七鳃鳗的基因组为1007.98 Mb, 含有213.53 Mb的MITEs序列; 斑马鱼的基因组为1427.29 Mb, 含有237.76 Mb的MITEs序列。同时, MITEs序列的AT含量较高,鱼类基因组中的MITEs转座子也是如此, 它们的AT含量在51.75%(猬鳐)到61.68%(斑马鱼)之间, 均多于GC核苷酸在基因组的占比。而MITEs在鱼类基因组中存在丰富的拷贝[32,33], 其中MITEs拷贝数最多的是斑马鱼(88337个全长拷贝), 最少的是红鳍东方鲀(500个全长拷贝)。
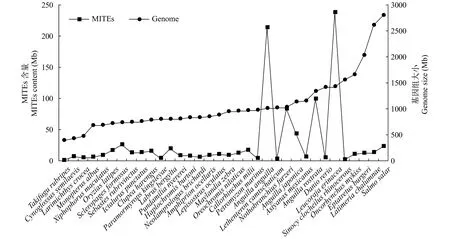
图1 33种鱼类基因组大小和MITEs含量的关系Fig. 1 The relationship between genome size and MITEs content of the 33 fish species
2.2 鱼类基因组中的MITEs超家族
将得到的MITEs一致序列根据各自的TSD和TIR序列特征进行分类[12], 进一步计算各个超家族在基因组的占比。分析结果表明, 鱼类基因组中MITEs超家族有: TC1-Mariner、PHIS、P、Kobolok、PiggyBac、hAT、Ginger、CMC、Merlin、Sola2, 不同鱼类基因组中的MITEs超家族的分布存在较大差异(图2)。TC1-Mariner超家族(TSD:TA)存在于本研究所有的鱼类基因组中, 其中斑马鱼的TC1-Mariner全长拷贝数最多, 有42501个(占基因组中MITEs序列的48.11%), 红鳍东方鲀的TC1-Mariner全长拷贝序列最少, 只有19条(3.8%)。PHIS超家族(TSD: TWA, CTWAG, CWG)存在29个物种中, 海七鳃鳗的PHIS全长拷贝数最多, 为12868个(20.44%)。P超家族(TSD: 7—8 bp, TIR:CANRG)存在于30个鱼类物种中, 斑马鱼P超家族的全长拷贝最多, 为22414个(25.37%)。Kobolok超家族(TSD: TTAA, TIR: RG)存在于23个鱼类物种中, 墨西哥丽脂鲤的Kobolok超家族全长拷贝最多,为4647个(8.69%)。PiggyBac超家族(TSD: TTAA,TIR: CMY)存在于27个物种基因组, 日本青鳉全长拷贝最多, 有16951个(70.74%)。hAT超家族(TSD:5—8 bp, TIR: YARNG)除了象鲨和斑点叉尾鮰以外, 存在于31个物种基因组中, 全长拷贝数最多的是斑马鱼, 有6586个(7.46%)。Ginger超家族(TSD:4—5 bp, TIR: TGT)存在于除了半滑舌鳎以外的32个物种基因组中, 大西洋鲑的Ginger拷贝数最多,为4320个(6.39%)。其他的MITEs超家族, 比如Merlin, 只存在于犀角金线鲃的基因组, 拷贝数为547个(7.51%)。Sola2超家族只存在于海七鳃鳗、蒲氏盲鳗、猬鳐三个物种基因组中, 拷贝数最多的是海七鳃鳗, 有3084个(4.90%)。除了上述的MITEs超家族, 还有部分MITEs拷贝由于TSD和TIR无任何规律, 无法鉴定出家族, 全部归为unknown。总而言之, 鱼类基因组中TC1-Mariner超家族占比最大, 与之前的研究结果相符合[16]。
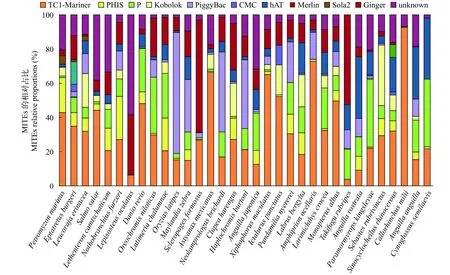
图2 33种鱼类基因组中MITEs超家族分布情况Fig. 2 Distributions of MITEs superfamily in the genome of the 33 fish species
2.3 MITEs插入时间估计
根据K2P模型[26]和公式T=K/2r[27], 使用软件DAMBE计算不同全长拷贝与一致序列之间的分化度(K), 并用perl脚本统计各物种中MITEs的插入时间。结果表明, 大多数鱼类的MITEs全长拷贝(79.58%)插入基因组的时间约为4百万年前, 也有少数的MITEs全长拷贝插入时间更为古老(20.42%,图3)。
在鱼类进化过程中, 基因组的转座子数量也在发现改变, 从得到的MITEs插入时间结果分析,7.5百万年前大部分鱼类基因组中的MITEs拷贝较少, 随着时间的推移, 部分物种出现了MITEs数量激增的现象, 鱼类基因组的MITEs插入时间相对更集中在2百万—0.5百万年前, 如日本七鳃鳗、海七鳃鳗、猬鳐、矛尾鱼、斑点雀鳝、斑马鱼、小丑鱼、贝氏隆头鱼在此期间都有一个MITEs集中扩增的表现。其中斑马鱼的MITE拷贝数就曾在7.5百万—6.5百万年前有短暂的数量上升, 到6.5百万—6百万年前出现MITEs数量减少(仅有1889个全长拷贝), 而后的4百万年间, 斑马鱼的MITEs数量又缓慢的开始增加, 直到2百万—1.5百万年前达到一个扩增的峰值(11596个全长拷贝), 到近期MITEs数量又逐步降低。同样, 大西洋鲑鱼的MITEs数量也是缓慢的上升, 从7.5百万—7百万年前的435个MITEs全长拷贝, 经过5百万年, 到2.5百万—2百万年前时达到数量峰值(10841个全长拷贝), 然后MITEs序列又逐渐减少。不同的是, 有些物种在进化过程中,MITEs会经历两次扩增“爆发”期(拷贝数量达到峰值, 而后下降), 比如墨西哥丽脂鲤、斑马鱼、罗非鱼、象鼻鱼和犀角金线鲃。除此之外的其他物种都仅有一次MITEs扩增“爆发”。
2.4 MITEs在基因组中的分布
鱼类基因组中的MITEs转座子具有较高的丰度, 为了保证结果的准确性, 仅选取有完整TIR的MITEs全长拷贝进行插入位置分析。结果显示(表2),鱼类基因组的MITEs全长拷贝插入到基因内部的数量十分丰富(计算插入时, 以转座子所处位置为准, 较长的转座子可能出现重复计数的现象, 对结果无影响), 例如斑马鱼插入到基因的有12885个。其中矛尾鱼、斑点雀鳝、象鼻鱼、大西洋鲱、斑马鱼、犀角金线鲃、墨西哥丽脂鲤、斑点叉尾鮰、大西洋鲑鱼、红鳍东方鲀、黄鳝、半滑舌鳎、贝氏隆头鱼、大黄鱼、罗非鱼、布氏新亮丽鲷、斑马宫丽鱼、红丽鱼、伯氏朴丽鱼、小丑鱼、花斑剑尾鱼、弗氏假鳃鳉和日本青鳉基因组中的MITEs都有插入到外显子的情况。对这部分基因进行注释, 发现象鼻鱼、斑马鱼、墨西哥丽脂鲤、弗氏假鳃鳉、黄鳝、大黄鱼、小丑鱼、罗非鱼的MITEs都插入到能够编码具有锌指结构的蛋白(Zinc finger)的序列中, 这些序列编码的蛋白大多与基因表达相关; 墨西哥丽脂鲤有一条MITEs插入到能编码视蛋白对应的序列中; 斑点雀鳝的有一条MITEs插入到编码脂肪包被蛋白(与肥胖相关)的序列中等等。但后续还需要更详细的分析和实验验证, 才能明确MITEs是否对基因表达有影响。
3 讨论
鱼类占现有脊椎动物总数的50%以上, 它丰富的物种多样性, 广阔的物种分布以及古老的进化地位都使它成为生物进化历史上不可或缺的一部分。随着测序技术的发展, 越来越多的鱼类基因组数据被公布, 基因组中的转座子序列也得到了生物学家的关注, 目前已完成52种鱼类的转座子鉴定工作[21], 鱼类特有的转座子数据库也已经发布[34]。MITEs属于非自主型的DNA转座子, 已有研究表明MITEs起源于自主DNA转座子, 并且其在基因组中存在丰富的拷贝[35], MITEs的插入对“宿主”的基因功能也会造成影响[36—38]。我们使用de novo预测和结构预测相结合的方法对33种鱼类基因组进行MITEs转座子预测, 然后用Bioedit手工鉴定, 有效降低了预测结果的假阳性, 为后续插入时间和插入位置分析提供保障。
鱼类基因组中MITEs含量差异较大(0.11%—21.18%), 比植物基因组(0.01%—9.98%)[10]和昆虫基因组(0.00%—6.24%)[12]中的MITEs含量更高。MITEs在基因组中的拷贝数较多, 斑马鱼中全长拷贝可达88337个, 其原因可能有两个, 一方面, 辐鳍鱼纲的鱼类比软骨鱼纲的含有更多DNA转座子; 另一方面, 斑马鱼是模式物种, 基因组质量高(基因组组装到染色体, Contig N50为854399 bp), 预测结果也更好。在本研究中, 猬鳐基因组鉴定的MITEs含量很低(0.11%), 这可能与软骨鱼基因组中DNA类转座子总体含量偏低及基因组组装质量不高(基因组Contig N50为665 bp)有关。同时, 发现鱼类基因组大小和基因组中MITEs含量呈正相关(r=0.124,P<0.05), 与植物和昆虫的情况类似[10,12], 因此可知MITEs的高拷贝数、高含量对基因组大小的进化有一定贡献。而鱼类基因组中的MITEs超家族主要是TC1-Mariner (TSD: TA), 它在植物[10,30]、昆虫[12]、病毒[14]等基因组中都含有, 也是最初发现的MITEs之一[30], 在MITEs含量中占主导地位。

图3 33种鱼类基因组中MITEs插入时间估计Fig. 3 MITEs insertion time estimation in the 33 fish genomes
MITEs与其他转座子不同, 它在基因组中倾向插入到基因内部或附近(基因侧翼5 kb区域内)[39],本研究结果显示, 大部分MITEs全长拷贝都插入到“宿主”的基因内部, 符合MITEs的插入特征。但MITEs的插入可能会使基因表达发生变化, 例如水稻的mPing转座子插入Rurm1基因的内含子中, 使水稻颖叶更加细长[40]。在鱼类基因组中, 很多MITEs序列插入到基因的内含子, 甚至有23种鱼类的MITEs序列插入到基因的外显子, 这在昆虫和植物中很少见。所以, 还需要对插入到基因的MITEs序列进行深入研究, 确定它们对“宿主”的基因表达和功能是否有影响。
MITEs能够以“剪切-粘贴”模式高效率转座[35],依靠对应的自主转座子编码的转座酶, 在短时间内增加拷贝数。MITEs插入时间分析结果显示, 鱼类基因组的MITEs大多在2百万—0.5百万年前出现了一次扩增“爆发”期, 这期间MITEs的数量快速增加并达到峰值, 之后数量又逐渐减少。转座子“生命周期(Lifecycle)”假说[41]认为, 转座子进入“宿主”后有一个快速扩增时期, 这期间转座子拷贝数急剧增加, 同时产生突变导致部分转座子失活。接着进入成熟期, 转座子拷贝数增加和丢失的速率达到平衡。最后进入衰老期, 自主型和非自主型转座子丢失, 不再扩增, 衰老期持续的时间最长可达几百万年。我们得到的MITEs扩增结果与这个假说相符合, 这种情况也说明MITEs虽然能通过转座增加拷贝, 但并不能一直扩增下去, 若这种扩张给“宿主”带来负面影响, “宿主”会有一些沉默机制阻止MITEs扩增, 如piRNA(非编码小RNA)通过诱导特殊位点的组蛋白修饰能对转座子转座进行有效抑制[42]。若这类MITEs对转座子有利, 则会继续留存在基因组中, 例如斑马鱼基因组中MITEs序列有两次扩增的迹象。因此在鱼类基因组中, MITEs的扩增会影响基因组大小, 同时“宿主”也有相应的机制控制MITEs的数量, 而MITEs在基因组中的插入会对物种的进化、基因的结构和功能产生影响, 因此鉴定鱼类的MITEs是研究鱼类基因组中转座子功能的基础。

表2 MITEs在鱼类基因组中的分布Tab. 2 Distributions of MITEs in the fish genomes
4 结论
对33种鱼类基因组的MITEs进行预测、鉴定和分类, 发现鱼类的MITEs含量差异显著, MITEs含量与鱼类基因组大小呈正相关。在33种鱼类中, 均含有大量TSD为TA的MITEs序列, 与昆虫、植物类似。鱼类基因组中的MITEs插入时间大多在4百万年前, 更倾向于插入在基因内部, 这可能会对基因表达调控产生影响。我们阐述了鱼类基因组中MITEs的分布情况, 为后续鱼类基因组中MITEs功能的研究奠定了基础。

