多参数预测人巨细胞病毒临床病毒株UL145基因B细胞表位
伍苑宾 胡兢晶 王波 苏海浩 谭琪琪

[摘要] 目的 应用多参数预测及分析人巨细胞病毒(Human cytomegalovirus,HCMV)临床病毒株UL145基因B细胞表位。 方法 根据人巨细胞病毒UL145基因氨基酸序列预测其二级结构,结合跨膜结构、亲水性、可及性及抗原性方案等参数综合预测UL145基因B细胞表位。 结果 HCMV pUL145氨基酸等电点为6.64,二级结构以无规则卷为主,1~130位氨基酸均为膜外区域,结合多种预测方法分析提示,88~99位氨基酸序列内可作为HCMV UL145基因的B细胞候选表位。 结论 多参数预测提示88~99位氨基酸序列为HCMV UL145基因B细胞表位预测序列,为进一步制备相应的抗体提供了重要依据。
[关键词] 人巨细胞病毒;UL145;B细胞表位;生物信息学分析
[中图分类号] R39;R71 [文献标识码] A [文章编号] 1673-9701(2020)35-0027-04
[Abstract] Objective To predict and analyze B cell epitope of UL145 gene of human cytomegalovirus (HCMV) clinical strain by using multi-parameter method. Methods According to the amino acid sequence of HCMV UL145 gene, the secondary structure was predicted. Combined with transmembrane structure, hydrophilicity, accessibility, antigenicity scheme and other parameters, the B cell epitope of UL145 gene was predicted comprehensively. Results The amino acid isoelectric point of HCMV pUL145 was 6.64, the secondary structure was dominated by random curling, and amino acids 1-130 were all extra-membranous regions. Combined with various prediction methods, it was suggested that amino acid sequences at positions 88-99 could be used as B cell candidate epitopes of HCMV UL145 gene. Conclusion Multi-parameter prediction indicates that the amino acid sequences at positions 88-99 are predicted sequences of B cell epitopes of HCMV UL145 gene, which provide an important basis for further preparation of corresponding antibodies.
[Key words] Human cytomegalovirus; UL145; B cell epitope; Bioinformatics analysis
人巨細胞病毒(Human cytomegalovirus,HCMV)属疱疹病毒β亚科,为双链DNA病毒,在多数人群中为隐性感染或潜伏感染,但在新生婴儿、免疫低下或抑制的人群中可引起症状性感染,可导致较高的死亡率[1]。HCMV是新生儿最常见的病原体,也是宫内感染的常见病原体,约0.5%的婴儿会感染HCMV,其中有8%~10%的婴儿在出生后出现症状性感染,感染后患儿可出现黄疸、肝脾肿大、发育延迟等临床症状,并且对中枢神经系统造成不可逆损伤,如感音神经性听力损失、小头畸形及脑瘫[2]。且有研究显示,HCMV感染与动脉粥样硬化、格林-巴利综合征、胶质母细胞瘤及癌症的发生相关[3]。目前治疗HCMV感染的药物主要有更昔洛韦(GCV)、膦甲酸钠(PFA)、缬更昔洛韦(VCG),但由于耐药性及其毒副作用,在临床上应用具有一定的限制[4]。疫苗方面,在最近的Ⅱ期临床试验中,以水包油乳MF59佐剂(gB/MF59)配制的重组gB疫苗产生的抗体效价与自然感染所产生的效价相当。然而,该疫苗在预防HCMV血清学阴性的妇女感染及减轻移植受体者的病毒血症方面仅显示出中等效力[5-6]。
HCMV基因组大小约为236 kb,编码750个开放阅读框(ORF)[7]。研究发现,与实验室AD169病毒株相比,HCMV临床病毒株存在一个包含至少有19个ORF的UL/B'区域,其大小约为15 kb[8],而AD169病毒株中并不存在此区域,并且有研究表明,HCMV UL/B'区域内的基因具有调节NK细胞的功能,从而实现免疫逃避[9]。这些研究显示,此区域内的基因功能可导致HCMV产生致病性感染。目前已有发现UL148与HCMV免疫逃避有关[10-11],UL146能诱导中性粒细胞的趋化[12],但UL145基因功能仍不明确。课题组前期已对UL145基因进行初步研究,发现其基因及编码的氨基酸序列极为保守,存在多个翻译后修饰位点,因此,UL145基因可能在感染或免疫逃避过程中起重要作用[13]。本研究通过应用多参数预测UL145基因抗原表位,为研究其蛋白功能提供基础,现报道如下。
1 材料與方法
1.1 材料来源
HCMV UL145氨基酸序列:检索自NCBI数据库临床低传代HCMV D3病毒株,序列号为DQ180367.1。D3病毒株为本课题组前期对UL145基因全序列进行了测序研究,提交给GenBank并被收录。
1.2 方法
1.2.1 二级结构预测 对UL145二级结构进行预测,β-转角及无规则卷区域作为预测区域。
1.2.2 跨膜结构预测 对UL145进行跨膜结构预测,非跨膜结构区域作为预测区域。
1.2.3 亲水性、可及性、极性、柔韧性、抗原性参数预测 选取多参数进行综合分析,以高于阈值的区域作为候选表位。
1.2.4 综合分析 通过在线软件预测抗原性区域;同时结合吴玉章等[14]建立的抗原指数分析UL145基因的B细胞候选表位。
2 结果
2.1 pUL145氨基酸序列分析
pUL145包含130个氨基酸残基,PI为6.64,相对分子质量为14 KD。序列如下:MYGVLAHYYS FISSPSVMVN FKHHNAVQLL CARTRDGTTG WERLTHHASY HANYGAYAVL MATSQRKSLV LHRYSAVTAV ALQLMPVEML RKLDQSDWVR GAWIVSETFP TSDPKGFWSD DDSSMGGSDD。
2.2 pUL145二级结构分析
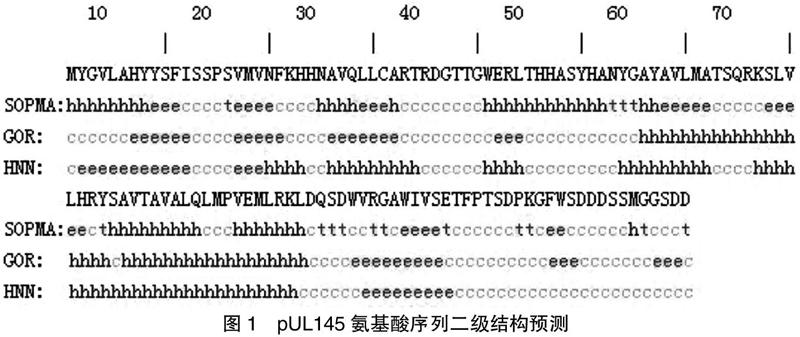
结合多种预测方案,无规则卷及β-转角区域多位于35~40、108~116、119~124位氨基酸。见图1。
2.3 pUL145跨膜结构预测
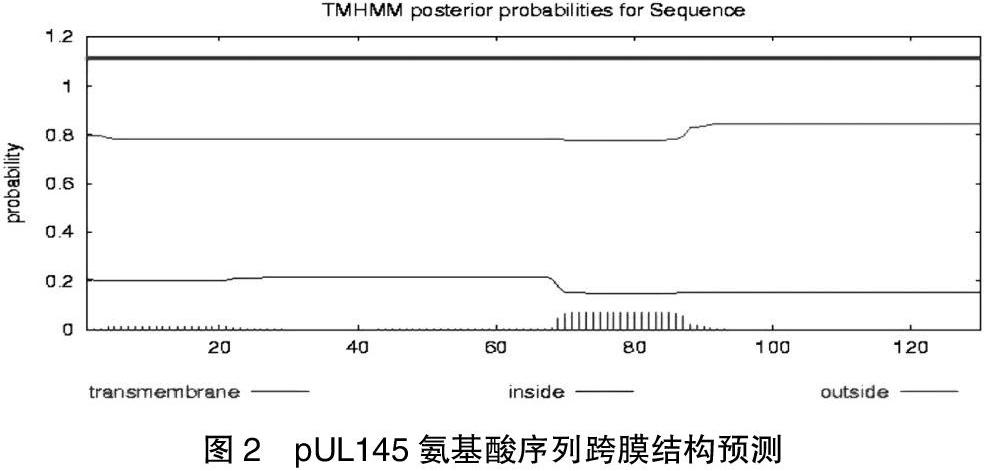
1~130位氨基酸均为膜外区域,作为抗原表位预测区域。见图2。
2.4 pUL145氨基酸的多参数预测
高于阈值4~6个氨基酸可作为候选区域。见图3、表1。
2.5 pUL145 抗原指数预测
通过ABCpred服务器对pUL145进行抗原指数分析,结果显示表位区域在115~130、80~95、37~52、108~123可能性较大;结合其二级结构、跨膜区域以及多参数预测,32~40、88~99、109~126区域可能为B细胞候选区域;将候选的3段区域计算各氨基酸残基的抗原性总值,取其平均抗原指数,取分值高区域作为候选表位。可见88~99氨基酸区域分值较高,考虑pUL145 B细胞表位位于88~99位氨基酸区域内或附近。见表2。
3 讨论
在健康人群中,受自身免疫系统的调控,HCMV感染通常是无症状的。但在孕期及免疫受损的人群(如移植受体者或HIV感染者)中可能具有严重的致病性。HCMV的免疫调控通过下调人类白细胞抗原(HLA)-Ia分子,使被感染的细胞无法被T细胞识别从而形成免疫逃避[15]。UL/b'区编码多种干扰NK细胞识别的免疫逃避蛋白,例如UL135可破坏免疫突触,UL141和UL142可阻止NK细胞活化配体的表达[16],UL148通过下调MICA,从而避免NK细胞杀死HCMV感染的细胞[17]。课题组前期研究发现,UL145基因及其序列极为保守,一般保守基因与病毒生存关系更为密切,但关于其相关的功能研究较少。
B细胞表位(B cell epitope)是能被B细胞抗原受体或抗体特异性识别并相互结合的特殊化学基团,是引起免疫应答和免疫反应的基本单位。目前B细胞表位在疾病的诊断及疫苗开发中有一定的优势。抗原表位可分为B细胞抗原表位和T细胞表位;并且根据表位结构的不同可分为连续性及不连续性表位[18]。B细胞抗原表位的预测方法有肽探针扫描技术、蛋白质“切割”法、噬菌体展示技术等[19],但总体这些方法具有昂贵、费时及准确率低等劣势。目前已经开发了多种计算方法来辅助或替代实验方法,包括BepiPred、DiscoTope、CBtope和ABCpred,其中ABCpred其敏感性、特异性、阳性率分别为67.14%、64.71%、65.61%[20-21]。但单参数预测方法准确率不高,通过上述结果可见,各方法预测B细胞表位结果有所不同,因此,需要结合多种方法进行综合分析以避免局限性。
目前,B细胞表位的预测主要结合二级结构、亲水性、可及性、抗原性等进行综合预测。二级结构预测可见pUL145二级结构中α-螺旋占33.8%,β-折叠占20.0%,无规则卷占34.6%,β-转角占11.5%,结果以无规则卷为主;位于表面的亲水性区域能更好地与抗原表位接触;抗原与抗体结合时蛋白构象会出现改变,因此抗原表位需同时具备一定的柔韧性。应用多种方案综合分析pUL145抗原表位,能提高正确性。
目前,对UL145基因所编码的蛋白功能及在HCMV感染中的作用研究较少。通过对UL145氨基酸序列进行多参数预测,各方案预测的候选表位存在差异性。对各方案进行综合分析,可见3段候选表位,分别为32~40、88~99、109~126区域。将这3段潜在的抗原表位通过抗原指数法分析,认为HCMV UL145 B细胞抗原表位存在于88~99位氨基酸区域内,为研究UL145基因功能提供了重要依据。
[参考文献]
[1] Griffiths P,Baraniak I,Reeves M. The pathogenesis of human cytomegalovirus[J].J Pathol,2015,235(2):288-297.
[2] Britt W. Maternal immunity and the natural history of congenital human cytomegalovirus infection[J].Viruses,2018,10(8):405.
[3] Simanek AM,Dowd JB,Pawelec G,et al. Seropositivity to cytomegalovirus,inflammation,all-cause and cardiovascular disease-related mortality in the United States[J]. PLoS One,2011,6(2):e16103.
[4] Amina A.Antiviral treatment of cytomegalovirus infection[J]. Infect Disord Drug Targets,2011,11(5):475-503.
[5] Nelson CS,Huffman T,Jenks JA,et al. HCMV glycoprotein B subunit vaccine efficacy mediated by nonneutralizing antibody effector functions[J]. Proc Natl Acad Sci USA,2018,115(24):6267-6272.
[6] Baraniak I,Kropff B,Ambrose L,et al.Protection from cytomegalovirus viremia following glycoprotein B vaccination is not dependent on neutralizing antibodies[J]. Proc Natl Acad Sci USA,2018,115(24):6273-6278.
[7] Stern-Ginossar N,Weisburd B,Michalski A,et al. Decoding human cytomegalovirus[J]. Science,2012,338(6110):1088-1093.
[8] Cha TA,Tom E,Kemble GW,et al. Human cytomegalovirus clinical isolates carry at least 19genes not found in laboratory strains[J]. Journal of Virology,1996,70(1):78-83.
[9] Patel M,Vlahava VM,Forbes SK,et al. HCMV-encoded NK modulators:Lessons from in vitro and in vivo genetic variation[J]. Front Immunol,2018,9:2214.
[10] Nightingale K,Lin KM,Ravenhill BJ,et al. High-definition analysis of host protein stability during human cytomegalovirus infection reveals antiviral factors and viral evasion mechanisms[J]. Cell Host Microbe,2018,24(3):447-460.
[11] Wang ECY,Pjechova M,Nightingale K,et al. Suppression of costimulation by human cytomegalovirus promotes evasion of cellular immune defenses[J]. Proc Natl Acad Sci USA,2018,115(19):4998-5003.
[12] Saederup N,Mocarski ES Jr.Fatal attraction:Cytomegalovirus-encoded chemokine homologs[J]. Curr Topic Microbiol Immunol,2002,269:235-256.
[13] 胡兢晶,王波,蘇海浩,等. 广州地区先天性感染婴儿人巨细胞病毒UL145基因多态性研究[J]. 中国妇幼保健,2010,25(20):2862-2865.
[14] 吴玉章,朱锡华. 一种病毒蛋白B细胞表位预测方法的建立[J]. 科学通报,1994,39(24):2275-2279.
[15] Raj A,Kumar P. Subversion of immune response by human cytomegalovirus[J]. Front Immunol,2019,10:1155.
[16] Patel M,Vlahava VM,Forbes SK,et al. HCMV-encoded NK modulators:Lessons from in vitro and in vivo genetic variation[J]. Front Immunol,2018,9:2214.
[17] Dassa L,Seidel E,Oiknine-Djian E,et al. The human cytomegalovirus protein UL148A downregulates the NK cell-activating ligand MICA to avoid NK cell attack[J]. Journal of Virology,2018,92(17):e00162-18.
[18] Barlow DJ,Edwards MS,Thornton JM.Continuous and discontinuous protein antigenic determinants[J].Nature,1986,322(6081):747-748.
[19] Xu XL,Sun J,Liu Q,et al. Evaluation of spatial epitope computational tools based on experimentally-confirmed dataset for protein antigens[J]. Chin Sci Bull,2010,55:2169-2174.
[20] Ansari HR,Raghava GP. Identification of conformational B-cell Epitopes in an antigen from its primary sequence[J].Immunome Res,2010,6:6.
[21] Saha S,Raghava GP. Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J]. Proteins,2006,65(1):40-48.
(收稿日期:2020-07-07)

