一种植物基因组DNA快速提取方法的建立与评估
李 琳,罗淋淋,3,罗光宇,3,莫蓓莘,刘 琳
1)深圳大学生命与海洋科学学院,广东省植物表观遗传学重点实验室,广东深圳518060;2)深圳大学龙华生物产业创新研究院,广东深圳518060;3)深圳大学物理与光电工程学院,广东深圳518060
植物基因组脱氧核糖核酸(deoxyribonucleic acid, DNA)的提取是分子生物学实验的基础,然而植物基因型鉴定等需要大样本量基因组DNA的分子生物学实验,往往需要耗费大量时间和精力进行DNA提取.传统的DNA提取方式,如十二烷基硫酸钠(sodium dodecyl sulfate, SDS)法[1-2]、十六烷基三甲基溴化铵(cetyltrimethylammonium ammonium bromide, CTAB)法[3]和提取试剂盒等都存在步骤繁琐、耗时过长和试剂有毒有害等问题,且在提取过程中还会产生大量废液,不能满足大批量实验和现代分子生物学发展的要求.现已报道了多种优化的快速基因组提取方法,常规做法是更换个别试剂或简化特定步骤[4-5],但对大规模实验来说,这些方法仍较繁琐,且适用面较窄,主要用于水稻[6-7]和拟南芥[8]等少数模式植物.为此,本研究借鉴已有的快速提取植物基因组DNA的方法[9-14],经过大量实验和对体系不断的调整优化,最终确立了一套适用于聚合酶链式反应(polymerase chain reaction, PCR)鉴定的植物基因组DNA快速提取方法.该方法只需将少量植物叶片(以水稻为例,取样量仅为3 mm×5 mm~3 mm×10 mm)放入96孔板中,加入120~150 μL提取液和2~3粒直径为2 mm的氧化锆珠,在高通量组织研磨仪上震荡破碎1~2 min后得到植物提取液,即可直接应用于PCR反应.本方法成本低、PCR产物扩增效率高,且结果稳定,适于多种处于不同时期和状态的植物基因组DNA的提取,尤其适于大样本量的实验并能显著缩短实验时间.
1 材料与方法
1.1 实验材料
本研究以水稻(OryzasativaL.)日本晴、玉米(ZeamaysL.)B73、拟南芥(ArabidopsisthalianaL.)Columbia生态型、马铃薯(SolanumtuberosumL.)成熟期的叶片为实验材料.同时,将水稻和拟南芥的地上及地下部分分别取材进行实验.
1.2 提取缓冲液的配置
CTAB法提取缓冲液配方及提取方法见文献[14].本实验所使用DNA 10×提取缓冲液母液按100 mmol/L Tris (pH=9.5)、5 mmol/L 乙二胺四乙酸(ethylened diaminetetraacetic acid, EDTA)、5 mmol/L NaCl和1 mmol/L KCl配方配置,使用时使用去离子水稀释至1×即可.该浓缩型提取液储存于4 ℃冰箱可保存6~10个月.
1.3 植物基因组DNA的提取和检测
用剪刀剪取3 mm×5 mm~3 mm×10 mm的植物叶片,置于已加入2~3颗直径为2 mm的氧化锆珠和120~150 μL提取缓冲液的96孔板中.将叶片压于管底,勿紧贴管壁.盖好配套硅胶盖后上下摇晃检查密封性,在高通量组织研磨仪(上海净信 Tissuelyser-192)上以60 Hz频率震荡破碎10~120 s(叶片越新鲜幼嫩振荡时间越短),再以4 000 r/min速率低速离心60 s(离心目的是沉淀杂质,该步骤也可省略),缓慢揭去硅胶盖,清理96孔板表面防止样品交叉污染,所得上清可直接用于PCR扩增或于-20 ℃储存.如样本数目较少,可选择0.2 mL八联排PCR管或1.5/2.0 mL的离心管作为提取容器,但需根据要求调整研磨条件,研磨时间过久可能击碎离心管壁,导致样品流失.
得到植物基因组DNA后,使用Invitrogen Qubit 3.0荧光定量仪和Qubit dsDNA HS Assay 试剂盒(Q32854)对DNA浓度进行测定,检测方法参照说明书.同时,本研究从DNA的保存时间、植物组织部位和DNA完整度方面检测了该提取体系的适用性,并对结果的可靠性进行评测.
1.4 PCR反应体系和条件
在实验操作过程中,PCR反应体系可根据物种和实验用途进行稍作调整.一般情况下,扩增所用的模板量为0.5~1.0 μL,可根据不同植物类型及实验需求进行适当调整.本实验使用的Taq酶为Vazyme 2×Taq master mix,扩增体系为:10× PCR buffer: 2 μL;引物(10 nmol/L): 0.8~1.0 μL;dNTP (10 mmol/L): 0.5~1.0 μL;DNA提取液: 0.5~1.0 μL;Taq酶: 0.3~0.5 μL;双蒸水补足至20 μL.PCR反应程序:95 ℃预变性3 min,95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,35~38个循环,最后72 ℃延伸5 min.对于扩增引物的设计无特殊要求,遵循正常扩增反应的设计规则即可.由于使用该方法提取的植物基因组DNA浓度较低,建议扩增循环数设为35个以上,以保证获得足量的产物.
1.5 SNP和SSR检测
以水稻为例,通过RiceVarMap(http://ricevarmap.ncpgr.cn/v1/contact/)查找单核苷酸多态性(single nucleotide polymorphisms, SNP)位点,设计引物对多个水稻品种进行PCR鉴定和测序;通过Gramene(http://archive.gramene.org/markers/microsat/50_ssr.html)提供的水稻简单重复序列标记(simple sequence repeat, SSR)标准引物序列,随机挑选引物对多个水稻品种进行PCR鉴定和琼脂糖凝胶电泳检测.所检测的水稻品种信息见表1,引物设计和对应基因信息见表2.

表1 SNP和SSR检测所用水稻品种信息表

表2 检测引物表
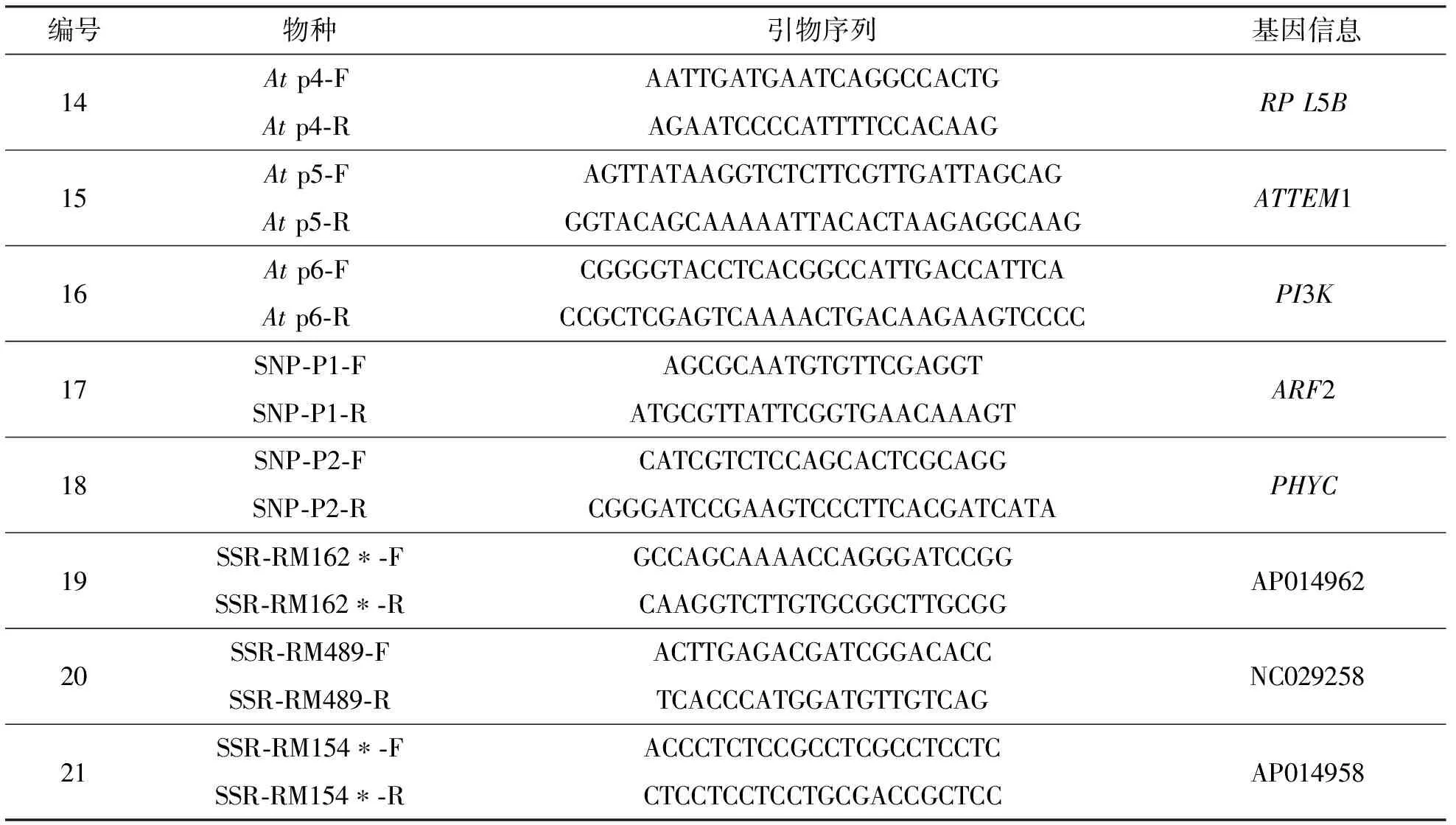
(续表2)
1.6 引物信息
表2列出了实验所用的引物.其中,编号1~4的引物用于检测不同物种中利用快速提取方法得到的基因组DNA保存时间的实验;编号3~4的引物同时也用于检测在拟南芥和水稻中快速提取法所得基因组为模板的PCR扩增产物测序质量和对植物不同组织的适用性实验;编号5~16的引物用于检测快速提取法所得植物基因组DNA的完整性实验;编号17~21的引物用于检测快速提取法对SNP和SSR分析适用性实验.
2 结 果
2.1 快速提取法获得的DNA质量满足PCR扩增及测序鉴定的要求
用本研究提取法对水稻、拟南芥、玉米和马铃薯进行测试.由于采用微量提取方法,样品的取样量少,所得基因组DNA浓度低,难以被琼脂糖凝胶电泳检测到.通过Qubit荧光定量仪可对提取所得基因组DNA进行定量,测定浓度为3~4 ng/μL(150 μL体系),总质量为450~600 ng.
该快速提取方法对PCR体系无特殊要求,采用常规体系即可.由于植物基因组DNA快速提取法是粗提DNA,未经纯化,为保证检测结果准确、稳定且可重复,PCR添加模板量不宜太多.20 μL体系中,不同物种PCR反应模板量建议请扫描论文末页右下角二维码查看表S1.
拟南芥和水稻样品利用本研究方法和CTAB法所提取DNA为模板的PCR扩增测序结果请扫描论文末页右下角二维码见图S1.4个物种利用两种提取法所做的PCR扩增结果无明显区别,所得测序峰图都尖锐且单一,表明两种提取法所得基因组DNA的PCR扩增产物测序质量相似.利用该方法所得的基因组DNA质量完全满足测序鉴定等分子实验的要求,可进行进一步的实验研究.
2.2 快速提取法获得的DNA可长时间保存
以拟南芥、水稻、玉米和马铃薯为材料,利用快速提取法获得基因组DNA,对提取当天的样品和-20 ℃下保存了7、30、60和120 d的样品,分别进行PCR检测,并设置CTAB法提取的DNA作为对照,结果如图1所示.其中,bp为碱基对(base pair).从图1可见,所有样品都有清晰整齐的条带,表明该方法所得基因组DNA可在-20 ℃下完整保存至少4个月,能够满足一般分子生物学实验的需要.

图1 利用植物基因组DNA快速提取法获得的植物基因组DNA在不同保存时间下的PCR检测
2.3 快速提取法的适用性
2.3.1 快速提取法适用于植物不同组织和不同生长状态的叶片
为检测该基因组提取方法对植物不同组织和生长状态的适用性,本研究以拟南芥和水稻不同组织和不同状态的叶片为材料,提取基因组DNA进行PCR扩增检测,结果如图2.本研究选取了叶片状态相对较差的老叶、黄叶及根部作为实验材料,对样品研磨后进行PCR检测.结果显示,所有样本皆能成功提取到基因组DNA,且检测条带明亮、单一,表明该方法适用于植物不同组织和生长状态基因组DNA的提取鉴定.
2.3.2 快速提取法获得的基因组DNA完整性高
使用本研究方法获得的PCR产物片段都少于1 000 bp,为验证所提取基因组DNA的完整性,以拟南芥和水稻为例,通过扩增不同长度的DNA片段进行验证,如图3.分别选取拟南芥和水稻多个不同基因,设计引物扩增100(p1)、450(p2)、750(p3)、1 000(p4)、2 000(p5)和4 000 bp(p6)基因片段,PCR检测结果显示所有条带清晰单一稳定,表明利用本研究方法所提取的基因组DNA具有较高完整性,适用于各种长度片段的常规扩增实验.

图2 植物基因组DNA快速提取法对水稻、拟南芥不同状态叶片和不同组织部位的适用性检测

图3 植物基因组DNA快速提取法所得植物基因组DNA完整性检测
2.3.3 快速提取法适用于SNP和SSR分析
为进一步检测该快速提取方法得到的基因组DNA是否适用于SNP和SSR分析,本研究针对实验室现有的多个水稻品种设计引物进行检测(水稻品种和引物相关信息见表1和表2).
选取8个水稻品种进行检测,图4(a)和图4(b)分别展示了使用快速提取法和CTAB法得到的粳稻CAU2和籼稻金南特B两个品种基因组DNA对ARF2和PHYC基因的PCR扩增测序结果,限于篇幅,其他6个水稻品种的SNP测序结果请扫描论文末页右下角二维码见图S2.其中,序列上方数字为此段序列在基因中的位置.结果显示,检测的不同水稻品种在该基因中的单核苷酸突变与在RiceVarMap提供的基因突变位点相同,用CTAB法提取的植物基因组DNA和快速提取方法得到的测序结果无差异.针对SSR分析实验,参考Gramene网站(http://archive.gramene.org/markers/microsat/50_ssr.html)提供的水稻SSR标记标准引物序列,随机挑选3对引物对8个水稻品种进行PCR鉴定,结果如图5.琼脂糖凝胶结果显示,不同品种之间的SSR序列是有区别的,且用CTAB法提取的植物基因组DNA和本研究提出的快速提取方法得到的结果无差异,表明该植物基因组DNA快速提取法稳定可靠,适用于SNP和SSR的分析检测.

图4 快速提取法和CTAB法获得的水稻基因组DNA的SNP检测结果

图5 利用快速提取法获得的水稻基因组DNA 进行SSR分析
3 结论和讨论
在实验方法不断优化调整的过程中,总结出如下植物基因组DNA快速提取法在操作中的注意事项:① 由于样品采取量少,植物组织未完全破碎,提取到的基因组DNA浓度与CTAB法相比较低,使用琼脂糖凝胶电泳检测微量基因组DNA时无明显条带产生,但并不影响PCR检测;② 植物叶片薄小易紧贴在管壁,氧化锆珠可能不易完全接触并破碎,因此样品破碎后需检查破碎情况,个别样本可能需重复破碎操作,根据实际情况选择是否需要离心,针对比较干燥不易破碎的样品,可先加水浸泡后再操作;③ 在揭去硅胶盖前应先离心,将管壁上的液体离心至管底,同时揭开硅胶盖时须缓慢小心;④ 在PCR过程中吸取模板时要尽量吸取上清,避免吸到杂质,以保证不影响Taq酶的活性和PCR产物的质量;⑤ 对于不同的物种和同一物种不同部位的基因组DNA提取,可根据实际情况调整氧化锆珠数量、高通量组织破碎仪频率及破碎时间等参数,以制备高质量的DNA;若所得样品较少,可使用八联管代替96孔板,由于材料不同,破碎结果差别也较大,用八联管经过高通量组织研磨仪与96孔板相比,样品破碎程度更好;⑥ 由于不同物种组织的特异性,取样量和破碎时间稍有差别,做大批量实验前可先做预实验来确定最佳实验方案;⑦ 为保证实验的准确性,样品应避免多次反复冻融,减小对基因组DNA保存时间的影响.
本研究提出的基因组DNA快速提取方法适用于多种植物以及植物不同组织和生长状态基因组DNA的提取,且提取到的基因组DNA片段完整,PCR扩增结果准确且重复性好.该植物基因组DNA快速提取法可大批量、快速的对多种植物进行基因型鉴定和品种鉴定,利用96孔板操作可提高每批次样品检测的通量.与CTAB法相比,该方法操作步骤简单、耗时少,不需要使用氯仿等有毒有害的有机溶剂,对样本的质量要求较低,所需样本量少,可大幅节约时间和物质成本,提高工作效率.与XU等[15-18]提出的植物基因组DNA快速提取方法相比,本方法利用96孔板和配套的硅胶盖能够提高检测通量,同时减少交叉污染.本研究对所提基因组DNA的质量、保存时间和片段完整性进行了检测,并与CTAB法提取结果对比,确定两种方法的扩增结果无明显差异.利用本研究方法提取不同植物物种和不同组织的基因组DNA,检测结果均显示良好,表明该方法对不同植物物种和不同组织具有普遍适用性,可满足大多数实验的要求.但该体系也存在一定的局限性,除本研究所检测的水稻、玉米、拟南芥和马铃薯4个物种,我们也在番茄(SolanumlycopersicumL.)、花生(ArachishypogaeaL.)、白菜(BrassicarapaL.)、大豆(GlycinemaxL.)等物种中利用此方法提取了它们的基因组DNA,但实验重复性不理想.因此,针对组织中酚和糖等含量较高植物,仍需进一步优化实验条件[19-20],以期提高结果的稳定性.通过调整和优化提取体系,该方法有望用于更多的植物物种,为遗传多样性研究和植物基因型鉴定等基础研究提供有效技术手段.

