花生AhbHLH63基因的克隆与表达分析
崔维佩 谷朝阳 唐桂英 徐平丽 柳展基 单雷


摘要:bHLH转录因子在调节植物生长发育中起着重要作用。本研究利用转录组测序结果从栽培种花生丰花1号中克隆得到AhbHLH63基因。序列分析显示,AhbHLH63基因有两个剪接体AhbHLH63-1.1和AhbHLH63-1.2,其ORF分别长1 188 bp和1 152 bp ,分别编码395个氨基酸和383个氨基酸组成的蛋白。亲缘关系分析表明,AhbHLH63蛋白含有一个预测的bHLH结构域,与其他植物的bHLH63具有较高的同源性,与苜蓿等的同源蛋白亲缘关系较近。实时荧光定量PCR分析表明,AhbHLH63-1.1和AhbHLH63-1.2表达模式基本一致,均为组成型表达,在花中表达量最高,茎和叶中次之,在根中表达量最低;在花生种子的不同发育时期,发育中期表达量较高。
关键词:花生;bHLH转录因子;基因克隆;表达分析
中图分类号:S565.2:Q785文献标识号:A文章编号:1001-4942(2019)09-0035-07
Cloning and Expression Analysis of [WTHX][STHX]AhbHLH63[WTHZ][STHZ] Gene in Peanut
Cui Weipei , Gu Chaoyang1,3, Tang Guiying1, Xu Pingli1, Liu Zhanji4, Shan Lei1, 2, 3
(1. Biotechnology Research Center, Shandong Academy of Agricultural Sciences/ Shandong Provincial Key Laboratory of
Crop Genetic Improvement Ecology and Physiology, Jinan 250100, China; 2. College of Life Science, Shandong Normal
University, Jinan 250014, China; 3. College of Life Sciences, Shandong University, Qingdao 266237, China;
4. Shandong Cotton Research Center, Jinan 250100, China)
Abstract bHLH transcription factors play the important roles in regulating plant growth and development. In this study, an AhbHLH[STBX]63 gene was cloned from cultivated peanut variety Fenghua 1 based on the results of transcriptomic sequencing. Sequence analysis showed that the AhbHLH[STBX]63 gene had two kinds of splicesome, AhbHLH[STBX]63-1.1 and AhbHLH[STBX]63-1.2[STBZ], which had the open reading frame as 1 188 bp and 1 152 bp and encoded 395 and 383 amino acids respectively. Phylogenetic analysis indicated that the AhbHLH63 protein contained a predicted bHLH domain, which had high sequence identity with bHLH63 or homologous proteins from other plants, and had the highest similarity with that of alfalfa. Quantitative RT-PCR analysis showed that the expression profile of AhbHLH[STBX]63-1.1 was consistent with that of AhbHLH[STBX]63-1.2[STBZ], which displayed the constitutive patterns. AhbHLH[STBX]63[STBZ] gene had the highest expression in flower, followed by stem and leaves, and the lowest in root. The highest expression level of AhbHLH[STBX]63 gene appeared at the mid-developmental stage during the development of peanut seed.
Keywords Peanut (Arachis hypogaea L.); bHLH transcription factor; Gene cloning; Expression analysis
bHLH(basic helix-loop-helix)转录因子广泛存在于真核生物,因含有bHLH结构域而得名。bHLH 结构域由大约50 ~ 60个氨基酸组成,N端约含10 ~ 15个氨基酸的碱性氨基酸区,C端约含40个氨基酸左右的α-螺旋-环-α-螺旋区(HLH区)[1],N端的碱性氨基酸区有DNA识别位点及DNA结合位点;约50%的HLH 区含高度保守的 H5-E9-R13 序列(His5-Glu9-Arg13) ,此结构对 bHLH 与DNA 的结合不可或缺,且HLH区两个相连的螺旋结构富含疏水氨基酸[2]。普遍认为bHLH转录因子以同源二聚體或异源二聚体的形式发挥功能[3],且其二聚体的特性由HLH 区内的疏水氨基酸及带电荷氨基酸残基之间的相互作用决定[4]。bHLH众多成员在调控植物生长发育的很多过程中都起着十分重要的作用[5],如光信号传导、激素合成[6]、抗逆以及根毛发育[7,8]等。大量研究证实,植物bHLH转录因子可与其他转录因子共同发挥功能,如bHLH类转录因子PIF3 和HFR1形成异源二聚体,进一步与光敏色素形成具有功能的三元复合体,调控植物生长萌发过程[9];bHLH1与MYB3转录因子相互作用协调调控类黄酮代谢途径[10]。植物 bHLH 转录因子发挥作用时既可起转录激活作用也可起抑制作用。Heisler等[11]通过对基因功能缺失突变株的研究证实bHLH转录因子基因SPATULA可以通过抑制下游基因的转录抑制种子的萌发。
近年来的研究发现,选择性剪接(alternative splicing)在植物生长发育调控中发挥着重要作用。拟南芥中61%具有内含子的基因存在选择性剪接[12];花生62%以上的脂肪酸合成与油脂积累相关基因存在选择性剪接[13]。mRNA通过选择性剪接可以增加转录本的多样性和编码蛋白的丰富度,从而精细调控许多生物学过程。
本研究根据花生转录组测序结果,克隆了花生AhbHLH63基因,发现该基因存在两种剪接方式。利用生物信息学详细分析该基因及编码的氨基酸序列,并利用qRT-PCR分析其在栽培种花生丰花1号不同组织及其不同发育时期的表达情况,以期为该基因的功能研究提供依据。
1 材料与方法
1.1 植物材料
花生品种丰花1号(由本实验室保存),种植于山东省农业科学院饮马泉实验基地。发育中的花生种子从果针入土开始计算,每间隔10 d取样一次,取10 ~ 70 d的去种皮种子为样品;采取盛开期的整朵花及出土后20 d的主根、主茎和幼嫩叶片为材料。取材后迅速冻存于液氮并于-80℃冰箱保存,用于RNA或DNA提取。
1.2 菌株、质粒和试剂
大肠杆菌(Escherichia coli)T1感受态细胞、pEASY-T1克隆载体、2×TransTaq High Fidelity(HiFi)PCR SuperMix,购自北京全式金生物技术有限公司;荧光实时定量PCR检测试剂盒PrimeScriptTM RT reagent Kit with gDNA Eraser、TB Green Premix Ex TaqTMⅡ、限制性内切酶,购于宝生物工程(大连)有限公司;凝胶回收试剂盒、质粒小提试剂盒、DL2000分子量标准,购自 GENERAY 生物工程(上海)有限公司;TRIzol Reagent,购自赛默飞世尔科技公司(上海);植物总RNA提取试剂盒,购自北京华越洋生物科技有限公司;由上海生工生物工程技术服务有限公司合成扩增引物;DNA测序由山东省农业科学院测序中心完成。
1.3 AhbHLH63基因cDNA和基因组DNA(gDNA)的克隆
以丰花1号幼嫩叶片为材料,选用CTAB法[14]提取DNA;利用华越洋(多糖多酚)试剂盒提取果针入土70 d的花生种子总 RNA。使用 PrimeScriptTM1st Strand cDNA Synthesis Kit反转录合成 cDNA 第一条链,置于-20℃备用。结合转录组数据,利用 Primer Premier 5.0 软件设计引物,经过 PCR检测筛选最终引物(表1)。
PCR反应体系包括HiFi Ⅱ/Ⅰ Mix 10 μL,cDNA/gDNA 1 μL,上下游引物(10 μmol/L)各 0.5 μL,加ddH2O补足至20 μL。反应程序:94℃ 5 min;94℃ 30 s,56.5℃ 30 s,72℃ 2 min (gDNA为4 min),30个循环;72℃ 7 min。琼脂糖电泳检测PCR产物,GENERAY试剂盒回收目的片段,连接到克隆载体pEASY-T1上,转化后,挑取阳性单克隆,进行酶切鉴定和序列测定。
1.4 AhbHLH63基因的序列分析
AhbHLH63蛋白的氨基酸组成、理论分子量和等电点利用https://web.expasy.org/protparam/在线软件分析;采用Expasy的在线工具ProtScale(https://web.expasy.org/protscale/)进行AhbHLH63蛋白的亲疏水性分析;通过NCBI的CDD(Conserved Domain Database,https://www.ncbi.nlm.nih.gov/cdd)进行结构域预测;AhbHLH63蛋白的亚细胞定位利用Cell-PLoc 2.0 (http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/ ) 进行预测;通过DNAMAN软件进行不同物种间氨基酸序列同源性分析,系统进化树由MEGA 6.0软件构建,采用Neighbor-Joining 法进行1 000次Boot-strap分析。
1.5 AhbHLH63基因的表达模式分析
以丰花1号花生的根、茎、叶、花和不同发育时期的种子为材料提取RNA,反转录合成cDNA第一条链;[JP2]以qRT-bHLH63-1.1F和qRT-bHLH63-1.1R及qRT-bHLH63-1.2F和qRT-bHLH63-1.2R为引物,以AhActin7为内参基因(表 1),进行荧光定量 PCR扩增,反应体系采用 TransStart Green qPCR SuperMix UDG说明书中20 μL体系,[JP]每个样品3个重复。采用ABI 7500 PCR仪器的两步法进行,程序如下:95℃ 10 min;95℃ 10 s,退火及延伸60℃ 30 s,40个循环。ABI 7500自带软件自动生成PCR扩增曲线。采用2-ΔΔCt方法进行基因表达量的计算。
2 结果与分析
2.1 AhbHLH63基因cDNA和gDNA克隆
[JP2]以提取的豐花1号果针入土70 d的种子第一链cDNA为模板,利用特异性引物AhbHLH63-orf F 1.1和AhbHLH63-orf[JP] R 1.1扩增到约12 kb的片段(图1)。测序分析可知,所扩得的片段大小分别为1 219 bp和1 237 bp,分别命名为AhbHLH63-1.1和 AhbHLH63-1.2。
以特异性引物AhbHLH63-gDNA F和AhbHLH63-gDNA R扩增AhbHLH63基因的gDNA 序列,得到一段约2 kb的产物(图2)。测序结果显示,片段大小为2 078 bp。
2.2 AhbHLH63基因序列分析
AhbHLH63基因的两种cDNA序列AhbHLH63-1.1和AhbHLH63-1.2的ORF分别长1 188 bp和1 152 bp,分别编码 395和383个氨基酸,理论相对分子量分别为41.69 kD和43.1 kD,等电点分别为8.07和7.07。
对gDNA序列及结构进行分析,结果(图3)显示,该基因包含7个外显子和6个内含子。两cDNA的差异是由第6个内含子的选择性剪接导致。AhbHLH63-1.1剪接体在转录后剪切掉了较大第6内含子;而AhbHLH63-1.2则选择性地剪切掉较小的内含子,致使第6外显子109 bp处引入1个终止密码TAA提前终止了翻译。
2.3 AhbHLH63的生物信息学分析
利用CDD分析AhbHLH63蛋白的保守结构域,结果显示,AhbHLH63蛋白含有保守结构域bHLH,推测该蛋白是bHLH家族成员(图 4)。
利用Expasy在线软件分析氨基酸组成,结果(表2)显示,AhbHLH63基因两剪接体编码的蛋白氨基酸组成中Ser含量最高,其次为Ala,Pro、Asn及Leu所占比例较为接近,其余15种氨基酸含量均低于6%。AhbHLH63-1.1和AhbHLH63-1.2蛋白中疏水性氨基酸分别占43.4%、43.2%,亲水性氨基酸分别占56.6%、56.8%。利用Expasy的ProtScale工具在线预测AhbHLH63的疏水性,结果表明AhbHLH63蛋白属于不稳定性蛋白,该蛋白存在明显的疏水区和亲水区;在AhbHLH63保守结构域(约第200 ~ 270个氨基酸之间)的N端主要为碱性氨基酸,具有亲水性,而保守结构域的C端两个相连的螺旋结构则主要由疏水性氨基酸组成(图5,以AhbHLH63-1.1蛋白为例)。Carretero-Paulet[15]等的研究发现,α 螺旋-环-α螺旋结构多借助疏水氨基酸的作用形成二聚体,而疏水氨基酸与带电氨基酸残基之间的作用决定二聚体的特性,转录因子从而发挥其功能[4]。由此推测AhbHLH63蛋白的碱性氨基酸及疏水氨基酸区域可能与其功能有关。利用 Cell-PLoc 2.0在线软件,预测AhbHLH63基因编码的两蛋白质均位于细胞核。
利用 DNAMAN 软件对花生bHLH63的氨基酸与大豆、苜蓿、赤豆等19个物种相关序列进行同源性分析,并利用MEGA 6.0软件构建系统发育树,结果(图6)显示, AhbHLH63蛋白与同属蝶形花科的大豆、密花豆、狭叶羽扇豆、蒺藜苜蓿等物种的bHLH63蛋白亲缘关系较近,聚为一簇;单子叶植物水稻、小麦、玉米等的同源蛋白聚为一簇。AhbHLH63与SsbHLH63(密花豆)、LaHBI1-like(狭叶羽扇豆)、MtbHLH63(蒺藜苜蓿)、VabHLH63(赤豆)和GmCIB1(大豆,cryptochrome interaction bHLH1)等序列一致性均达55%以上;与GrHBI1-like(棉花)序列一致性也接近50%;而与已知功能的AtCIB1(拟南芥)亲缘关系相对较远,序列相似性仅为39%。AhbHLH63的保守结构域序列与其他物种相关序列比对发现,AhbHLH63蛋白与其他已知bHLH保守结构域仅存在个别氨基酸的差异(图7);其中AhbHLH63序列的碱性氨基酸位于bHLH的N端,且含高度保守序列H5-E9-R13,进一步表明AhbHLH63属于bHLH转录因子。
2.4 AhbHLH63的基因表达分析
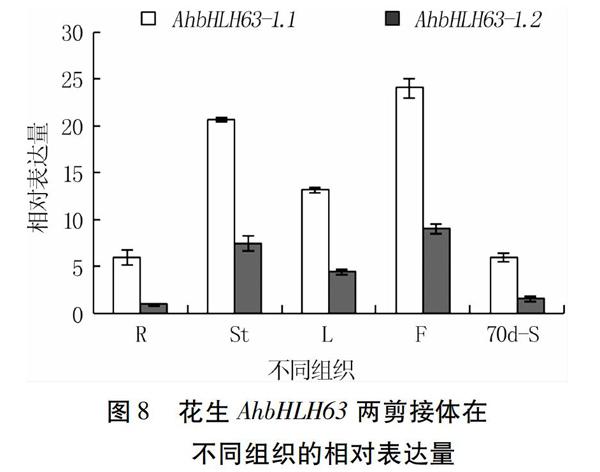
以AhActin7作为内参基因,对AhbHLH63基因在丰花1号根(R)、茎(St)、叶(L)、花(F)的表达模式进行qRT-PCR分析,以果针入土70 d的种子(70 d-S)中AhbHLH63基因表达量作为对照。结果显示,花生不同组织中,AhbHLH63-1.1和AhbHLH63-1.2两剪接体的表达模式较为一致,均为花中的表达量最高,其次是茎和叶,AhbHLH63-1.1在根中的表达量仅高于果针入土70 d的种子,AhbHLH63-1.2基因在根中表达量低于果针入土70 d的种子。不同组织中AhbHLH63-1.1表达量明显高于AhbHLH63-1.2(图8)。
在不同发育时期的花生种子中,发育中期即果针入土30 ~ 50 d时AhbHLH63基因表达量明显高于发育前期及后期。两剪接体表达模式基本相同,果针入土10 d时AhbHLH-1.1表达量高于AhbHLH-1.2,但整体看来二者表达量均不高;果针入土20 d时,AhbHLH-1.2表达量略高于AhbHLH-1.1;果针入土30 d两剪接体表达量均继续明顯升高,且AhbHLH-1.1表达量重新高于AhbHLH-1.2;两剪接体表达量达到峰值均在果针入土40 d时;果针入土50 d时AhbHLH-1.1基因表达量略微下降,而AhbHLH-1.2表达量下降显著,表达水平与30 d时基本持平;此后基因表达量继续逐步降低,且两剪接体的相对表达量水平呈现维持一致的趋势(图9)。
3 讨论与结论
bHLH转录因子作为植物中仅次于 MYB 的第二大转录因子家族[16],在植物生长发育的方方面面均起重要调控作用。本研究根据前期的转录组数据,[JP2]克隆得到花生bHLH转录因子基因AhbHLH63的cDNA 序列,AhbHLH63蛋白含有螺旋-环-螺旋结构,属于bHLH转录因子家族。对其理化性质进行分析发现,AhbHLH63蛋白包含明显的疏水区和亲水区,其位于疏水区的螺旋-环-[JP]螺旋结构可能与其功能结构域相关。系统进化树分析显示,与AhbHLH63蛋白亲缘关系较近的除了大豆、苜蓿、赤豆等植物的bHLH63蛋白外,还包括已知功能的狭叶羽扇豆、棉花的HBI1-like蛋白及大豆的CIB1蛋白等。研究发现,CIB1参与拟南芥[17]、小麦[18]和大豆[19]等植物开花过程的调控。Liu等[20]的研究证实大豆中的CIB1蛋白与隐花色素蛋白CRY2a 相互作用能够激活蓝光依赖的叶衰老;CRY2与CIB1之间的相互作用还参与调控蓝光依赖的开花起始及胚轴伸长过程。拟南芥中HBI1 基因是CIB1的同源基因,外源生长素能够诱导HBI1在幼苗根中的表达[21],且拟南芥HBI1通过响应外源激素赤霉素、油菜素内酯的信号,从而调控细胞伸长相关基因的表达[17,22]。此外,HBI1作为生长激素与免疫信号相互作用的关键节点,参与拟南芥的免疫调节[8]。本研究获得的AhbHLH63转录因子具体参与调控植物生长发育的哪些过程,有待进一步研究。
选择性剪接是真核生物转录后水平上调控基因表达的重要手段。有些基因通过可变剪接赋予不同剪接体不同的功能,有些则是多个剪接体共同促进某一功能的发挥[23]。DOG1(delay of germination 1)基因是种子休眠的重要调控因子,拟南芥种子的休眠性与不同生态型间DOG1表达水平的差异密切相关。DOG1通过选择性剪接能产生5种剪接变体,共编码3种蛋白异構体。尽管单个DOG1蛋白有功能,但该蛋白需要与自身或其他异构体结合防止自身降解。DOG1-β在种子发育过程中的积累量明显高于其他异构体,而DOG1-δ在种子成熟期丰度相对增加。因此,选择性剪接可能是精细调控DOG1蛋白积累的一种机制[24]。PP2C类磷酸酶基因HAB1的两个剪接体HAB1.1和HAB1.2分别编码全长蛋白和C端截短的蛋白,在ABA介导的种子萌发过程中起相反作用。HAB1.1负调控ABA信号途径,而HAB1.2由于缺少催化结构域,丧失了磷酸酶活性,从而正调控ABA信号途径。当ABA存在时,HAB1.2为主要异构体,HAB1.1与HAB1.2比例明显下降,对下游SnRK2激酶活性的抑制作用减弱,进而防止种子萌发[25]。不同的细胞类型及生理状态等可能会影响产生剪接体的方式以及不同剪接体之间的相互作用[26]。本研究克隆了花生AhbHLH63基因的两个剪接体AhbHLH63-1.1和AhbHLH63-1.2,并分析了其在栽培品种丰花1号不同组织及不同发育时期种子中的表达情况,结果显示AhbHLH63基因呈组成型表达模式,两剪接体均在花中表达量最高,根中表达量较低;种子发育中期的表达量高于前期及后期。虽然此过程中剪接体AhbHLH63-1.1和AhbHLH63-1.2总体趋势较为一致,但也略有差异。无论是不同组织还是不同发育时期种子中AhbHLH63-1.1的表达量整体高于AhbHLH63-1.2,推测是因为第6号内含子的剪切方式不同导致AhbHLH63-1.1和AhbHLH63-1.2蛋白功能有所差异。AhbHLH63两剪接体的具体功能及作用机制异同仍需进一步研究。深入研究相同基因中不同的剪接体,将有望进一步阐明植物内含子的剪接机理及其调控基因表达的作用。
参 考 文 献:
[1]Atchley W R, Fitch W M. A natural classification of the basic helix-loop-helix class of transcription factors[J].Proceedings of the National Academy of Sciences, 1997, 94(10): 5172-5176.
[2]Pires N, Dolan L. Origin and diversification of basic-helix-loop-helix proteins in plants[J].Molecular Biology & Evolution, 2010, 27(4): 862-874.
[3]Heim M A, Jakoby M, Werber M. The basic helix-loop-helix transcription factor family in plants: a genome-wide study of protein structure and functional diversity[J].Molecular Biology and Evolution, 2003, 20(5): 735-747.
[4]Toledo-Ortiz G. The Arabidopsis basic/helix-loop-helix transcription factor family[J].The Plant Cell, 2003, 15(8): 1749-1770.
[5]王勇, 陈克平, 姚勤. bHLH转录因子家族研究进展[J].遗传, 2008, 30(7):821-830.
[6]Qi T, Huang H, Wu D, et al. Arabidopsis DELLA and JAZ proteins bind the WD-repeat/bHLH/MYB complex to modulate gibberellin and jasmonate signaling synergy[J].Plant Cell, 2014, 26(3): 1118-1133.
[7]Liu W, Tai H, Li S, et al. bHLH122 is important for drought and osmotic stress resistance in Arabidopsis and in the repression of ABA catabolism[J].New Phytologist, 2014, 201(4): 1192-1204.
[8]Fan M,Bai M Y,Kim J G,et al.The bHLH transcription factor HBI1 mediates the trade-off between growth and pathogen-associated molecular pattern-triggered immunity in Arabidopsis[J].The Plant Cell,2014,26(2):828-841.
[9]Brownlie P, Ceska T A, Lamers M, et al. The crystal structure of an intact human Max-DNA complex: new insights into mechanisms of transcriptional control[J].Structure (London), 1997, 5(4): 509-520.
[10] [JP2]Nakatsuka T, Haruta K S, Pitaksutheepong C, et al. Identification and characterization of R2R3-MYB and bHLH transcription factors regulating anthocyanin biosynthesis in gentian flowers[J].Plant and Cell Physiology, 2008, 49(12): 1818-1829.[JP]
[11] Heisler, Atkinson M G, Bylstra A, et al. SPATULA, a gene that controls development of carpel margin tissues in Arabidopsis, encodes a bHLH protein[J].Development, 2001, 128(7): 1089-1098.
[12] Kianianmomeni A, Ong C S, Rtsch G, et al. Genome-wide analysis of alternative splicing in Volvox carteri[J].BMC Genomics, 2014, 15(1): 1117-1137.
[13] Jian R, Guo F, Wang Y, et al. Transcriptome analysis of alternative splicing in peanut (Arachis hypogaea L.)[J].BMC Plant Biology, 2018, 18(1): 139.
[14] 梁雪莲, 郑奕雄, 陈晓玲, 等. 花生DNA提取方法比较[J].生物技术, 2007, 17(1): 41-44.
[15] Carretero-Paulet L, Galstyan A, Roig-Villanova I, et al. Genome-wide classification and evolutionary analysis of the bHLH family of transcription factors in Arabidopsis, poplar, rice, moss, and algae[J].Plant Physiology, 2010, 153(3): 1398-1412.
[16] 杨鹏程, 周波, 李玉花. 植物花青素合成相关的bHLH转录因子[J].植物生理学报, 2012(8): 747-758.
[17] Sheng W, Ling L, Pengbo X, et al. CRY1 interacts directly with HBI1 to regulate its transcriptional activity and photomorphogenesis in Arabidopsis[J].Journal of Experimental Botany, 2018, 69(16): 3867-3881.
[18] 孫扬. 小麦bZIP基因TaGBF调节光形态建成与抗逆的作用机制研究[D].济南:山东大学, 2014.
[19] Yang D G, Zhao W, Meng Y Y, et al. A CIB1-like transcription factor GmCIL10 from soybean positively regulates plant flowering[J].Science China Life Sciences, 2015, 58(3): 261-269.
[20] Liu H, Yu X, Li K, et al. Photoexcited CRY2 interacts with CIB1 to regulate transcription and floral initiation in Arabidopsis[J].Science, 2008, 322(5907): 1535-1539.
[21] Radoeva T, Lokerse A S, Llavata-Peris C I, et al. A robust auxin response network controls embryo and suspensor development through a basic helix loop helix transcriptional module[J].The Plant Cell, 2019, 31: 52-67.
[22] Bai M Y,Fan M,Oh E,et al.A triple helix-loop-helix/basic helix-loop-helix cascade controls cell elongation downstream of multiple hormonal and environmental signaling pathways in Arabidopsis[J].The Plant Cell,2012,24(12):4917-4929.
[23] [JP2]Graveley B R. Alternative splicing: increasing diversity in the proteomic world[J].Trends in Genetics, 2001, 17(2): 100-107.[JP]
[24] Nakabayashi K, Bartsch M, Ding J, et al. Seed dormancy in Arabidopsis requires self-binding ability of DOG1 protein and the presence of multiple isoforms generated by alternative splicing[J].PLoS Genetics, 2015, 11(12): e1005737.
[25] Wang Z, Ji H, Yuan B, et al. ABA signalling is fine-tuned by antagonistic HAB1 variants[J].Nature Communications, 2015, 6: 8138-8149.
[26] Blencowe B J. Alternative splicing: new insights from global analyses[J].Cell, 2006, 126(1): 37-47.
收稿日期:2019-08-24
基金项目:山东省农业科学院农业科技创新工程项目(CXGC2018E13)
作者简介:崔维佩(1996—),女,硕士研究生,研究方向为植物分子遗传。
通讯作者:单雷(1965—),女,研究员,研究方向为植物分子遗传。E-mail: shlei1025@sina.com

