莱茵衣藻type I metacaspase基因克隆及其参与调控程序性细胞死亡研究*
张恒宇 柳逸群 葛 源
莱茵衣藻type I metacaspase基因克隆及其参与调控程序性细胞死亡研究*
张恒宇1柳逸群2葛 源1①
(1. 中国海洋大学海洋生命学院 青岛 266003;2. 中国海洋大学水产学院 青岛 266003)
Metacaspase是在原生动物、真菌和植物中发现的一种具有底物精氨酸/赖氨酸特异性的半胱氨酸蛋白酶。根据蛋白质结构特征可将metcaspase分为typeⅠ与typeⅡ两种类型,均参与调控多种植物与原生动物程序性细胞死亡。本研究根据GenBank数据库莱茵衣藻() type I metacaspase基因(GenBank No. XM_001696904)序列,采用巢式PCR克隆获取type I metcaspase基因开放阅读框(ORF)序列并命名为CrMC1。CrMC1 ORF全长987 bp,推测编码1个包含405个氨基酸的蛋白质。通过与已知物种type I metacaspase氨基酸序列进行同源序列比对发现,CrMC1具有高度保守的p20、p10、连接区结构域以及精氨酸、半胱氨酸活性中心位点。研究显示,在H2O2诱导的莱茵衣藻程序性细胞死亡过程中,CrMC1表达量显著提高(<0.05),2 h后达到峰值,3 h时下降至对照组同一水平。结果表明,莱茵衣藻type I metacaspase基因CrMC1参与H2O2诱导的莱茵衣藻程序性细胞死亡调控。
Metacaspase;莱茵衣藻;程序性细胞死亡
莱茵衣藻()是绿藻门(Chlorophyceae)、团藻目(Volvocales)、衣藻属()的一种单细胞真核微藻,具有良好的环境适应能力、繁殖能力以及生长速率,能在固体培养基上形成单藻落,也可在液体中培养,有“绿色酵母”之称(谢传晓等, 2003; 孙晓梅等, 2004; 耿予欢等, 2007)。目前,莱茵衣藻全基因组测序已经完成(Merchant, 2007),已成功建立针对莱茵衣藻线粒体、叶绿体与细胞核基因组的遗传操作系统和CRISPR/Cas9介导的基因组精确编辑系统(Greiner, 2017)。此外,目前已建立莱茵衣藻光合作用、呼吸作用、细胞分裂、养分吸收、辐射、紫外线和化学诱变下的基因组稳定性和适应性反应、耐重金属植物修复、盐度、水温、渗透压和氧化胁迫应激等重要生理过程研究的模型(Harris, 2001; Hanikenne, 2003; Hema, 2007; Dimova, 2008)。因此,莱茵衣藻已经成为藻类遗传学、生物化学和分子生物学研究的重要模式物种(徐田枚, 2007; Harper, 1999; Wu, 1997)。
植物程序性细胞死亡(Programmed cell death)是指与动物细胞凋亡(Apoptosis)同源的植物细胞死亡方式,普遍存在于植物发育和应对病原感染、干旱等生物或非生物胁迫过程中(Reape, 2008)。然而,动物细胞凋亡通路中关键调控与执行基因家族胱冬肽酶(Cysteine aspartic acid specific protease, caspase)家族在植物中并不存在(van Doorn, 2005; Ge, 2016)。研究表明,植物中存在一种caspase同系蛋白metacaspase。Metacaspase是非后生动物完成正常生理学反应所必需的多功能蛋白酶,且植物和原生动物中氧化应激诱导的程序性细胞死亡过程都依赖于metacaspase(Vercammen, 2007)。根据其结构特征,metacaspase又可以分为type I与typeⅡ两类(马聪等, 2012)。其中,type I metacaspase具有富含脯氨酸重复基序的N末端前结构域,并且包含锌指结构。而type Ⅱ metacaspase缺乏前结构域,但在大(p20)、小(p10)亚基之间具有较长连接区。在原生动物和真菌中,目前仅发现了type I metacaspase,而高等植物基因组中同时具有type I与type Ⅱmetacaspase。Metacaspase活性中心普遍具有保守的组氨酸–半胱氨酸结构,其中半胱氨酸残基作为水解底物肽键的亲核体。
近年的研究表明,单细胞藻类在生物和非生物胁迫条件下,亦可发生程序性细胞死亡。莱茵衣藻经被用于程序性细胞死亡研究,并获得了成功(Moharikar, 2006; Zuo, 2012; Pérezpérez, 2010; Yordanova, 2013; Sirisha, 2014)。全基因组序列预测显示,莱茵衣藻基因组中同时存在type I与typeⅡ metacaspase基因,其结构与功能尚不明确。本研究通过巢式PCR克隆获得莱茵衣藻type I metacaspase ORF序列,利用生物信息学技术预测其氨基酸序列并进行同源性分析,继而利用荧光定量PCR技术分析type I metacaspase基因在H2O2诱导的莱茵衣藻程序性细胞死亡过程中的表达量变化。本研究为更好地阐明植物与微藻程序性细胞死亡的分子机制及其生物演化关系提供理论依据,同时为高品质微藻藻种选育和微藻增养殖工业及应用(何舟等, 2015; 乔洪金等, 2016)提供技术支持。
1 材料与方法
1.1 藻种培养
野生型莱茵衣藻藻种cc-125 mt+购自美国明尼苏达大学莱茵莱茵衣藻种质库(Chlamycollection, University of Minnesota, 美国)。藻种接种至含100 μmol/L氨苄青霉素的固体TAP培养基平板,挑取单克隆接种于液体TAP培养基中,置于25℃、光照强度2000 lx的光照培养箱振荡培养(100 r/min)至对数生长期(OD434 nm=1),光/暗周期为12 h/12 h。
1.2 莱茵衣藻总RNA提取与cDNA合成
藻种离心收集后,采用液氮反复冻融法破壁,采用QIAGEN RNeasy Plant Mini Kit试剂盒提取莱茵衣藻总RNA。采用琼脂糖凝胶电泳法和Qubit 3.0分光光度计鉴定总RNA质量与浓度。cDNA合成采用M-MLV反转录酶试剂盒进行。利用莱茵衣藻18 rRNA (GenBank No. AY665726)作为内参。
1.3 CrMC1 ORF巢式PCR扩增
根据NCBI GenBank数据库中莱茵衣藻type I metacaspase cDNA序列(GenBank No. XM_001696904)设计引物,进行两步法巢式PCR克隆,引物序列见表1。使用Nucleospin Gel and PCR Clean-up试剂盒纯化第二步PCR产物,并进行poly(A)-tailing添加粘末端反应,进而将其连接到pGEM-T载体中进行蓝白斑筛选。筛选后提取质粒并送至生工生物工程(上海)有限公司进行测序、组装并输入BLAST进行验证,证实所获序列为莱茵衣藻type I metacaspase ORF序列,命名为CrMC1。
表1 莱茵衣藻metacaspase type I ORF克隆引物

Tab.1 Primers for C. reinhardtii metacaspase type I ORF cloning
1.4 生物信息学分析
利用在线SMS生物软件(http://www.bio-soft.net/ sms/),根据CrMC1 ORF的DNA序列推测CrMC1氨基酸序列,获得包含405个氨基酸的序列。进而利用Vector NTI 11.5软件ClustalX方法将CrMC1与从NCBI数据库获得的其他物种metacaspase构建系统进化树,物种名与蛋白编号见表2。此外,通过将CrMC1氨基酸序列与自uniprot数据库(http://www.uniprot. org/)获得的3种拟南芥() type I metacaspase (AtMC1, No. Q7XJE6; AtMC2, No. Q7XJE5; AtMC3, No. Q9FMG1)氨基酸序列的同源比对,预测其功能域。
1.5 H2O2诱导莱茵衣藻细胞死亡率测定
将处于生长对数期(OD434 nm=1)的莱茵衣藻cc125 mt+分别置于实验组(1 mmol/L H2O2)与对照组进行培养,并测定0~480 min内的死亡率。死亡率测定采取SYTOX green染料进行。利用正置荧光显微镜观察计数至少200个细胞,根据SYTOX green标记阳性为死亡细胞、隐性为存活细胞进行细胞计数,并计算死亡率。
表2 进化树所用metacaspase基因序列

Tab.2 Metacaspase gene sequence of phylogenetic tree
1.6 荧光定量PCR
提取不同时间点实验组与对照组莱茵衣藻总RNA,并反转录获得cDNA,利用荧光定量PCR法进行CrMC1表达量分析。荧光定量PCR反应使用SensiFAST SYBR No-ROX试剂盒在ABI Prism 7000型荧光定量PCR仪上进行。荧光定量PCR所得数据利用18S rRNA作为内参基因,按照2-DD法处理数据。所得数据利用Microsoft Excel 2010软件进行平均值、标准差和差异显著性分析(<0.05)并作图。CrMC1与内参基因18S rRNA (Nguyen, 2011)引物见表3。
表3 荧光定量PCR引物

Tab.3 Primers used in qRT-PCR
2 结果
2.1 CrMC1基因克隆及进化分析
如图1A所示,本研究通过巢式PCR克隆到莱茵衣藻type I metacaspase基因ORF全长序列,包含987 bp,推测编码405个氨基酸,命名为CrMC1。为分析CrMC1进化关系,本研究从NCBI数据库中筛选出已分类的植物metacaspase蛋白氨基酸序列,包括拟南芥、小麦()、鸡()、挪威云杉()、团藻()等(表1),使用Vector NTI 11.5软件的ClustalX法进行同源性分析,并构建系统进化树(NJ)。如图1B所示,系统进化树中的19个metacaspase成员形成了2个大分支,其中,CrMC1与所有已知type I metacaspase位于同一分支内,故可推测CrMC1属于type I metacaspase。此外,在type I metacaspase分支中,CrMC1与团藻目的metacaspase具有最高相似性,与植物例如拟南芥type I metacaspase AtMC2亲缘关系较远,提示其位于生物演化的初级地位。
2.2 CrMC1蛋白结构域预测
蛋白多序列比对分析显示(图2),CrMC1具有metacaspase家族典型的、高度保守的p20 (氨基酸44~ 195)结构域、p10 (氨基酸229~328)结构域以及中间连接区(氨基酸196~228)。与拟南芥type I metacaspase 蛋白AtMC1/2/3比较,虽然CrMC1的N末端区域较为简单,然而其脯氨酸含量较高,为典型的富含脯氨酸区域(23.2%),符合type I metacaspase的结构特征。CrMC1蛋白酶活性中心包含高度保守的组氨酸(132) (图2*处)与半胱氨酸(188) (图2#处)结构,推测其具有已知metacaspase类似的蛋白酶活性。
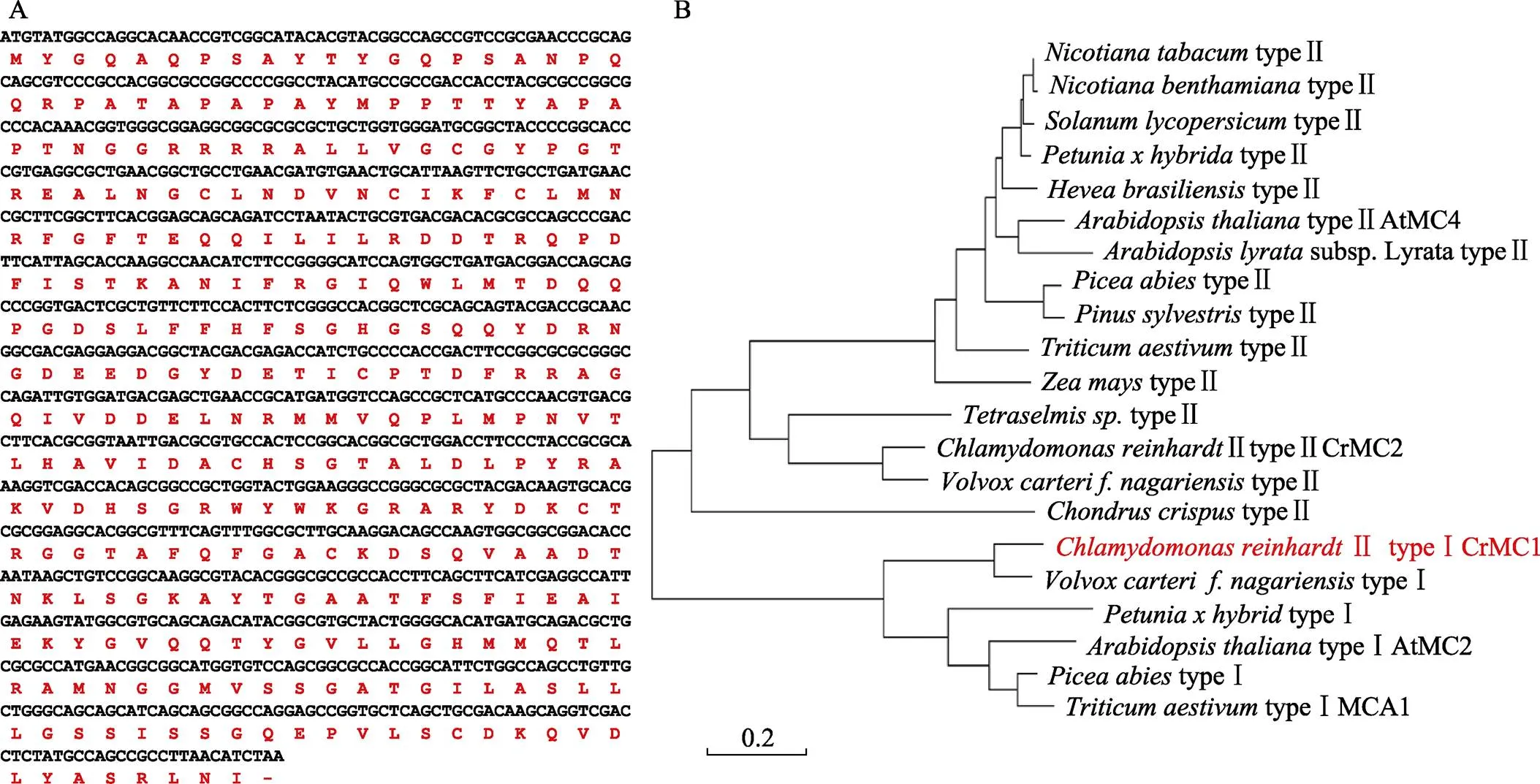
图1 CrMC1结构分析
A.通过利用SMS生物软件(http://www.bio-soft.net/sms/)推测CrMC1氨基酸序列;B. 利用Vector NTI 11.5软件ClustalX方法将CrMC1与从NCBI数据库获得的其他物种metacaspase构建系统进化树,物种名与蛋白编号见表2
A.CrMC1 ORF was deduced to CrMC1 protein sequence using online SMS software (http://www.bio-soft.net/sms/); B. Phylogenetic tree was constructed using metacaspase protein sequence of different species obtained from NCBI database. Latin names and protein IDs are presented in Table 2

图2 CrMC1蛋白质结构预测
利用Vector NTI 11.5软件ClustalX方法将CrMC1与自uniprot数据库(http://www.uniprot.org/)获得的3种拟南芥I型metacaspase (AtMC1, NO. Q7XJE6; AtMC2, NO. Q7XJE5; AtMC3, NO. Q9FMG1)氨基酸序列进行同源比对。图中蓝色背景标示为p20结构域,红色背景标示为p10结构域,黑框标示区域为连接区。活性中心分别以*和#标出
CrMC1 was aligned with type I metacaspase from Arabidopsis: AtMC1, NO. Q7XJE6; AtMC2, NO. Q7XJE5; AtMC3, NO. Q9FMG1. Arabidopsis sequences were obtained from uniprot database. The domain of p20, p10 and internal linker is highlighted in blue background, red background and black frame, respectively. The conserved His-Cys residues are labelled by * and # respectively
2.3 莱茵衣藻CrMC1参与H2O2诱导细胞死亡调控
由图3A可见,1 mmol/L H2O2处理可诱导处于对数生长期的莱茵衣藻出现明显的细胞死亡现象。根据Vavilala等(2015)报道,H2O2诱导的莱茵衣藻细胞死亡方式为程序性细胞死亡。本研究进而利用荧光定量PCR方法检测处于对数生长期的莱茵衣藻CrMC1基因在1 mmol/L H2O2诱导后1~8 h内表达水平的变化情况。如图3B所示,1 mmol/L H2O2诱导后,莱茵衣藻CrMC1基因的表达量自1 h起显著高于对照组(<0.05),2 h后达到最高,约为对照组1.77倍,5 h时下降至对照组同一水平,与程序性细胞死亡发生时间基本同步,推测CrMC1处于莱茵衣藻程序性细胞死亡调控通路的上游。

图3 CrMC1参与H2O2诱导的莱茵衣藻程序性细胞死亡调控
A. H2O2处理后1~480 min内莱茵衣藻细胞死亡率。莱茵衣藻培养与H2O2处理如前所述;B. CrMC1基因在H2O2诱导的莱茵衣藻程序性细胞死亡中的表达量变化。实验均进行3次取平均值
A. The mortality ofcells within 1~480 min after H2O2treatment.culture and H2O2treatment as previously described; B. CrMC1 gene expression changes in H2O2-induced PCD in. Each experiment was performed three times and averaged
3 讨论
Madeo等(2002)首次报道了使用酵母Yca1突变菌株在氧化应激导致的细胞死亡对metacaspase的依赖性,证明Yca1在酵母细胞中参与病毒毒素和非生物相关因素应激以及细胞衰老过程中的细胞死亡(Madeo, 2009)。此外,还有研究表明,脂毒性应激(Low, 2008)和肌醇饥饿(Guérin, 2009)等部分酵母细胞致死条件下,metacaspase是酵母实现细胞死亡的必要的条件;白念珠菌的metacaspase,CaMCA1,参与介导氧化应激诱导的细胞死亡(Cao, 2009);而构巢曲霉的2个metacaspase的其中之一,CasA在ER应激相关的细胞死亡起促进作用(Colabardini, 2010)。
目前,在模式生物拟南芥中分别鉴定出3个type I metacaspase基因(AtMC1~3)与6个typeⅡ metacaspase基因(AtMC4~9)(Vercammen, 2004)。Type I metacaspase AtMC1显示为拟南芥中HR细胞死亡的正调节物,而AtMC2则能拮抗AtMC1的这种细胞死亡效应。而且AtMC2作为抗凋亡调节剂,其蛋白水解活性是显性的。通过对2种metacaspase的遗传操作,几乎可以使植物细胞内的免疫受体消除HR活性(Coll, 2010)。与高等植物相似,莱茵衣藻中同时存在1个type I metacaspase基因与1个typeⅡ metacaspase基因。由此推测,与酵母相比,莱茵衣藻程序性细胞死亡调控与高等植物具有更高的同源性。因此,对莱茵衣藻metacaspase基因及其功能的研究,对更好地阐释植物程序性细胞死亡调控通路以及type I与typeⅡ metacaspase基因相互作用有着极为重要的意义。
本研究首次报道了莱茵衣藻type I metacaspase基因ORF序列及其参与调控H2O2诱导程序性细胞死亡作用。通过巢式PCR从野生型莱茵衣藻藻种中克隆到1个type I metacaspase基因并命名为CrMC1。序列分析表明,CrMC1具有type I metacaspase特征性的N末端脯氨酸富含区、保守的p20与p10结构域以及组氨酸–半胱氨酸活性中心结构,推测其在演化上与已知高等植物metacaspase具同源性。Type I metacaspase在高等植物与酵母程序性细胞死亡中所扮演的角色尚存在争议。例如,AtMC2表达量在拟南芥程序性细胞死亡过程中调高而AtMC1表达量调低或无变化。本研究的实验数据证实,与AtMC2类似,CrMC1表达量在过氧化物胁迫导致的莱茵衣藻程序性细胞死亡早期显著提高,证实其参与早期程序性细胞死亡的调控。
综上所述,本研究结果初步描述了莱茵衣藻中type I metacaspase基因的结构与功能,为植物程序性细胞死亡研究和藻类增养殖工业提供了理论基础和技术支持。
Cao Y, Huang SB, Zhu Z,. Candida albicans cells lacking CaMCA1-encoded metacaspase show resistance to oxidative stress-induced death and change in energy metabolism. Fungal Genetics and Biology, 2009, 46(2): 183–189
Colabardini AC, De Castro PA, De Gouvêa PF,. Involvement of theprotein kinase C with farnesol tolerance is related to the unfolded protein response. 2010, 78(5): 1259–1279
Coll NS, Vercammen D, Smidler A,. Arabidopsis type I metacaspases control cell death. Science, 2010, 330(6009): 1393–1397
Dimova EG, Bryant PE, Chankova SG,Adaptive response: Some underlying mechanisms and open questions. Genetics and Molecular Biology, 2008, 31(2): 396–408
Ge Y, Cai YM, Bonneau L,. Inhibition of cathepsin B by caspase-3 inhibitors blocks programmed cell death in. Cell Death and Differentiation, 2016, 23: 1493–1501
Geng YH, Wei D, Li GJ,. Research progress on. Natural Product Research and Development, 2007, 19(1): 175–179 [耿予欢, 魏东, 李国基, 等. 极地雪衣藻的研究进展. 天然产物研究与开发, 2007, 19(1): 175–179]
Greiner A, Kelterborn S, Evers H,Targeting of photoreceptor genes invia zinc-finger nucleases and CRISPR/Cas9. Plant Cell, 2017, 29(10): 2498–2518
Guérin R, Beauregard PB, Leroux A,. Calnexin regulates apoptosis induced by inositol starvation in fission yeast. PLoS One, 2009, 4(7): e6244–e6244
Hanikenne M., as a eukaryotic photosynthetic model for studies of heavy metal homeostasis and tolerance. New Phytologist, 2003, 159(159): 331–340
Harper JDI., cell cycle mutants. International Review of Cytology, 1999, 189(08): 131–150
Harris EH.as a model organism. Annual Review of Plant Physiology and Plant Molecular Biology, 2001, 52(52) 363–406
He Z, Song J, Chang YQ,. Effects of addingL. meal in diets on the growth, body composition and digestive enzymes activities of juvenile sea cucumber(Selenka). Progress in Fishery Sciences, 2015, 36(4): 122–127 [何舟, 宋坚, 常亚青, 等. 穗花狐尾藻(L.)饲喂对刺参()幼参生长、体成分及消化酶的影响. 渔业科学进展, 2015, 36(4): 122–127
Hema R, Senthil-Kumar M, Shivakumar S,., a model system for functional validation of abiotic stress responsive genes. Planta, 2007, 226(3): 655–670
Low CP, Shui G, Liew LP,. Caspase-dependent and -independent lipotoxic cell-death pathways in fission yeast. Journal of Cell Science, 2008, 121: 2671–2684
Ma C, Kong WW. Research progress in plant metacaspase. Chinese Bulletin of Botany, 2012, 47(5): 543–549 [马聪, 孔维文. 植物Metacaspase研究进展. 植物学报, 2012, 47(5): 543–549]
Madeo F, Carmona-Gutierrez D, Ring J,. Caspase- dependent and caspase-independent cell death pathways in yeast. Biochemical and Biophysical Research Commu- nications, 2009, 382(2): 227–231
Madeo F, Herker E, Maldener C,. A caspase-related protease regulates apoptosis in yeast. Cell, 2002, 9(4): 911–917
Merchant SS, Prochnik SE, Vallon O,Thegenome reveals the evolution of key animal and plant functions. Science, 2007, 318(5848): 245–250
Moharikar S, D¢Souza JS, Kulkarni AB,. Apoptotic-like cell death pathway is induced in unicellular chlorophyte(chlorophyceae) cells following UV irradiation: Detection and functional analyses. Journal of Phycology, 2006, 42(2): 423–433
Nguyen AV, Toepel J, Burgess S,Time-course global expression profiles ofduring photo-biological H2production. PLoS One, 2011, 6(12): e29364
Pérezpérez ME, Florencio FJ, Crespo JL. Inhibition of target of rapamycin signaling and stress activate autophagy in. Plant Physiology, 2010, 152: 1874–1888
Qiao HJ, Wang JY, Zhang LM,. Effects of dietary fish oil replacement by microalgae in microdiets on growth performance, survival and fatty acid profile of Japanese flounder () juveniles. Progress in Fishery Sciences, 2016, 37(5): 56–63 [乔洪金, 王际英, 张利民, 等. 微粒饲料中以微藻粉替代鱼油对牙鲆()稚鱼生长存活和脂肪酸组成的影响. 渔业科学进展, 2016, 37(5): 56–63]
Reape TJ, Molony EM, McCabe PF. Programmed cell death in plants: distinguishing between different modes. Journal of Experimental Botany, 2008, 59(3): 435–444
Sirisha VL, Sinha M, D¢Souza JS. Menadione-induced caspase-dependent programmed cell death in the green chlorophyte. Journal of Phycology, 2014, 50(3): 587
Sun XM, Hu HJ, Li YG. New material of family Chlamydomonaceae (Volvocales) in China. Journal of Wuhan Botanical Research, 2004, 22(6): 503–506 [孙晓梅, 胡鸿钧, 李夜光. 中国衣藻科(团藻目)新资料. 武汉植物学研究, 2004, 22(6): 503–506]
van Doorn WG, Woltering EJ. Many ways to exit? Cell death categories in plants. Trends in Plant Science, 2005, 10(3): 117–122
Vavilala S, Gawde K, Sinha M,. Programmed cell death is induced by hydrogen peroxide but not by excessive ionic stress of sodium chloride in the unicellular green alga. European Journal of Phycology, 2015, 50(4): 422–438
Vercammen D, Cotte BVD, Jaeger GD,. TypeⅡ metacaspases Atmc4 and Atmc9 ofcleave substrates after arginine and lysine. Journal of Biological Chemistry, 2004, 279(44): 45329–45336
Vercammen D, Declercq W, Vandenabeele P,. Are metacaspases caspases? Journal of Cell Biology, 2007, 179(3): 375–380
Wu L, Hepler PK, John PCL. The met 1 mutation in, causes arrest at mitotic metaphase with persisting p34 cdc2 -like H1 histone kinase activity that can promote mitosis when injected into higher-plant cells. Protoplasma, 1997, 199(3): 135–150
Xie CX, Han W, Yu ZL. Progress ofas a model organism. Hereditas, 2003, 25(3): 350–354 [谢传晓, 韩伟, 余增亮. 模式生物衣藻及其研究进展. 遗传, 2003, 25(3): 350–354]
Xu TM. Analysis of the mutational character ofNfr-4 mutant strain. Master´s Thesis of Xinjiang University, 2007 [徐田枚. 莱茵衣藻() Nfr-4突变株的突变性质分析. 新疆大学硕士研究生学位论文, 2007]
Yordanova ZP, Woltering EJ, Kapchinatoteva VM,. Mastoparan-induced programmed cell death in the unicellular alga. Annals of Botany, 2013, 111(2): 191–205
Zuo Z, Zhu Y, Bai Y,. Acetic acid-induced programmed cell death and release of volatile organic compounds in. Plant Physiology and Biochemistry, 2012, 51(2): 175–184
Cloning of the Metacase Type I Gene ofand Its Involvement in the Regulation of Programmed Cell Death
ZHANG Hengyu1, LIU Yiqun2, GE Yuan1①
(1. College of Marine Life Science, Ocean University of China, Qingdao 266003; 2. College of Fisheries, Ocean University of China, Qingdao 266003)
The metacaspase is a cysteine protease with the substrate of arginine/lysine, specifically found in protozoa, fungi, and plants. The metacaspase is generally believed to be closely related to the caspase of the metazoan. Previous studies have shown that metacaspases can be divided into type I and typeⅡ according to the differences in their structural characteristics, and both types have been found to be involved in the regulation and the control of programmed cell death in various plants and protozoa. Based on the information of the sequence of thetype I metacaspase cDNA, obtained from the Genbank database (Genbank NO. XM_001696904), this study was able to use the method of two-step nested PCR cloning to retrive the Open Reading Frame (ORF) of the sequence of thetype I metacaspase cDNA. The ORF sequence, mentioned above, was named as the CrMC1 in this study. The length of the CrMC1 ORF, in its entirety, is 987 bp, from which can be inferred that there are 405 amino acids encoded in this sequence. By the method of homologous sequence comparative analysis with the already known type I metacaspase protein, it was then found that the CrMC1 has conservative p20, p10, linker domain, and arginine and cysteine active center sites. Furthermore, this study is also able to show that, during the H2O2induced programmed cell death of, the gene transcripts of the CrMC1 was increased at a statistically significant level (<0.05). The gene transcripts of the CrMC1 then reached its peak after 2 h. Eventually, the gene transcripts of the CrMC1 decreased to the same level as it was in the control group at 3 h. In conclusion, the results as discussed above have indicated the following: theCrMC1 was involved in the regulation and the control of the H2O2induced programmed cell death of.
Metacaspase;; Programmed cell death
S9
A
2095-9869(2019)06-0131-07
10.19663/j.issn2095-9869.20180507002
http://www.yykxjz.cn/
张恒宇, 柳逸群, 葛源. 莱茵衣藻type I metacaspase基因克隆及其参与调控程序性细胞死亡研究. 渔业科学进展, 2019, 40(6): 131–137
Zhang HY, Liu YQ, Ge Y. Cloning of the metacase type I gene ofand its involvement in the regulation of programmed cell death. Progress in Fishery Sciences, 2019, 40(6): 131–137
* 863计划生物医药领域青年科学家专项基金(2015AA020903)和青岛市科技计划基础研究项目(13-1-4-253-jch)共同资助 [This work was supported by China National High-Tech Research and Development Programme (863 Programme) (2015AA020903), and Qingdao Municipal Science and Technology Project Basic Science Research Programme (13-1-4-253-jch)]. 张恒宇, E-mail: 854636605@qq.com
葛 源,E-mail: geyuan@ouc.edu.cn
2018-05-07,
2018-05-17
GE Yuan, E-mail: geyuan@ouc.edu.cn
(编辑 冯小花)

