形态学和SNP标记分析马氏珠母贝杂交子代及其亲本群体的遗传结构
黄景, 潘肖兰, 许濛, 刘文广, 张华, 何毛贤
海洋生物学
形态学和SNP标记分析马氏珠母贝杂交子代及其亲本群体的遗传结构
黄景1,2,3, 潘肖兰1,2,3, 许濛1,2,3, 刘文广1,3, 张华1,3, 何毛贤1,3
1. 中国科学院南海海洋研究所, 中国科学院热带海洋生物资源与生态重点实验室, 广东 广州 510301; 2. 中国科学院大学, 北京 100049; 3. 中国科学院南海海洋研究所, 广东省应用海洋生物学重点实验室, 广东 广州 510301
马氏珠母贝是重要的海水养殖贝类, 为了探究繁育亲本及其子代的遗传结构和关系, 本研究运用多元性形态学和SNP标记对马氏珠母贝母本深圳群体、父本海南群体和其杂交子代F1的外部形态和分子遗传结构进行分析。结果发现, 3个群体的综合判别率为72%, F1和母本群体的形态差异最小, 父本群体与母本、F1的形态差异较大。采用HRM法(high resolution melting) 应用4个SNP位点对这3个群体进行分型, 3个群体的平均观测杂合度o和期望杂合度e分别为0.2110~0.2879和0.3317~0.4685, F1的杂合度高于两个亲本; 平均多态信息含量PIC值为0.2643~0.3556, 呈现中等程度遗传多样性。F1与母本之间的基因流N最大(7.7701), 遗传距离最小(0.0546), 亲缘关系最近; 两个亲本之间的N最小(1.9662), 遗传距离最大(0.1759)。rs8位点可以判别两个亲本群体, 可作为特异性的标记。该结果可以为马氏珠母贝群体遗传结构鉴别、育种群体管理提供指导。
马氏珠母贝; 遗传学; 遗传结构; 形态学; SNP
马氏珠母贝 () 又称合浦珠母贝, 是中国海水珍珠产业主要养殖贝类, 具有极高的经济价值, 在中国广东、广西和海南省沿海分布广泛。然而, 近年来马氏珠母贝养殖产业面临着疾病暴发(Miyazaki et al, 1999)、环境污染(Liu et al, 2012)、高死亡率(张莉, 2007)、近亲繁殖(Wada et al, 1994)等一系列问题, 海水珍珠产业的发展受阻, 造成极大的损失(Hine et al, 2000)。上述问题的有效解决有助于实施种质创制和良种培育, 而对群体遗传结构的监测、鉴别则是育种过程的重要环节。
在马氏珠母贝传统育种中, 常常通过外部形态差异对群体进行判别并指导育种, 杜晓东等(2002)发现北部湾种群和大亚湾种群在壳长、壳宽、壳宽系数上差异显著; 顾志峰等(2009)通过壳宽系数和壳色对三亚野生种群和印度养殖种群进行鉴别。谷龙春等(2010)利用壳长、壳高、壳宽对不同群体(海南三亚群体、广西北海群体和广东徐闻群体)双列杂交子代家系进行判别, 筛选出优良亲本。而仅仅通过形态区分群体指导育种时常常受到多基因调控和环境影响, 因此有必要引入分子标记辅助育种。
近年来, 越来越多的分子标记被用来分析马氏珠母贝的种质资源和遗传结构, 例如内部转录间隔序列(internal transcriptional spacing sequence, ITS) (喻达辉等, 2005)、扩增片段长度多态性(amplified fragment length polymorphism, AFLP) (Yu et al, 2006)、随机扩增多态性DNA (randomly amplified polymorphic DNA, RAPD) (苏天凤等, 2002; Miyake et al, 2016)、单序列间重复(inter-simple sequence repeat, ISSR) (姜因萍等, 2007)、微卫星 (simple sequence repeats, SSR) (沈恩健, 2010) 和单核苷酸多态性 (single nucleotide polymorphism, SNP) (李耀国等, 2016)。在各类分子标记中, SNP因其在基因组中分布泛、多样性高、易于分型等特点, 成为基因分型最理想的分子标记 (Ganal et al, 2009)。目前, SNP分型主要通过以下方法: 低等通量分型方法, 包括等位基因特异 PCR (Newton et al, 1989)和限制性内切酶酶切法 (Neff et al, 1998); 中通量分型方法, 包括变性高效液相色谱技术 (Nickerson et al, 2000)、质谱法 (Hong et al, 2008)、SNaPshot技术 (Sobrino et al, 2005)和高分辨溶解曲线法(high resolution melting, HRM) (Montgomery et al, 2007); 高通量分型方法, 如SNP芯片技术(赵杰等, 2018)。HRM方法不需要任何特异性探针, 而且灵敏度高, 操作简便(赵琼一等, 2010)。基于HRM方法的SNP标记常常用于品种鉴定(王家丰, 2013)、基因分型(王艳等, 2015)、分子诊断(胡笑蓉等, 2012)、疾病相关基因突变扫描(李梅等, 2012)和动物育种(陈亮等, 2014)。在海洋经济贝类中, HRM方法已被用于SNP标记开发、遗传结构分析和亲缘关系鉴定(童晓飞等, 2012; 金玉琳, 2014), 而在马氏珠母贝中, 利用HRM对SNP进行分型并进行遗传结构分析的研究较少。
本研究利用HRM技术对马氏珠母贝2个亲本群体及其杂交子代进行SNP分型, 同时结合形态学分析它们的遗传结构, 探究群体亲缘关系并判别群体。研究结果可为马氏珠母贝育种群体的管理和育种实践提供理论和技术指导。
1 材料与方法
1.1 实验材料构建和样品采集
繁育用亲本分别来源于2龄深圳群体和海南群体, 二者都为深圳大鹏澳海区的养殖群体。以双列杂交的方式构建群体, 群体内自繁作为亲本群体; 杂交一代(F1)以深圳群体为母本, 海南群体为父本; 反交群体因数量太少而放弃。亲本群体和F1在相同的条件下进行管理和养殖。12月龄贝时, 分别从每个群体 (2个亲本群体和 F1)中随机抽样50只贝, 测量每只贝的壳长、壳宽、壳高、铰合线长、软体部重、壳重和总重, 并计算出壳宽指数、壳重指数、肥满度指数、壳高/壳长、壳宽/壳长、铰合线/壳长, 共计 13个形态性状参数。长度精确到 0.01mm; 重量精确到 0.01g。每个群体从中挑取30只贝的闭壳肌组织于90%乙醇中固定, 放置于–20℃保存, 用于后续SNP分型实验。壳宽指数(Wada et al, 1991)、壳重指数(杜晓东等, 2002)和肥满度指数(Yokogawa, 1998)计算公式如下:
壳宽指数 = 壳宽/(壳长+壳高+壳宽) (1)
壳重指数 = [壳重/(壳长×壳高×壳宽)]×105(2)
肥满度指数 = [软体部重/(壳长×壳高×壳宽)]×105(3)
1.2 DNA提取
采用 HiPure Universal DNA Kit (Magen, China) 试剂盒对每个样本的闭壳肌基因组DNA进行提取, 相关操作步骤参照试剂盒说明书。使用微量分光光度计(Quawell Q5000, San Jose, CA, USA) 测量DNA的浓度, 并用琼脂糖凝胶电泳分析DNA质量, 于–20℃储存。
1.3 SNP分型
从前期的研究中筛选出在深圳群体和海南群体中基因型频率差异显著的4个SNP位点(rs8、rs33、rs35、rs44)。结合文献并采用Primer premier 6.0 软件进行引物设计(表1), 并送由华大公司(中国)合成。对引物进行验证后, 利用罗氏LC-480定量PCR仪(Roche, 瑞士), 使用高分辨率溶解曲线法(high resolution melting, HRM)对亲本群体和 F1 中的4个位点进行SNP分型。10μL HRM反应: 384孔板的每个加样孔中加入 5μL 2× Fast Super EvaGreen Master Mix(US EVERBRIGHT INC), 0.5μL上游引物(5μmol·L–l), 0.5μL下游引物(5μmol·L–l), 0.5μL DNA (25ng·μL–1), 3.5μL ddH2O混匀。HRM扩增反应程序为: 94℃预变性10min, 94℃变性20s, 退火60℃至52℃(降落PCR: 每个循环降低0.5℃)10s, 72℃延伸10s, 扩增45个循环。扩增程序结束后运行高分辨率溶解曲线程序: 95℃变性1min, 40℃1min, 65~95℃, 每秒上升 0.02℃(每上升 1℃收集25次荧光信号)。利用LightCycler ® 480 Gene Scanning Soft-ware 分析软件, 分析各个群体的HRM曲线。

表1 HRM法基因分型引物
注: F为正向引物, R为反向引物
1.4 数据分析
利用SPSS 24.0.0.0软件(http://www.spss.com.cn) 对3个群体的13个形态参数进行单因素方差分析、主成分分析、判别分析和聚类分析。单因素方差分析采用Duncan方法进行多重比较。主成分分析通过因子分析处理13个形态参数, 获得主成分贡献率以及累计贡献率。判别分析通过逐步判别法建立判别函数, 在此基础上利用典则判别函数对每个个体进行预测分类。聚类分析通过系统聚类法, 计算平方欧式距离进行聚类。利用 POPGENE version1.32软件(Yeh et al, 1999)计算每个群体SNP位点的基因型频率、观测杂合度(observed heterozygosity,o)、期望杂合度(expected heterozygosity,e)、香农多样性指数()、基因流(N)、遗传分化系数(st)和遗传距离, 并进行哈迪-温伯格平衡检验(Hardy-Weinberg equilibrium, HWE)。4个位点的多态信息含量(polymorphism information content, PIC)利用 PIC- Cale(Shao et al, 2013)软件计算。根据每个群体间的Nei’s遗传距离, 使用MEGA 5(Tamura et al, 2011)软件, 利用NJ法绘制3个群体的群体聚类图。
2 结果
2.1 马氏珠母贝3个群体形态学分析
3个群体形态参数见表2, 父本群体的壳高、总重、壳重和软体部重显著高于母本群体, 母本群体的铰合线/壳长显著高于父本群体; F1的壳宽、壳高、软体部重等多个形态参数与母本相似, 与父本形态差异较大; F1在壳重指数显著高于两个亲本群体。
通过因子分析, 选择主成分贡献率较大的3个主成分进行负荷值计算(表3)。结果显示前3个主成分的贡献率分别为43.2%、27.6%和12.8%, 累计贡献率为83.6%。其中主成分1主要受总重、软体部重、壳高、壳重、铰合线长和壳长的影响, 主成分2主要受壳宽指数、壳宽和壳宽/壳长的影响, 结果表明这9个独立性状参数最能反映亲本群体和 F1间的外部形态差异。
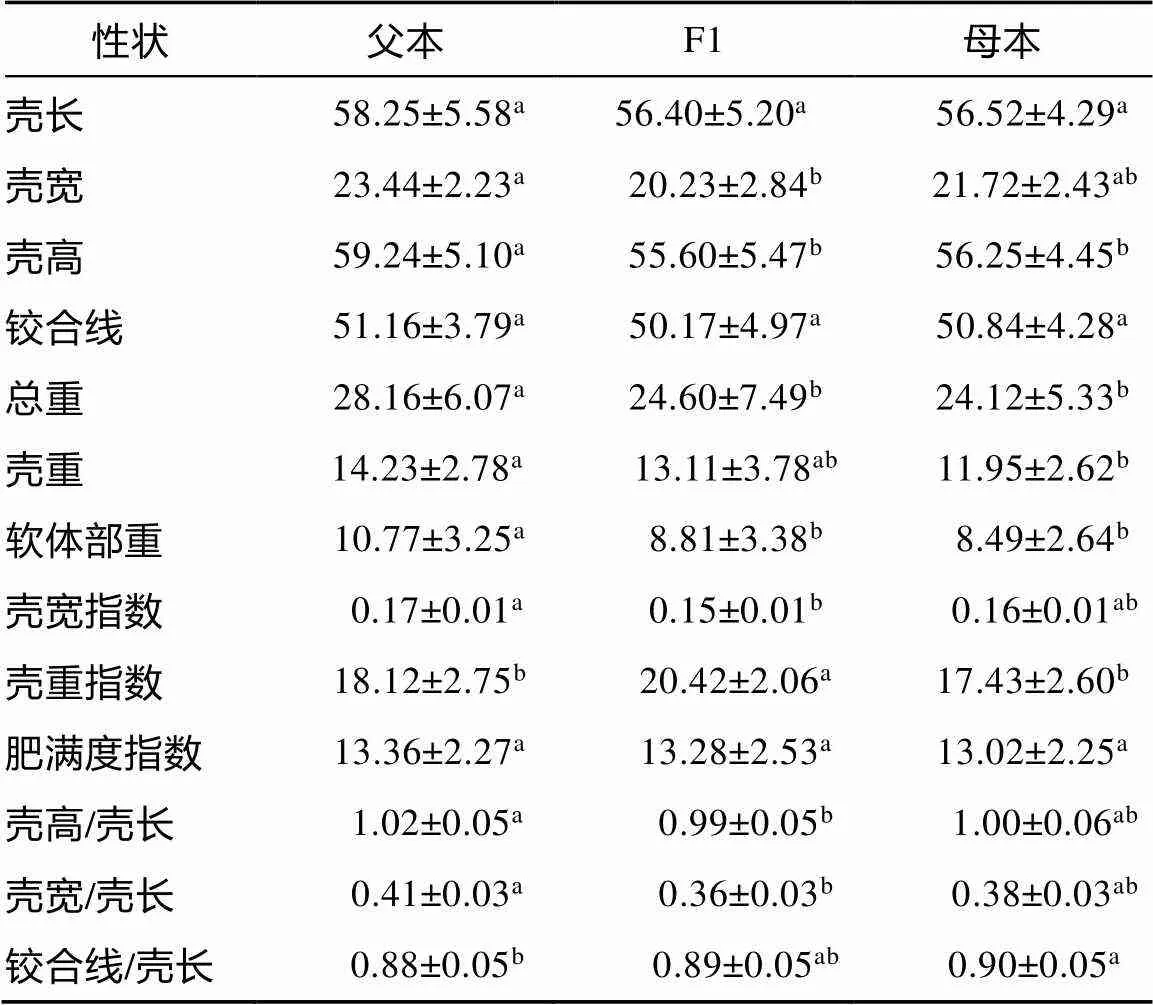
表2 马氏珠母贝3个群体主要形态参数的平均值
注: 上标不同字母之间表示存在显著性差异(<0.05)
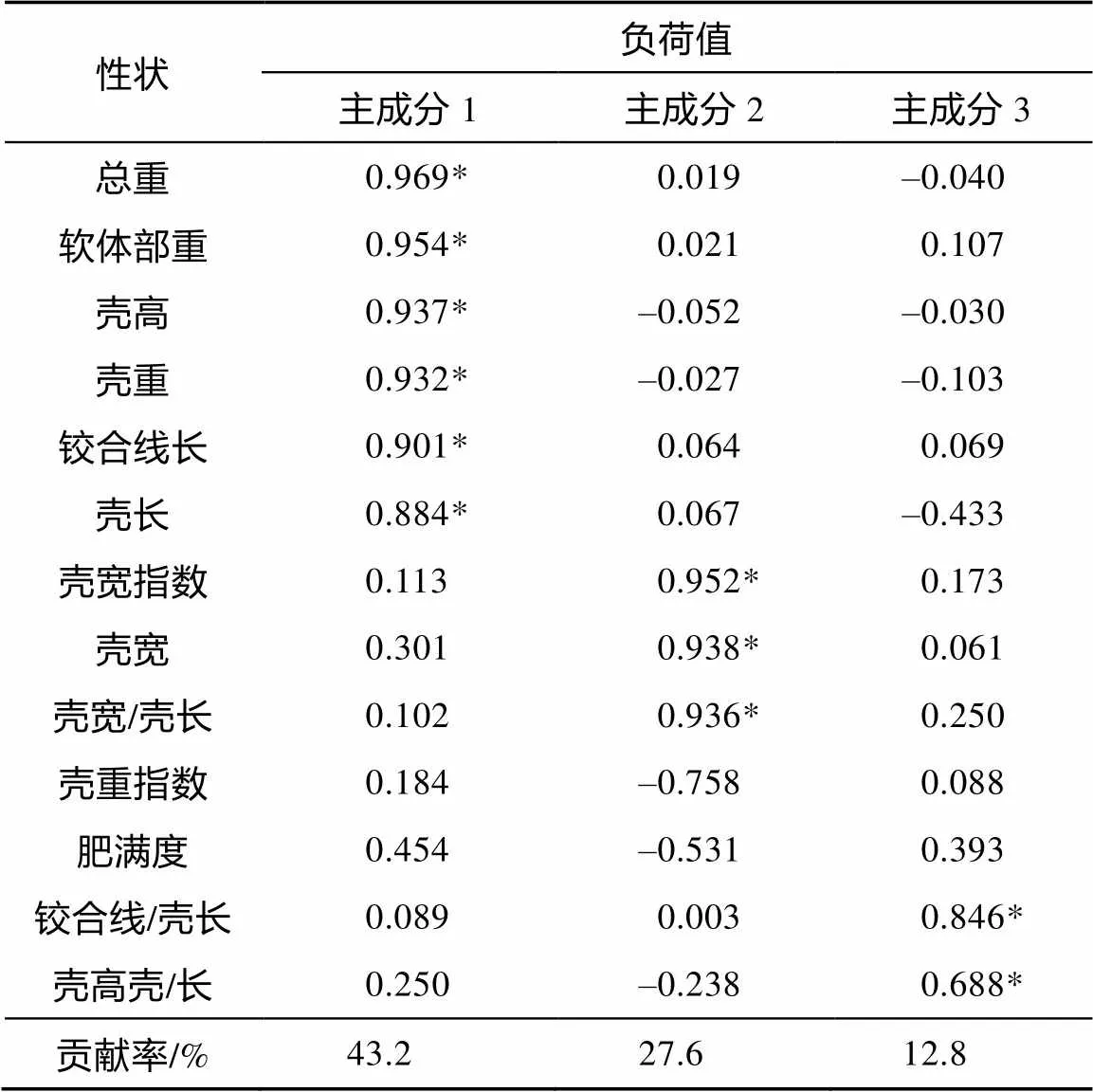
表3 马氏珠母贝3个群体形态参数主成分分析
注: *表示负荷值大于0.6(表示该因素可以有效的代表其主成分, 主成分贡献率越大表示该主成分越能反映群体之间的形态差异)
13个性状参数通过逐步判别分析经F检验, 结果表明除了壳宽/壳长, 其余12个形态形状可对群体进行判别。从马氏珠母贝3个群体的判别分析结果(表4)可知父本、F1、母本群体的判别准确率分别为72%、64%、80%, 综合判别率为72%, 3个群体的综合判别率不高。
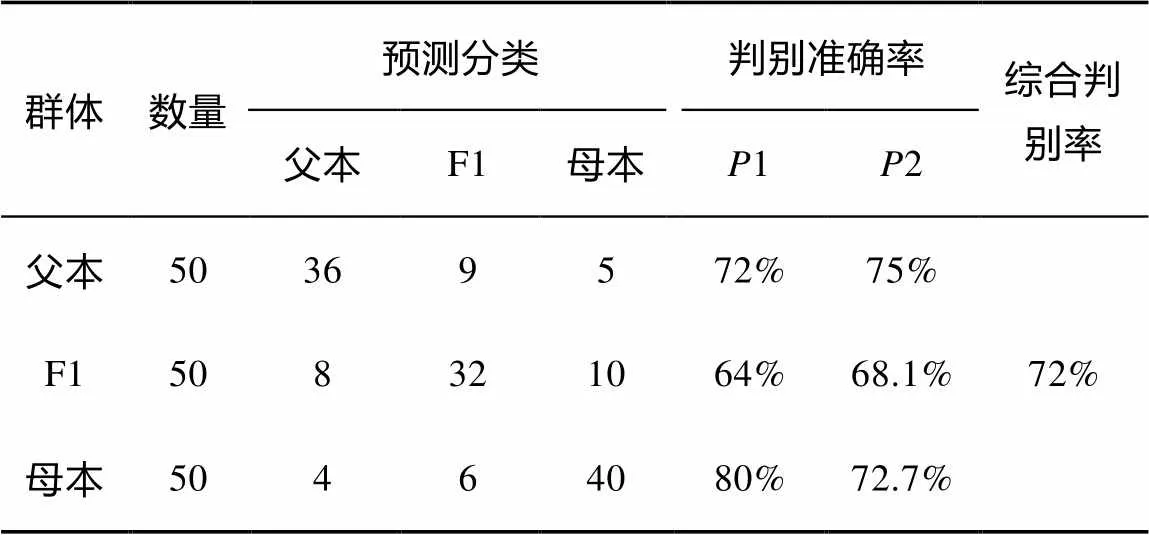
表4 判别函数对马氏珠母贝3个群体观测样本的预测分类及准确率

马氏珠母贝3个群体的形态聚类分析结果见图1。由图可知, 母本群体和F1最先聚为一类, 而父本群体单独聚为一支。该结果说明母本群体和F1的个体形态差异较小, 而父本群体与其他两群体之间形态差异较大。

图1 马氏珠母贝3个群体形态聚类图
2.2 SNP标记的遗传多样性参数分析
将获得的rs8、rs33、rs35、rs44标记的基因分型数据用软件分析后, 3个群体的e、o、a、e、、PIC和哈迪-温伯格平衡检验(Hardy-Weinberg equilibrium, HWE)值等遗传多样性参数见表5。结果显示, 这4个SNP标记的在3个群体中的香农多样性指数()为0.3111~0.6881, 多态信息含量(PIC)为 0.1555~0.3747, 表现为中度多态性(除了父本群体的rs8和rs35标记表现为低度多态性)。4个SNP标记在3个群体中平均杂合度o和e分别为0.2110~0.2879 和 0.3317~0.4685。3个群体总体呈现中等程度遗传多样性。
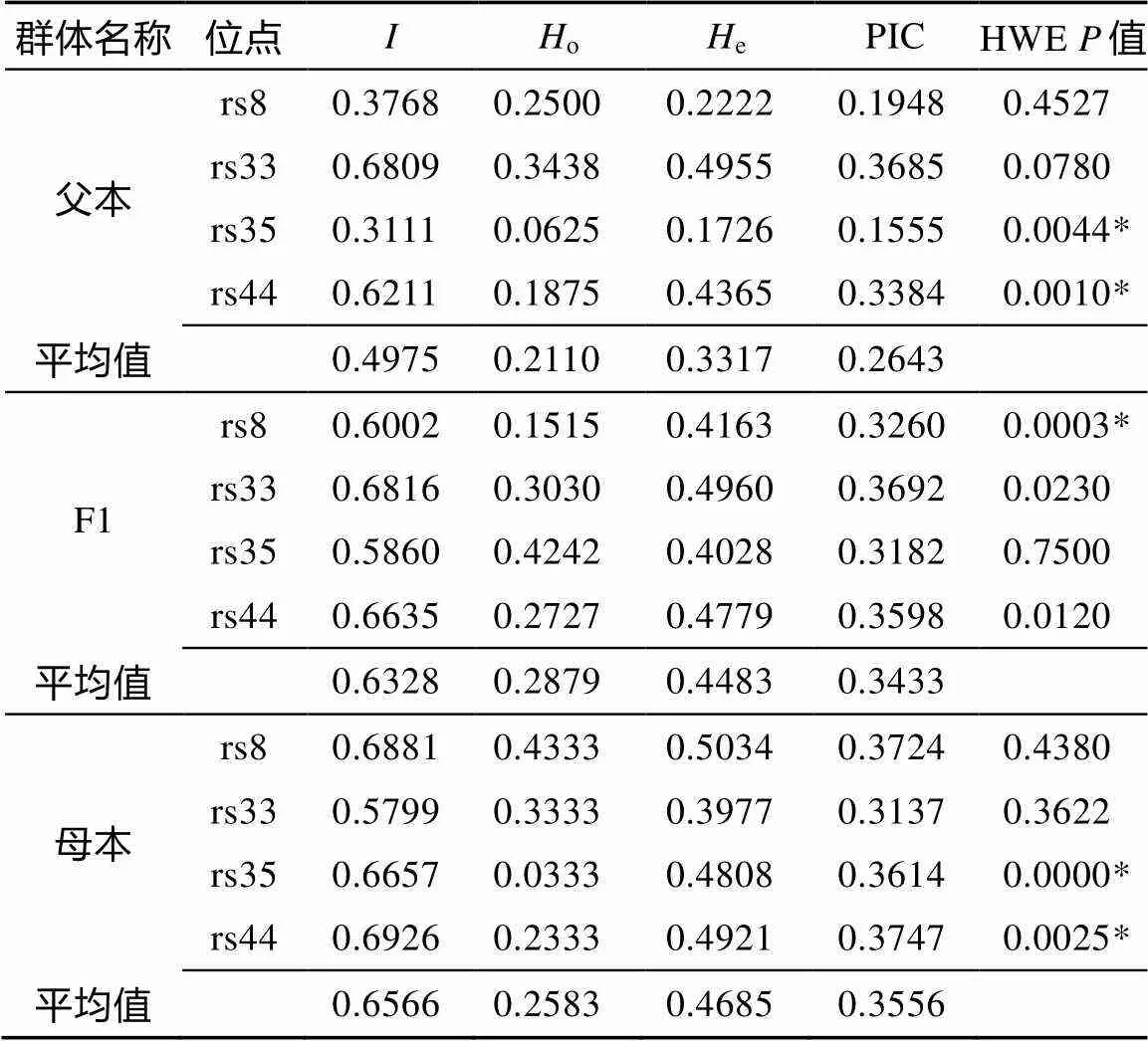
表5 马氏珠母贝3个群体遗传多样性参数
注:为香农多样性指数,o为观测杂合度,e为期望杂合度, PIC为多态信息含量, HWE为哈迪-温伯格平衡检验; *表示经过校正后仍显著偏离哈迪-温伯格平衡的位点(<0.01)
2.3 SNP标记的遗传结构分析
3个群体4个SNP位点(rs8、rs33、rs35、rs44)利用HRM方法成功分型, 分型结果见图2。计算出各个群体的基因型频率(表6)。从计算结果可知, 父本在rs8位点中基因型CC的基因型频率为0, 父本在该位点主要表现为纯合子TT(0.75), 而母本在该位点存在纯合子CC(0.33), 子代在该位点中基因型C来源于母本。母本群体在rs35位点的基因型CA的基因型频率(0.03)最小, 而父本在该位点的基因型CA、AA的基因型频率也较小(均为0.06)。
马氏珠母贝3个群体之间的遗传距离、遗传相似系数和基因流(m)、遗传分化系数(st)的分析数据见表7, 分析结果表明亲本群体之间的遗传距离最大(0.1759), 遗传相似系数最小(0.8387); F1和母本群体之间的遗传距离最小(0.0546), 遗传相似系数最大(0.9947)。3个群体之间的基因流N值均大于1, 说明3个群体之间存在基因流动; 亲本群体之间的基因流m值最小(1.9662), 表明亲本之间基因流动较小, 遗传分化较大; 而F1和母本群体之间的基因流m值最大(7.7701), 表明该两群体之间基因流动较大, 遗传分化较小。3个群体之间的遗传分化系数(st)在0.0166~0.1128之间, 表明1.66%~11.28%的遗传变异存在于群体之间, 88.72%~98.34%的遗传变异存在于群体内。
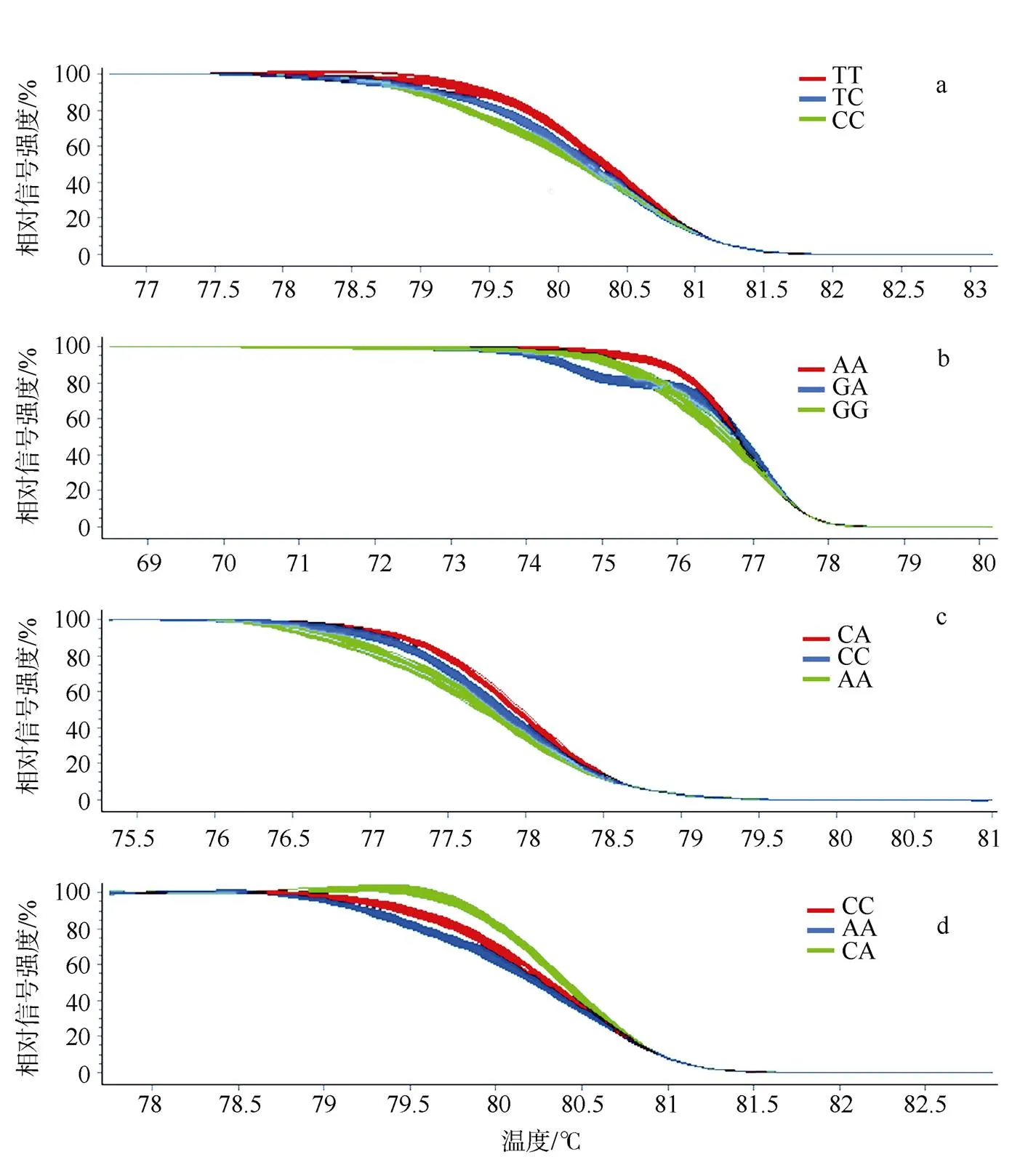
图2 马氏珠母贝3个群体4个位点分型结果图
a、b、c、d分别为rs8、rs33、rs35、rs44的HRM分型结果图, 不同颜色代表不同基因型
Fig. 2 The results of four loci typing in tree populations ofa, b, c and d are the results of HRM typing for rs8, rs33, rs35, and rs44, respectively, different colours represent different genotypes
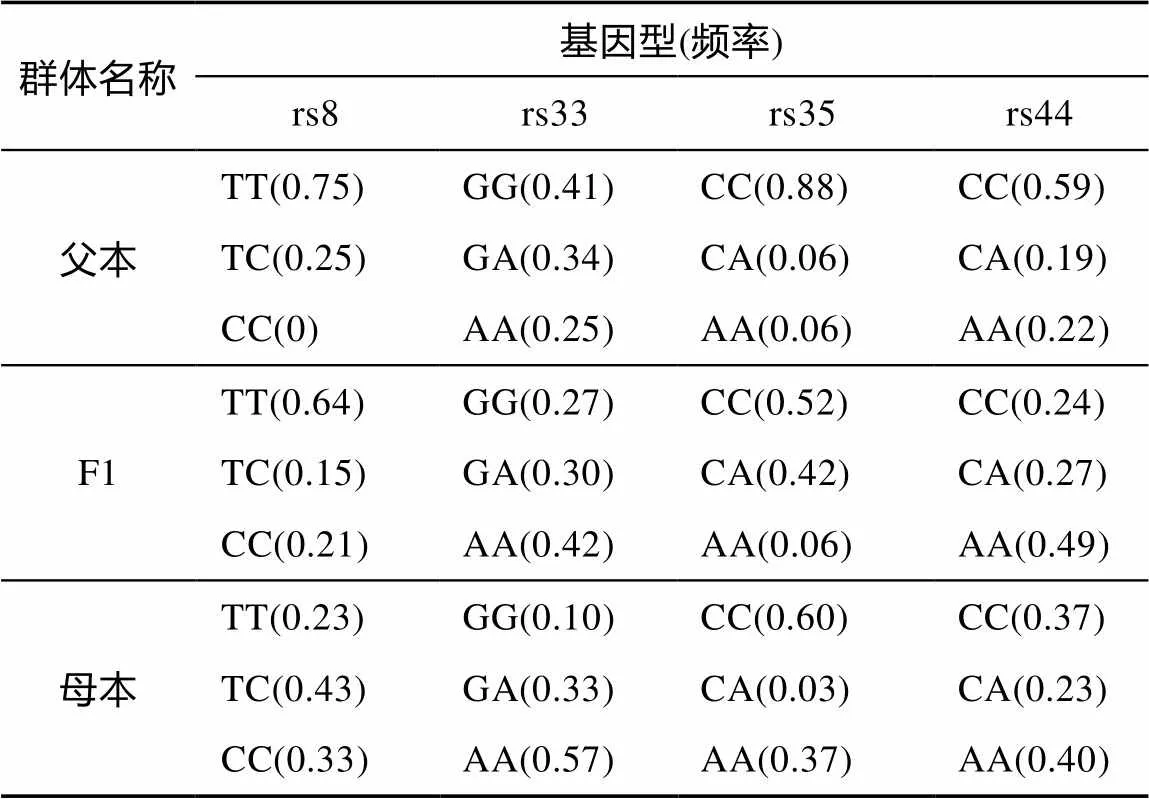
表6 马氏珠母贝3个群体SNP的基因型和频率

表7 马氏珠母贝3个群体遗传结构参数
注: 遗传距离和遗传相似系数中, ****下方数据为Nei's遗传距离, ****上方数据为遗传相似系数; 基因流和遗传分化系数中, ****下方数据为基因流N值, ****上方数据为遗传分化系数。基因流N值 = 0.25(1–st) /st,st为遗传分化系数
将获得的rs8、rs33、rs35、rs44标记的基因分型数据, 利用软件进行聚类分析, 绘制NJ进化树(图3)。母本群体最先与F1聚为一类, 父本群体单独聚为一支, 表明F1与母本群体之间亲缘关系最近, 而亲本群体之间亲缘关系最远。
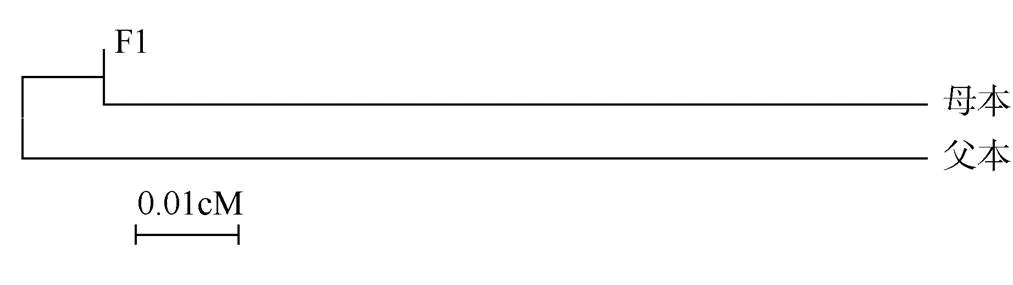
图3 马氏珠母贝3个群体基于分子标记的群体聚类图(NJ树)
3 讨论
3.1 亲本间遗传结构差异
一般来说, 表型性状常常用来对马氏珠母贝的不同群体和家系进行区分和判别, 例如壳长、壳宽、壳高、总重等(顾志峰等, 2009), 但对形态相似的群体进行分类难度较大。本研究通过测定的13个形态性状进行多元形态分析, 反映了3个群体形态上的差异和相似性。从外部形态数据可知, 亲本群体在壳高、总重、壳重和软体部重差异显著。从主成分分析可以看出亲本群体间的外部形态差异主要受总重、软体部重、壳高、壳重、铰合线长和壳长的影响, 这6个性状是区分2亲本外部形态的主要依据, 这一结果与外部形态分析较为一致。从判别分析结果可以看出母本群体的判别率(80%)要高于父本群体的判别率(72%), 判别效果较好, 说明母本群体内个体差异较小。而综合判别率为72%, 判别效果不高, 这可能与养殖过程中环境因素和人为主观选择因素有关(刘泽浩等, 2010)。罗会等(2013)曾对马氏珠母贝不同地理群体的形态差异进行研究, 结果发现广西、三亚、雷州和越南的4个地理群体综合判别率为57%, 认为较低的判别率可能受到主观选择因素影响, 导致群体间差异较小。聚类分析的结果发现亲本群体之间形态距离最远, 结合亲本群体间不高的判别率, 我们猜测亲本群体可能存在一定的基因流动(陈蓉等, 2009)。亲本群体的多元形态学分析的结果基本上可以区分两个群体, 但是进一步探究3亲本群体的遗传结构还需结合分子标记进行研究。
本实验室前期的研究中筛选出了在深圳群体和海南群体中基因型频率差异显著的位点: 海南群体rs8位点的基因型CC频率为0, rs35位点的基因型AA频率为0, rs44位点的基因型CA频率为0, 而深圳群体在rs8、rs35、rs44位点均存在3种基因型; 深圳群体rs33位点的基因型AA频率为0, 而海南群体存在3种基因型。本研究的SNP分型结果显示父本群体在rs8位点的基因型CC频率为0, 而母本群体存在3种基因型, 这一结果与前期工作的结果基本一致; 而父本群体在rs35位点出现了少量的AA基因型, 这可能是由于实验室前期工作研究样本量不足结果导致。分型结果表明rs8位点可以区分亲本群体, 成为特异性标记。
st(Wright, 1978)、遗传距离 (Weir et al, 1984) 和基因流是研究种群遗传分化的重要参数。Wright (1978)认为, 当st< 0.05时, 种群间分化很小; 0.05≤st≤ 0.15, 遗传分化程度中等。汤健等(2013)通过6个微卫星位点分析了9个马氏珠母贝养殖家系的遗传结构, 发现家系间st为0.0749, 处于中等遗传分化。和上述研究相较, 本次研究中亲本群体的st最大(0.1128), 说明亲本群体存在中等程度遗传分化,
当基因流(N) >1, 基因流可以阻止种群之间分化 (Wright, 1931)。本次研究结果中, 亲本群体之间的N最小(1.9662), 遗传距离最大(0.1759), 遗传分化较大, 和聚类分析结果一致。本实验室前期对海南群体和深圳群体遗传结构研究结果中发现, 海南群体和深圳群体的m为1.4114, 遗传距离为0.1595 (另文发表), 与本次的研究结果相似。在马氏珠母贝其他群体研究中, 侯战辉等(2008)用17个 EST-SSR 位点得出印度种群和三亚种群的st值为0.486, 遗传距离为1.119, 群体之间分化程度很高。王爱民等(2007), 利用6个SSR位点对印度群体和三亚群体杂交子代的遗传结构进行研究, 发现4组子代的st值为0.357, 存在较大的遗传差异和遗传分化。和上述结果相较, 本次研究的亲本群体的st值和遗传距离均偏小, 遗传分化中等。结合形态分析结果, 我们猜测可能是由于亲本群体之间存在基因交流导致种质资源受到污染, 使遗传距离偏小; 也可能本次研究SNP位点数目偏少, 导致遗传信息偏少。
3.2 杂交对后代遗传结构影响
通过形态分析发现, F1在多个形态参数上与父本差异显著, 而与母本没有显著性差异。F1判别分析的判别率为64%, 比亲本群体低。从个体分布来看, 50个个体中有10个个体错判给母本, 略高于父本群体(8个)。综合判别率不高的原因也与F1的低判别率有关, 董志国等(2008)对三角帆蚌3个地理种群杂交进行判别分析, 综合判别率为68.75%~ 79.78%, 认为该判别效果较为理想, 而且杂交群体的部分性状还需经过多代遗传才能稳定。形态聚类分析结果发现母本群体最开始与F1聚为一类, F1再与父本群体聚为一类, 结合判别分析结果可以得出F1在形态上与母本群体最相似, 也就是说表观遗传结构上更接近母本群体。
遗传多样性分析结构表明这4个位点呈现中度多态性(0.25 本次研究中F1和其他群体之间遗传分化很小, 这一结果与3个群体的系谱关系一致。其中, F1与母本群体的st最小(0.0166),m最大(7.7701), 遗传距离最小(0.0546)。因此相较于父本群体, F1与母本群体的基因流动更频繁, 遗传分化更小。利用NJ法对3个群体进行聚类分析, 结果显示F1与母本群体最先聚为一类, 亲缘关系最近, 而与父本群体亲缘关系最远, 这一结果和形态聚类结果较为一致。无论从形态还是分子遗传结构上, F1遗传结构上都更偏向母本群体。曲艳波等(2006) 用RAPD分析马氏珠母贝以印度群体为父本、三亚群体为母本进行杂交, 发现杂交子代与亲本之间的遗传距离不等, 且偏向母本。在其他贝类中也出现这种杂交子代与亲本遗传结果有差异的现象(李太武等, 2002), 这可能与亲本的遗传结构间的相互作用有关。 本研究中发现HRM分型方法在3个群体4个位点上成功区分了3种基因型, 结果说明HRM方法可以用于马氏珠母贝的SNP基因分型。传统的基因分型技术存在各式各样的缺点, 如AS-PCR的检测通量较低、灵敏度差错误率高(杨润婷等, 2013); SSCP检测成功率低、假阳性高; DHPLC法无法确定碱基突变位置; 高通量的测序技术成本相对较高。相较而言, HRM方法通量较高, 灵敏度也更高, 操作便捷。对于HRM进行分型, 需注意PCR产物特异性及其大小、GC含量、SNP类型和反应体系优化(Wittwer, 2009)。本研究使用罗氏所用的HRM法也有需要注意的地方, 如为了增加灵敏度, 产物长度应在80~100bp最优(Reed et al, 2004); HRM对SNP类型(A/T、G/C)的分型灵敏度不高, 而对G/A进行区分最容易(李纪勤, 2012), 因此选取SNP位点应严谨; 引物需提前进行PCR验证, 应无二聚体结构; 在扩增片段区域内出现多个SNP位点, 会干扰分型结果, 应避开SNP密集区域(Liew et al, 2004); 分型的DNA模板要求纯度较好, 进行反应的模板初始浓度在20~30ng·μL–1最优, 模板浓度过高会造成非特异性扩增(白牡丹等, 2012); 使用不同饱和染料时, 根据燃料特性调整扩增循环数, 例如SYBR Green I染料对DNA扩增存在一定抑制性, 应适当延迟5个循环数。由于HRM法反应灵敏, 对反应条件和模板要求较高, 因此在利用HRM法对马氏珠母贝的遗传结构和亲缘关系进行探究时, 可进一步优化反应体系, 探究反应条件, 达到准确分型结果。 总的来说, 结合形态学和SNP标记可以对两个亲本群体(深圳群体和海南群体)进行判别和区分, F1在形态和遗传结构上与母本群体更相似。 白牡丹, 王彩虹, 殷豪, 等, 2012. 苹果不同HRM反应体系分析效果评价[J]. 分子植物育种, 10(1): 115–120. BAI MUDAN, WANG CAIHONG, YIN HAO, et al, 2012. Evaluation of different reaction systems for HRM analysis in apple[J]. Molecular Plant Breeding, 10(1): 115–120 (in Chinese with English abstract). 陈亮, 赵柏淞, 李玲, 等, 2014. 人工诱导栉孔扇贝雌核发育胚胎的SNP标记分析[J]. 中国海洋大学学报, 44(2): 48–52. CHEN LIANG, ZHAO BOSONG, LI LING, et al, 2014. Genotyping artificially induced gynogenetic embryos of Zhikong scallop () with SNP markers[J]. Periodical of Ocean University of China, 44(2): 48–52 (in Chinese with English abstract). 陈蓉, 刘建勇, 唐连俊, 等, 2009. 中国5个海区不同群体毛蚶形态差异分析[J]. 海洋科学, 33(7): 64–69. CHEN RONG, LIU JIANYONG, TANG LIANJUN, et al, 2009. Morphological variations analysis of five different populations ofin China[J]. Marine Sciences, 33(7): 64–69 (in Chinese with English abstract). 董志国, 李家乐, 郑汉丰, 2008. 三角帆蚌3个地理种群自交与杂交F1代的形态差异分析[J]. 大连水产学院学报, 23(2): 92–97. DONG ZHIGUO, LI JIALE, ZHENG HANFENG, 2008. Morphological variability in original parents and their reciprocal hybrids F1 of fresh water musselfrom three geographical populations in China[J]. Journal of Dalian Fisheries University, 23(2): 92–97 (in Chinese with English abstract). 杜晓东, 李广丽, 刘志刚, 等, 2002. 合浦珠母贝2个野生种群的遗传多样性[J]. 中国水产科学, 9(2): 100–105. DU XIAODONG, LI GUANGLI, LIU ZHIGANG, et al, 2002. Genetic diversity of two wild populations in[J]. Journal of Fishery Sciences of China, 9(2): 100–105 (in Chinese with English abstract). 谷龙春, 李金碧, 喻达辉, 等, 2010. 合浦珠母贝双列杂交家系的建立与遗传分析[J]. 水产学报, 34(1): 26–31. GU LONGCHUN, LI JINBI, YU DAHUI, et al, 2010. Establishment and genetic analysis of complete diallel cross families of pearl oyster ()[J]. Journal of Fisheries of China, 34(1): 26–31 (in Chinese with English abstract). 顾志峰, 王嫣, 石耀华, 等, 2009. 马氏珠母贝两个不同地理种群的形态性状和贝壳珍珠质颜色比较分析[J]. 渔业科学进展, 30(1): 79–86. GU ZHIFENG, WANG YAN, SHI YAOHUA, et al, 2009. Comparison of morphometrics and shell nacre colour between two geographical populations of pearl oyster(Dunker)[J]. Marine Fisheries Research, 30(1): 79–86 (in Chinese with English abstract). 侯战辉, 王嫣, 石耀华, 等, 2008. 马氏珠母贝(Pinctada martensii)2个不同地理种群遗传变异的EST-SSR分析[J]. 海洋与湖沼, 39(2): 178–183. HOU ZHANHUI, WANG YAN, SHI YAOHUA, et al, 2008. EST-SSR analysis of genetic variation between two geographical populations of pearl oyster,(Dunker)[J]. Oceanologia et Limnologia Sinica, 39(2): 178–183 (in Chinese with English abstract). 胡笑蓉, 毛雄英, 王卫华, 等, 2012. 一种基于HRM技术的快速DNA甲基化检测方法[J]. 中国卫生检验杂志, 22(6): 1336–1338. HU XIAORONG, MAO XIONGYING, WANG WEIHUA, et al, 2012. A novel and reliable method of DNA methylation analysis based on HRM technology[J]. Chinese Journal of Health Laboratory Technology, 22(6): 1336–1338 (in Chinese with English abstract). 姜因萍, 何毛贤, 林岳光, 2007. 马氏珠母贝群体内遗传多样性的ISSR分析[J]. 海洋通报, 26(5): 62–66. JIANG YINPING, HE MAOXIAN, LIN YUEGUANG, 2007. Genetic diversity of a cultured population onDunker by ISSR marker[J]. Marine Science Bulletin, 26(5): 62–66 (in Chinese with English abstract). 金玉琳, 2014. 长牡蛎SNP标记开发及其在家系分析和物种鉴定中的应用[D]. 青岛: 中国海洋大学: 32–38. JIN YULIN, 2014. Development and characterization of SNP markers and their application in family analysis and species identification for Pancific oyster[D]. Qingdao: Ocean University of China: 32–38 (in Chinese with English abstract). 李纪勤, 2012. 栉孔扇贝()EST-SNP的开发及其应用[D]. 青岛: 中国海洋大学: 53–54. LI JIQIN, 2012. Development, characterization and application of EST-SNP markers in Zhikong scallop ()[D]. Qingdao: Ocean University of China: 53–54 (in Chinese with English abstract). 李梅, 吕伟朋, 韦鸿, 等, 2012. HRM方法检测非小细胞肺癌患者胸水标本癌细胞基因突变的临床意义[J]. 大连医科大学学报, 34(4): 321–323, 347. LI MEI, LÜ WEIPENG, WEI HONG, et al, 2012. Feasibility of HRM to detect gene mutations in hydrothorax of patients with non -small cell lung cancer[J]. Journal of Dalian Medical University, 34(4): 321–323, 347 (in Chinese with English abstract). 李太武, 孙修勤, 刘艳, 等, 2002. 中日栉孔扇贝杂交子一代群体的遗传变异[J]. 高技术通讯, 12(6): 101–105, 100. LI TAIWU, SUN XIUQIN, LIU YAN, et al, 2002. Allozyme variation of two subspecies ofand their reciprocal hybrids[J]. High Technology Letters, 12(6): 101–105, 100 (in Chinese with English abstract). 李耀国, 刘文广, 林坚士, 等, 2016. 马氏珠母贝SNP标记开发及家系遗传多态性分析[J]. 海洋通报, 35(1): 96–102. LI YAOGUO, LIU WENGUANG, LIN JIANSHI, et al, 2016. Development of SNP markers inand its application for family genetic analysis[J]. Marine Science Bulletin, 35(1): 96–102 (in Chinese with English abstract). 刘泽浩, 李捷, 杨学玲, 等, 2010 中国北方沿海潮间带常见镜蛤的种类[J]. 海洋科学, 34(12): 30–35. LIU ZEHAO, LI JIE, YANG XUELING, et al, 2010. Species of commonfrom intertidal zones of northern China[J]. Marine Sciences, 34(12): 30–35 (in Chinese with English abstract). 罗会, 刘宝锁, 黎火金, 等, 2013. 合浦珠母贝不同地理种群的形态差异和判别分析[J]. 广东农业科学, 40(12): 171–174. LUO HUI, LIU BAOSUO, LI HUOJIN, et al, 2013. Morphological variations and discriminant analysis on four populations of[J]. Guangdong Agricultural Sciences, 40(12): 171–174 (in Chinese with English abstract). 吕林兰, 杜晓东, 王嫣, 等, 2008. 马氏珠母贝3个野生种群及种群间杂交后代遗传多样性的ISSR分析[J]. 水生生物学报, 32(1): 26–32. LÜ LINLAN, DU XIAODONG, WANG YAN, et al, 2008. Genetic diversity of three popultions and the first generations of hybridization between different populations of pearl oyster,(Dunker)[J]. Acta Hydrobiologica Sinica, 32(1): 26–32 (in Chinese with English abstract). 曲艳波, 叶翚, 石耀华, 等, 2006. 马氏珠母贝杂交选育群体遗传变异的RAPD分析[C]//海南生物技术研究与发展研讨会论文集. 三亚: 中国生物工程学会: 27–34. QU YANBO, YE HUI, SHI YAOHUA, et al, 2006. RAPD analysis of genetic variation among cross breeding system of pearl oyster[C]//Proceedings of the Workshop on Research & Development of Biotechnology in Hainan. Sanya: Chinese Society of Biotechnology: 27–34 (in Chinese with English abstract). 沈恩健, 2010. 利用AFLP和SSR对南海区域马氏珠母贝群体的遗传结构分析[D]. 湛江: 广东海洋大学. SHEN ENJIAN, 2010. Genetic diversrty analyses of Pinctada fucata (dunker) in south China sea using AFLP and SSR[D]. Zhanjiang: Guangdong Ocean University (in Chinese with English abstract). 苏天凤, 蔡云川, 张殿昌, 等, 2002. 合浦珠母贝3个养殖群体的RAPD分析[J]. 中国水产科学, 9(2): 106–109. SU TIANFENG, CAI YUNCHUAN, ZHANG DIANCHANG, et al, 2002. RAPD analysis of three cultured populations of[J]. Journal of Fishery Sciences of China, 9(2): 106–109 (in Chinese with English abstract). 汤健, 管云雁, 刘文广, 等, 2013. 马氏珠母贝家系遗传结构的微卫星分析[J]. 海洋科学, 37(8): 35–41. TANG JIAN, GUAN YUNYAN, LIU WENGUANG, et al, 2013. Microsatellite DNA analysis of genetic structures about 9 families of[J]. Marine Sciences, 37(8): 35–41 (in Chinese with English abstract). 童晓飞, 郭莹, 丛晓霏, 2012. 高分辨溶解曲线及其在检测海洋经济贝类SNPs的应用前景[J]. 北京农业, (15): 139. TONG XIAOFEI, GUO YING, CONG XIAOFEI, 2012. The high-resolution melting and the application foreground used in detecting SNPs of the biomonitor[J]. Beijing Agriculture, (15): 139 (in Chinese with English abstract). 王爱民, 石耀华, 2007. 马氏珠母贝的遗传改良技术[M]//王清印. 海水养殖生物的细胞工程育种. 北京: 海洋出版社: 133–164. 王爱民, 王嫣, 顾志峰, 等, 2010. 马氏珠母贝()2个地理群体杂交子代的杂种优势和遗传变异[J]. 海洋与湖沼, 41(1): 140–147. WANG AIMIN, WANG YAN, GU ZHIFENG, et al, 2010. Heterosis and genetic variation of hybrids from two geographical populations of pearl oyster,[J]. Oceanologia et Limnologia Sinica, 41(1): 140–147 (in Chinese with English abstract). 王家丰, 2013. 长牡蛎基因区SNP标记规模开发及其在遗传育种研究中的应用[D]. 青岛: 中国科学院研究生院(海洋研究所). WANG JIAFENG, 2013. Large-scale development of SNP marker and its potential application in genetic breeding of[D]. Qingdao: Institute of Oceanology, Chinese Academy of Sciences (in Chinese with English abstract). 王艳, 赵雪, 姜振峰, 等, 2015. 大豆小片段法HRM基因分型体系优化[J]. 中国油料作物学报, 37(4): 453–461. WANG YAN, ZHAO XUE, JIANG ZHENFENG, et al, 2015. Optimization of small amplicon of HRM genotyping system in soybean[J]. Chinese Journal of Oil Crop Sciences, 37(4): 453–461 (in Chinese with English abstract). 杨润婷, 吴波, 李翀, 等, 2013. 两种SNP分型方法的比较及其在柚品种鉴定中的应用[J]. 园艺学报, 40(6): 1061–1070. ZHAO RUNTING, WU BO, LI CHONG, et al, 2013. Comparison of allele-specific PCR and high resolution melting analysis in SNP genotyping and their application in Pummelo cultivar identification[J]. Acta Horticulturae Sinica, 40(6): 1061–1070 (in Chinese with English abstract). 喻达辉, 李有宁, 吴开畅. 2005. 中国、日本和澳大利亚珍珠贝的ITS2序列特征分析[J]. 南方水产, (02): 4–9. YU DAHUI, LI YOUNING, WU KAICHANG, 2005. Analysis on sequence variation of ITS 2 rDNA infrom China, Japan and Australia[J]. South China Fisheries Science, 1(2): 1–6 (in Chinese with English abstract). 张莉, 2007. 中国珍珠产业的问题、困境与出路[J]. 农业现代化研究, 28(4): 443–445. ZHANG LI, 2007. Economical analysis of dilemma of Chinese pearl industry and outlet[J]. Research of Agricultural Modernization, 28(4): 443–445 (in Chinese with English abstract). 赵杰, 游新勇, 徐贞贞, 等, 2018. SNP检测方法在动物研究中的应用[J]. 农业工程学报, 34(4): 299–305. ZHAO JIE, YOU XINYONG, XU ZHENZHEN, et al, 2018. Review on application of SNP detection methods in animal research[J]. Transactions of the Chinese Society of Agricultural Engineering, 34(4): 299–305 (in Chinese with English abstract). 赵琼一, 李信, 周德贵, 等, 2010. 后基因组时代下作物的SNP分型方法[J]. 分子植物育种, 8(1): 125–133. ZHAO QIONGYI, LIN XIN, ZHOU DEGUI, et al, 2010. SNP genotyping methods for crops in post-genomic era[J]. Molecular Plant Breeding, 8(1): 125–133 (in Chinese with English abstract). BOTSTEIN D, WHITE R L, SKOLNICK M, et al, 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 32(3): 314–331. GANAL M W, ALTMANN T, RÖDER M S, 2009. SNP identification in crop plants[J]. Current Opinion in Plant Biology, 12(2): 211–217. HINE P M, THORNE T, 2000. A survey of some parasites and diseases of several species of bivalve mollusc in northern Western Australia[J]. Diseases of Aquatic Organisms, 40(1): 67–78. HONG S P, SHIN S K, LEE E H, et al, 2008. High-resolution human papillomavirus genotyping by MALDI-TOF mass spectrometry[J]. Nature Protocols, 3(9): 1476–1484. LIEW M, PRYOR R, PALAIS R, et al, 2004. Genotyping of single-nucleotide polymorphisms by high-resolution melting of small amplicons[J]. Clinical Chemistry, 50(7): 1156–1164. LIU WENGUANG, HUANG XIANDE, LIN JIANSHI, et al, 2012. Seawater acidification and elevated temperature affect gene expression patterns of the pearl oyster[J]. PLoS One, 7(3): e33679. MIYAKET, ISOWA K, ISHIKAWAT, et al, 2016. Evaluation of genetic characteristics of wild and cultured populations of the Japanese pearl oysterby using AFLP markers[J]. Aquaculture International, 24(2): 537–548. MIYAZAKI T, GOTO K, KOBAYASHI T, et al, 1999. Mass mortalities associated with a virus disease in Japanese pearl oysters[J]. Diseases of Aquatic Organisms, 37(1): 1–12. MONTGOMERY J, WITTWER C T, PALAIS R, et al, 2007. Simultaneous mutation scanning and genotyping by high- resolution DNA melting analysis[J]. Nature Protocols, 2(1): 59–66. NEFF M M, NEFF J D, CHORY J, et al, 1998. dCAPS, a simple technique for the genetic analysis of single nucleotide polymorphisms: experimental applications ingenetics[J]. The Plant Journal, 14(3): 387–392. NEWTON C R, GRAHAM A, HEPTINSTALL L E, et al, 1989. Analysis of any point mutation in DNA. The amplification refractory mutation system (ARMS)[J]. Nucleic Acids Research, 17(7): 2503–2516. NICKERSON M L, WEIRICH G, ZBAR B, et al, 2000. Signature-based analysis of MET proto-oncogene mutations using DHPLC[J]. Human Mutation, 16(1): 68–76. REED G H, WITTWER C T, 2004. Sensitivity and specificity of single-nucleotide polymorphism scanning by high- resolution melting analysis[J]. Clinical Chemistry, 50(10): 1748–1754. SHAO KE, XIONG MEIHUA, XU NIAN, et al, 2013. Characterization of microsatellite loci inand cross-amplification in four other Chinese cyprinid species[J]. Conservation Genetics Resource, 5(1): 9–13. SOBRINO B, BRIÓN M, CARRACEDO A, 2005. SNPs in forensic genetics: a review on SNP typing methodologies[J]. Forensic Science International, 154(2–3): 181–194. TAMURA K, PETERSOND, PETERSON N, et al, 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 28(10): 2731–2739. WADA K T, KOMARU A, 1991. Estimation of genetic variation in shell traits of the Japanese pearl oyster[J]. Bulletin of National Research Institute of Aquaculture. (Japan)/Yoshokukenho, (20): 19–24. WADA K T, KOMARU A, 1994. Effect of selection for shell coloration on growth rate and mortality in the Japanese pearl oyster,[J]. Aquaculture, 125(1–2): 59–65. WEIR B S, COCKERHAM CC, 1984. Estimating-statistics for the analysis of population structure[J]. Evolution, 38(6): 1358–1370. WITTWER C T, 2009. High-resolution DNA melting analysis: Advancements and limitations[J]. Human Mutation, 30(6): 857–859. WRIGHT S, 1931. Evolution in Mendelian populations[J]. Genetics, 16(2): 97–159. WRIGHT S, 1978. Evolution and the genetics of populations, vol. 4. variability within and among Populations[M]. Chicago: University of Chicago Press. YEHF C, YANG R C, BOYLE T, et al, 1999. POPGENE, version 1.32: The user friendly software for population genetic analysis[CP]. Canada: University of Alberta. YOKOGAWA K, 1998. Morphological variabilities and genetic features in Japanese common clam[J]. Venus(Japanese Journal of Malacology), 57(2): 121–132. YUDAHUI, CHU KAHOU, 2006. Low genetic differentiation among widely separated populations of the pearl oysteras revealed by AFLP[J]. Journal of Experimental Marine Biology and Ecology, 333(1): 140–146. Morphological and SNP markers for analysis of genetic structure of hybrid progeny and their parental populations of HUANG Jing1,2,3, PAN Xiaolan1,2,3, XU Meng1,2,3, LIU Wenguang1,3, ZHANG Hua1,3, HE Maoxian1,3 1. Key Laboratory of Tropical Marine Bio-resources and Ecology, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, China;2. University of Chinese Academy of Sciences, Beijing 100049, China;3. Guangdong Provincial Key Laboratory of Applied Marine Biology, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, China The pearl oyster,, is a primary marine bivalve species. To study the genetic structure and relationship of cultured parents and their hybrid generation, external morphology and genetic structure were assessed in the three populations offrom Shenzhen population (female), Hainan population (male) and their hybrid generation F1, based on morphological multivariate analysis and SNP markers. The results showed that the average discriminant accuracy was 72%, and the morphological characteristics of F1 and female were similar, while male was the most different from the other two populations. Four SNP markers of the three populations were genotyped by the HRM (high resolution melting) method. The results showed that the average observed (o) and expected (e) heterozygosity were 0.2110 to 0.2879 and 0.3317 to 0.4685, respectively; and the heterozygosity of F1 was higher than that of the parent populations. The average polymorphism information content (PIC) values of the three populations were in the range of 0.2643 to 0.3556, which showed moderate genetic diversity. Gene flow (N) between F1 and female was the largest (7.7701), with minimum genetic distance being 0.0546; thus, their genetic relationships were similar. Gene flow (N) between the parent populations was minimum (1.9662), with largest genetic distance being 0.1759. Finally, we found that rs8 marker can be used as a specific marker to discriminate parent populations. These results should assist the discriminate of genetic structure and management of selective breeding of. ; genetics; genetic structure; morphology; SNP 10.11978/2019012 http://www.jto.ac.cn Q953; Q958.8 A 1009-5470(2019)06-0080-10 2019-01-21; 2019-02-27。 林强编辑 国家贝类产业技术体系建设专项(CARS-49) 黄景(1993—), 男, 湖北省黄石市人, 硕士研究生, 研究方向为马氏珠母贝分子遗传育种。E-mail: 1152524871@qq.com 何毛贤。E-mail: hmx2@scsio.ac.cn, 电话: 020-89023144 2019-01-21; 2019-02-27. Editor: LIN Qiang Modern Agro-industry Technology Research System(CARS-49) HE Maoxian. E-mail: hmx2@scsio.ac.cn, Tel: 020-890231443.3 HRM方法优化

