基于级联全卷积神经网络的颈部淋巴结自动识别算法
秦品乐 李鹏波 曾建潮 朱辉 徐少伟


摘 要:针对现有算法自动识别颈部淋巴结效率不高、存在大量假阳性且整体假阳性去除效果不理想的问题,提出一种基于级联全卷积神经网络(FCN)的颈部淋巴结识别算法。首先,结合医生的先验知识采用级联FCN进行初步识别,即第一个FCN从头颈部计算机断层扫描图像(CT)中提取淋巴结医学分区;然后,第二个FCN从分区内提取候选样本并在三维层面合并这些样本以生成三维图像块;最后,将提出的特征块平均池化引入到三维分类网络中,对输入的不同尺度三维图像块进行二分类以去除假阳性。
在颈部淋巴结数据集中,采用级联FCN识别颈部淋巴结的召回率可达97.23%;引入特征块平均池化的三维分类网络的分类准确率可达到98.7%。在去除假阳性之后的准确率可达93.26%。实验结果分析表明,所提算法能有效实现颈部淋巴结的自动识别并取得较高的召回率和准确率,优于目前相关文献报道的算法;且算法简单高效,易于扩展到其他三维医学图像的目标检测任务中。
关键词: 颈部淋巴结检测;计算机辅助诊断;全卷积神经网络;假阳性去除;三维医学影像
中图分类号:TP391.3
文献标志码:A
Abstract: The existing automatic recognition algorithms for cervical lymph nodes have low efficiency, and the overall false positive removal are unsatisfied, so a cervical lymph node detection algorithm using cascaded Fully Convolutional Neural Networks (FCNs) was proposed. Firstly, combined with the prior knowledge of doctors, the cascaded FCNs were used for preliminary identification, that was, the first FCN was used for extracting the cervical lymph node region from the Computed Tomography (CT) image of head and neck. Then, the second FCN was used to extract the lymph node candidate samples from the region, and merging them at the three-dimensional (3D) level to generate a 3D image block. Finally, the proposed feature block average pooling method was introduced into the 3D classification network, and the 3D input image blocks with different scales were classified into two classes to remove false positives. On the cervical lymph node dataset, the recall of cervical lymph nodes identified by cascaded FCNs is up to 97.23%, the classification accuracy of the 3D classification network with feature block average pooling can achieve 98.7%. After removing false positives, the accuracy of final result reaches 93.26%. Experimental results show that the proposed algorithm can realize the automatic recognition of cervical lymph nodes with high recall and accuracy, which is better than the current methods reported in the literatures; it is simple and efficient, easy to extend to other tasks of 3D medical images recognition.Key words: cervical lymph node detection; computer-aided diagnosis; Fully Convolutional Neural (FCN) network; false positive removal; Three-Dimensional (3D) medical imaging
0 引言
頸部淋巴结引流丰富,全身约800枚淋巴结,其中约300枚位于头颈部。淋巴结作为人体非常重要的免疫器官,起着过滤淋巴、清除细菌和异物、产生淋巴细胞和抗体等作用。头颈部肿瘤种类多,约占全身恶性肿瘤的6%,而其中颈部淋巴结转移癌约占头颈部恶性肿瘤总数的75%[1]。全身的癌肿一经侵犯淋巴系统,均有可能转移至颈部淋巴结,同时头颈部区域的原发肿瘤也极易引发颈部淋巴结转移,颈部淋巴结的异常往往表示其引流范围内存在感染灶。大量的手术与病理资料证实,头颈部肿瘤的淋巴结转移存在规律,即仅向其邻近区域进行转移。就特定肿瘤而言,还存在淋巴结转移的高危区域[2]。研究报告显示,如果及早发现和治疗,颈淋巴癌患者的5年存活率将提高40%左右[3]。因此,检测和识别颈部淋巴结对于及早发现和治疗颈部淋巴癌具有重要意义。
在颈部淋巴结诊断中,传统的触诊对肿瘤是否发生转移具有很高的假性结论,需借助医学影像辅助检查。计算机断层扫描(Computed Tomography, CT)是目前检测颈部淋巴结最可靠的方法,它较好地对软组织或器官成像,能良好地显示出病变的区域。在CT图像中,颈部区域的淋巴结与周围组织界限模糊,CT值与周围的软组织相近,且淋巴结的病理复杂、形态小、不规则。颈部淋巴结的生理特征和图像的复杂性使得放射科医师提取非常困难。医生根据各自经验进行判断,费时费力且结论会随各自主观经验判断而存在误差,这导致结果存在大量的假阳性。对淋巴结检测和分析的常用手段是传统的机器学习算法。魏骏等[4]提出基于统计学的半自动分割算法,采用遍历阈值提取淋巴结种子点,加入基于统计学的强制停止条件,对种子点进行区域生长,实现淋巴结分割及其体积的计算。Kan等[5]提出基于支持向量机的淋巴结判决算法,依据淋巴结及周围软组织生理特征来识别淋巴结区域。
深度学习作为机器学习的一个重要分支,近几年得到了迅猛发展。随着强大高效的图形处理器(Graphic Processing Unit, GPU)以及大量可用的训练数据出现,卷积神经网络(Convolutional Neural Network, CNN)在视觉识别领域取得多项突破[6-7],并且在医疗图像上也取得了不错的成就[8-9]。与传统的机器学习算法相比,卷积神经网络的优势在于特征提取过程无需人工参与,通过端到端的形式自动提取深层次特征。
目前深度学习应用于医学影像的检测和分析通常采用两阶段:1)提取候选样本;2)去除假阳性。针对第一阶段,Lee等[10]提出CNN-GAP算法,将VGG(Visual Geometry Group)网络与全局平均池化(Global Average Pooling, GAP)[11]相结合,在超声图像上绘制热图来定位转移性淋巴结区域并区分良恶性。虽然该算法在超声影像中识别转移性淋巴结存在优势,但不同影像类型的淋巴结形态表现及特征差异较大,且该算法的定位结果较为粗糙,对于CT图像中识别淋巴结的问题并不适用。目前最先进的通用物体检测模型Faster R-CNN(Faster Region-Based Convolutional Networks)在自然图像上表现优异,在医学影像上也有广泛应用。
Ding等[12]采用改进的Faster R-CNN,在特征提取网络的最后一层加入反卷积层以恢复更多细粒度特征,能有效提高对CT图像中较小尺寸肺结节的识别能力。该算法虽然在识别小尺寸目标方面存在优势,但易受预设锚框的影响,导致同时识别大小目标的效果较差,且颈部区域杂质较多,容易导致大量假阳性样本,对于CT图像中识别淋巴结的效果并不理想。相对于目标检测算法,基于像素的分割算法能够利用目标周围的上下文信息实现更精确的定位,在算法复杂度和识别结果等方面往往能取得不错的效果。Christ等[13]提出全卷积神经网络(Fully Convolutional Neural network, FCN)与迁移学习相结合,对CT图像中肝脏内部病变区域进行分割,并使用条件随机场进行后处理得到分割结果。由于颈部区域的淋巴结与周围组织间的界限模糊、血管等杂质较多且淋巴结目标较小,直接分割原始CT图像容易产生较多假阳性样本。为避免这种情况,需要在前处理阶段提取淋巴结感兴趣区域。针对第二阶段,Xie等[14]提出训练三个模型来分别提取样本的三个轴切面信息,最后对分类结果进行投票,用于去除肺部结节的假阳性。然而该算法中每个模型仅学习二维平面信息,丢失了空间信息,且多网络投票机制消耗资源,造成去假阳性效果不理想。通过单一网络在特征层面融合三个轴切面信息,有助于提高准确率。Roth等[15]提出了新型的2.5D算法,采用样本的三个轴切面组成三通道伪彩图像,输入到二维卷积神经网络进行分类,用于去除腹部及纵膈淋巴结的假陽性。该算法虽然整合了样本的轴切面信息,但对于样本的整体空间信息利用不足,导致对难样例的区分效果不好。将样本输入三维(Three Dimension, 3D)分类网络,网络充分利用样本的完整空间信息学习到更具代表性的判别特征,有助于提高对难样例的判别能力。Ding等[12]与苗光等[16]在去假阳性部分的算法较为相似,以所有候选样本中心点为基准裁剪为相同尺寸,输入到3D网络进行分类,用于去除肺部结节的假阳性。该类算法虽然能保留样本完整的空间信息,取得明显效果,但由于网络中全连接层的影响,仅限于输入固定大小的样本,而淋巴结尺度变化明显,在传统3D网络上表现不佳。为避免这种情况,就要求网络同时兼顾大小目标的识别。
颈部淋巴结与周围组织间的界限模糊,具有病理复杂、形态小、不规则、分布区域广泛等特点。目前深度学习应用于识别颈部淋巴结领域的研究较少,仍需解决以下两个关键问题:1)由于受到血管等无关组织的干扰,利用深度学习直接从CT图像中提取,将产生大量假阳性样本;2)针对形态尺度差异较大的样本,采用传统3D分类网络同时识别较大目标与较小目标的难度较大,整体去假阳性的效果并不理想。
针对颈部淋巴结自动识别的关键问题,本文提出基于级联全卷积神经网络的识别算法。针对第一个关键问题,本文提出级联FCN的实现方式,首先提取淋巴结医学分区,继而从分区内提取候选样本。针对第二个关键问题,本文提出特征块平均池化理念,替代传统3D网络的全连接层以便允许网络输入不同尺度样本。与传统方法相比,本文算法能有效解决深度学习应用于识别淋巴结的两个关键问题,并大幅提高识别的召回率和准确率。该算法可作为颈部淋巴结的预筛查工具,对医生的辅助诊断治疗具有重要的意义。
1 颈部淋巴结识别
1.1 总体框架
整体框架如图1所示。
本文算法主要分为两个阶段:1)提取淋巴结候选样本;2)去除假阳性。第一阶段:采用级联FCN,首先从CT图像中分割出肌肉等无关组织,得到淋巴结医学分区,进而从分区内提取候选样本;第二阶段:将特征块平均池化引入到3D分类网络中,对输入不同尺度的候选3D样本进行二分类,得到分类结果。
1.2 提取候选
提取淋巴结候选样本作为整个模型中的关键步骤,其目的在于保持高灵敏度的同时限制候选样本的数量。由于两方面的原因,本文采用先提取淋巴结医学分区,然后从分区内提取候选样本的方案:1)颈部淋巴结形态各异且与周围组织间的界限模糊,血管、食道、肌肉等组织干扰淋巴结的识别;2)虽然颈部淋巴结分布广泛,但是分布具有规律性,医学上根据淋巴结相邻的组织器官及血管,将颈部淋巴结分布区域划分为七大分区。故本文采用先提取淋巴结医学分区,然后从分区内提取候选样本的方案。
1.2.1 改进U-Net网络
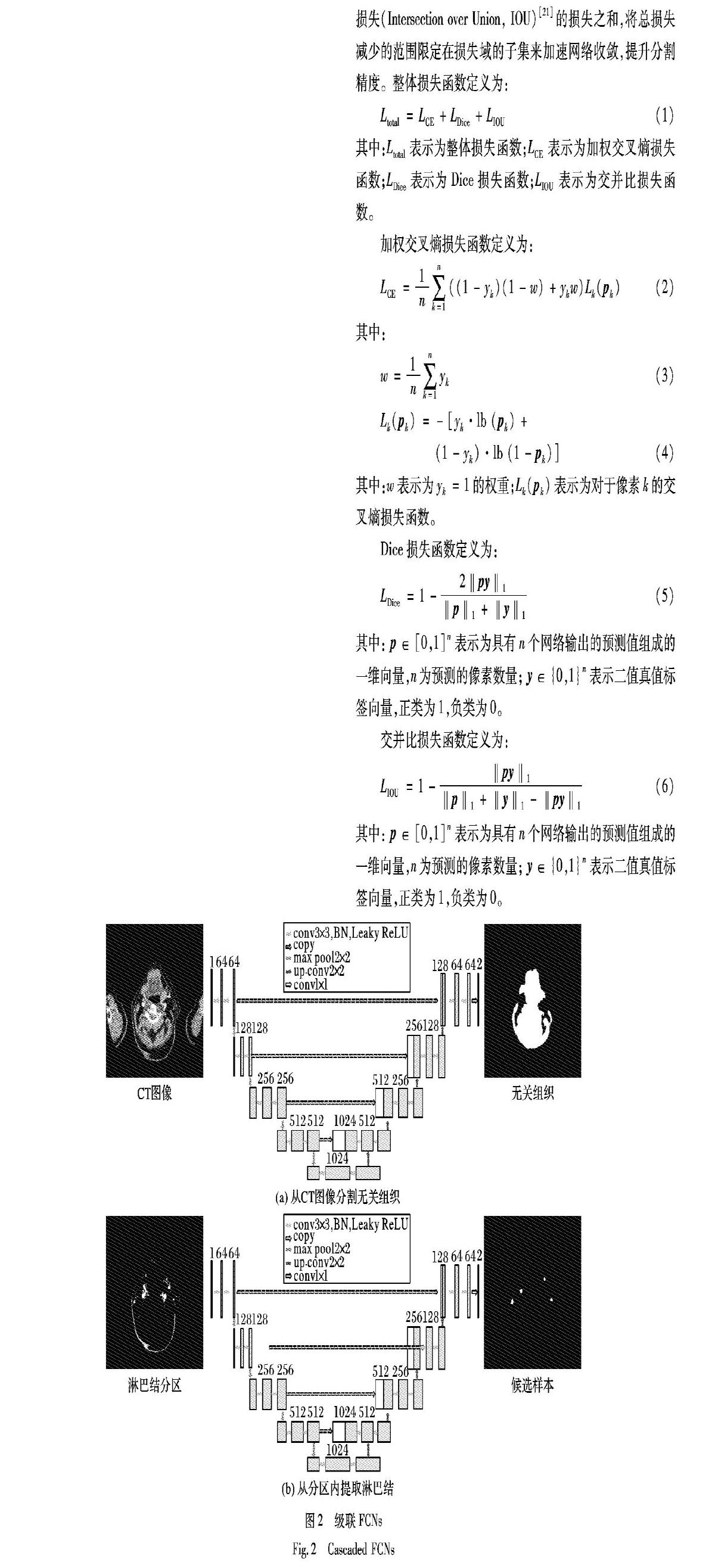
本文采用级联FCN提取淋巴结分区及候选样本,如图2所示,具体FCN选用改进的全卷积神经网络U-Net。由于原始的全卷积神经网络U-Net[17]在提取淋巴结问题上表现不佳,故采用改进的U-Net网络:首先将网络输入大小调整为CT图像的大小;其次,网络层数较深导致计算量过大,使得网络只能接受较小批次的图像,网络训练速度较慢,故在网络中添加批量归一化(Batch Normalization, BN)[18],激活函数替换为Leaky-ReLU(Leaky Rectified Liner Uints)[19],加速网络收敛,并提高网络的泛化能力,减少过拟合;最后,对于分割医学影像时面临的类别严重不平衡问题,损失函数采用联合损失,即加权交叉熵损失(Cross Entropy, CE)、Dice损失[20]、交并比损失(Intersection over Union, IOU)[21]的损失之和,将总损失减少的范围限定在损失域的子集来加速网络收敛,提升分割精度。整体损失函数定义为:
从CT图像中提取淋巴结分区如图3所示。由于颈部淋巴结医学分区的形状变化过于明显,不易直接提取,故选择根据医生的先验知识,从CT图像上剥离肌肉等无关组织,进而得到淋巴结分区。具体实现为:1)将图像范围调整为-140~260HU(Hounsfield),采用改进U-Net分割出肌肉等无关组织;2)将图像范围调整为20~80HU后减去无关组织部分,得到淋巴结医学分区。经过咨询放射科医师,将图像范围调整为-140~260HU的原因为该范围下肌肉等组织与淋巴结之间的界限相对明显,有助于剥离无关组织的同时,尽可能保留所有真实淋巴结,保证淋巴结的高召回率。之后将调整为20~80HU的原因为该范围下淋巴结显示效果最为明显且干扰因素较少,以便更精确地提取淋巴结候选样本。
从分区内提取淋巴结候选样本如图4所示。在20~80HU范围的淋巴结分区图像中,采用第二个改进U-Net基于淋巴结的标注真值进行训练,从分区内提取淋巴结候选样本,之后将候选样本的掩码轮廓转化为对应的最小外接矩形框,达到提取淋巴结候选样本的目的。
1.2.3 合并候选样本
为了获取候选样本的空间信息,需要合并CT序列中多层切片之间的候选样本,通过坐标之间的欧氏距离合并相近的候选样本,并利用3D非极大值抑制(Non Maximum Suppression,NMS)减少候选样本数量。通过判断候选3D样本与真值3D样本之间的体素交并比[22]来确定候选样本的正
1.3 去除假阳性
提取候选阶段之后,生成较多的候选3D样本,主要由血管与淋巴结组成,两者在二维平面上灰度值接近,形态大小相似,无法区分,而在三维空间中,血管呈现柱状结构,而淋巴结呈现为结状,两者的立体形态截然不同,故去除假阳性时必须结合样本的空间信息。
1.3.1 网络结构
3D分类网络以VGG网络结构为基础,其结构如图5所示,由6个三维卷积层、3个三维最大池化层、特征块平均池化层及二分类层组成。第一、二层是连续的两个卷积层,由32个3×3×3的卷积核组成,第三、四层是连续的两个卷积层,由64个3×3×3的卷积核组成,第五、六层是连续的两个卷积层,由128个3×3×3的卷积核组成,其中每个三维卷积层的步长均为1、填充均为0。连续卷积之后加入空间批量归一化、修正线性单元,以提取淋巴结内部的空间信息。第一个三维最大池化层由32个1×2×2的最大池组成,第二个三维最大池化层由64个2×2×2的最大池组成,第三个三维最大池化层由128个2×2×2的最大池组成,其中每个三维最大池化层后均加入参数为0.5的Dropout层,以避免过拟合,提高模型稳定性。之后特征块平均池化取代传统3D网络的全连接层,以允许网络输入不同尺度3D样本进行训练;最后接入二分类层,采用二值交叉熵(Binary Cross Entropy, BCE)作为损失函数,输出分类结果。
1.3.2 特征块池化
不同尺度3D样本x×y×z输入网络后,首先通过特征提取网络,结合样本原始空间信息提取潜在的深层特征,输出128個m×n×k的三维特征块。通过特征块池化方式提取每个特征块的一个值,代表该特征块的深层次特征,最终计算得到128个神经元的值,用以代表输入的3D样本整体的深层次特征。最后将128维深层次特征输入到二分类层进行判别,输出分类结果。最终实现基于特征块池化的3D网络允许输入不同尺度的3D样本进行训练的目的。
对于特征块池化方式,本文提出两种:特征块最大池化(Feature Block Maximum Pooling, FBMP)和特征块平均池化(Feature Block Average Pooling, FBAP)。前者仅提取每个特征块内部的最大激活值,容易导致过拟合,不能代表该特征块的整体激活信息;后者通过计算每个特征块内部激活值的均值,汇总整体激活信息,起到正则化作用,防止过拟合,代表该特征块的整体激活信息。
对比传统3D网络,基于特征块池化的3D网络优势在于:1)基本不改变3D样本大小,完整保留样本的原始空间信息;2)针对形态尺度差异较大的3D样本,同时兼顾较大目标与较小目标的识别;3)在不增加计算量的前提下使得网络训练时间更短、精度更高。
2 实验设计与结果分析
2.1 实验数据集
实验数据集来源于山西省肿瘤医院CT放射科,影像类型为静脉增强的头颈部CT序列图像,原始图像大小为512×512pixel,切片厚度为0.625mm。参照标准由4名经验丰富的专家手工注释。为了保证注释的完整性和准确性,4名放射科医师对每张头颈部CT图像标注两遍,结果包含两种真值:组织区域掩码、淋巴结掩码。第一遍由4名专家手动标注组织区域掩码,以便提取淋巴结医学分区;第二遍由4名专家独立标注淋巴结掩码。图6为数据集中不同形态及分布的颈部淋巴结样本。最终挑选出直径大于3mm并由3名及以上专家共同注释的颈部淋巴结。最终获得70例,共计2220张CT图像,其中包含1140张颈部淋巴结图像。
2.2 模型训练方法
实验数据分为两部分:80%用于训练级联FCN模型和训练3D分类模型;剩余的20%作为测试数据来测试淋巴结的召回率、准确率等。针对数据增强,级联FCN模型的数据集使用随机镜像翻转、随机正负30°内旋转、随机裁剪及弹性形变等数据增强策略;3D CNN模型训练的数据集,由于正样本数量较少,为保持正负样本均衡,首先在(x, y,z)三个方向进行[-3,3]内的随机平移,然后根据3D样本的中心坐标进行[90°,180°,270°]三个角度的随机旋转进行增广,最后保持正负样本的比例接近1∶1。针对模型优化,级联FCN模型采用随机梯度下降(Stochastic Gradient Descent, SGD)算法,学习率初始化为0.01,衰减率设为0.1,批处理大小设置为8,动量设置为0.9,训练轮次为100,并在第30、60、90轮次时衰减学习率;三维分类模型除批处理大小设置为16以外,其余训练参数均与FCN模型保持一致。所有训练的网络模型均在GPU Tesla M40上使用Pytorch深度学习框架实现,全部模型训练约为12.5h,测试所有病例的总时间约为120s(15例CT序列,包含450张CT图像)。
2.3 结果分析
2.3.1 评价指标
针对级联FCN模型,采用召回率来分析提取候选样本的性能。召回率r的计算公式为:其中:Tt为所有被检测出的真实颈部淋巴结的个数;Ff为所有未检测出的真实颈部淋巴结的个数;Tt+Ff为理论上检测到的所有真实颈部淋巴结的个数。
针对3D分类模型,由于在去除假阳性过程中需要同时保证正负类的分类精度,而准确率(Accuracy, ACC)可以充分反映分类网络准确识别真阳性和假阴性的比率,故采用准确率来分析其对候选样本的分类性能。准确率ACC的计算公式为其中:Tt为所有被正确分类的淋巴结数量;Tf为所有被正确分类的负类数量;Ft为被误分类为淋巴结的负类数量;Ff为被误分类的淋巴结数量。
2.3.2 候选识别结果与分析
在候选识别阶段,通过对比召回率、每次扫描假阳性数、耗时等因素,分析不同提取算法对于提取候选的影响。为保证公平比较,除对比算法不同之外,采用同一批颈部淋巴结数据集并使用相同的数据增强策略。
该阶段对提取候选的主流方法及本文方法进行对比实验,包括:Ding等[12]提出的改进Faster R-CNN算法,Ronneberger等[17]提出的U-Net算法,苗光等[16]提出的改进U-Net算法。基线算法为对比测试,在FCN提取淋巴结医学分区的基础上形态学提取候选。本文算法为级联FCN算法。
作为主流的医学影像目标检测框架,凭借设计优秀的区域候选网络(Region Proposal Network, RPN)及锚框设置取得相对较高的召回率,但由于CT图中颈部区域血管、食道、肌肉等无关组织的干扰,產生大量假阳性样本,对最终识别结果造成很大影响。分割模型U-Net通过分析目标周围上下文信息来限制假阳性样本数量,相较Faster R-CNN网络结构简单,在假阳性样本数量及运行时间上优势明显,但召回率相对较低。
2)第2~3行为改进分割网络的对比。U-Net虽然限制假阳性样本数量,且相对耗时最少,但由于网络过深,导致网络训练速度较慢,召回率较差。Improved U-Net在网络结构中加入批量归一化、Leaky-Relu等,并采用联合损失进行训练,使得网络训练快速收敛,并提高网络的泛化能力,在其他指标表现相当的前提下召回率相较于U-Net有一定提升。
3)第3~5行为单阶段与两阶段提取的对比。
Improved U-Net虽然在运算耗时较少的前提下达到较高的召回率,但由于颈部区域众多干扰因素影响,候选样本数量仍然较高。而基线算法和本文算法通过两阶段的方式,先提取淋巴结感兴趣区域,避免无关组织干扰,在提高召回率的同时大幅减少假阳性样本数量,但由于多阶段的影响造成耗时增加。
4)第4~5行为传统形态学方法与分割网络的对比。
基线算法通过FCN提取淋巴结分区来避免无关组织干扰,使得召回率及假阳性样本数量均保持较好的水平,但在分区内仅依据淋巴结形态特点筛选符合条件的轮廓来提取候选样本,存在部分杂质轮廓被错误提取为候选样本。本文算法在基线算法基础上,采用FCN取代形态学,网络能够学习更深层次且更丰富的特征来提取候选,达到保持高召回率的同时限制候选样本数量,但相比其他所有算法耗时最长。
2.3.3 去除假阳性结果与分析
在去除假阳性阶段,通过对比准确率、耗时等因素,分析不同算法对于分类识别性能的影响。训练时为保证公平比较,除对比算法不同之外,采用同一批淋巴结候选样本数据并使用相同的数据增强策略。该阶段对去假阳性的主流方法及本文方法进行对比实验,包括:Xie等[14]提出的多网络投票算法,Roth等[15]提出的2.5D CNN,苗光等[16]提出的3D CNN算法。
基线算法为对比测试,基于特征块最大池化的3D CNN算法。本文算法为基于特征块平均池化的3D CNN算法。
1)第1~2行为轴切面信息融合方式的对比。2D CNN算法训练三个模型分别提取三个轴切面信息,最后融合分类结果,其中每个模型仅学习单个轴切面的二维信息,导致其在准确率和运行时间等方面均表现不佳;2.5D CNN算法采用单一的二维网络将三个轴切面信息在特征层面融合,使得准确率和运行时间等方面相较于2D CNN提升明显,它相比其他所有算法耗时最少。
2)第2~3行为不同网络类型的对比。2.5D CNN算法将轴切面组成三通道伪彩图像,仅利用轴切面信息,对于样本整体的空间信息利用不足,导致对难样例的区分效果较差,准确率难以继续提升。而3D CNN算法充分利用样本的完整空间信息学习到更具代表性的判别特征,有助于提高对难样例的识别能力,但由于网络参数及计算量增加,导致相较于2D CNN耗时增加。
3)第3~5行为全连接与特征块池化的对比。3D CNN算法由于全连接层的存在,网络训练时需输入固定大小的样本,导致损失一些对分类结果非常重要的深层次特征,相较于基于特征块池化的网络准确率不佳,且全连接层导致耗时增加。基线算法与本文算法将3D CNN的全连接层替换为特征块池化,允许网络输入不同尺度的样本,兼顾较大目标与较小目标的识别,在网络参数减少的同时起着正则化作用,使得在准确率和耗时等方面相较于3D CNN有一定提升。
4)第4~5行为不同特征块池化的对比。基线算法将3D CNN的全连接层替换为特征块最大池化,由于最大池化仅提取每个三维特征块内部的最大激活值,容易导致过拟合。
相比基线算法,本文算法通过特征块平均池化替换特征块最大池化,计算每个三维特征块内部激活值的均值,汇总整体激活信息,起到正则化作用,防止过拟合,相比其他所有算法在运行耗时相对较少的情况下取得最高的准确率。
2.3.4 整体结果与分析
整体算法流程分为两阶段:第一阶段,采用级联FCN提取淋巴结医学分区及分区内提取候选样本,然后合成对应的候选3D样本。第二阶段,针对输入数据,若候选3D样本的深度过小,则插值到网络输入的最小尺度;否则不调整,最大限度保留样本的空间信息。针对网络,3D CNN_FBAP对输入的不同尺度候选样本进行分类,得到分类结果。从图8~9中可看出,在去除假阳性之后,最终准确率稳定在93.26%。
本文算法在自动识别颈部淋巴结时,仍然存在一些局限性:
1)在提取候选阶段,如图10所示,易识别样本分布独立、易于识别;部分难样例为与周围组织存在粘连且灰度值相近,在提取分区时需要很高的识别精度。而级联FCN的识别精度有限,可能导致某些淋巴结被误认为肌肉等多余组织而产生错误分割的情况。针对这种情况,将探索更适合这种情况下的识别算法。
2)在生成候选3D样本阶段,依据坐标之间欧氏距离将距离接近的样本进行合并,由于可能出现正样本和负样本的欧氏距离更接近的情况,导致合并后的候选3D样本并非理想的3D样本。针对这种情况,将尝试提出更适合的距离度量算法来克服这种局限性。
3)相比其他算法,本文算法虽然在召回率及准确率等方面存在优势,但整体识别耗时相对较长。针对这种情况,将尝试端到端的网络来保持识别优势的同时缩短耗时。
3 结语
针对现有算法识别颈部淋巴结效率不高,存在大量假阳性且整体假阳性去除效果不理想的问题, 本文提出了一种新的基于级联全卷积神经网络的识别算法。实验结果表明,针对提取候选样本,采用级联FCN可以保持高灵敏度的同时限制候选样本的数量;针对目标形态尺度差异较为明显的3D样本,基于特征块平均池化的3D分类网络允许输入不同尺度的3D样本,充分利用3D样本的原始空间信息去学习更深层次的判别特征,获得最优的分類精度。在3D医学图像的研究中,该算法相对简单高效,且能够更为精确地执行检测任务,对临床的诊断和治疗具有重要的意义。下一步将利用PSPNet(Pyramid Scene Parsing Network)、Mask R-CNN(Mask Region-Based Convolutional Networks)等较为复杂但精度更高的分割网络来提取淋巴结样本,并探索将多阶段网络融合为端到端网络,以便提高精度的同时减少耗时。
参考文献(References)
[1] PATEL N U, LIND K E, McKINNEY K, et al. Clinical validation of a predictive model for the presence of cervical lymph node metastasis in papillary thyroid cancer[J]. American Journal of Neuroradiology, 2018, 39(4): 756-761.
[2] SPINELLI C, TOGNETTI F, STRAMBI S, et al. Cervical lymph node metastases of papillary thyroid carcinoma, in the central and lateral compartments, in children and adolescents: predictive factors[J]. World Journal of Surgery, 2018, 42(8): 1-10.
[3] BEJNORDI B E, VETA M, van DIEST P J, et al. Diagnostic assessment of deep learning algorithms for detection of lymph node metastases in women with breast cancer[J]. JAMA, 2017, 318(22): 2199-2210.
[4] 魏駿, 何凌, 车坤, 等. CT 图像的颈部淋巴结半自动分割算法[J]. 计算机工程与设计, 2015, 36(11): 3014-3018. (WEI J, HE L, CHE K, et al. Semi-automatic detection and segmentation of neck lymph nodes in CT image[J]. Computer Engineering and Design, 2015, 36(11): 3014-3018.)
[5] KAN Y, DONG D, ZHANG Y, et al. Radiomic signature as a predictive factor for lymph node metastasis in early-stage cervical cancer[J]. Journal of Magnetic Resonance Imaging, 2019, 49(1): 304-310.
[6] ESTEVES C, ALLEN-BLANCHETTE C, MAKADIA A, et al. Learning SO (3) equivariant representations with spherical CNNs[EB/OL]. [2019-01-10]. http://openaccess.thecvf.com/content_ECCV_2018/papers/Carlos_Esteves_Learning_SO3_Equivariant_ECCV_2018_paper.pdf.
[7] ELSAYED G F, SHANKAR S, CHEUNG B, et al. Adversarial examples that fool both computer vision and time-limited humans [EB/OL]. [2019-01-10]. http://papers.nips.cc/paper/7647-adversarial-examples-that-fool-both-computer-vision-and-time-limited-humans.pdf.
[8] RAJPURKAR P, IRVIN J, ZHU K, et al. CheXNet: radiologist-level pneumonia detection on chest X-rays with deep learning[EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1711.05225.pdf.
[9] RAJPURKAR P, HANNUN A Y, HAGHPANAHI M, et al. Cardiologist-level arrhythmia detection with convolutional neural networks[EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1707.01836.pdf.
[10] LEE J H, BAEK J H, KIM J H, et al. Deep learning-based computer-aided diagnosis system for localization and diagnosis of metastatic lymph nodes on ultrasound: a pilot study[J]. Thyroid, 2018, 28(10): 1332-1338.
[11] ZHOU B, KHOSLA A, LAPEDRIZA A, et al. Learning deep features for discriminative localization[C]// Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Piscataway: IEEE, 2016: 2921-2929.
[12] DING J, LI A, HU Z, et al. Accurate pulmonary nodule detection in computed tomography images using deep convolutional neural networks[C]// Proceedings of the 2017 International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 10435. Berlin: Springer, 2017: 559-567.
[13] CHRIST P F, ETTLINGER F, GRN F, et al. Automatic liver and tumor segmentation of CT and MRI volumes using cascaded fully convolutional neural networks[EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1702.05970.pdf.
[14] XIE H, YANG D, SUN N, et al. Automated pulmonary nodule detection in CT Images using deep convolutional neural networks[J]. Pattern Recognition, 2018, 85: 109-119.
[15] ROTH H R, LU L, SEFF A, et al. A new 2.5D representation for lymph node detection using random sets of deep convolutional neural network observations[C]// Proceedings of the 2014 International Conference on Medical Image Computing and Computer-Assisted Intervention. Berlin: Springer, 2014: 520-527.
[16] 苗光, 李朝鋒. 二维和三维卷积神经网络相结合的CT图像肺结节检测方法[J]. 激光与光电子学进展, 2018, 55(5): 129-137. (MIAO G, LI C F. Detection of pulmonary nodules CT images combined with two-dimensional and three-dimensional convolution neural networks[J]. Laser & Optoelectronics Progress, 2018, 55(5): 129-137.)
[17] RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[C]// Proceedings of the 2015 International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 9351. Berlin: Springer, 2015: 234-241.
[18] IOFFE S, SZEGEDY C. Batch normalization: accelerating deep network training by reducing internal covariate shift[EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1502.03167.pdf.
[19] HE K, ZHANG X, REN S, et al. Delving deep into rectifiers: Surpassing human-level performance on ImageNet classification[C]// Proceedings of the 2015 IEEE International Conference on Computer Vision. Piscataway: IEEE, 2015: 1026-1034.
[20] SHEN C, ROTH H R, ODA H, et al. On the influence of dice loss function in multi-class organ segmentation of abdominal CT using 3D fully convolutional networks[EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1801.05912.pdf.
[21] RISTER B, YI D, SHIVAKUMAR K, et al. CT organ segmentation using GPU data augmentation, unsupervised labels and IOU loss[EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1811.11226.pdf.
[22] CHOY C B, XU D, GWAK J, et al. 3D-R2N2: a unified approach for single and multi-view 3D object reconstruction[C]// Proceedings of the 2016 European Conference on Computer Vision, LNCS 9912. Berlin: Springer, 2016: 628-644.

