大豆类黄酮糖基转移酶基因UGT73C19的功能研究
狄少康,尹青岗,夏亚迎,,庞永珍
大豆类黄酮糖基转移酶基因的功能研究
狄少康1,尹青岗2,夏亚迎1,2,庞永珍1
(1中国农业科学院北京畜牧兽医研究所,北京 100193;2中国科学院植物研究所,北京 100093)
类黄酮是大豆中积累的一类重要的植物次生代谢产物,参与大豆的生长、发育和抗逆等诸多生理活动。由UDP-糖基转移酶(UGT)催化的糖基化修饰是类黄酮生物合成的关键步骤。【】通过系统研究大豆9编码重组酶的体外酶活特性和体内特性,完善大豆黄酮类化合物合成和积累的机制,为大豆品质的遗传改良提供基因资源和理论基础。通过高效液相色谱(HPLC)的方法检测大豆核心种质资源叶片中类黄酮的种类和含量,通过qRT-PCR的方法检测了的表达水平。以大豆Williams 82叶片cDNA为模板,克隆得到的编码区序列。使用MEGA5和DNAMAN软件进行多重序列比对,并构建进化树。通过原核表达系统获得UGT73C19的重组蛋白,分析UGT73C19重组蛋白对各种类黄酮苷元的糖基转移活性,并通过高效液相色谱-质谱(HPLC-MS)对产物进行鉴定,确定重组蛋白的糖基化位点。利用qRT-PCR技术对在大豆不同组织的表达水平进行分析。构建植物过量表达载体,通过花序浸染法转化拟南芥,获得表达量高的纯合株系,检测转基因株系叶片和种子中类黄酮的种类和含量。通过HPLC分析大豆核心种质资源叶片类黄酮成分,发现不同品种中类黄酮的成分和含量存在明显差异。根据类黄酮成分的不同,将大豆核心种质分为12种不同的类型。大豆核心种质资源叶片中总黄酮的含量与的表达水平呈正相关关系。克隆得到的编码区序列,全长1 482 bp,编码493个氨基酸,UGT73C19蛋白在C-端有一个保守的PSPG结构域。体外酶活分析表明,重组的UGT73C19蛋白对6种类黄酮苷元(山奈酚、槲皮素、杨梅素、芹黄素、大豆苷元和染料木素)都具有糖基转移活性,其中对槲皮素的催化效率最高;糖基化位点分别位于类黄酮的5位和7位羟基上,重组UGT73C19蛋白的糖基化底物和位点具有多样性。过量表达的拟南芥叶片和种子中的类黄酮总量明显升高,其中叶片中总黄酮含量提高49%—70%,种子中总黄酮的含量提高34%—37%;尤其是种子中槲皮素3-鼠李糖的含量显著增加。UGT73C19蛋白是催化合成大豆中多个类黄酮糖苷的关键糖基转移酶,过量表达可以提高转基因植物中黄酮醇糖苷和类黄酮的含量。
大豆;糖基转移酶基因;类黄酮;黄酮醇
0 引言
【研究意义】大豆()是一种重要的经济作物,不仅储存了大量的蛋白质与油脂,而且富含多种次生代谢产物,特别是黄酮、黄酮醇、异黄酮等类黄酮化合物[1-2]。大豆类黄酮广泛参与共生、防御和抗性等生理过程[3-5]。此外,大豆类黄酮还具有抗氧化、清除氧自由基和抗癌等对人类健康有益的功能[6-9]。因此,阐明大豆类黄酮的未知生物合成途径,将有利于提高大豆类黄酮的含量,改良大豆的品质。【前人研究进展】尽管类黄酮的基本骨架结构十分保守,然而糖基化、甲基化和酰基化等多种修饰反应使类黄酮的结构更加复杂和多样化。其中,由UDP-糖基转移酶(UDP-glycosyltransferase,UGT)催化的糖基化反应是类黄酮生物合成途径末端的关键步骤,糖基化可以提高类黄酮的稳定性、多样性和生物活性[10-12]。Yonekura-Sakakibara等[13]通过预测植物UGT家族的序列相似性,发现大豆中存在200多个,但是绝大多数大豆的功能还未被阐明。研究表明,在大豆种皮中特异表达仅对花青素和黄酮醇具有催化活性[14]。体外表达的重组蛋白IF7GT可以糖基化异黄酮,生成异黄酮7--葡萄糖苷(isoflavone 7--β-D-glucosides)[15]。另外4个UGT88家族的重组蛋白(UGT88E14、UGT88E15、UGT88E16和UGT88E18)可以特异催化异黄酮(主要是genistein和daidzein)形成不同类型的异黄酮糖苷。特别是重组UGT73F2蛋白对3种异黄酮(daidzin、genistin和glycitin)均具有糖基转移活性[16]。近期,YIN等[17]从更新的大豆基因组中对进行了全基因组水平的鉴定和分析,共鉴定212个候选基因,发现它们以成簇的方式广泛分布于20个染色体,对其中的188个表达谱进行了分析。从中克隆并详细研究了11个的特征,发现其中6个重组UGT蛋白(UGT72X4、UGT72Z3、UGT73C20、UGT88A13、UGT88E19和UGT92G4)具有糖基化黄酮醇、黄酮、异黄酮的活性,它们在大豆毛状根和拟南芥中的过量表达可以显著提高黄酮醇和/或异黄酮的含量[17]。【本研究切入点】虽然在大豆中已经鉴定了若干个糖基化类黄酮的[14-17],但是相对于大豆中存在的上百种糖基化的类黄酮[1,18]和200多个大豆而言[17,19],目前已经被功能鉴定的仅仅是很少的一部分。由于大豆及其功能的多样性、普遍性和复杂性[13,17],还需要更深入地研究的特性及其功能,以期系统阐明大豆类黄酮的生物合成机制。【拟解决的关键问题】本研究利用大豆基因组和表达谱数据,筛选到一个与大豆叶片类黄酮积累水平相关的候选(),发现该基因的表达水平与大豆核心种质资源中的类黄酮含量正相关。通过克隆该基因并分析其重组蛋白的底物及其酶动力学参数;在拟南芥中异源过量表达,阐明对拟南芥叶片和种子黄酮醇的种类和含量的影响。对于系统研究大豆类黄酮的糖基化机制、大豆分子辅助育种和改良大豆类黄酮的品质提供参考依据。
1 材料与方法
1.1 试验材料
供试材料为大豆Williams 82和大豆核心种质资源,均由中国农业科学院作物所大豆育种和资源课题组提供。转基因拟南芥为哥伦比亚生态型(Columbia-0)。大肠杆菌()DH5α和Novablue、农杆菌GV3101、原核表达载体pMAL-C2X均由庞永珍实验室保存。植物表达载体pCXSN由西南大学罗克明教授提供。
1.2 UGT73C19的克隆与进化分析
从2周苗龄的大豆Williams 82叶片中提取总RNA(TRIzol A+,天根生化),使用M-MLV逆转录酶(promega corporation)进行反转录反应合成cDNA。根据前期预测的数据[9]以及phytozome公布的大豆的CDS序列设计引物,使用高保真酶Phusion(Thermo Fisher Scientific Inc.)进行PCR扩增,克隆的开放阅读框并进行测序鉴定。通过软件MEGA5 version 5.2.2(http://www.megasoftware. net/)分析UGT73C19蛋白序列和其他植物来源UGT的同源性。
1.3 类黄酮的提取和分析
收集大豆叶片、拟南芥叶片和拟南芥种子,在液氮中研磨,冷冻干燥,称取50 mg冻干样品,加入1 ml甲醇混匀。超声30 min,4℃过夜,12 000 r/min转速,4℃离心10 min,取100 μl上层溶液,置于200 μl内衬管中,使用液相色谱ACQUITY UPLC HSS C18 column(1.7 μm,100×2.1 mm i.d.,Waters)进行分离。色谱柱柱温为30℃,流速为0.4 ml·min-1,其中A流动相为水(含0.1%甲酸),B流动相为乙腈,洗脱梯度为:0—30 min,B相从95%线性梯度降至70%。上样体积为2 μl。
1.4 体外酶活试验
经HⅠ和Ⅰ双酶切的与含有MBP(麦芽糖结合蛋白)标签的pMAL-C2X载体连接,转化大肠杆菌(DH5α),鉴定序列正确后,转入表达菌株Novablue,使用0.5 mmol·L-1的IPTG诱导表达目的蛋白,参照pMAL融合蛋白和纯化系统(New England BioLab Inc.)纯化重组蛋白。
分析重组蛋白对各种类黄酮苷元的糖基转移活性,酶活反应体系为50 μl(1 mmol·L-1DTT,100 mmol·L-1Tris-HCl(pH7.5),10 μg重组蛋白,4 mmol·L-1UDP-glucose);30℃反应0.5 h后,加入等体积的甲醇终止反应。通过UPLC对酶活产物进行初步分析,再通过HPLC-MS对产物进行鉴定,确定产物糖基化的位点。通过酶动力学分析,确定重组蛋白的最适底物和催化效率。质谱分析条件为:电喷雾离子化,全离子扫描,利用Analytical software(MassLynx,version4.1)控制Xevo TQ-MS spectrometer(Waters,Milford,MA,USA)在正负离子模式(PI和NI)下进行质谱分析。脱溶剂气温度为400℃;脱溶剂气流速率(N2)为800 L·h-1;气帘气流量,50 L·h-1;PI:锥孔电压,30 V;毛细管电压,3 kV;NI则分别是-60 V和2 kV;采集质谱范围:100—1 000 m·z-1。
1.5 转基因拟南芥的获得和分析
将与由CaMV35S启动子驱动的植物过量表达载体pCXSN连接,转入农杆菌GV3101,通过花序侵染法转化拟南芥。使用qRT-PCR确认转基因植株中的表达水平,从中筛选表达量最高的3个株系进行深入分析。获得T3纯合的拟南芥,分析其21 d幼苗(地上部分)和成熟种子类黄酮的成分与含量。
1.6 实时荧光定量PCR分析
根据全长序列,利用Primer3.0在线工具(http://primer3.ut.ee/)设计1对实时荧光定量PCR引物(F:5′-CCTGTACCAGATACACACATC-3′;R:5′-CATTAGAGAGTCCTGCAGGTA-3′),以大豆为内参基因。对不同大豆组织中的表达量进行检测,反应体系和条件参照Yin等[17]方法,每个材料均具有3次技术重复,以2-ΔΔCT法进行数据分析。对转基因拟南芥材料中的表达量进行分析时,以拟南芥为内参基因,方法同上。
1.7 数据统计与分析
采用Microsoft Excel 2016进行了数据统计和分析,采用-test法进行差异性分析。
2 结果
2.1 UGT73C19的表达水平与大豆叶片黄酮醇含量的关联分析
大豆叶片中类黄酮主要以黄酮醇糖苷的形式存在[18],通过对大豆核心种质中类黄酮成分进行分析,发现不同品种中类黄酮的成分和含量存在明显差异(图1)。故根据类黄酮成分的不同,将大豆核心种质分为12种不同的类型(图1)。在这12种类型中,其中第3和12类型分别与已经报道的大豆材料Nezumisaya和Harosoy的类黄酮组成一致[20-21],第8和11类型分别与Kitakomachi和Koganejir的类黄酮组成一致[22],这4个大豆材料中的主要类黄酮成分已经被鉴定。大豆中不同类黄酮类型的差异主要是由黄酮醇糖基化的不同位置和数量造成的,而糖基转移酶UGT正是造成这种差异的主要原因。即使在相同类型的品种中,类黄酮(主要是黄酮醇)含量的差异也非常大,而的表达水平和活性很有可能是决定不同黄酮醇糖苷含量的关键因素。
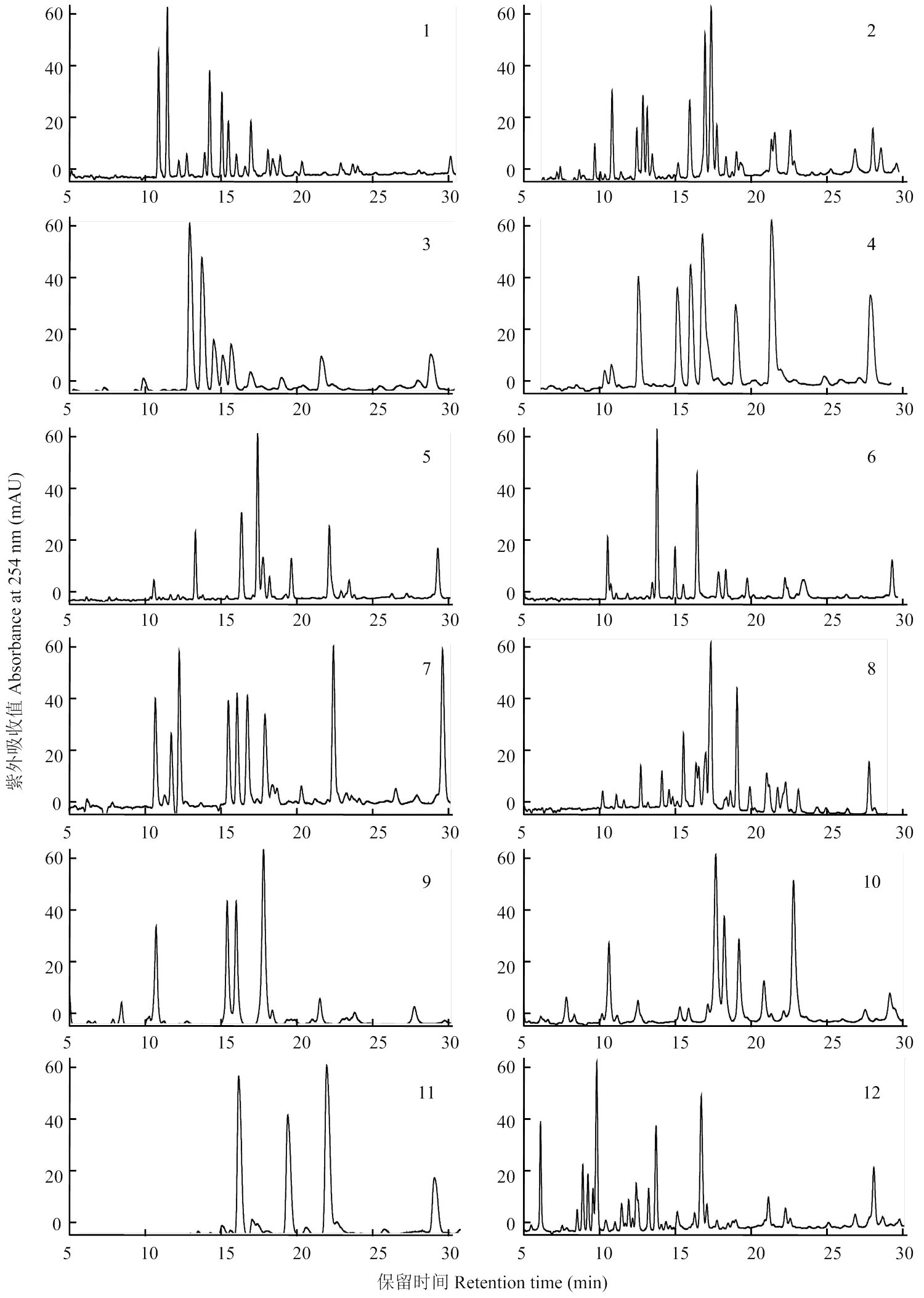
图1 大豆核心种质资源叶片中12种不同黄酮醇糖苷类型分析

图2 大豆类黄酮含量与UGT73C19表达水平的关联分析
分别选择同一类型(第5号类型含有最丰富的大豆种质资源,其中还包括基因组序列已知的大豆材料Williams 82和Clark)中类黄酮总量不同的20个大豆材料(高含量和低含量的品种各10个),检测类黄酮的含量和不同在大豆叶片中的表达水平。结果表明,在类黄酮含量高的材料中,的表达水平也相对较高;而在类黄酮含量较低的材料中,的表达水平很低或者基本不表达(图2)。表明在类黄酮类型相同的大豆材料中,类黄酮的含量与的表达水平呈正相关关系。
2.2 UGT73C19的克隆与进化分析
从Williams82叶片中克隆得到(Glyma03g187000)序列。编码区全长1 482 bp,编码493个氨基酸。与大豆中已知功能的UGT蛋白的多重序列比对表明,UGT73C19和功能已知的UGT73C20[17]的氨基酸序列相似度最高,达到82%,而与其他植物UGT蛋白,如Nt7Gt、Db7Gt、Sb7Gt和At7Rt的相似性分别为46%、44%、43%和28%[23-26]。UGT73C19保守的植物糖基转移酶结构域(PSPG,Putative secondary plant glycosyltransferase)出现在第331—351位氨基酸位点,UGT73C19与其他蛋白在PSPG结构域内的相似性较高,在其他区域的相似性较低(图3)。
通过对含有多个类黄酮UGT蛋白的进化分析,表明UGT73C19与来源的Db7Gt[23]、来源的Nt7Gt[24]、来源的Sb7Gt[25]等7位羟基糖基化的蛋白聚成一簇(图4),推测UGT73C19很可能催化类黄酮7-位的糖基化反应;同时,UGT73C19与UGT73C20的进化关系最近,推测它们可能具有相似的功能。相反,UGT73C19并不与催化大豆叶片中合成黄酮醇多糖苷的蛋白Fg1(GmF3G6Gt)或Fg3(GmF3G6Rt)[20-21]聚成一簇,说明UGT73C19可能并没有催化合成多糖苷的功能。
2.3 UGT73C19的组织表达分析
通过qRT-PCR对在不同组织中的表达水平进行分析。结果(图5)表明,在各组织均有表达,但在叶片和发育晚期的种子(开花后80 d)中的表达水平相对较高,说明可能主要在叶片和种子发育晚期中发挥作用。

黑色背景的氨基酸相似度为100%,深灰色背景的相似度大于75%,浅灰色背景的相似度在50%—75%。虚线方框位置为PSPG结构域
2.4 重组UGT73C19蛋白的体外酶活分析
将与蛋白表达载体pMAL-C2X连接,通过原核表达系统,表达并纯化重组蛋白,发现含麦芽糖结合蛋白标签的重组蛋白大小约为95.6 kD。以UDP-葡萄糖为糖基供体,以代表性的类黄酮苷元(黄酮醇、黄酮和异黄酮共16种)等为糖基供体,对重组UGT73C19蛋白进行体外酶活分析。结果表明,重组的UGT73C19蛋白对黄酮醇(山奈酚(Kaempferol)、槲皮素(Quercetin)、杨梅素(Myricetin))、黄酮(芹黄素(Apigenin))和异黄酮(大豆苷元(Daidzein)、染料木素(Genistein))具有活性(图6)。
HPLC结果显示,含有UGT73C19重组蛋白的反应产生了新的化合物峰(图6-a—图6-f,上图),而含失活蛋白的对照反应并没有产生新的化合物峰(图6-a—图6-f,下图)。通过分析对应化合物裂解的碎片的质核比并与文献中标准品进行比对[17,27],发现糖基化的位点分别在山奈酚(Kaempferol)和芹黄素(Apigenin)的5位羟基(图7-A Ka:285.1、K5G:446.8;图7-D Ap:269.1、A5G:431.0)、杨梅素(Myricetin)和大豆苷元(Daidzein)的7位羟基(图7-C My:316.7、M7G:479.1;图7-E Da:253.0、D7G:415.1)、槲皮素(Quercetin)的4/5/7位羟基(图7-B Qu:300.8、Q4G:463.4)、染料木素(Genistein)的5/7位羟基(图7-F G:269.0、G7G:431.1),说明UGT73C19的糖基化底物和位点具有多样性。
进一步以具有活性的6种类黄酮为底物、以UDP-葡萄糖为糖基供体进行酶动力学分析发现,重组UGT73C19蛋白对这6种底物的亲和程度不同,其中对杨梅素(Myricetin)的K最低(为34.69 μmol·L-1)(表1),说明杨梅素可能是UGT73C19的最适底物。重组UGT73C19蛋白对Kaempferol、Quercetin、Myricetin、Apigenin、Daidzein和Genistein的K/K值分别为297.62、302.50、291.258、101.60、70.01和97.92(s-1·M-1)(表1),结果表明,在这些底物中,UGT73C19蛋白对Quercetin的催化效率最高。综上所述,重组UGT73C19蛋白在体外对3种黄酮醇(Kaempferol、Quercetin和Myricetin)、2种异黄酮(Daidzein和Genistein)和1种黄酮(Apigenin)具有糖基化活性,其中对3种黄酮醇的催化效率相对较高,对黄酮醇中的杨梅素(Myricetin)的亲和度最高。
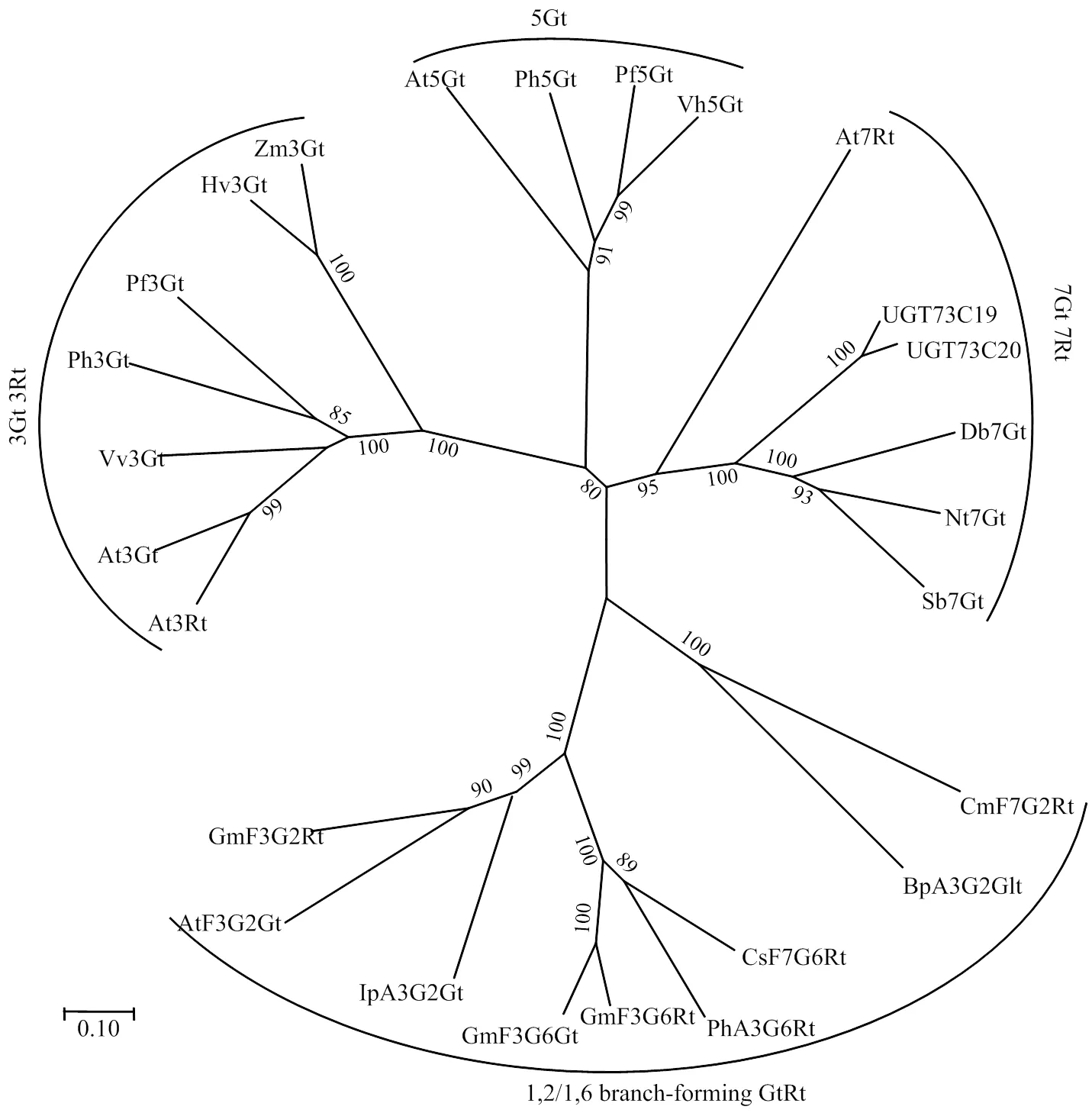
蛋白名称和Genbank登录号如下:At3Rt:NM_102790;At3Gt:NM_121711;Vv3Gt:AF000371;Ph3Gt:AB027454;Pf3Gt:AB002818;Hv3Gt:X15694;Zm3Gt:X13501;At5Gt:NM_117485;Pf5Gt:AB013596;Vh5Gt:BAA36423;Ph5Gt:AB027455;Db7G:CAB56231;Nt7Gt:AAB36653;Sb7Gt:BAA83484;At7Rt:AY093133;CmF7G2Rt:AAL06646;CsF7G6Rt:ABA18631;IpA3G2Gt:AB192315;PhA3G6Rt:X71059;BpA3G2Glt:AB190262;AtF3G2Gt:Q9FN26;GmF3G2Gt:NP_001345948;GmF3G6Gt:NP_001345940;GmF3G6Rt:I1LCI8;UGT73C19:XM003521374.1;UGT73C20:XM003554355.1。G:葡萄糖苷转移酶glucosyltransferase;Rt:鼠李糖苷转移酶rhamnosyltransferase;Glt:葡萄糖醛酸转移酶 glucuronosyltransferase
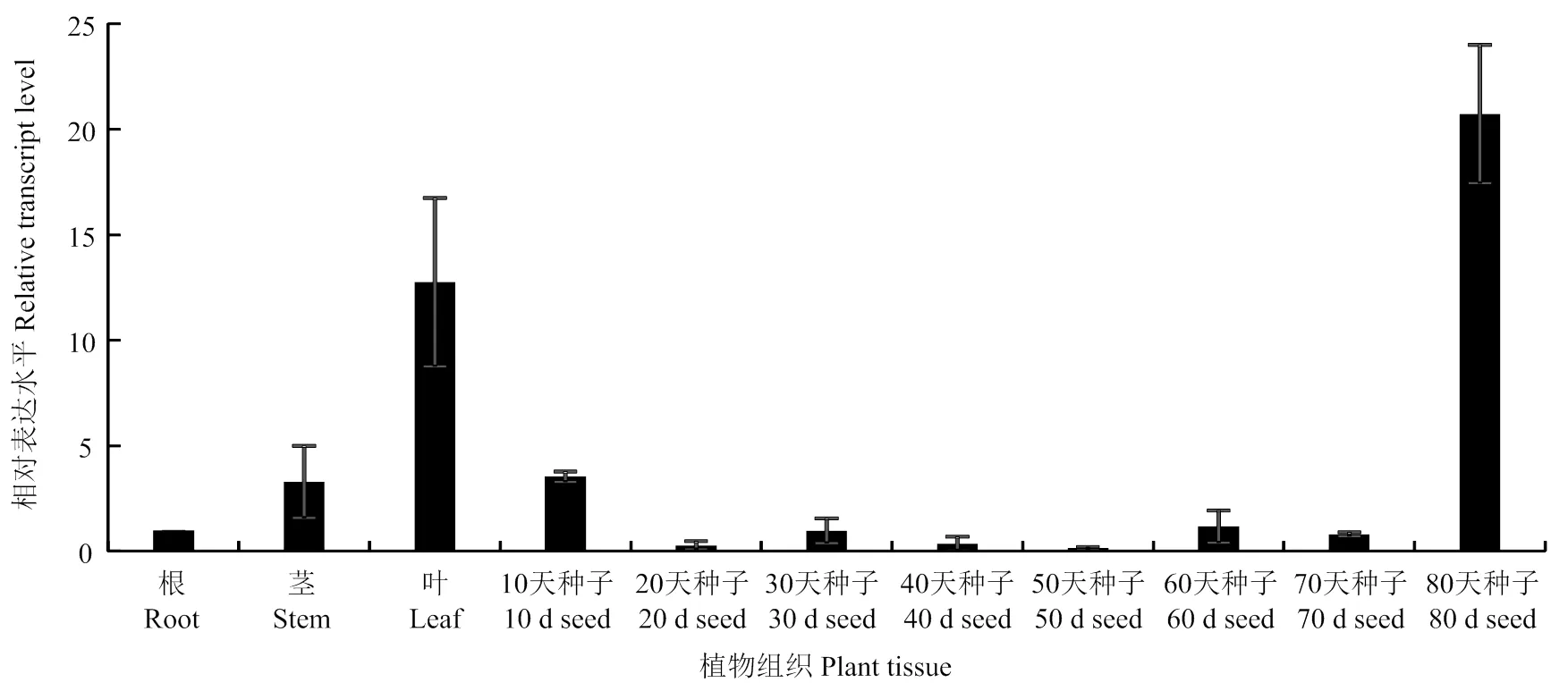
图5 大豆UGT73C19的组织表达分析
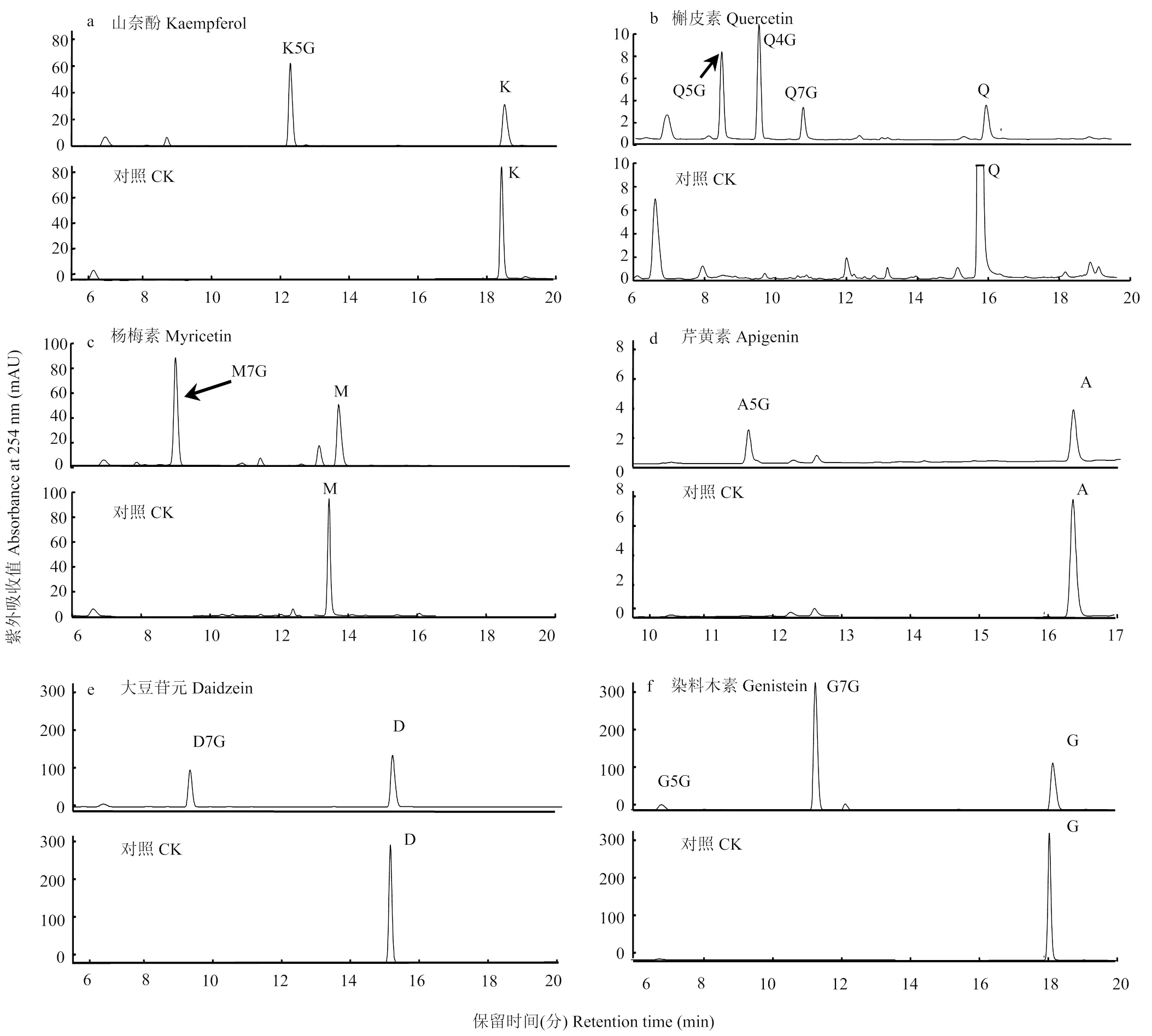
上图为UGT73C19重组蛋白的酶活产物,下图为失活蛋白的对照产物;a:山奈酚;b:槲皮素;c:杨梅素;d:芹黄素;e:大豆苷元;f:染料木素。K:山奈酚;A:芹黄素;M:杨梅素;D:大豆苷元;Q:槲皮素;G:染料木素;K5G:3-O-葡萄糖苷山奈酚;A5G:5-O-葡萄糖苷芹黄素;Q5G:5-O-葡萄糖苷槲皮素;Q4G:4-O-葡萄糖苷槲皮素;Q7G:7-O-葡萄糖苷槲皮素;D7G:7-O-葡萄糖苷大豆苷元;M7G:7-O-葡萄糖苷杨梅素;G7G:7-O-葡萄糖苷染料木素;通过保留时间、紫外吸收谱、质谱结构和文献综合比对分析
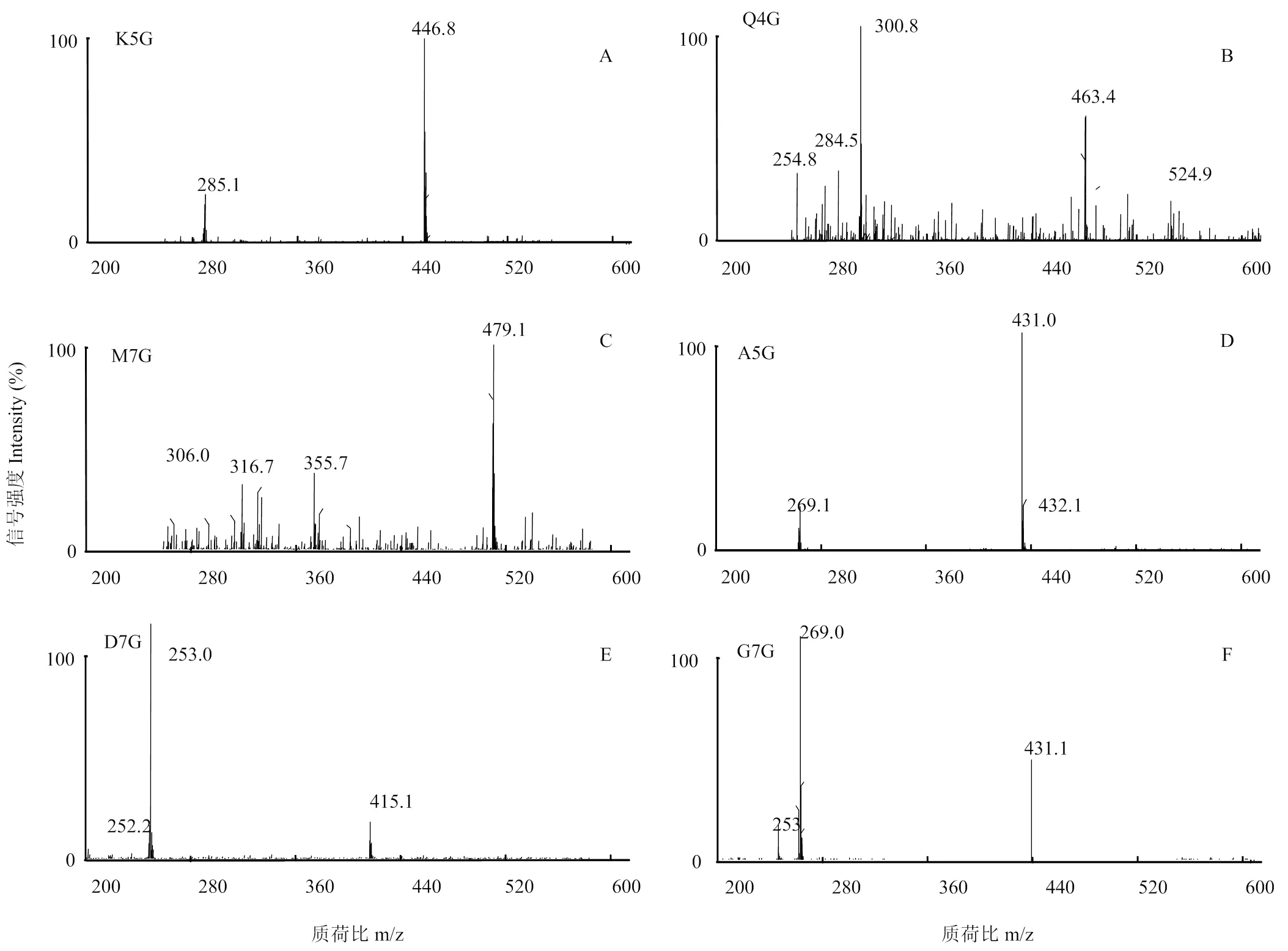
A:UGT73C19+Ka(K5G);B:UGT73C19+Qu(Q4G);C:UGT73C19+My(M7G);D:UGT73C19+Ap(A5G);E:UGT73C19+Da(D7G);F:UGT73C19+G(G7G)
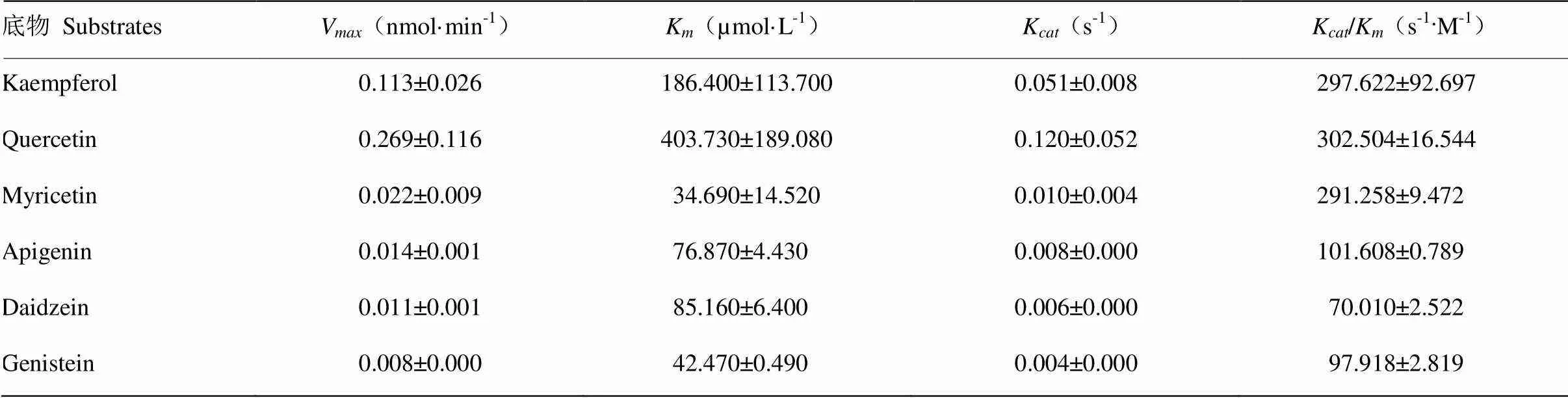
表1 重组UGT73C19蛋白的酶动力学分析
2.5 UGT73C19在拟南芥中的过量表达
为了验证的体内功能,通过花序侵染法转化拟南芥,利用qRT-PCR的方法筛选出转基因植株中表达量相对较高的3个株系OE-1、OE-2和OE-5(图8-A)。野生型拟南芥幼苗中主要存在3种黄酮醇的山萘酚糖苷,分别为kaempferol 3--rhamnoside---rhamnoside (KR3R7)、kaempferol 3--glucoside- 7--rhamnoside(KG3R7)和kaempferol-3--[rhamnosyl (1→2glucoside)]-7--rhamnoside(KGR3R7)[28](图8-B)。通过高效液相色谱(HPLC)分析发现,转基因拟南芥幼苗中这3种山萘酚糖苷都明显降低,分别降低了59%、56%和66%(图8-C)。
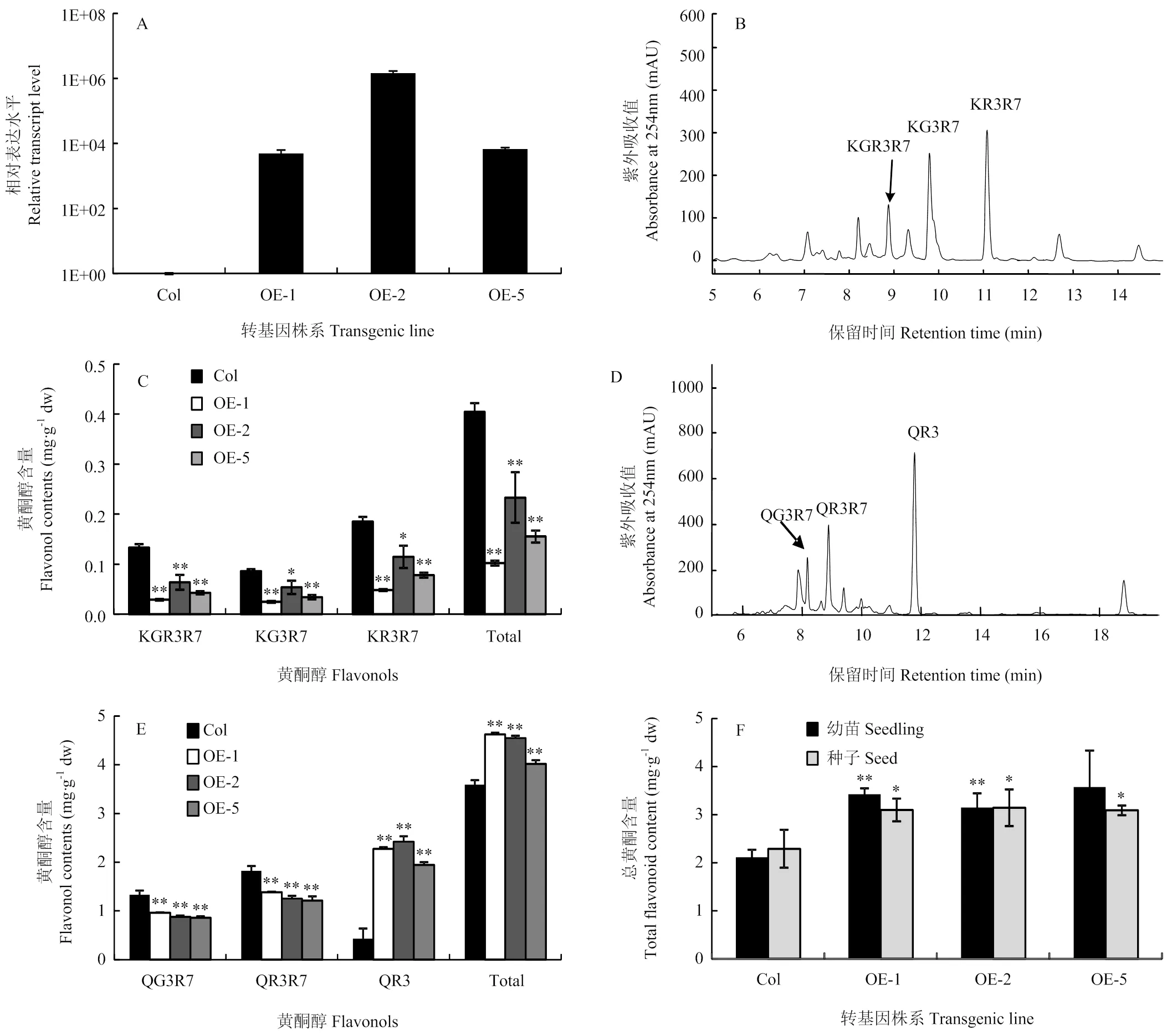
*:P<0.05;**:P<0.01
野生型拟南芥种子中含有3种主要的黄酮醇糖苷,分别为槲皮素糖苷[28](图8-D):Quercetin 3--glucoside-7--rhamnoside(QG3R7)、Quercetin 3--rhamnoside-7--rhamnoside(QR3R7)和Quercetin 3--rhamnoside(QR3)。定量分析发现,3个转基因株系相对于野生型而言,种子中槲皮素二糖苷QG3R7、QR3R7分别降低了33%和30%;而单糖苷QR3含量则极显著地增加了4.15倍,最终使槲皮素糖苷总量显著升高了22%(图8-E)。
综合检测转基因幼苗和种子中总黄酮的含量,发现3个转基因株系幼苗中总黄酮的含量分别增加为62%、49%和70%;种子中总黄酮的含量与对照相比也显著提高,分别提高了34%、37%和35%(图8-F)。
3 讨论
3.1 UGT73C19的表达特性
UDP糖基转移酶是在植物体内广泛存在的一类蛋白质,在植物次生代谢物的修饰方面具有重要的作用。对于类黄酮而言,由UGT催化的糖基化反应可以增加类黄酮的多样性,使植物更好适应复杂多变的环境[13]。本研究中基因的组织特异性表达谱分析表明,主要在叶片中表达,这与叶片中积累大量的黄酮醇是一致的[22],同时该重组蛋白具有糖基化黄酮醇苷元的作用,所以可能在叶片中负责黄酮醇糖苷的合成。另外,还在种子成熟后期表达,大豆种子中主要积累异黄酮[28],表明可能也负责大豆种子中异黄酮糖苷的积累。大豆中存在200多个不同的,存在基因冗余的现象[17],其中被验证具有糖基化类黄酮功能的编码基因仅10余个,这些分别在不同的组织部位表达,这些蛋白很有可能具有不同的分工,分别负责不同类黄酮的一次或者多次糖基化,形成多样化的类黄酮衍生物,以适应不同的环境变化。
3.2 UGT73C19蛋白的底物特异性
本研究通过体外表达重组蛋白的方法,对重组蛋白UGT73C19的酶活特性进行了分析,发现该重组蛋白可以催化黄酮醇、黄酮和异黄酮共3类6种苷元的糖基化反应,说明该重组蛋白并非对单一底物具有活性,而是具有底物多样性。底物多样性这一特点与UGT73C20、UGT88A13[17]和IF7GT[15]相似,这几个UGT都具有相似的底物谱。同时,底物多样性这一特征与大豆中鉴定的其他UGTs也具有相似性。其中大豆UGT73F2对大豆种子主要存在的3种异黄酮(daidzin、genistin和glycitin)具有糖基化活性[15];重组的UGT72X4、UGT72Z3和UGT92G4蛋白可以特异地催化黄酮醇的糖基化[17]。另外,UGT79B30[20]和UGT79A6[22]还可以催化黄酮醇糖苷的二次糖基化,产生黄酮醇二糖糖苷。相对而言,UGT73C19催化的底物种类更多,但是其并不具有UGT79B30催化类黄酮糖苷的二次糖基化的功能。
UGT73C19和UGT73C20具有较高的序列相似性,在进化树上与UGT73C20聚类于7--糖基转移酶簇,体外的酶活分析也证明,UGT73C19也具有在7位羟基产生糖苷的功能,与UGT73C20的底物谱最为接近(对黄酮和异黄酮具有活性)[17],说明可以通过序列和进化关系大致预测UGT蛋白的催化底物。
3.3 UGT73C19的体内外功能
虽然UGT73C19重组蛋白在体外对3种黄酮醇(Kaempferol、Quercetin和Myricetin)、2种异黄酮(Daidzein和Genistein)和1种黄酮(Apigenin)单体都具有糖基化活性,但是在转基因拟南芥中,由于黄酮醇积累的组织特异性,转基因幼苗中以山奈酚为主的(Kaempferol)并没有提高,反而降低,这一结果很有可能是由于内源对应的被抑制造成的。而在种子中,过表达可以明显提高黄酮醇中的槲皮素(Quercetin)单糖苷QR3的含量,这与重组蛋白对槲皮素的相对较高的催化活性是一致的,同时说明UGT73C19在体内也可以利用鼠李糖作为糖基供体。总体而言,UGT73C19蛋白的体内外功能并不完全一致,许多植物UGT都存在这一现象,造成这一现象的原因可能是植物体内最适底物浓度或者是其它底物的抑制作用造成的[10,17,27-28]。同时,过量表达时,拟南芥中总黄酮的含量明显提高,说明UGT73C19在体内的产物可能会通过反馈作用使代谢流导向类黄酮途径。
4 结论
是参与大豆叶片黄酮醇糖基化的关键基因。UGT73C19重组蛋白具有底物多样性和糖基化位点多样性的特征,是一个多功能的糖基转移酶。的异源过量表达可以提高植物类黄酮的含量,是进行植物类黄酮代谢工程的候选基因。
[1] HO H M, CHEN R Y, LEUNG L K, CHAN F L, HUANG Y, CHEN Z Y. Difference in flavonoid and isoflavone profile between soybean and soy leaf., 2002, 56: 289-295.
[2] DUEÑAS M, HERNÁNDEZ T, ROBREDO S, LAMPARSKI G, ESTRELLA I, MUÑOZ R. Bioactive phenolic compounds of soybean (cv. Merit): modifications by different microbiological fermentations., 2012, 62(4): 241-250.
[3] SUBRAMANIAN S, STACEY G, YU O. Distinct, crucial roles of flavonoids during legume nodulation., 2007, 12(7): 282-285.
[4] 庄炳昌, 岳德荣, 王玉民, 李建平, 谢雪菊, 徐豹. 大豆不同品种次生代谢产物及相关酶类含量与抗食心虫的关系. 中国油料, 1992(3): 20-22.
ZHUANG B C, YUE D R, WANG Y M, LI J P, XIE X J, XU B. The correlation of content of flavonoid, total phenolate and enzyme in soybean with resistance level to, 1992(3): 20-22. (in Chinese)
[5] Liu C W, Murray J D. The role of flavonoids in nodulation host-range specificity: an update.,2016, 5(3): 33.
[6] Dixon R A, Steele C L. Flavonoids and isoflavonoids - a gold mine for metabolic engineering., 1999, 4(10): 394-400.
[7] Maggioni D, Biffi L, Nicolini G, Garavello W. Flavonoids in oral cancer prevention and therapy., 2015, 24(6): 517-528.
[8] HOSTETLER G L, RALSTON R A, SCHWARTZ S J. Flavones: food sources, bioavailability, metabolism, and bioactivity., 2017, 15; 8(3): 423-435.
[9] 杨硕, 徐元庆, 邢媛媛, 史彬林. 植物源黄酮类化合物对动物免疫和抗氧化功能影响的研究进展. 动物营养学报, 2019, 31(7): 2958-2964.
YANG S, XU Y Q, XING Y Y, SHI B L. Research advances on effects of plant-based flavonoids on immune and antioxidative functions in animals.,2019, , 31(7): 2958-2964. (in Chinese)
[10] JONES P, VOGT T. Glycosyltransferases in secondary plant metabolism: tranquilizers and stimulant controllers., 2001, 213: 164-174.
[11] GACHON C M, LANGLOIS-MEURINNE M, SAINDRENAN P. Plant secondary metabolism glycosyltransferases: the emerging functional analysis., 2005, 10: 542-549.
[12] CAPUTI L, MALNOY M, GOREMYKIN V, NIKIFOROVA S, MARTENS S. A genome-wide phylogenetic reconstruction of family 1 UDP-glycosyltransferases revealed the expansion of the family during the adaptation of plants to life on land., 2012, 69: 1030-1042
[13] Yonekura-Sakakibara K, Hanada K. An evolutionary view of functional diversity in family 1 glycosyltransferases., 2011, 66(1): 182-193.
[14] Kovinich N, Saleem A, Arnason J T, Miki B. Functional characterization of a UDP-glucose: flavonoid 3--glucosyltransferase from the seed coat of black soybean ((L.) Merr.).,2010, 71(11/12): 1253-1263.
[15] NOGUCHI A, SAITO A, HOMMA Y, NAKAO M, SASAKI N, NISHINO T, TAKAHASHI S, NAKAYAMA T. A UDP-glucose: isoflavone 7--glucosyltransferase from the roots of soybean () seedlings. Purification, gene cloning, phylogenetics, and an implication for an alternative strategy of enzyme catalysis., 2007, 282(32): 23581-23590.
[16] FUNAKI A, WAKI T, NOGUCHI A, KAWAI Y, YAMASHITA S, TAKAHASHI1 S AND NAKAYAMA T. Identification of a highly specific isoflavone 7--glucosyltransferase in the soybean ((L.) Merr.)., 2015, 56(8): 1512-1520.
[17] Yin Q, Shen G, Di S, Fan C, Chang Z, Pang Y. Genome-wide identification and functional characterization of UDP-glucosyltransferase genes involved in flavonoid biosynthesis in.,2017, 58(9): 1558-1572.
[18] Kim E H, Kim S L, Kim S H, Chung I M. Comparison of isoflavones and anthocyanins in soybean [(L.) Merrill] seeds of different planting dates., 2012, 60(41): 10196-10202.
[19] MAMOON REHMANA H, AMJAD NAWAZA M, BAO L, HUSSAIN SHAHB Z, LEE J M, AHMAD M Q, CHUNG G, YANG S H. Genome-wide analysis of Family-1 UDP-glycosyltransferases in soybean confirms their abundance and varied expression during seed development., 2016, 206: 87-97.
[20] Rodas F R, Di S, Murai Y, Iwashina T, Sugawara S, Mori T, Nakabayashi R, Yonekura-Sakakibara K, Saito K, Takahashi R. Cloning and characterization of soybean geneencoding flavonol 3--glucoside/galactoside (1→6) glucosyltransferase., 2016, 92(4/5): 445-456.
[21] Di S, Yan F, Rodas F R, Rodriguez T O, Murai Y, Iwashina T, Sugawara S, Mori T, Nakabayashi R, Yonekura-Sakakibara K, Saito K, Takahashi R. Linkage mapping, molecular cloning and functional analysis of soybean geneencoding flavonol 3--glucoside/galactoside (1→2) glucosyltransferase., 2015, 15: 126.
[22] Rodas F R, Rodriguez T O, Murai Y, Iwashina T, Sugawara S, Suzuki M, Nakabayashi R, Yonekura- Sakakibara K, Saito K, Kitajima J, Toda K, Takahashi R. Linkage mapping, molecular cloning and functional analysis of soybean geneencoding flavonol 3--glucoside (1→6) rhamnosyl transferase., 2014, 84(3): 287-00.
[23] VOGT T, GRIMM R, STRACK D. Cloning and expression of a cDNA encoding betanidin 5--glucosyltransferase, a betanidin- and flavonoid-specific enzyme with high homology to inducible glucosyltransferases from the Solanaceae., 1999, 19(5): 509-519.
[24] Horvath D M, Chua N H. Identification of an immediate- early salicylic acid-inducible tobacco gene and characterization of induction by other compounds., 1996, 31(5): 1061-1072.
[25] HIROTANI M, KURODA R, SUZUKI H, YOSHIKAWA T. Cloning and expression of UDP-glucose: flavonoid 7--glucosyltransferase from hairy root cultures of., 2000, 210(6): 1006-1013.
[26] YONEKURA-SAKAKIBARA K, TOHGE T, NIIDA R, SAITO K. Identification of a flavonol 7--rhamnosyltransferase gene determining flavonoid pattern inby transcriptome co-expression analysis and reverse genetics., 2007, 282(20): 14932-14941.
[27] Su X, Shen G, Di S, Dixon R A, Pang Y. Characterization of UGT716A1 as a multi-substrate UDP: flavonoid glucosyltransferase gene in., 2018, 8: e2085.
[28] YIN Q, SHEN G, CHANG Z, TANG Y, GAO H, PANG Y. Involvement of three putative glucosyltransferases from the UGT72 family in flavonol glucoside/rhamnoside biosynthesis inseeds., 2017, 68(3): 597-612.
Functional Characterization of a UDP: Flavonoid Glycosyltransferase Gene
DI Shaokang1, YIN Qinggang2, XIA Yaying1,2, PANG Yongzhen1
(1Institute of Animal Sciences, Chinese Academy of Agricultural Sciences, Beijing 100193;2Institute of Botany, Chinese Academy of Sciences, Beijing 100093)
【】 Flavonoids are a group of important plant secondary metabolites accumulate in soybean, which are involved in many physiological activities, including soybean growth, development and stress resistance. Glycosylation catalyzed by UDP-glycosyltransferase is a key step in flavonoid biosynthesis. 【】The objective of the present study is to investigate theenzymatic activity andfunction of a soybean glycosyltransferase protein encoded by thegene, the achievement of which will deep our understanding on the mechanism of the flavonoid biosynthesis in soybean. This study will provide gene resource and theoretical basis for the genetic modification in soybean. 【】 Flavonoids in the leaves of soybean core germplasm resources were detected by HPLC, and the expression level ofgenes were detected by qRT-PCR. The coding region of thegene was cloned from cDNA of soybean leaf (Williams 82). The amino acid sequences of UGT73C19 were searched in the NCBI database, and the software MEGA5 and DNAMAN were used for multiple sequence alignment and the construction of a phylogenetic tree. The recombinant UGT73C19 protein was expressed inand its enzymatic activity was determined towards various flavonoid aglycones. All the enzymatic products were identified by HPLC-MS. The expression profile of thegene in soybean was analyzed by qRT-PCR.was over-expressed inby floral dipping method. Flavonoid content and composition were determined in seedlings and seeds in homozygous lines that showed the relatively highexpression level. 【】 Flavonoids in the leaves of soybean core germplasm showed significant differences in flavonoid composition and content in different varieties. Soybean core germplasm can be divided into 12 different types according to flavonoid composition. There was a positive correlation between the content of flavonoids and the expression level ofgene in the leaves of soybean core germplasm resources. The coding sequence ofgene was cloned,and the coding region was found to be 1482 bp, encoding a protein of 493 amino acids. The deduced UGT73C19 protein was found to have a conserved PSPG domain at the C-terminal.enzymatic activity analysis revealed that the recombinant UGT73C19 protein exhibited glycosyltransferase activity toward six flavonoid aglycones (kaempferol, quercetin, myricetin, apigenin, daidzein and genistein), and it showed the highest catalytic efficiency toward quercetin. The glycosylation sites were at the 5 and 7 hydroxy groups of flavonoid substrates, and the glycosylation substrates and sites of the recombinant UGT73C19 protein showed high diversity. It was found that the total flavonoid contents in the seedlings and seeds of the transgenicincreased significantly, by 49% to 70% in leaves and 34% to 37% in seeds, in particular quercetin 3-rhamnose in the seeds. 【】 The recombinant UGT73C19 protein can catalyze the glycosylation of a group of flavonoid compounds and over-expression ofgene can increase the content of flavonols in plants like.
soybean; UDP-glucosyltransferase; flavonoids; flavonols
10.3864/j.issn.0578-1752.2019.20.002

2019-04-25;
2019-06-12
国家重点研发项目(2016YFD0101005)
狄少康,E-mail:dishaokang@caas.cn。
庞永珍,E-mail:pangyongzhen@caas.cn
(责任编辑 李莉)

