基于复杂网络的胃癌关键基因筛选与分析
王晓曼 刘文奇
(昆明理工大学理学院,昆明,650093)
引 言
近些年来,复杂性科学的实践研究迅速发展,分析方法不断更新,技术应用范围日益扩展。对于一个复杂系统,单独研究系统中的个体已经不能反映系统的性质。往往把系统中的个体视为节点,个体之间的关系抽象成边,这样就可以用复杂网络来研究系统的整体性质。目前,复杂网络理论应用的范围越来越广泛,不仅应用到军事[1]、政治[2]和交通[3],且已被用来理解生物系统中的基因功能[4]。
近些年来,癌症患者的数量逐步上升,中国每年就新增400多万癌症患者。胃癌作为如今最常见的恶性肿瘤之一,发病率仅次于肺癌、乳腺癌、结/直肠癌和前列腺癌。早期胃癌病人的生存率能达到90%,但是大部分胃癌患者确诊时就已经丧失治疗机会。杨维良等[5]的研究表明胃癌为一种高发、高恶性度、预后不良的恶性肿瘤,长期以来包括手术切除及其他综合性治疗的应用,并没有带来满意的生存率。胃癌本身的生物学过程归根结底也是基因突变产生的,因此基因水平的治疗成为胃癌治疗学取得新突破的希望。如果能应用科学的数据分析方法,找到胃癌恶化过程中的关键基因,就能为癌症患者带来意想不到的收益。
王频等[6]的研究已经通过RNA测序建立的癌症数据(The cancer genome atlas,TCGA)和Affymetrix芯片(Affymetrix genechip arrays,HG-U133 Plus 2.0)产生的GSE307207这两个数据库中正常胃组织和癌症组织之间的基因表达,筛选出存在显著差异的胃癌相关基因。这样就大量剔除了与胃癌无关的基因,为分析关键基因缩小了范围。
周晖杰[7]与李星[8]研究表明,基因与蛋白质很少单独起作用,而是通过网状的相互作用而影响生物表达,因此基因组的研究也从结构基因组转向了功能基因组的研究,通过对基因网络的分析来了解生物系统的功能。Laura等[9]的研究表明,网络生物学是解释健康与疾病背景下基因组数据的一种系统性方法。其次王华等[10]已经将复杂网络的分析方法应用到高血压特征基因的筛选中。由于高血压疾病在细胞、组织与生理水平上都表现出复杂性,因此应用网络分析方法来研究高血压疾病。根据基因表达数据的相关性构建网络模型,网络中的枢纽基因显示出与具有不同表达水平的大多数基因相同的变化趋势,并且具有相近的表达模式。所以枢纽基因比其他基因更具代表性,这些枢纽基因可成为病情分析的关键依据。
本文将复杂网络与胃癌测序数据相联系,将胃癌相关基因抽象为节点,依据胃癌ⅡB期样本与胃癌ⅢA期样本间的基因变化率构建胃癌基因表达复杂网络。通过分析该胃癌基因表达网络的相关拓扑性质,发现网络是稀疏的且具有小世界性。通过计算节点的度中心性、介数中心性和紧密度中心性,并引入综合中心性指标,筛选出中心性较高的基因并做出了验证,因此建议了胃癌恶化过程中的枢纽基因,为胃癌恶化提供了良好的预警信号。
1 数据的预处理
本文使用的原始数据来自TCGA数据库,包括7种胃癌分期共408个样本,每个样本包含60484个基因测序。由于原始数据量非常庞大,应对数据进行初步筛选和降维。
首先使用两个可公开的国际肿瘤数据库:由RNA测序建立的癌症数据TCGA和Affymetrix芯片产生的GSE307207。比较这两个数据库中正常胃组织和癌症组织之间的基因表达,确定了存在显著差异的基因。TCGA和GSE30727分别有688个和3239个基因达到了标准(2倍变化和校正后p值<0.05)。TCGA和GSE30727数据集之间存在275个重叠基因,将这275个基因保留,用于后续的分析[6]。这样就筛选出与胃癌相关的基因,剔除了大量无关基因,为分析关键基因缩小了范围。
其次,原始数据包含胃癌不同分期不同分形共408个样本,样本数量较大。根据癌症TNM分期标准(依据肿瘤浸润的深度、是否存在淋巴结以及是否远处转移。T代表肿瘤浸润胃壁的深度,N表示局部淋巴结的转移情况,M则代表有没有远处转移的情况,可将胃癌分为Ⅰ,Ⅱ,Ⅲ,Ⅳ,4个临床病理分期,每期又具有不同的分形,本文筛选的样本应用第七版胃癌TNM分期标准如表1所示。根据大宗临床报道,实行规范治疗的1期胃癌患者5年生存率为82%~95%,2期为55%,3期为15%~30%,4期为2%[11]。在临床意义上讲,当胃癌处于ⅡB期时,肿瘤未侵及胃的外部或者侵浸胃的外部但是肿瘤未转移。处于此时的胃癌患者仍属于传统意义上的胃癌前期,有很大的治愈希望。当胃癌处于ⅢA期时,肿瘤侵及腹腔脏层腹膜或侵浸到胃的外部[12]。处于这阶段的胃癌患者,肿瘤已经侵浸胃的外部并发生淋巴结转移,在临床上已经发展为胃癌的中期,治愈希望较低(表2)。因此筛选处于胃癌ⅡB期病人的基因序列(40个样本)与处于胃癌ⅢA期病人的基因序列(36个样本)做为对照,分析胃癌从ⅡB期到ⅢA期转变过程中的关键基因,致力于为胃癌恶化提供良好的预警信号。

表1 第7版胃癌TNM分期分型Tab.1 Seventh edition gastric cancer TNM staging classification
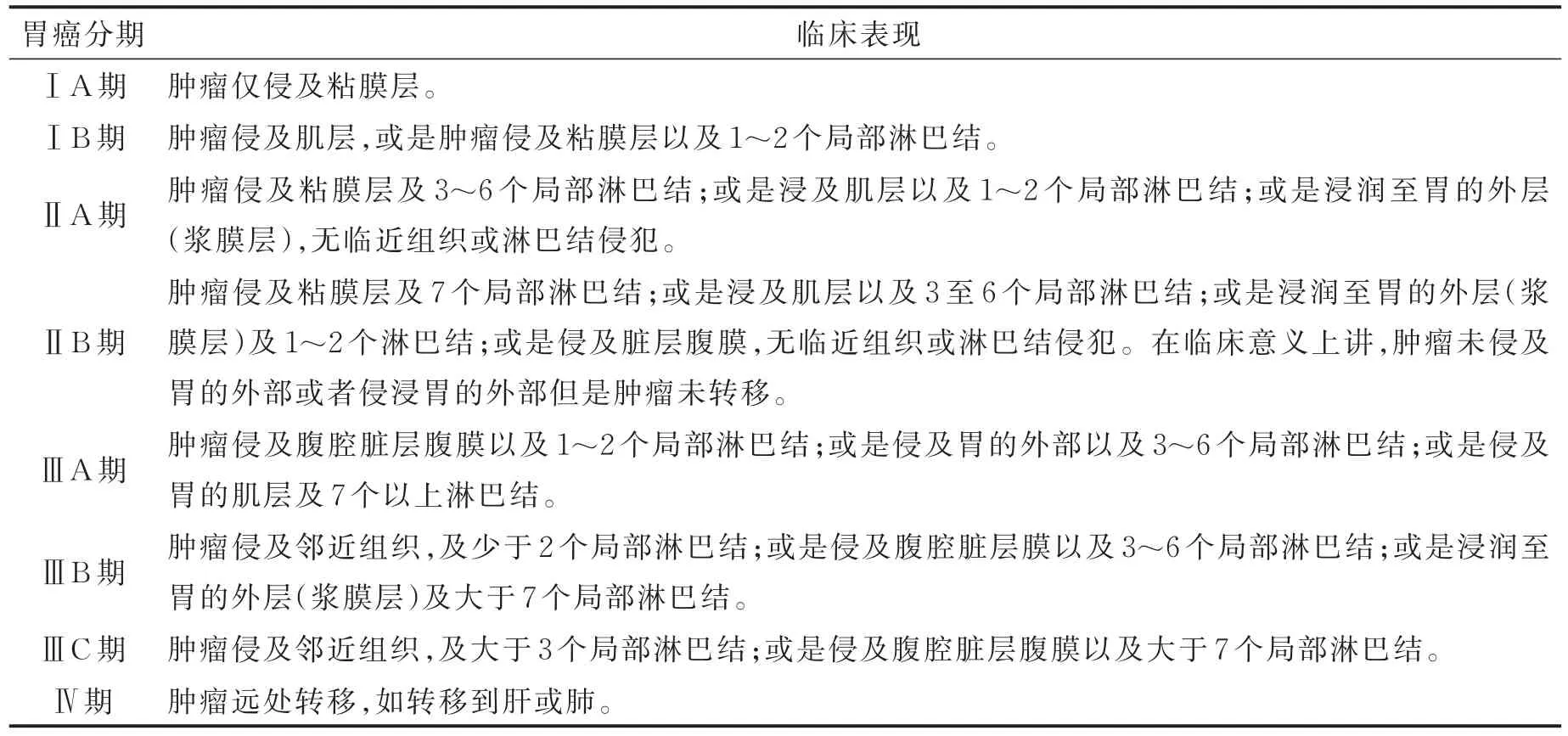
表2 胃癌不同分期的临床表现Tab.2 Clinical manifestations of different stages of gastric cancer
2 胃癌基因表达网络模型
本文选取了两个样本组的基因表达数据,分别是处于ⅡB期的胃癌患者,共40个样本,记为H;以及处于ⅢA期的胃癌患者,共36个样本,记为S。通过构建胃癌基因复杂网络模型,来研究胃癌基因表达数据的变换趋势与胃癌表型之间的关系。
2.1 基胃癌计算胃癌基因表达数据变化率
选取第2部分中初步筛选出的275个胃癌相关基因,将它们定义为网络节点,网络规模N=275。其中每1个节点都代表1个基因,用基因名称进行标注。首先,对于每个节点i,定义胃癌ⅡB期样本到胃癌ⅢA期样本的基因变化率为

式中:i=1,2,…,275,S=36,h=40。接下来计算出每个节点对应的基因变化率Ri,通过式(2)很容易得到基因变化率的平均值T=0.3617。这个平均值可以为下一步的阈值选取作为参考。
2.2 基因复杂网络的连边规则
由于胃癌基因变化率的平均值较大,应选择一个较小的阈值,增大基因间的关联性,降低节点间的扰动对网络的影响。通过多次实验,选定0.008T作为阈值,此时网络结构较为稳定,基因间的关联性较强,节点间的扰动较小[13]。建立连边的规则如下:比较节点i与节点j的基因变化率差值,当差值小于0.008T时,两个节点间的变化趋势相近,则在这两个节点间建立一条边。式(3)为胃癌基因复杂网络连边规则的数学表达

应用上述思路,构建了1个包含275个节点(基因)和828条边的胃癌基因表达网络。通过Pajek软件将该网络可视化。图1为胃癌基因表达网络示意图,图中间是网络的密集部分,有较高的连接数,图2是网络密集部分的放大图。
下面对该胃癌基因复杂网络的相关拓扑性质进行简单分析。
该胃癌基因表达网络包含275个节点,假设该网络是一个完全连通网络,则网络的最大连通度为

图1 胃癌基因表达网络示意图Fig.1 Schematic diagram of gastric cancer gene network

图2 胃癌基因网络示意图密集部分放大图Fig.2 Enlarged view of the gastric cancer gene network
然而,该网络共有828条边,平均度为6.02,其平均度远远小于该网络的最大连通度,故可以认为该胃癌基因表达网络是稀疏的。这表明,每个基因的表达趋势可能只与少量的基因表达数据变化趋势一致,也就是说,每个基因受其他基因调控的平均数量较小。
平均路径长度是指网络中所有顶点对之间最短路径的平均值,可通过平均路径衡量网络中不同基因数据变化的差异[14]。通过计算,该胃癌基因表达网络的平均路径长度约为9.48,平均路径较小,有

通过式(5),可计算得到该胃癌基因表达网络的聚类系数C=0.641。该网络的聚类系数约是同规模随机网络的28倍。综上所述,胃癌基因表达网络具有较大的聚类系数与较小的平均路径长度,满足小世界特性。
3 胃癌关键基因
构建出胃癌基因表达网络后,发现该网络具有稀疏性和小世界特性,这意味着平均每个基因可能受较少的基因调控,这些起到关键作用的基因可能具有较强的中心性。那如果能找到基因表达网络中的关键基因,就能为胃癌恶化提供良好的预警信号。下面将分析胃癌基因复杂网络中节点的3种中心性,包括介数中心性、紧密度中心性和度中心性,应用定量的方法对每个节点的中心度进行描述,进而确定胃癌基因表达网络中的关键基因。
度中心性[15]

式中:ki为节点i的度,N为网络节点总数。度中心性定义为节点的度与网络中其他节点总数的比值。通过度中心性可以迅速找到该网络中的关键节点。
介数中心性[15]

式中:gjk(i)为节点j与节点k的最短路径经过节点i的数量,gjk为节点j到节点i的最短路径数。介数中心性定义为节点i与节点k之间经过节点i的最短路径数与节点i与节点k之间的最短路径总数之比。介数中心性衡量的是经过该节点最短路径数的度量。通过介数中心性可以迅速找到该网络中的重要节点。
紧密度中心性[15]

式中:Li节点i到网络中每个节点距离的平均值。紧密度中心性定义为节点i到网络中每个节点距离的平均值的倒数。一般观点认为,网络的拓扑中心是网络中其他节点到该节点距离最小的节点,因此,紧密度中心性可以作为复杂网络中的中心性度量。
分别应用式(1-3)对度中心指标、介数中心性指标和紧密度中心性指标进行计算,各中心指标结果靠前的基因分别列在了表3中。由于有多个基因的某个中心性指标相同,所以选定度中心性排名前32的基因、介数中心性排名前20的基因以及紧密度中心性排名前22的基因列在表3中。
从表3中可以得出,度中心性大的节点往往它们的紧密度中心性也较大,介数中心性大的节点其紧密度中心性也是相对较大的。但是介数中心性的值普遍较小,如果直接分析不能发挥介数中心性的重要性。
图3给出了基于度中心性、介数中心性、紧密度中心性的重要胃癌基因的分布示意图,可以发现,3种中心性指标均靠前的是MMP11基因。但是由于中心性指标间的差异性,并没有太多的重叠基因。

表3 胃癌度中心性、介数中心性、紧密度中心和综合中心性指标靠前的基因及数值Tab.3 Top genes and values for three central and comprehensive central indicators of gastric cancer
图4绘出了胃癌基因度中心性、介数中心性和紧密度中心性3种指标分布的三维图,从图4中可以看出同时具有较高的3种中心性指标的节点应分布于图的最上部,这些节点就有可能是网络的枢纽节点。
引用加权分析的方法将这3种指标定量化,来计算每个节点的综合中心性指标。根据式(9),将度中心性、介数中心性、紧密度中心性分别归一化为一个相对性中心性指标:Ca(i)/Ca.max,Cb(i)/Cb.max和Cc(i)/Cc.max,将综 合中心性指标定义为3个中心性指标的平均值[10]。筛选出综合中心指标前20的基因,将其列在表3当中。
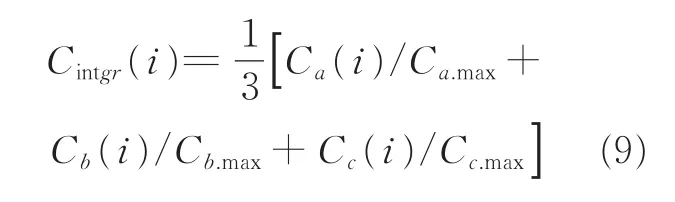
通过对表3中的4种中心性指标进行分析,发现,综合性中心指标排名第1的基因MMP11,其3种中心性指标均是较高的。由于度中心性差值较小,其他综合性中心指数较高的基因其度中心性不一定排名靠前,但是相对拥有较高的介数中心性和紧密度中心性,这并不意味着其度中心性是较低的。通过对表3进行分析发现基因INTS8到基因SGSM3的综合中心性指标下降过快,选定综合性指标Cintgr>0.72的16个基因作为关键基因,此外,由于ALDH6A1基因的度中心较高(排名第3位),在基因表达网络中拥有较高的度,连接着较多的节点,因此也应考虑其为关键基因。应说明的是,选择基因是否为关键基因的阈值可大可小,若想增大或减小关键基因的数量,应将阈值调小或调大。
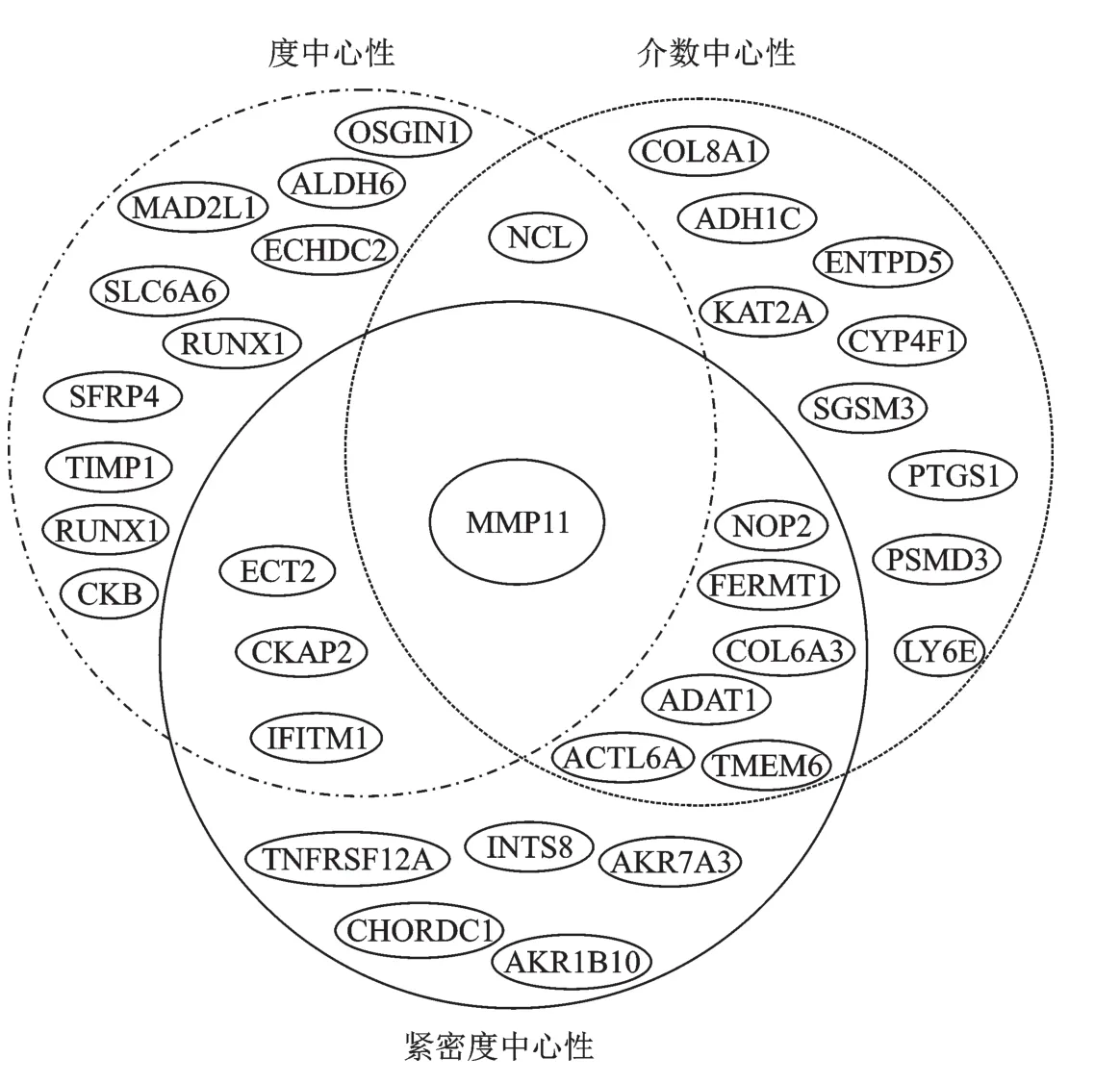
图3 基于3种中心性指标的重要胃癌基因分布图Fig.3 Gene map of important gastric cancer based on three central indicators

图4 度中心性、介数中心性和紧密度中心性分布的三维图Fig.4 Three-dimensional map of three central distributions
将该胃癌基因复杂网络进行分区视图,如图5所示,文献[16]指出发现度中心性较小的节点往往分布更加广泛。而大多数基因都集合在一个完全连通的子网络中,在此子网络中,中心性较高的基因链接着更多的基因,在基因调控网络中处于关键地位。通过式(9)选取的中心度较高的17个基因全部落在该子网络中,这与网络中关键基因定量考虑的结果一致。
因此可以确定出胃癌基因表达网络中的17个重要的枢纽基因,分别是:MMP11,TMEM63A,CKAP2,IFITM1,NOP2,ADAT1,ACTL6A,FERMT1,ECT2,SLC6A6,TNFRSF12A,CHORDC1,COL6A3,YEATS2,NCL,INTS8和ALDH6A1。
依据胃癌基因在ⅡB与ⅢA样本间的表达变化率构建胃癌基因复杂网络,将每个基因抽象为节点,分析了该网络的拓扑特征。通过Pajek软件对基因的3种中心性指标进行计算,进一步引入综合中心性指标,进而筛选出上述17个关键的胃癌基因。

图5 胃癌基因复杂网络社区结构图Fig.5 Gastric cancer gene complex network community structure
4 结束语
本文对TCGA胃癌数据进行初步的筛选和降维,筛选出与胃癌相关的275个胃癌基因,为分析关键基因缩小了范围。原始数据包含胃癌不同分期不同分形共408个样本,样本数量较大。根据胃癌TNM分期,筛选胃癌ⅡB期(40个样本)与ⅢA期(36个样本)做为对照,分析胃癌从ⅡB期到ⅢA期转变的关键基因,致力于发现胃癌恶化过程中的特征基因。其次应用复杂网络的方法,将网络分析的方法与胃癌基因表达数据结合起来。将胃癌基因抽象为节点,依据两组样本组间基因的变化率,选择合理的阈值来建立连边关系。然后分析了该网络的拓扑特征,发现胃癌基因表达网络是稀疏的,具有小世界特性。计算了该复杂网络节点的3种中心度指标,分别是度中心性、介数中心性和紧密度中心性,然后引入综合中心性指标,筛选出17个中心指标较高的基因。最后对该胃癌基因复杂网络进行社区划分,发现这17个中心度较高的基因都在一个规模较大的连通子网络中,进而验证了结论。因此建议了胃癌恶化过程中的关键基因,为胃癌恶化提供了良好的预警信号。
本文研究的问题是基于复杂网络理论和数据分析方法,筛选出胃癌恶化过程中17个的关键基因,但基因间的链路关系还需进一步的研究。另外,本文是基于病例数据做出的筛选与分析,缺乏动物实验模型验证,这需要继续研究学习。

