蓝玉簪龙胆ITS序列克隆与遗传分化分析
孙姗姗 王廷峰

摘 要:蓝玉簪龙胆为龙胆属的常见种之一,青藏高原特有物种。本研究通过对蓝玉簪龙胆6个个体进行ITS序列的克隆与测序,得到8条序列,长度为644~651bp,包含48个碱基突变和11个缺失/插入,共鉴定出7个单倍型。通过克隆序列构建蓝玉簪龙胆及其近缘物种线叶龙胆的系统发育树,2个物种可形成2个支系,但蓝玉簪龙胆大部分单倍型仍镶嵌在线叶龙胆支系内。本研究获得的蓝玉簪龙胆ITS序列及其分化分析为龙胆属进化研究提供了参考。
关键词:蓝玉簪龙胆;ITS;克隆;遗传分化
中图分类号:S-3 文献标识码:A DOI:10.19754/j.nyyjs.20191030005
引言
龙胆属植物是典型的高山植物,以青藏高原及其周边地区为分布中心和分化中心[1,2]。蓝玉簪龙胆(Gentiana veitchiorumHemsl.)多年生草本,生长于海拔4000~5000m的草甸或灌丛中,隶属于龙胆属多枝组,是青藏高原特有植物,也是多枝组中的常见物种,是高山草甸的重要组成部分。蓝玉簪龙胆花色素雅,形态优美,具有较高的园艺观赏价值,已被用于园艺栽培。进化研究表明,蓝玉簪龙胆的叶绿体基因组存在基因缺失[3],目前缺乏其群体遗传学研究[4]。
青藏高原是全球生物多样性热点地区之一,是维管束植物的多样性中心之一,是我国植物资源重要的“基因库”,有很高的生物多样性。同时,青藏高原具有数量众多的特有种,比如龙胆属、虎耳草属、杜鹃花属和马先蒿属等类群。此外,青藏高原的植物在研究植物区系起源和演化具有重要价值,整个东亚植物区系至少在其西部是以横断山至喜马拉雅为中心而发展起来的。
随着分子生态学的发展,结合遗传结构与历史地质事件,解决物种的种群历史演化过程及成因。在进化生物学中,ITS序列,即18S-5.8S-26S 核糖体DNA的基因内转录间隔区(nuclear ribosomalInternal Transcribed Spacer),是十分常用的分子标记。高等植物的核糖体DNA由18S rDNA、ITS1、5.8S rDNA、ITS2和26S rDNA组成,再由成百上万个基本单元串联成重复序列。ITS序列作为核基因分子标记有如下优点:被子植物的ITS长度稳定,一般在600~700bp间;基因序列比较保守,设计的通用引物在不同物种中均可扩增;进化速率较大,可提供丰富的系统发育信息,是重要的Barcoding标记,能较好的反应种间关系。其在种内居群也往往存在丰富的变异,可用于群体遗传学研究。因此,ITS已經成为被子植物分子系统学和群体遗传研究中应用最广的核基因分子标记。此外,ITS还可以用于检测杂交事件,推测亲本序列,在揭示杂交等复杂进化过程中具有重要作用。
本研究以蓝玉簪龙胆的3个居群为研究对象,通过对部分个体进行单克隆测序,探讨蓝玉簪龙胆ITS序列的基本特征及其与近缘种的遗传分化,研究结果将为龙胆属植物的进化研究提供参考。
1 材料与方法
1.1 样品采集
在四川和西藏的高山草甸上采集蓝玉簪龙胆3个居群(Fu2016036,四川色达;Fu2016175,西藏芒康;Fu2017207,西藏林周)6个个体,凭证标本保存在洛阳师范学院生命科学学院标本馆。样品采集,保证居群内每个个体间距至少10m,以防止克隆体;选择开花的成熟植株,选取幼嫩的叶子用干燥硅胶快速干燥,密封保存于-20℃中。
1.2 DNA提取、PCR扩增与序列克隆
通过CTAB法[5]从干燥的植物叶片中提取总DNA,用1%琼脂糖检测提取质量。选用通用的ITS1a-ITS4引物[6],扩增体系和扩增反应见表1。将PCR产物用EZ-10柱式胶回收试剂盒(Omega,广州)回收纯化,1%琼脂糖电泳检测,用分光光度计NanoDrop 2000 (Thermo Scientific)测定浓度后,进行pMD 18-T载体(Takara, 大连)在16℃连接4h,5ul反应体系为:载体50ng,PCR纯化产物20ng, Solution I 1.0ul。连接后的产物与E. coli DH5α感受态细胞(Takara, 大连)感受态细胞按说明书进行标准的转化程序,随后在平板上做蓝白斑筛选,37℃过夜培养。挑取阳性单克隆在LB培养基中培养3~5h后对菌液做PCR检测,反应体系和扩增程序见表1。将阳性克隆摇菌,用EZ-200 柱式试剂盒(生工,上海)提取质粒,用BigDye v3.1(Applied Biosystems, USA)和ABI3730xl(Applied Biosystems, USA)测序。
1.3 数据分析
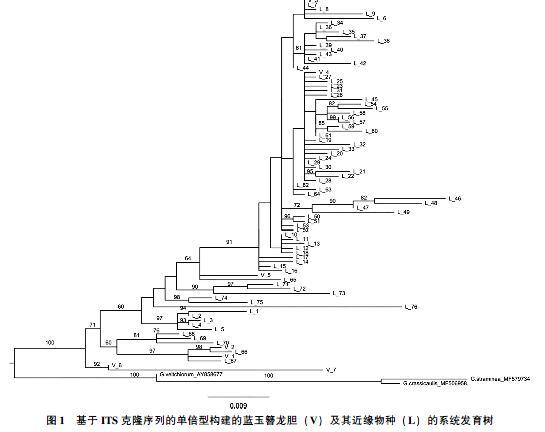
所得测序结果在软件GENEIOUS PRO 3.5.6[7]中查看测序峰图并逐一进行人工校对,通过比对去掉载体序列,随后进行数据整理和比对。将比对后的序列经格式转换后,在软件DnaSP 5.1[8]中统计单倍型个数。从GenBank下载蓝玉簪龙胆的近缘种线叶龙胆的76条ITS克隆数据(MN124292-MN124367)与蓝玉簪龙胆现有序列(AY858677),采用最大似然法,在IQ-TREE[9]中基于单倍型构建蓝玉簪龙胆和线叶龙胆的系统发育树。系统树构建采用的核苷酸替换模型由IQ-TREE自动计算。选用Ultrafast方法[10]进行bootstrap计算,设为5000次。以龙胆属秦艽组的麻花艽(MF579734)、粗茎秦艽(MF506958)为外类群。
2 结果
2.1 序列特征
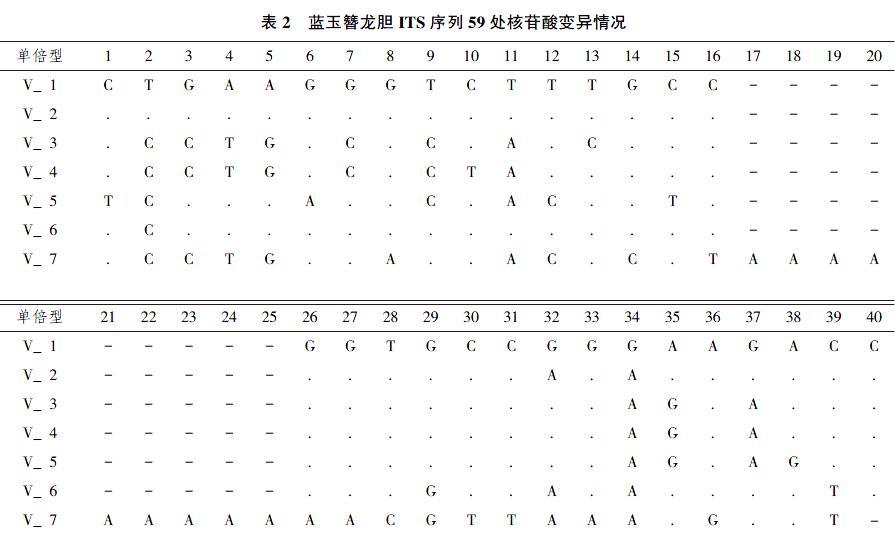
本研究对来自3个居群的6个个体进行基因克隆与测序,共得到8条ITS序列,长度为644~651bp。其在59个位置存在变异,包含48个碱基突变,11个缺失/插入(表2),共鉴定出7个单倍型(GenBank No. MN128583-MN128589)。在48处碱基突变中,2种碱基间的突变有47处,3种碱基间的突变有1处;缺失/插入的碱基片段长度从1~9bp。在鉴定出的8个单倍型中,仅有1个单倍型在多个克隆中共享,剩余单倍型均为单个克隆特有。共享的1个单倍型分布于6207种群的2个个体中。
2.2 遗传分化
本研究中,共得到蓝玉簪龙胆8条ITS序列,共鉴定出7个单倍型,表现很高的杂合性。与线叶龙胆76条ITS序列构建系统发育树可分为2个支系,一个支系包括蓝玉簪龙胆的2个单倍型(V_6,V_7),另一个支系为线叶龙胆的全部以及蓝玉簪龙胆剩余的5个单倍型(图1)。系统树基部节点的支持较高,内部的支持率大部分均低于60%(图1中未标注部分)。此外,值得注意的是从GenBank上下载的蓝玉簪龙胆ITS序列,其在系统发育树中与外类群聚在一起,而并未与该物种及其近缘种线叶龙胆的序列聚成一个大支。
3 总结
ITS序列是最常用的分子标记之一,被广泛用于系统发育树的构建和群体遗传结构分析。由本研究可知,蓝玉簪龙胆ITS序列存在高度杂合性。虽然本研究的样品有限,但在651bp长度的序列中,仍然存在48个碱基突变和11个缺失/插入。然而,如果不经过序列克隆直接进行测序,在居群内能鉴定出的变异位点非常有限。此外,系统发育树重建的结果表明,不经过序列克隆直接测序得到的序列在系统发育树中与外类群聚在一起,并未与蓝玉簪龙胆及其近缘种线叶龙胆的克隆序列聚成一个大支,虽然可能是由于样品产地不同导致的地区间的遗传分化,但也从一定程度说明克隆测序得到的序列与直接测序得到的序列在进化分析中可能存在结果不一致的情况。因此,对于ITS序列存在高度杂合的物种,深入的单克隆测序工作是有必要的。
蓝玉簪龙胆与线叶龙胆是2个形态分化明显的物种,具有显著的形态分化,例如線叶龙胆的莲座叶丛小或者没有、茎生叶为线形、花为天蓝色,而蓝玉簪龙胆的莲座叶丛大、茎生叶为椭圆形、花为深蓝色。然而,用分子片段ITS 难以将这2个物种区分开,说明2个物种的遗传分化尚不完全,是近期物种形成的产物,符合龙胆属在近期快速分化的事实[2]。虽然蓝玉簪龙胆形成了1个支系,且系统发育树基部节点的支持率较高,但多数蓝玉簪龙胆的单倍型仍内嵌在线叶龙胆支系之中。由于龙胆属是一个近期快速分化的类群[2],物种间的遗传分化较小,给近缘物种的区分增加了难度,有待用更大数据量如二代测序技术加以区分。
参考文献
[1] Ho T N , Liu S W. A worldwide monograph of Gentiana[M]. Beijing: Science Press, 2001.
[2]Favre A , Michalak I , Chen C H , et al. Out-of-Tibet: the spatio-temporal evolution of Gentiana (Gentianaceae)[J]. J. Biogeogr. 43: 1967-1978.
[3]Sun S S , Fu P C , Zhou X J , et al. The complete plastome sequences of seven species in Gentiana sect. Kudoa (Gentianaceae): insights into plastid gene loss and molecular evolution[J]. Front. Plant Sci., 2018, 9: 493.
[4]孙姗姗,付鹏程.龙胆族(龙胆科)分类与进化研究进展[J]. 西北植物学报,2019, 39(2): 0363-0370.
[5]Doyle J J , Doyle J L.A rapid DNA isolation procedure for small quantities of fresh leaf material [J]. Phytochemical Bulletin, 1987, 19: 11-15.
[6]Taberlet P . Universal primers for amplification of three non-coding regions of chloroplast DNA[J]. Plant Mol. Biol, 1991, 17(5):1105-1109.
[7]Kearse M , Moir R , Wilson A , et al. Geneious Basic: An integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics, 2012, 28(12):1647-1649.
[8]Librado P , Rozas J . DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11):1451-1452.
[9]Nguyen LT , Schmidt H A , von Haeseler A , et al. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Mol. Biol. Evol., 2015, 32: 268-274.
[10]Minh B Q , Nguyen M A T , Von Haeseler A . Ultrafast Approximation for Phylogenetic Bootstrap[J]. Mol. Biol. Evol., 2013, 30(5):1188-1195.
作者简介:
孙姗姗,讲师。研究方向:植物适应性进化。
基金项目:洛阳师范学院培育基金(项目编号:2018-PYJJ-009)

