马氏珠母贝DNA甲基化转移酶Dnmt1的克隆及表达
章佳斌,卢晓文,罗少杰,焦 钰,邓岳文,2
马氏珠母贝DNA甲基化转移酶的克隆及表达
章佳斌1,卢晓文1,罗少杰1,焦 钰1,邓岳文1,2
(1. 广东海洋大学水产学院,广东 湛江 524088;2. 广东省珍珠养殖与加工工程技术研究中心,广东 湛江,524025)
对马氏珠母贝()DNA甲基化转移酶1(DNA methyltransferase 1,Dnmt1)的基因全长及组织表达作分析,探究马氏珠母贝甲基转移酶Dnmt1()参与贝壳矿化调控机理。利用RACE技术获得的cDNA序列全长,并对的序列特征进行分析,同时利用逆转录-聚合酶链反应RT-PCR技术检测马氏珠母贝不同组织中的表达情况。基因cDNA长4 818 bp,其中,开放阅读框为4 623 bp,编码1 540个氨基酸。预测分子质量约为174.55 ku、理论等电点为5.89。结构域分析显示,PmDnmt1具有DMAP 结合结构域、DNMT1-RFD结构域、锌指结构、BAH结构域和C5胞嘧啶DNA甲基化酶结构域等。多序列比对结果发现,Dnmt1在不同物种间具有较高保守性。实时荧光定量PCR分析显示在所有检测组织中均有表达,在外套膜中央膜部分的表达量最高(< 0.05)。可通过调控外套膜的DNA甲基化修饰参与贝壳的形成。
马氏珠母贝;DNA甲基化转移酶;基因克隆;表达模式
DNA甲基化是目前在表观遗传学中研究最为成熟的一种表观遗传修饰,能够引起DNA稳定性、构象、染色体结构等多方面的改变,在调控基因表达[1]、维持染色体结构[2]以及基因印迹[3]等方面扮演着重要角色。通常来说,DNA甲基化是由甲基转移酶(DNA methyltransferase,Dnmt)起介导作用,并利用腺苷酰--甲硫氨酸(SAM)为甲基供体,将胞嘧啶(C)转变为5-甲基胞嘧啶(5mC)[4]。哺乳动物中,3种DNA甲基转移酶(Dnmt1、Dnmt2和Dnmt3)参与到DNA的甲基化修饰中。Dnmt3分为Dnmt3a和Dnmt3b两种,能够识别半甲基化和未甲基化序列,与DNA序列的从头甲基化有关;而Dnmt1能够以Dnmt3a/b修饰产物单链甲基化为模板,进而形成双链甲基化,其主要功能是维持基因组已有的甲基化模式,在配子形成期和胚胎早期的甲基化模式的形成发挥重要作用[5-6]。
已有研究表明无脊椎动物基因组的甲基化修饰参与调控转录活性[7]、外显子的选择性剪接[8]和早期发育活动[9]等。近年来,贝类甲基化模式也受到关注。研究表明不同家系的马氏珠母贝闭壳肌的甲基化水平与模式存在差异,近交家系的组织甲基化水平显著高于杂交家系的组织甲基化水平[10]。马氏珠母贝外套膜不同区域的甲基化存在显著差异,其组织间的甲基化水平由高到底依次是中央膜>边缘膜>套膜[11]。对牡蛎发育过程的甲基化研究发现[12],在牡蛎的胚胎发育和幼虫变态过程中其基因组差异甲基化区域受到强有力的调控,罗少杰等[13]发现牡蛎在感染帕金虫之后,其鳃与消化道的DNA甲基化水平显著低于对照组,这表明DNA甲基化在贝类免疫中起到一定的作用。
近些年对贝类甲基化的研究逐渐深入,但贝类基因组甲基化形成与调控的具体机制还尚未阐明,对于在贝类基因组甲基化作用的研究也未见报道。因此,本研究通过首次克隆获得马氏珠母贝基因,并检测其组织表达情况,以期为探究基因与贝类基因组甲基化关系,进一步探索无脊椎动物DNA甲基化修饰对基因表达调控作用提供参考。
1 材料与方法
1.1 材料
选用养殖于广东省湛江市徐闻县大井村海域(海域盐度约为31,pH为8.2)的“海选1号”马氏珠母贝()为实验用贝,清除贝体表面附着物暂养2 d后取样,样品置于液氮中保存。RACE扩增所用材料为马氏珠母贝的外套膜,用于荧光定量PCR的材料取自马氏珠母贝外套膜的边缘膜(Mantle edge,ME)、套膜(Mantle pallial,MP)、中央膜(Mantle central,MC)、闭壳肌(Adductor muscle,A)、鳃(Gill,Gi)、足(Foot,F)、肝胰脏(Hepatopancreas,He)、性腺(Gonads,Go)、血细胞(Hemocyte,B)。感受态细胞DH5α由本实验室保存。
1.2 引物信息
见表1。
1.3 方法
1.3.1 总RNA提取和cDNA第一链的合成 将马氏珠母贝各组织参照Trizol试剂说明书进行RNA的抽提;参照宝生物工程(大连)有限公司的Reverse Transcriptase M-MLV(RNase H-)说明书进行cDNA第一条链的合成。
1.3.2 DNA甲基化转移酶基因的全长cDNA的获得 根据马氏珠母贝基因组数据[14],利用Primer Premier5.0 软件设计引物,用于中间片段验证和5′端和3′端的获得。参照TaKaRa公司SMARTTM RACE cDNA Amptification Kit的说明书获得用于RACE的cDNA模板。利用3′RACE 的巢式引物依次扩增3′末端序列,第1轮PCR反应体系:Premix TaqTM 5 μL,UPM引物0.4 μL,outer引物0.4 μL,cDNA第一链0.4 μL,ddH2O 3.8 μL;第2次PCR反应体系引物更换为NUP引物和inner引物。随不同引物理论m值调整与其相应的退火温度,PCR反应条件为94 ℃预变性 5 min;94 ℃变性 30 s,退火30 s,72 ℃延伸 3 min,35 个循环;72 ℃保温10 min;4 ℃保存。5′末端序列扩增同样同巢式PCR获得,方法同3′末端序列。
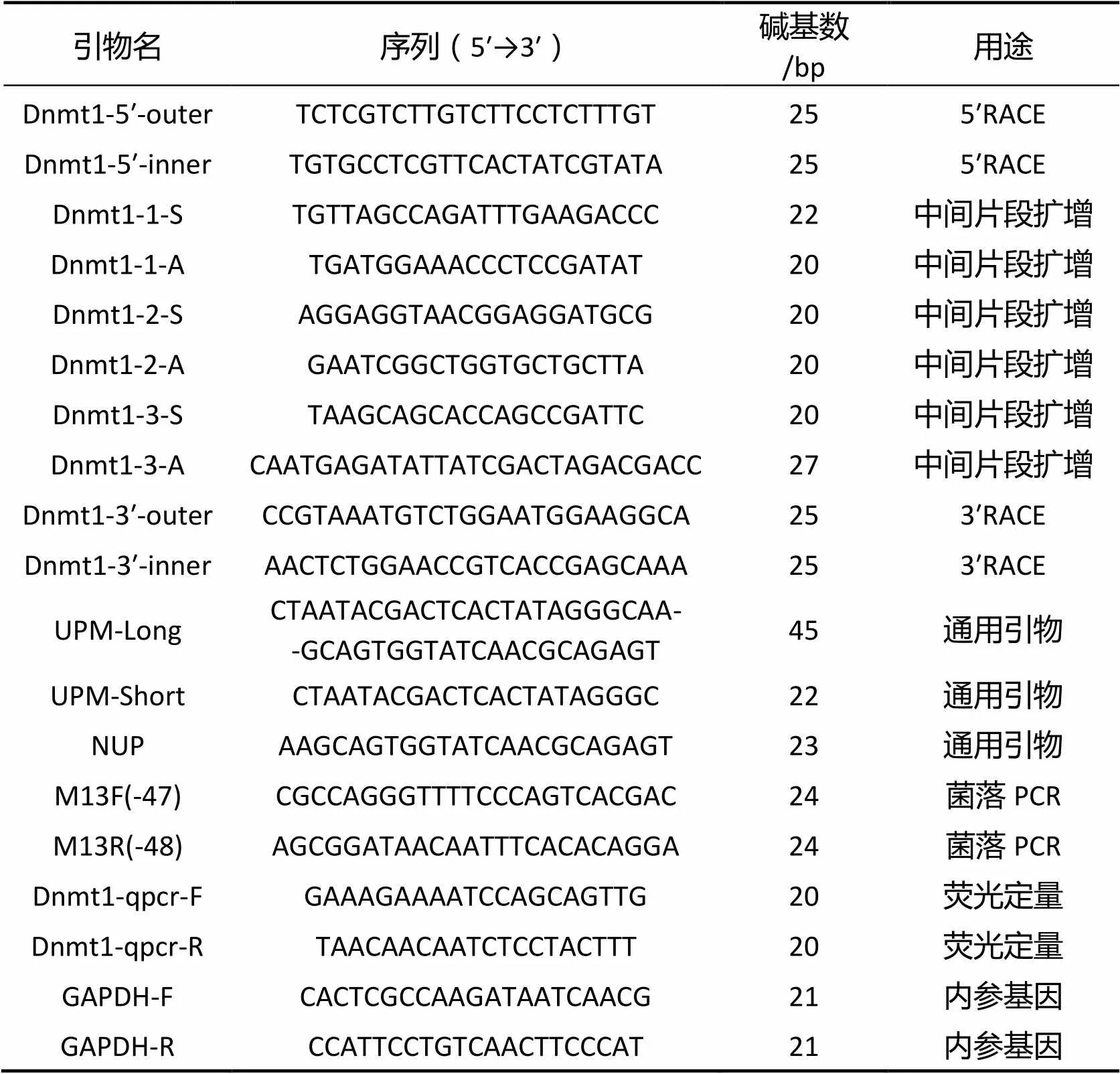
表1 研究所用引物
PCR产物先通过琼脂糖凝胶电泳检测产物,后根据纯化回收试剂盒说明书纯化目的基因片段;纯化后的基因片段与PMD19-T载体连接后转化到DH5α感受态细胞中,摇菌扩增后,经菌液PCR得到阳性单克隆,送至生工生物工程(上海)股份有限公司测序。
1.3.3 基因的生物信息学分析 利用DNAMAN软件将的中间片段与5′端和3′端片段进行拼接,即获得了其全长cDNA。根据所获得的基因全长,使用在线BLAST网站对基因的同源性和相似性进行比对;使用在线网站工具ORF Finder预测基因的开放阅读框;使用ExPASy分析蛋白的理化性质;使用PSITE V1分析氨基酸序列功能位点;使用SignalP 4.1和Cell-Ploc预测基因的信号肽与细胞定位;使用SMART和ScanProsite预测基因的结构域;使用SWISS-MODEL预测蛋白的空间结构;使用Prabi进行二级结构预测;使用EMBL在线工具Clustal Omega进行多序列比对;使用MEGAX软件进行进化树的构建。
1.3.4 荧光定量检测组织差异表达分析 以GAPDH作为内参基因,采用2-ΔΔCT相对定量分析法,检测在马氏珠母贝各组织中的表达,反应体系和程序参照Life Technologies Corporation 公司的SYBR Select Master Mix 说明书。荧光定量PCR结果用ABI step one Software 分析,样本重复性和组间差异性用SPSS19.0软件分析。
2 结果与分析
2.1 基因的基本特征
2.1.1序列特征分析 使用RACE技术获得基因序列(GenBank登录号:MN159188),图1可知,全长为4 818 bp,其中5′UTR为69 bp,3′UTR为126 bp,其中开放阅读框4 623 bp,共编码1 540个氨基酸。预测多肽分子质量174.54 ku、等电点为5.89。

起始密码子(atg)和终止密码子(tga)用小写字母表示;下划线部分代表DNMT1-RFD结构域;双下划线部分代表锌指结构;波浪线部分代表BAH结构域;虚下划线部分代表DMAP结合结构域;灰色背景部分表示C5胞嘧啶特异性DNA甲基化酶结构域;
2.1.2 PmDnmt1蛋白质理化性质分析 ExPASy预测带负电荷的氨基酸残基(Asp和Glu)为253个,带正电荷的氨基酸残基(Arg和Lys)为230个;不稳定系数为49.04,为不稳定蛋白;亲水性平均系数为-0.763,即为亲水性蛋白。通过PSITE V1对氨基酸序列的功能位点进行分析,发现该蛋白含有4个N-糖基化位点,3个cDNA和cGMP依赖蛋白激酶磷酸化位点,26个蛋白激酶C磷酸化位点,31个酪蛋白激酶Ⅱ磷酸化位点,9个酪氨酸激酶磷酸化位点,15个N-豆蔻酰化位点,2个酰胺化位点和23个微体C-端信号序列。SignalP 4.1分析显示没有信号肽,且通过Cell-Ploc分析发现该蛋白定位在细胞核。通过SMART和ScanProsite蛋白质分析工具和发现,该氨基酸序列在第10~57位上为DMAP结合结构域,第294~429位上为DNMT1-RFD结构域,第523~569位上为锌指结构(CXXC-type),第640~766位和821~989均为BAH结构域,第1 034~1 488位上为C5胞嘧啶特异性DNA甲基化酶结构域(图2)。在Prabi在线网站上对基因进行二级结构预测,结果显示:α螺旋30.97 %、β折叠4.61 %、延伸链15.06 %、无规则卷曲49.35 %。采用SWISS-MODEL软件预测PmDnmt1蛋白质分子的三维结构,与太平洋牡蛎Dnmt1结构存在较高的相似性(图3)。

图2 PmDnmt1结构域示意图
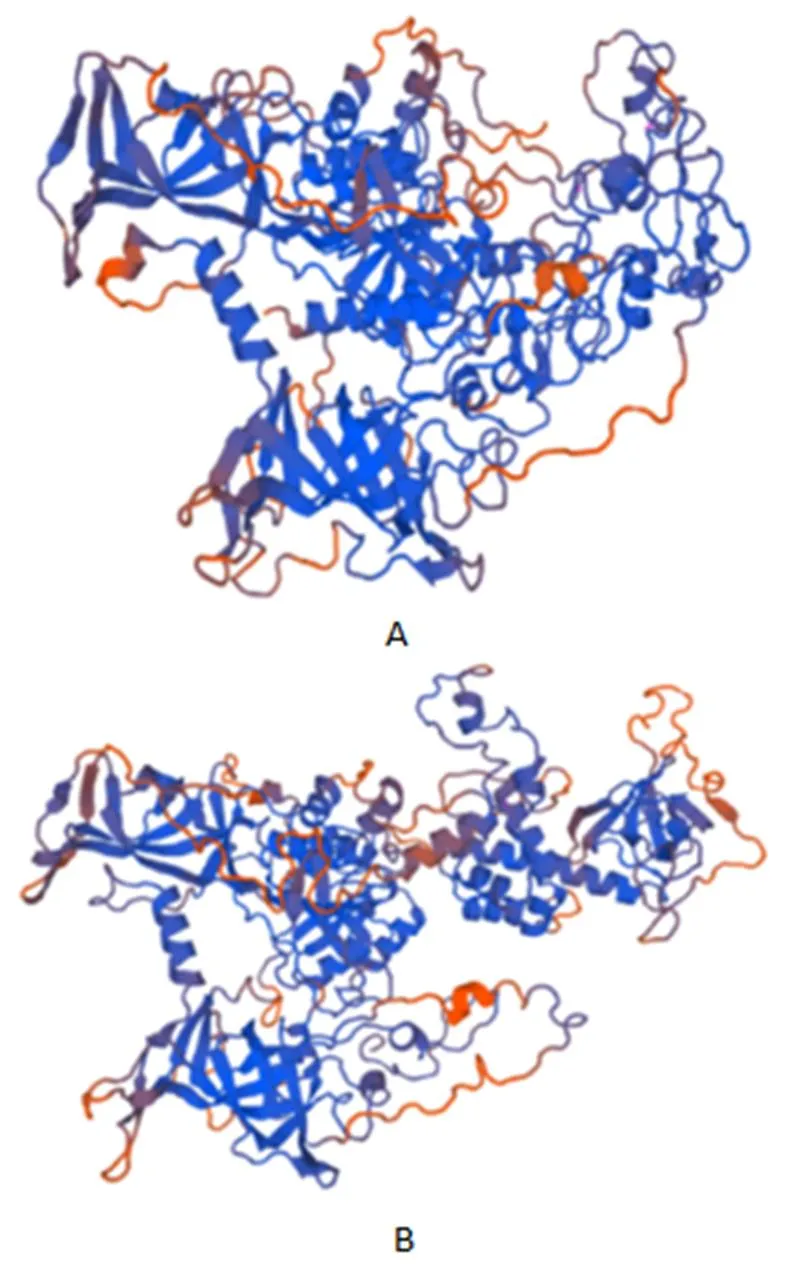
A:马氏珠母贝;B:太平洋牡蛎
2.1.3 PmDnmt1与其他物种的同源性分析 以EMBL在线工具Clustal Omega将PmDnmt1与太平洋牡()、美洲牡蛎()、虾夷扇贝()、拟球海胆()密西西比鳄()的Dnmt1的氨基酸进行多序列比对分析,结构显示物种间的Dnmt1具有较高的相似性(图4)。以马氏珠母贝、太平洋牡蛎、虾夷扇贝、拟球海胆、家蚕(、斑马鱼()、密西西比鳄、绿海龟()、褐家鼠()和人()的Dnmt1氨基酸序列为基础,使用NJ法构建系统进化树。结果显示,马氏珠母贝和太平洋牡蛎聚为一簇,并与虾夷扇贝聚在同一分支上;人和褐家鼠聚为一簇,并于密西西比鳄、绿海龟和斑马鱼聚在同一分支上(图5)。
2.2 组织表达差异分析
应用实时荧光定量PCR技术分析在组织中的表达量,所用组织分别为:外套膜的边缘膜(ME)、套膜(MP)、中央膜(MC)、闭壳肌(A)、鳃(Gi)、足(F)、肝胰脏(He)、性腺(Go)、血细胞(B)等九个组织。实验结果表明在马氏珠母贝的各个组织中均有表达,其中在中央膜上的表达量最高(< 0.05),在血淋巴中表达量最低(< 0.05),在余下7个组织中表达水平较为一致(如图6)。

保守氨基酸序列以蓝色阴影标准
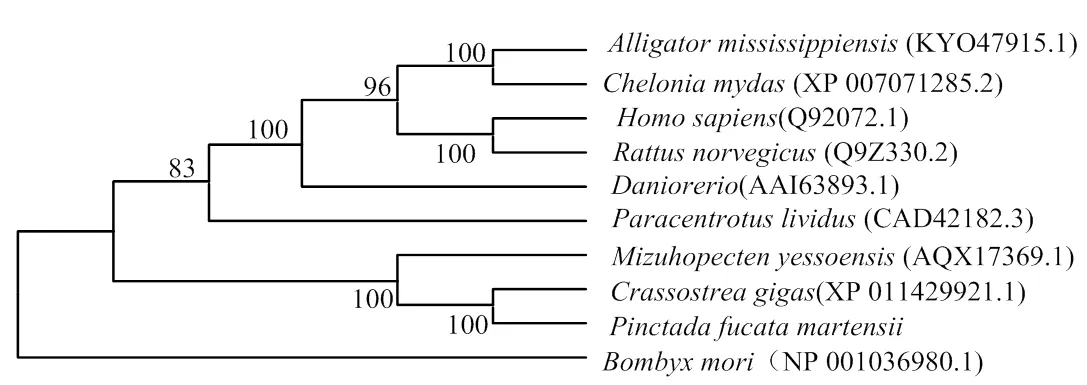
图5 PmDnmt1氨基酸序列聚类分析
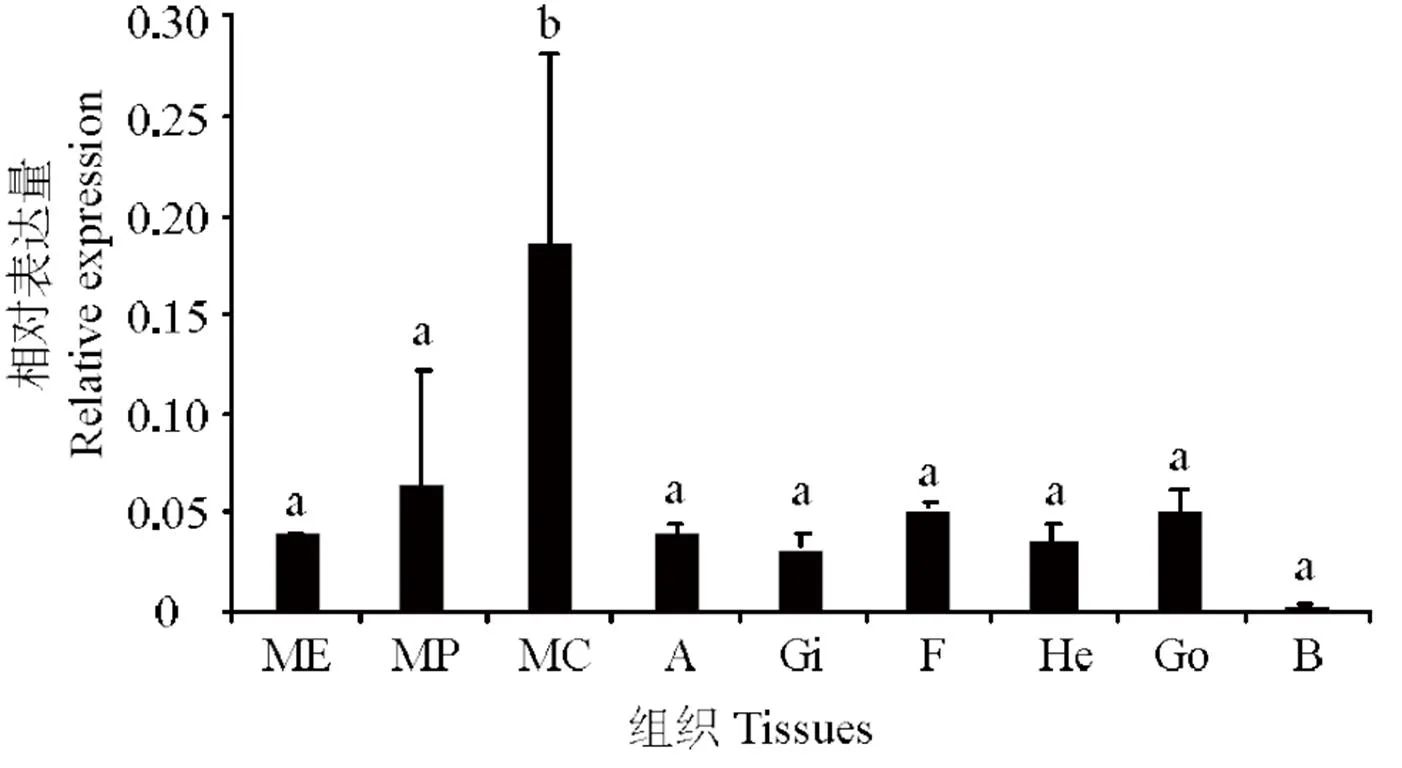
ME:边缘膜;MP: 套膜;MC:中央膜;A:闭壳肌;Gi:鳃;F:足; He: 肝胰脏;Go:性腺;B:血细胞;
3 讨论
DNA甲基化是一种常见的复制后修饰,参与生物机体内诸如调控基因表达、染色体结构维持、生长发育等多种重要生理过程的现象,主要由甲基转移酶行使DNA甲基化修饰功能。本实验通过RACE技术成功获得基因序列全长,为4 818 bp,共编码1 540个氨基酸。多序列比对分析结果显示PmDnmt1与不同物种中的Dnmt1的比对结果中呈现较高的保守性。一般认为Dnmt1序列可以分为两个区域N端蛋白识别区、C端催化区,其余位置上包含有半胱氨酸位点、BAH结构、锌离子结合点等起到锚定蛋白和形成DNA甲基化等作用[15-16]。SMART预测分析发现具有DMAP结合结构域,DNMT1-RFD结构域、锌指结构、BAH结构和C5胞嘧啶特异性DNA甲基化酶结构域等。DMAP结合结构域位于PmDnmt1的N端,在哺乳动物中,Dnmt1通过该位点与DMAP1(一种转录抑制因子)形成转录抑制复合物从而介导转录抑制[17],因此,可以推测PmDnmt1也具有转录抑制的功能。N端的DNMT1-RFD结构域与亚细胞定位、蛋白质结合和催化功能有关,研究表明,Dnmt1与裸露DNA和多核小体的结合活性受到DNMT1-RFD结构域的抑制,DNMT1-RFD结构域通过结合催化域而排除DNA发挥作用[18]。此外,PmDnmt1的锌指结构为CXXC类型,该结构域包含8个保守的半胱氨酸残基,能与2个锌离子结合,是与非甲基化CpG二核苷酸结合的位点[19]。这些典型结构域使得甲基转移酶具有稳定而广泛的作用位点,进而更好地调控基因表达。
为了更好探究马氏珠母贝甲基转移酶在组织中的作用,使用实时荧光定量PCR结果显示该基因mRNA在各个组织中均有表达,在中央膜中的表达量最高。外套膜是贝壳形成的主要器官,其不同区域所具有的结构特征和功能具有差异。贝壳按照形成的方式和组织的结构不同可分为3层,由内到外依次是珍珠质层,棱柱层及角质层。珍珠层是由中央的上皮细胞分泌形成,以文石为主;棱柱层结构由边缘膜分泌形成,以方解石为主;角质层结构由外套膜的外、中褶壳皮沟细胞分泌形成。研究发现与贝壳棱柱层形成有关的基因通常在边缘膜高表达,与贝壳珍珠层形成相关的基因则在中央膜和套膜区高表达[20]。马氏珠母贝外套膜不同区域其DNA甲基化水平不一[11],而在维持基因组的甲基化水平起到重要作用,且影响到胚胎发育[21]、器官功能分化[22]与转录调控。结合与贝壳珍珠层形成相关的矿化基因普遍在中央膜和套膜上高表达[22],推测矿化基因的高表达需要DNA甲基化等表观遗传修饰参与调控,因而有可能通过维持甲基化修饰参与调控贝壳珍珠层的形成。
4 结论
本研究首次利用RACE技术从马氏珠母贝克隆获得基因cDNA全长序列,该基因在马氏珠母贝外套膜中央膜中表达量最高,推测可能参与外套膜贝壳矿化基因的调控。
[1] MEEHAN R R. DNA methylation in animal development[J]. Seminars In Cell & Developmental Biology. 2003, 14: 53-65.
[2] RAZIN A. CpG methylation, chromatin structure and gene silencing-a three-way connection[J]. The EMBO Journal. 1998, 17(17): 4905-4908.
[3] AUTRAN D, HUANCA-MAMANI W, VIELLECA- LZADA J P. Genomic imprinting in plants: the epigenetic version of an Oedipus complex[J]. Current Opinion In Plant Biology. 2005, 8(1): 19-25.
[4] 张海燕, 赵洪斌, 田亚平, 等. DNA甲基化检测技术与应用前景[J]. 标记免疫分析与临床. 2013, 20(5): 348-351.
[5] SUBRAMANIAM D, THOMBRE R, DHAR A, et al. DNA methyltransferases: a novel target for prevention and therapy[J]. Frontiers In Oncology. 2014, 4: 80.
6] CUI D, XU X. DNA methyltransferases, DNA methylation, and age-associated cognitive function[J]. International Journal of Molecular Sciences. 2018, 19: 13155.
[7] SUZUKI M M, BIRD A. DNA methylation landscapes: provocative insights from epigenomics[J]. Nature Reviews Genetics. 2008, 9(6): 465-476.
[8] LYKO F, FORET S, KUCHARSKI R, et al. The honey bee epigenomes: differential methylation of brain DNA in queens and workers[J]. Plos Biology. 2010, 8(11): e1000506.
[9] RIVIERE G, WU G, FELLOUS A, et al. DNA methylation is crucial for the early development in the oyster[J]. Marine Biotechnology. 2013, 15(6): 739-753.
[10] 罗少杰, 张鹏飞, 焦钰, 等. 马氏珠母贝近交与杂交家系的 DNA 甲基化多态性比较[J]. 广东海洋大学学报. 2016, 36(6): 9-15.
[11] 罗少杰, 邓岳文, 郑哲, 等. 马氏珠母贝外套膜不同区域基因组DNA甲基化MSAP分析[J]. 中国水产科学. 2016, 23(6): 1227-1235.
[12] SONG K, LI L, ZHANG G. The association between DNA methylation and exon expression in the Pacific oyster[J]. Plos One. 2017, 12(9): e185224.
[13] FARIAS N D, DE OLIVEIRA N F P, DA SILVA P M. Perkinsus infection is associated with alterations in the level of global DNA methylation of gills and gastrointestinal tract of the oyster[J]. Journal of Invertebrate Pathology. 2017, 149: 76-81.
[14] DU X, FAN G, JIAO Y, et al. The pearl oystergenome and multi-omic analyses provide insights into biomineralization[J]. Gigascience. 2017, 6:1-12.
[15] 苏玉, 王溪, 朱卫国. DNA甲基转移酶的表达调控及主要生物学功能[J]. 遗传. 2009, 31(11): 1087-1093.
[16] 王志刚, 建新. DNA甲基转移酶分类、功能及其研究进展[J]. 遗传. 2009, 31(9): 903-912.
[17] ROUNTREE M R, BACHMAN K E, BAYLIN S B. DNMT1 binds HDAC2 and a new co-repressor, DMAP1, to form a complex at replication foci[J]. Nature Genetics. 2000, 25(3): 269-277.
[18] FARISA S, FAGAN R L, MATTHEW W, et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1[J]. Journal of Biological Chemistry. 2011, 286(17): 15344-15351.
[19] CROSS S H, MEEHAN R R, NAN X, et al. A component of the transcriptional repressor MeCP1 shares a motif with DNA methyltransferase and HRX proteins[J]. Nature Genetics. 1997, 16(3): 256-259.
[20] FUNABARA D, OHMORI F, KINOSHITA S, et al. Novel genes participating in the formation of prismatic and nacreous layers in the pearl oyster as revealed by their tissue distribution and rna interference knockdown[J]. Plos One. 2014, 9: e847061.
[21] RAI K, NADAULD L D, CHIDESTER S, et al. Zebra fish Dnmt1 and Suv39h1 regulate organ-specific terminal differentiation during development[J]. Molecular and Cellular Biology. 2006, 26(19): 7077-7085.
[22] ANDERSON R M, BOSCH J A, GOLL M G, et al. Loss of Dnmt1 catalytic activity reveals multiple roles for DNA methylation during pancreas development and regeneration[J]. Developmental Biology. 2009, 334(1): 213-223.
Molecular Cloning and Expression Analysis of DNA Methyltransferase 1 () from
ZHANG Jia-bin1, LU Xiao-wen1, LUO Shao-jie1, JIAO Yu1, DENG Yue-wen1,2
(1.,524088,;2.,524025,)
【】In order to offer a theoretical basis for exploring the regulation of DNA methyltransferase 1 of() in biomineralization, the full-length gene was obtained and the tissue expression pattern ofwas elucidated. 【】The cDNA sequence ofwas obtained by rapid amplification of cDNA ends technology (RACE). The characteristics ofwere analyzed, and the mRNA expression ofin different tissues ofwere detected by fluorescence quantitative PCR. 【】The results showed that the length ofwas 4 818 bp,including ORF 4 623 bp which encoded 1 540 amino acids. The predicted molecular weight was about 174.55 ku and the theoretical isoelectric point was 5.89. The result of domain prediction analysis showed thathad DMAP binding domain, Dnmt1RFD, zinc finger structure, BAH domain and c-5 cytosine-specific DNA methylase domain profile. The multi-sequence alignment results showed that Dnmt1 was highly conserved across different species. Real-time fluorescence quantitative PCR analysis showed thatgene was expressed in all test tissue, with the highest expression in the mantle central (< 0.05).may be involved in shell formation through regulating the DNA methylation.
; DNA methyltransferase; gene clone; expression pattern
Q78
A
1673-9159(2019)05-0016-08
10.3969/j.issn.1673-9159.2019.05.003
2019-04-20
国家自然科学基金(31672626)
章佳斌,男,硕士研究生,研究方向为珍珠培育与加工。E-mail:874788088@qq.com
邓岳文,教授,研究方向为珍珠贝遗传育种与养殖。E-mail:dengyw@gdou.edu.cn
章佳斌,卢晓文,罗少杰,等. 马氏珠母贝DNA甲基化转移酶的克隆及表达[J]. 广东海洋大学学报,2019,39(5):16-23.
(责任编辑:刘朏)

