药物靶标作用关系预测结果评价及查询验证
余冬华 郭茂祖,2,3 刘晓燕 程 爽
1(哈尔滨工业大学计算机科学与技术学院 哈尔滨 150001)2(北京建筑大学电气与信息工程学院 北京 100044)3(建筑大数据智能处理方法研究北京市重点实验室(北京建筑大学) 北京 100044)4(中国工程物理研究院材料所 四川绵阳 621900)
药物研发是医药界的驱动力,也引起了学术界的研究热潮,计算机辅助药物设计是对传统的实验型药物发现的补充,加速了新药开发进程,缩短药物研发时间,降低药物研发成本,同时提升药物研发成功率.
网络药理学(network pharmacology)[1-2]和药物重定位(drug repositioning)[3-4]颠覆了传统的药物研发理念,完善了药物靶标作用关系(drug-target interaction)预测的理论基础,为计算机辅助药物设计提供了新思路,加速了药物发现与设计.随着公开数据库KEGG[5],DrugBank[6],ChEMBL[7]等中的可用数据增多,很多预测药物靶标作用关系的计算方法也被提出[8-13].其主要思路为:建立合适的药物靶标数据,构建作用关系预测方法并做出合理的评价,然后进行实际作用关系预测.这些计算方法弥补了实验方法中的时间消耗和高昂成本投入的缺陷.
Yamanishi等人[14]在2008年基于KEGG数据库建立了Enzyme,IC(ion channel),GPCR(G -protein-coupled receptor),NR(nuclear receptor)四个药物靶标作用关系数据集(包括药物靶标作用关系矩阵、药物-药物相似度矩阵、靶标-靶标相似度矩阵),其中,已经验证的药物靶标作用关系用1表示,未知作用关系的药物靶标对(unknown interaction)用0表示,受到广泛接受及认同,Google学术显示引用次数为467次,被认为是药物靶标作用关系预测模型的标准检验数据集.2009年,Bleakley等人[15]在上述4个数据集的基础上,提出了经典的二分局部模型(bipartite local models, BLM)药物靶标作用关系预测方法,并把其当作二分类模型,采用ROC(receiver operating characteristic curve)曲线及曲线下方面积(area under curve, AUC)作为该方法的评价标准.
该数据集的建立促使了很多预测方法的诞生,而AUC评价标准也对后续方法产生了重要影响.然而,该评价需要将未知作用关系的药物靶标对视为负例,这是一种不合理的假设,因为很多未知的作用关系可能实际上存在相互作用关系,只是到目前为止,仍没有被实验所证实.况且,预测方法的目标就是需要将潜在的作用关系预测出来,即将作用关系0预测成1.尽管如此,这种不合理的评价方式也一直在延续.作为一种弥补手段,会将预测结果中极有可能的药物靶标对(比如TopN,即按照预测的药物靶标对作用关系可能性从大到小排序,取前N对)在数据库中查询验证.Laarhoven等人[16]提出GIP-RLS(Gaussian interaction profile-regularized least squares)方法后,用AUC及AUPR(area under precision-recall curve)进行了评价.对于需要预测的药物靶标作用关系,仅对每个数据集Top 20查验过KEGG,DrugBank,ChEMBL数据库.Gonen[17]提出的KBMF2K(kernelized Bayesian matrix factori-zation with twin kernels)预测方法,用AUC进行评价,且仅对每个数据集Top 5查验过KEGG,DrugBank,ChEMBL数据库.Mei等人[18]提出BLMNII(bipartite local models with neighbor-based interaction-profile)预测方法后,用AUC及AUPR进行了评价,而没有对预测的药物靶标对进行数据库的查询验证,也就没有验证所预测的作用关系中是否存在已经被实验验证的药物靶标对.Hao等人[19]提出的RLS-KF(regularized least squares with kernel fusing)预测方法,用AUC及AUPR进行评价,仅对NR数据集中预测Top 100对药物靶标进行数据库查询验证,虽然其AUC值接近于1,对于二分类模型来说,接近于1的AUC值表示该模型预测接近于完全正确,但对于NR数据集来说,直到Top 100时,才有31对药物靶标已经获得了生物化学实验验证,对于目前可以查询到的已验证的药物靶标对来说,仍然有9对不包含在内,这也表明,如此之高的预测精度,在实际预测中远未达到理想结果.Luo等人[20]提出DTINet(network integra-tion pipeline for drug-target interaction)预测方法,也用AUC及AUPR进行了模型评价,并对预测结果的Top 150药物靶标对进行了数据库查询验证,特别地,选择了极有可能具有作用关系的COX inhibitory进行了生物化学实验验证,实验结果证实了DTINet模型的预测准确.Hao等人[21]提出DNILMF(dual-network integrated logistic matrix factorization)预测方法,用AUC及AUPR评价该模型,采用与Yamanishi等人[14]相似的方式收集了一个新数据集,在最后的预测结果中,仅对Top 5的药物靶标对进行了查询验证.Olayan等人[22]提出DDR(novel method that improves the drug-target interaction prediction accuracy)预测方法,在方法的评价方面,舍弃了AUC评价,而选择加权平均AUPR评价,并且对预测中的Top 25进行了查询验证.Peng等人[23]提出PreNNDS(drug-target identification mode by integrating neighbor interac-tion profiles, nonnegative matrix factorization, discriminative low-rank representation, and sparse representation classification into a unified frame-work)预测方法,仅采用AUPR进行评价,与其他文章按照预测得分(或概率)选择TopN个药物靶标对进行查询验证不同,Peng 等人选择了2个靶标(ID分别为hsa1132及hsa1124)所对应的Top 20的药物进行查询验证,同样地,选择了2个药物(ID分别为D00255及D00195)所对应的Top 20的靶标进行查询验证.Liu等人[24]提出NRLMF(neighbor-hood regularized logistic matrix factorization)预测方法后,用 AUC及AUPR进行评价,该文在KEGG,DrugBank,ChEMBL,Matador数据库中查询验证了4个数据集中Top 1 000的药物靶标对,因此,其给出了Top 10,Top 30,Top 50的评价结果.然而,其并未提供可以快速高效地对如此大量的未知作用关系药物靶标对查询验证的方法,无法减轻后续学者研究该问题的工作量.实际上,对于Enzyme,IC,GPCR,NR四个数据集来说,Top 1 000仍然是未知药物靶标作用关系中很小的一部分.多种方法的查询验证TopN值总结见表1:

Table 1 The Checked Top N Values of Various Methods表1 各种方法查询验证Top N值
因此,如何验证这些预测结果的准确性是研究者面临的一个挑战,在目前有限条件下,考虑到时间消耗及实验成本,不可能对所有未知的药物靶标作用进行生物化学实验验证.综合现有的各种评价方法,一种可行的方式就是用AUC及AUPR验证预测方法可行性及预测精度,并利用数据共享,如公开数据库DrugBank,KEGG,ChEMBL,提供的很多药物靶标之间的作用关系数据,将预测的作用关系逐对在数据库中进行查询验证,确认该未知的作用关系是否已经被相关实验验证.这种方式得到了学术界普遍认可.然而,现有的方法也仅仅查询验证TopN药物靶标对,因为查询验证是一个重复且耗时的工作,且未知作用关系的药物靶标对占预测总数的绝大多数,而更为合理的验证预测结果的方式为:对所有未知作用关系的药物靶标对在数据库上查询验证一遍.
针对上述问题,本文不仅为药物靶标作用关系验证提供一种高效、便捷、可行的查询验证方法DTcheck(drug-target check),并利用DTcheck查询的结果对4个标准数据集及AUC评价做了分析,主要贡献归纳为
1) 结合网络爬虫特性,设计DTcheck查询验证方法并实现.
2) 基于KEGG,DrugBank,ChEMBL数据库,通过DTcheck将4个数据集中已经验证的药物靶标作用关系补充完整.
3) 对于(批量)提供KEGG DRUG ID及KEGG GENES ID的药物靶标对,DTcheck可以在线查询并返回其在KEGG,DrugBank,ChEMBL数据库中的验证结果.
4) 利用DTcheck查询的结果,重新分析了BLM方法的预测结果,探讨了ROC曲线评价药物靶标作用关系的不合理性,并引入TopN评价指标,实际结果表明AUC值低的BLMd在预测新的药物靶标作用关系时要优于AUC值高的BLMmax,给AUC评价的不合理性提供了依据.

Fig. 1 Technical route and flow of DTcheck图1 DTcheck 技术路线及流程
1 DTcheck查询验证关键问题及解决思路
Yamanishi等人[14]提供的标准数据集中采用KEGG ID标注药物及靶标,而DrugBank,ChEMBL有其自己的ID体系.如药物D02441(KEGG DRUG ID)在DrugBank对应的ID为DB00311,在ChEMBL中对应的Compound ID为CHEMBL18,而靶标hsa766(KEGG GENE ID)需要与UniProt数据库[25]中的P43166(UniProt ID)对应起来,通过DrugBank中的药物或者ChEMBL中的药物(或化合物)查询到的对应靶标,会提供对应的UniProt ID.因此,其中一个关键问题就是需要将3个数据库中提供的不同ID合理对应起来.
药物靶标作用关系是成对出现的,如D02441~hsa766,因此,在查询验证过程中,可通过D02441在KEGG DRUG中查找到对应靶标hsa766,也可在DrugBank中通过DB00311查找到对应靶标P43166(UniProt ID),而hsa766与P43166代表同一个靶标,即不同的ID代表同一个对象.相对应地,也可以从靶标hsa766出发,从KEGG GENE中查找到药物D02441.因此,另一个关键问题就是在查询验证中,需要对药物靶标对进行双向查询验证.
由于KEGG DRUG中提供了该药物在ChEMBL及DrugBank中的对应ID和相应的网页链接,因此,利用爬虫抓取数据的特性,读取该药物在KEGG DRUG中所提供的对应靶标ID后,需要额外读取该药物在其他2个数据库中的ID及链接.而在KEGG GENE中查询的数据仅是该靶标是否有对应的药物即可.因此,在药物靶标对的双向查询验证中,可以优先查询靶标,然后查询药物.
从KEGG DRUG中读取该药物在ChEMBL,DrugBank中的链接后,可以独立地进行查询,然后将查询结果合并,最后核对待验证的药物靶标对.KEGG DRUG提供的ChEMBL链接可能会有多个,需要遍历所有可能链接,读取该链接返回的ChEMBL Target信息及其跳转链接,就可以查询到该药物对应靶标的UniProt ID.从KEGG DRUG还可以读取该药物的DrugBank跳转链接,利用爬虫访问该链接,获取返回信息,从其返回信息中读取Targets,Enzymes,Carriers,Transporters四个版块信息所提供的UniProt ID即可.这样就可以获取该药物在ChEMBL,DrugBank两个数据库中所对应靶标的UniProt ID.
靶标的UniProt ID与KEGG GENE ID并不一样,后者也是4个标准数据集所提供的ID,因此,需要先将UniProt ID转换成KEGG GENE ID才能核查待验证的药物靶标对的作用关系是否已经被证实.此时可以借助UniProt数据库提供的ID mapping功能,将靶标的UniProt ID转换成KEGG GENE ID,这一步的实现需要借助爬虫发送数据请求并获取返回信息的特性.
2 DTcheck查询验证技术路线
解决了DTcheck查询验证中遇到的关键问题,本文设计了如图1所示的技术路线流程,其代码链接为https:github.comYu123456DTcheck.从图1中可以看出,双向查询分成2个部分独立查询,最终需要将2部分查询结果合并,而3个数据库之间,需要先从KEGG DRUG中获取DrugBank,ChEMBL数据库的跳转链接,然后依次获取相应数据库中的靶标信息,而从这2个数据库中返回的靶标是UniProt ID,为了与待验证的药物靶标对核对,需要增加一步ID转换,把UniProt ID转换成KEGG GENE ID.
3 预测结果评价及查询验证分析
Yamanishi等人[14]于2008年建立了Enzyme,IC,GPCR,NR四个标准数据集,如表2所示.其中,drug-target interactions代表当时已经验证的药物靶标作用关系;unknown interaction代表当时未知的药物靶标作用关系;从最后2行已知与未知药物靶标作用关系具体数值来看,未知作用关系远多于已知作用关系.该数据集从建立至今已有10年之久,在这期间有很多作用关系得到生物实验验证,本文采用DTcheck查询KEGG,DrugBank,ChEMBL三个数据库,将新增的作用关系补充完整.伴随这4个数据集,Bleakley等人[15]于2009年提出了经典的BLM预测方法,并用AUC进行评价,本文将依据DTcheck查询到的结果,重新探讨AUC评价的不合理性,并采用TopN中已经被验证的药物靶标对的数目重新分析预测结果.

Table 2 Drug-Target and Their Interactions表2 药物靶标及其作用关系信息
3.1 数据集已验证作用关系补充
经过DTcheck的查询,每个数据集都可以找到新验证的药物靶标对,如表3所示.从增长率(increased ratio)来看,新药物靶标关系增加最快的数据集为GPCR;新发现的药物靶标对数最多的却是Enzyme数据集,达到907对;虽然NR药物靶标对的数目最少,但也将近增加一半,达到40对.

Table 3 Drug-Target and Their Increased Interactions表3 药物靶标数据集新增作用关系
表4给出了NR数据集的DTcheck查询到的40对已经验证的药物靶标作用关系.其中,DrugID,TargetID分别表示药物、靶标的ID;C,D分别表示在ChEMBL,DrugBank数据库中查询到该药物靶标对作用关系;K需要做一个区分,在KEGG栏中,表示通过药物找到了对应的靶标,在KEGGh栏中,则表示通过靶标找到了对应的药物;空白表示对应数据库中未查询到相关信息.表4中出现大量空白,表明3个数据库之间的信息并没有完全同步,因此,必须查询每一个数据库,避免遗漏.对于NR数据集来说,DrugBank提供的已验证作用关系最多,其次是KEGG,最后才是ChEMBL.
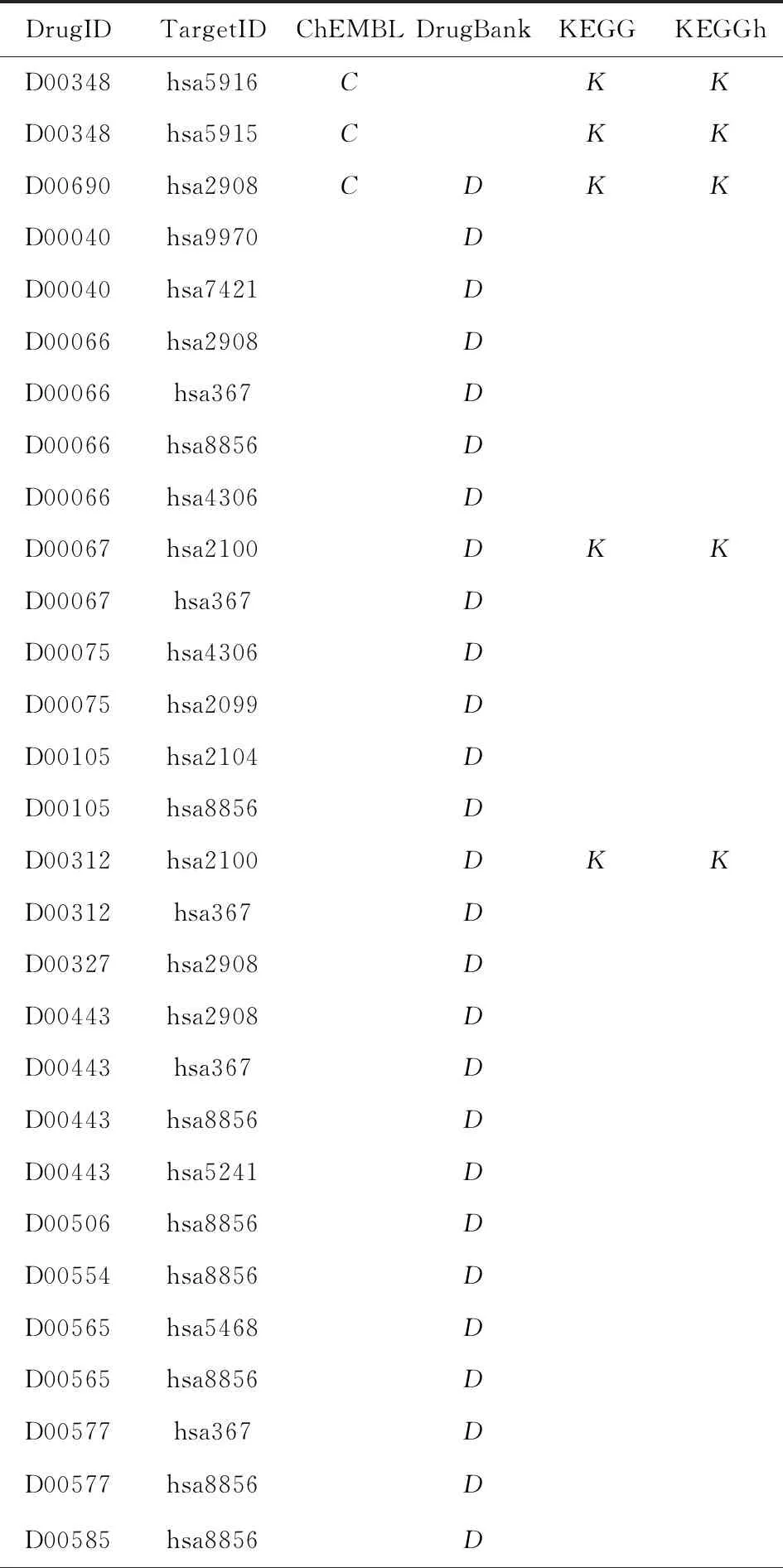
Table 4 New Increased Drug-Target Interactions of NR表4 NR数据集新增药物靶标关系
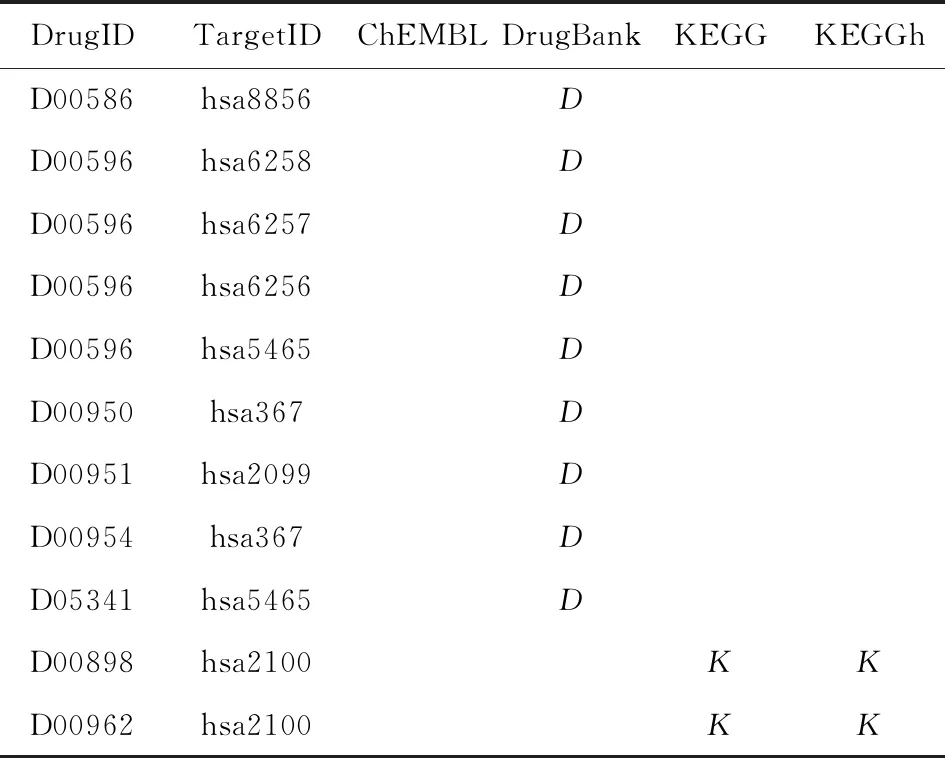
Continued (Table 4)
Note: The blank indicates that the interaction relationship has not been queried yet.
由于Enzyme,GPCR,IC这3个数据集查询到的新增作用关系数目较多,限于篇幅,不宜在文中直接给出,其查询的相应结果见https:github.comYu123456Drug-targetChecked.
3.2 BLM方法预测结果查询验证及评价分析
BLM可以细分为3个子方法,即BLMd,BLMt,BLMmax,其中BLMmax是取BLMd,BLMt二者预测的较大值.Bleakley等人[15]依据AUC(如表5所示,较优者黑体表示)评价指出,在3个方法中,BLMmax性能最好,BLMd性能最差.

Table 5 AUC of 4 Datasets表5 AUC 评价值
Note: The boldface represents the best one of the three methods.
从AUC评价来看,BLMmax非常显著地优于BLMd,因此,BLMmax对新药物靶标作用关系的预测结果也应该优于BLMd,然而,表6却展现出截然相反的结论.在TopN评价中,从相同的N中找到更多验证关系者性能更优,在表6中较优者已经加粗表示.在Top 50之前的每一个TopN中,BLMd预测正确的药物靶标对均多于BLMmax;在Top 50至Top 1 000之间,才出现后一种方法优于前一种方法的趋势,如Top 1 000时,BLMmax预测正确的药物靶标对的数目多于BLMd.然而,开发药物靶标作用关系预测方法的初衷是为实验生物化学家提供有实质指导作用的建议,即尽可能少的实验下找到真正的作用关系,因为用生物实验验证药物靶标作用关系既耗时又费钱,因此,Top 50评价会是一个更优的选择.从这个方面来说,BLMd优于BLMmax,这也表明仅用AUC评价药物靶标预测模型不合理.
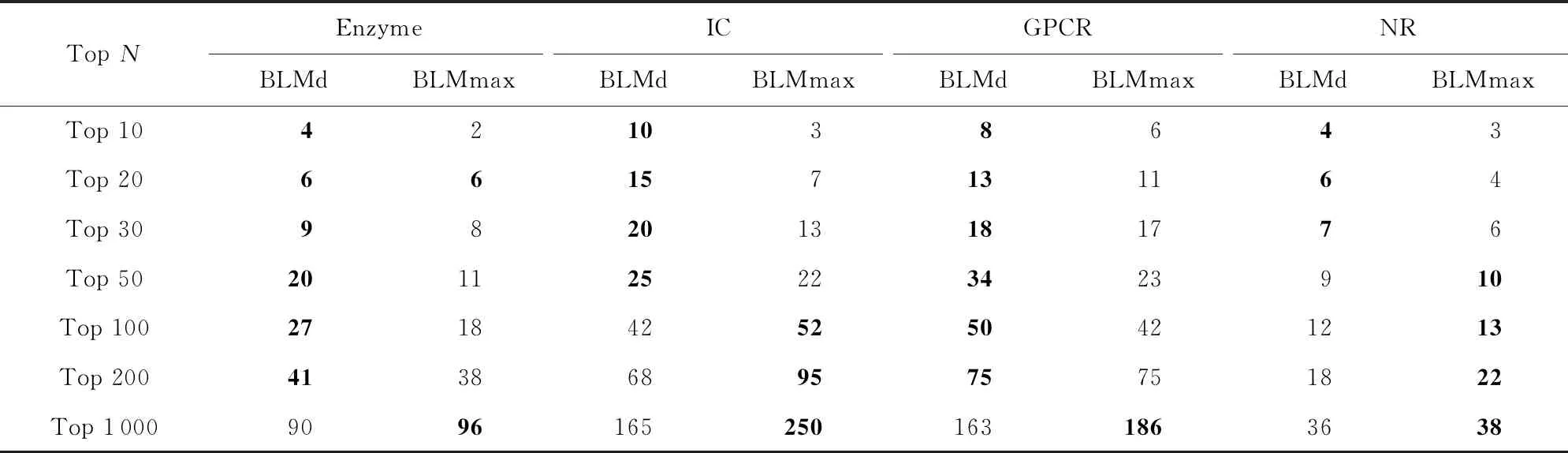
Table 6 Comparing New Interaction of Top N Between BLMmax and BLMd表6 BLMmax 与 BLMd 中Top N中新作用关系比较
Note: The boldface represents the better interaction between BLMd and BLMmax.
4 总 结
本文利用爬虫特性,设计并实现了DTcheck在KEGG,DrugBank,ChEBML数据库上高效、便捷的药物靶标作用关系查询验证,利用DTcheck实现对标准数据集新增作用关系的扩展补充,基于BLM方法的预测结果,说明了AUC评价药物靶标作用关系预测的不合理性,及TopN可以给出更加合理的评价.本文提供的药物靶标对在线查询验证思路及实现,大大节省研究者查询验证时间,使得在方法的验证中不仅仅限于每个数据集仅查询预测排序靠前的少数药物靶标对,有利于更准确地评价药物靶标预测方法.
DTcheck也有待于继续扩展,目前仅仅查询KEGG,DrugBank,ChEMBL三个数据库,实际上,Matador,SuperTarget等数据库也可以查询到一些相关作用关系.我们将会继续对此工作扩展,以期DTcheck更具有广泛性.

