CRISPR/Cas9技术在非模式植物中的应用进展
白英俊 李国瑞 黄凤兰 李威
摘要:基因组编辑技术的出现对植物遗传育种及作物性状的改良产生了深远意义。CRISPR/Cas(clusteredregularlyinterspacedshortpalindromicrepeat)是由成簇规律间隔短回文重复序列及其关联蛋白组成的免疫系统,其作用是原核生物(40%细菌和90%古细菌)用来抵抗外源遗传物质(噬菌体和病毒)的入侵。该技术实现了对基因组中多个靶基因同时进行编辑,与前两代基因编辑技术:锌指核酶(ZFNs)和转录激活因子样效应物核酶(TALENs)相比更加简单、廉价、高效。目前CRISPR/Cas9基因编辑技术已在拟南芥(Arabidopsisthaliana)、烟草(Nicotianabenthamiana)、水稻(Oryzasativa)、小麦(Triticumaestivum)、玉米(Zeamays)、番茄(tomato)等模式植物和多数大作物中实现了定点基因组编辑,其应用范围不断地向各类植物扩展。但与模式植物和一些大作物相比,CRISPR/Cas9基因编辑技术在非模式植物,尤其在一些小作物的应用中存在如载体构建、靶点设计、脱靶检测、同源重组等问题有待进一步完善。该文对CRISPR/Cas9技术在非模式植物与小作物研究的最新研究进展进行了总结,讨论了该技术目前在非模式植物、小作物应用的局限性,在此基础上提出了相关改进策略,并对CRISPR/Cas9系统在非模式植物中的研究前景进行了展望。
关键词:CRISPR/Cas9系统,植物遗传育种,基因组编辑,非模式植物
中图分类号:Q943
文献标识码:A
文章编号:1000-3142(2019)03-0419-08
遗传突变对于研究植物基因功能和作物遗传改良至关重要。在过去,自然突变体的表征已经揭示了遗传多样性的重要性。而且,许多研究已经使用了物理方法(γ辐射)、化学方法(甲磺酸乙酯)或生物方法(例如T-DNA/转座子)导致点突变、缺失、重排和基因重组得到突变体。但随机诱变会产生许多不希望的突变,以及大规模的筛查突变体不仅工作繁琐且成本昂贵。序列特异性核酸酶(SSNs)的出现在基因定点诱变方面实现了突破。SSN可以诱导特定的双链断裂(DSBs)染色体位点,此核酸酶可以引起的两种不同的DNA双链修复机制:非同源末端连接(nonhomologousend-joining,NHEJ)和同源重组型修复(homology-directedrepair,HDR)。锌指核酸酶(ZFNs)作为第一代SSNs(Bibikovaetal.,2002)被用于编辑植物基因组(Petolino,2015)。然而,ZFN结构难以操作且成本高昂,极大地阻碍了它们在各类植物当中的应用(Sanjanaetal.,2012)。之后TALEN的开发并在植物中应用(Boch&Bonas,2010),虽然在载体的构建及操作上比ZFN更容易但是使用TALEN仍然需要建造复杂的串联在TAL蛋白中的重复结构域且成本仍然很高。
CRISPR技术的问世加速了基因编辑工程的进程,突破了前两代基因编辑技术对每个靶基因确定不同的核酸酶的局限性(Sunetal.,2016;Eyquemetal.,2017;Svitashevetal.,2015)。相比与前两代基因编辑技术,CRISPR/Cas9系统只需要对每1个基因靶位点修饰合成1个靶标sgRNA(singleguideRNA),实现了对基因组精确定点编辑。除此之外CRISPR/Cas9还有系统操作简单、花费成本低、编辑效率高等优势。目前该技术已在拟南芥、水稻、玉米(Wangetal.,2017;Kleinstiveretal.,2016)等模式植物中实现了精确的定点突变,且正在进一步向更多的植物中实现其应用价值。
1CRISPR/Cas9系统的基本组成结构
一个完整的CRISPR序列组件应该含有重复序列区(repeat)和间隔区(spacer)。间隔区的功能在于外源DNA序列的识别与俘获(Demircietal.,2018)短而保守的重复序列区内存在回文序列,可以形成发卡结构实现对外源基因的降解。序列的上游存在一个启动CRISPR的前导区(Doudna&Charpentier,2014),破坏外源入侵基因时需要在前导区上游的Cas蛋白基因家族的共同作用。目前已经发现了Cas1-Cas10等多种类型的Cas基因。最后CRISPR序列和Cas蛋白基因组合成一个高度保守的CRISPR/Cas9系统(Osakabe&Osakabe,2015;Petolino,2015)。
CRISPR系统分成3个类型:Type1、Type2、Type3。Type1型在细菌和古生菌中均有分布(Puchta&Fauser,2014),Type1型較其他两型相比组成最为复杂,不仅有多个亚型还包含了多种Cas蛋白,最核心蛋白元件为Cas3蛋白,该蛋白是一个含有多结构域的蛋白,但每个结构域都有不同的功能,其主要功能是核酸酶和解旋酶;Type2型的核心蛋白是含有HNH和RuvC两个核酸酶结构域的Cas9蛋白(Panetal.,2016);其中HNH核酸酶结构域在CRISPR系统中可以加工产生crRNA,对外源基因的剪切、降解有着重要作用,目前运用最为广泛操作最为简单的就是Type2型CRISPR系统(Lander,2016);Type3型系统存在于少数细菌和古细菌,Cas6和Cas10蛋白元件包含在该系统中,其主要功能是参与crRNA的加工和入侵DNA的降解。
2CRISPR/Cas9系统的作用机理
Type2型CRISPR-Cas系统只需要三个组件,即Cas9、RNA(tracrRNA)、CRISPRRNA(crRNA)识别和定位该系统病原体的遗传物质通过三步过程,即获得、表达和干扰(Uncklessetal.,2017;Shenetal.,2013)。第一阶段俘获外源DNA:当噬菌体和病毒入侵时,细菌的间隔序列将俘获外源基因的一小片段DNA序列,外源基因与间隔序列识别的序列被称为protospaceradjacentmotif(PAM位点:NGG,N为任意碱基);第二阶段crRNA的表达:将临近PAM的间隔序列修饰加工并整合到自身CRISPR基因座中,转录为Crispr前体RNA(CrisprprecursorRNA,precrRNA),之后进一步形成crRNA(shortCRISPR-derivedRNA,crRNA)(Shenetal.,2014;Samantaetal.,2016);第三阶段靶向干扰:对外源基因的降解除了需要成熟的crRNA之外还需要一些特异性较好的小分子RNA(tracrRNA反式激活RNA)的帮助。crRNA与tracrRNA形成新的复合体RNA,在与Cas9蛋白经sgRNA的引导在PAM位点处与外源DNA互补实现降解(Puchta,2017;Xieetal.,2015)。
3CRISPR/Cas9系统在非模式植物、小作物当中的应用
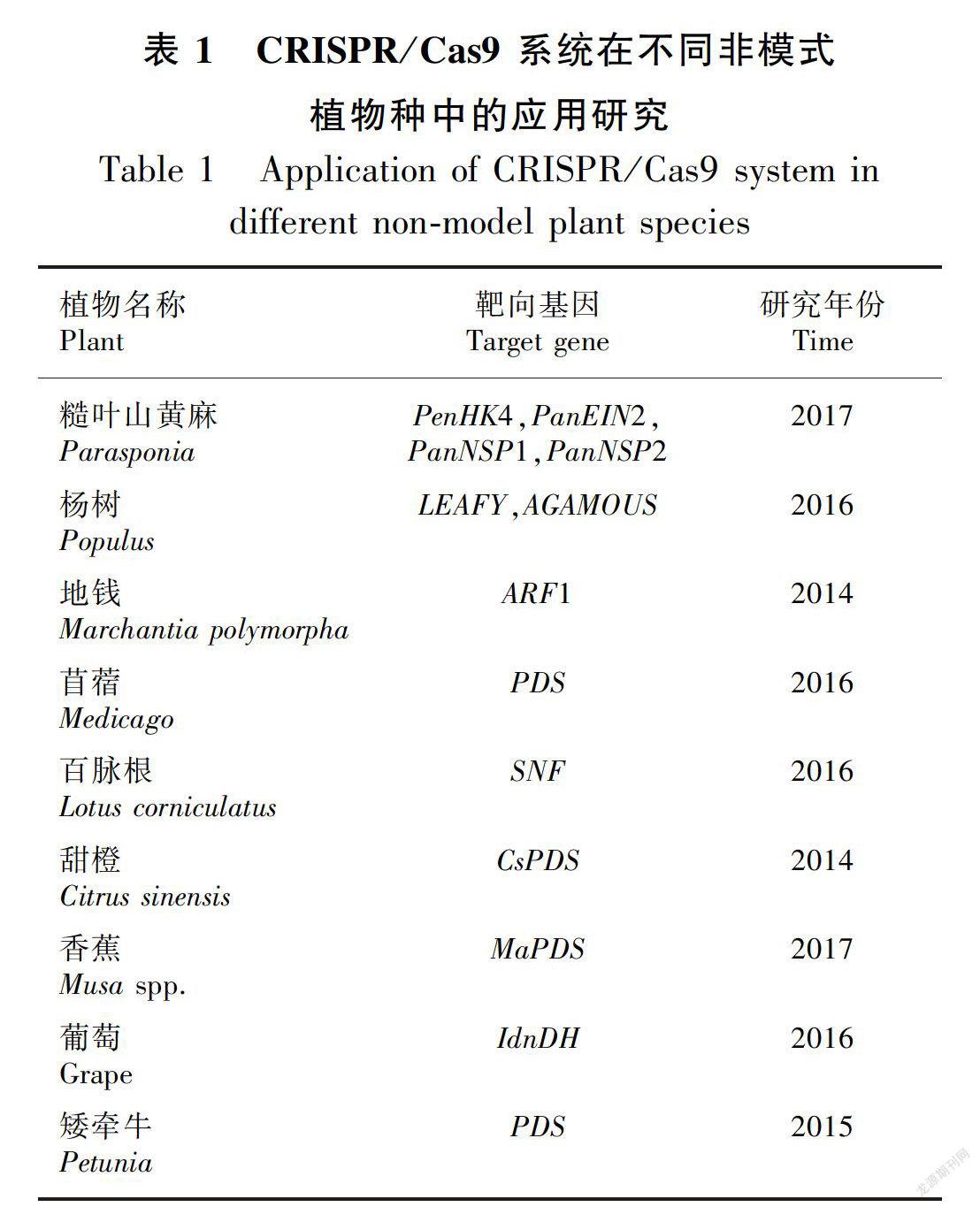
通常情况下非模式植物是指生长周期相对较长、且基因组尚未得到透彻研究的植物。CRISPR/Cas9基因编辑技术通过对基因的移码突变、片段删除及基因修改等功能,成为了植物遗传改良和分子设计的理想途径。CRISPR/Cas9基因编辑技术在更多非模式植物当中的应该不仅提高了生物多样性,而且对于开发更多植物的潜力有着重要意义。该技术已经逐渐在很多非模式植物中实现了基因组定点编辑(表1)。
3.1糙叶山黄麻(Parasponia)属于榆科的热带树种,被称为唯一可以与根瘤菌建立固氮内生真菌的非豆科植物
Vanetal.(2018)通过使用拟南芥AtU6启动子驱动sgRNA,其中Cas9蛋白用35s启动子驱动,使CRISPR/Cas9介导诱变PanHK4、PanEIN2、PanNSP1和PanNSP2四个基因,发现PanNSP1和PanNSP2是根瘤形成的必要条件。
3.2杨树(Populus)是杨属植物,全属有100多种
杨木除了用于制作家具、造纸和火柴之外可广泛用于生态防护林、三北防护林、农林防护林和工业用材林。杨树做为道路绿化,园林景观用也是一个非常优秀的树种。Elorriaga(2016)以pK2GW7为CRISPR/Cas9基因编辑载体定点突变LEAFY与AGAMOUS基因,发现该基因为杨树诱导雄性、雌性的必要基因。
3.3地钱(Marchantiapolymorpha)是苔藓类植物中分布最为广泛的物种之一
Suganoetal.(2014)以生长素调控因子ARF1为靶基因,用U6-1的启动子驱动sgRNA,以及35S启动子驱动的含核定位信号的Cas9蛋白,实现了CRISPR/Cas9在地钱当中的应用。
3.4苜蓿是苜蓿属(Medicago)植物的通称,俗称金花菜,是一种多年生开花植物
Mengetal.(2017)以pFGC5941为CRISPR/Cas9基因编辑载体,以苜蓿U6启动子驱动特定sgRNA,35s启动子驱动Cas9蛋白对蒺藜苜蓿的PDS基因进行定点敲除,并在T0代得到10.35%的纯合敲除突变体,为苜蓿等豆科牧草的功能基因组学研究提供了新的研究工具。
3.5百脉根(Lotuscorniculatus),豆科,属多年生草本植物
百脉根不仅可以作为饲料,还可以用于田地土壤的改良植物。Wangetal.(2016)利用CRISPR/Cas9基因编辑技术,对百脉根共生固氮SNF相关基因进行了编辑研究。
3.6甜橙(Citrussinensis),乔木,枝少刺或近于无刺
橙子中含量丰富的维生素C、P,有着能增加机体抵抗力的作用。Jia&Wang(2014)以pBI121为CRISPR/Cas9基因编辑载体,靶向编辑CsPDS基因,检测突变频率在3.2%~3.9%,为今后利用该技术进行柑橘的品种改良奠定了基础。
3.7香蕉(Musaspp.)是一种重要的热带水果和粮食作物
胡春华等(2017)构建了以MaPDS为靶标基因的pYLCRISPR/Cas9-sgRNA载体。该载体中的Cas9蛋白由PUbi启动子驱动,而sgRNA由水稻来源的U6a驱动,成功的获得了抗性植株。
3.8作为全球最重要的水果作物之一,葡萄(grape)具有巨大的经济价值
Renetal.(2016)以IdnDH为靶基因的pCACRISPR/Cas9二元载体。其中拟南芥U6启动子(AtU6)驱动sgRNA,Cas9蛋白基因又以35s启动子驱动。实现了CRISPR/Cas9系统在葡萄当中的应该最终获得了转基因植株。
3.9矮牵牛(Petunia),碧冬茄属,茄科,正式名碧冬茄,多年生草本植物
广泛用于花坛布置,花槽配置,景点摆设,窗台点缀,家庭装饰。Zhangetal.(2016a)以PGGE为载体,利用CRISPR/Cas9靶向编辑了类胡萝卜素生物合成中的关键酶PDS基因,成功获得了突变体植株。
4目前CRISPR/Cas9系統在非模式植物中的局限性
CRISPR/Cas9系统在模式作物和一些大作物中已经得到了充分的利用,如拟南芥、玉米、水稻、小麦等。但是扩展到非模式植物尤其是一些其开发潜力巨大实用价值有待提高的小作物,如蓖麻、荞麦、丝瓜、荠菜等,还存在着一定的局限性。
4.1非模式植物中CRISPR/Cas9载体构建问题
在进行CRISPR/Cas9系统的载体构建时,与非模式和小作物相比模式植物和大作物的研究较为透彻,可用其专属启动子驱动敲除载体。而大多数非模式植物尤其是小作物只能使用通用的强启动子(拟南芥U6)来驱动CRISPR/Cas9-sgRNA(Fauseretal.,2014;Zhang&Zhou,2014;D′halluink&Ruiter,2013)。经不断的研究一些科研人员构建了一套可以作为双子叶植物或单子叶作物的通用载体(Khatodiaetal.,2017)。这些通用的CRISPR/Cas9载体只需将物种进行单双子叶的区分便可以进行基因编辑。但这些通用载体对植物的适应性及敲除效率是否优于植物的专属敲除载体,其结果有待验证。
4.2CRISPR/Cas9系统在非模式植物中的靶点设计问题
自CRISPR/Cas9系统开创到应用,衍生出了包括人、动物、植物在内的在线和/或单机版共有20多种sgRNA设计软件。如CRISPRDesign、CRISPR-P等软件(Leietal.,2013)。按照操作平台、选择物种、输入序列、参数设置、结果输出等内容,通过制定30多个评测指标来确定靶基因的靶点。但是这些设计软件在智能涵盖的大多数模式植物和大的经济作物,还有很大一部分非模式植物及小作物目前没有相应的sgRNA设计软件。相关研究人员不得不根据常规靶点设计原则:
(1)靶点序列长度18~22bp,一般选择20bp;(2)选择的靶点序列应尽量靠近起始密码子。一般以第一个或第二个外显子为最佳;(3)选择特异性较好的靶点序列相,尽量避免重复序列、多联A序列,多联T序列;GC含量为40%~50%为最佳;(4)靶点5’端允许1~2bp的错配,但应保障靶点碱基匹配数不小于18bp。
为了确保敲除位点的准确性,通过基因外显子核苷酸序列找到PAM位点手动设计多个sgRNA。这为后期靶点检测确定特异性好的sgRNA增加了工作量。
4.3CRISPR/Cas9系统在非模式植物中的基因编辑效率与脱靶评估问题
对确定的靶点进行效率检测和脱靶评估时模式植物除了用实验方法外。如酶切法、SMART测序法、SSA报告载体活性检测法、Sanger测序法等(Yangetal.,2013;Hendeletal.,2014;Yinetal.,2015)。以综合各类脱靶检测方法为基础,衍生出了定量分析基因敲除效率的相关软件,如CRISPR-GA(CRISPRGenomeAnalyzer,http://54.80.152.219/)。目前非模式植物只能通过体外转录sgRNA及体外酶切靶点DNA片段反应通过与已知活性的标准sgRNA靶点进行比较来评价目标sgrna的特异性(Cradicketal.,2014;Sunetal.,2015)。此活性测定虽然不难但是过程较为繁琐加上需做重复试验来最终确定活性较高的sgRNA,耗时耗力。
4.4CRISPR/Cas9系统在非植物中的重组效率问题
重组效率低是CRISPR/Cas9系统在模式植物和在非模式植物的通病,常规CRISPR使用与核酸酶(最常见的是Cas9)偶联的指导RNA(gRNA),其一起连接到特定的DNA碱基区域;然后核酸酶剪切双螺旋(Wangetal.,2015)。细胞修复机制尝试重新加入切割的DNA末端,但偶尔会插入或删除一些碱基,这将使DNA代码变成乱码,并且可能误敲除靶基因。为了修复点突变,CRISPR/Cas9系统还必须引入具有正确碱基的“供体”DNA链,然后依靠称为同源性定向修复(HDR)的第二种细胞机制。但是此修复模式依赖于细胞分裂状态,细胞分裂差HDR的作用也就很差。
4.5CRISPR/Cas9系统在非模式植物中的遗传转化问题
利用遗传转化得到转基因植株,重点在于转化效率。目前相关研究表明转化效率与载体大小成反比。CRISPR/Cas9基因编辑系统中,通常Cas9蛋白原件大于5kb,所以遗传转化效率在一定程度会受到影响。虽然一些研究发现通过以连续转化的方式(即首先得到hCas9转基因植株,之后将卸载Cas9的小型sgRNA质粒转化至hCas9植株中)得到了与直接转化CRISPR/Cas9同样的突变体植株,但此方法多适用于以叶片为受体的遗传转化系统,且hCas9基因整合至植物基因组中有无毒害作用目前尚未可知(Lowderetal.,2015;Mikamietal.,2015)。
4.6单碱基基因编辑系统在非模式植物中的应用问题
CRISPR/Cas9单碱基编辑系统的应用实现了DNA雙链不发生断裂的情况下,通过sgRNA靶向定位,利用胞嘧啶核苷脱氨酶进行碱基编辑(魏瑜等,2017)。这项技术的成功纠正效率为23%~35%。目前该系统已经在模式植物中得到应用。此技术的不足之处在于,该系统只能实现单个碱基C→T或G→A的编辑。单碱基编辑系统的活性窗口仍然较大。单碱基编辑系统仍然会导致靶位点产生极少量DNA序列插入或缺失。
5提高CRISPR/Cas9系统在非模式植物中的应用效率相关策略
5.1完善非模式植物全基因组测序
全基因组测序的目的是获得物种的基因组序列,它可以研究基因组水平上物种的生长、发育、进化和起源加深对物种的了解,并在发现新基因和改进改良育种中发挥重要作用。CRISPR/Cas9系统用于基因组编辑研究,可以全面挖掘基因功能获得重要的遗传信息(Zhangetal.,2016b)。许多非模式植物,在各种极端生境中具有广泛的生态适应性和生长能力,CRISPR/Cas9技术的应用将促进在极端生境中生长的一些特殊性状基因的探索,如耐旱基因、耐盐基因、耐冷基因等。此外,CRISPR/Cas9系统的应用加速了探索基因代谢途径和转录调控机制,并可以通过基因敲除和插入突变,以实现对基因的改造。
5.2sgRNA和ssODNs的设计
在CRISPR/Cas9基因组编辑技术中,Cas9蛋白在特定靶位点上的裂解主要是由PAM序列和单链RNA(sgRNA)中的20nt序列决定的。因此,sgRNA序列的选择具有靶向性,直接决定了Cas9蛋白核酸酶的定位位置。首先,必须有PAM序列,其中PAM序列是NGG(N是任意碱基)序列。其次,为了减少切割过程中Cas9蛋白的缺失率,sgRNA序列应该与基因组上的靶序列高度匹配(Cencicetal.,2014)。此外,sgRNA与基因组其它位点的同源性最差,否则很容易导致错配。最后在设计sgRNA时将其长度从20nt缩短到17nt,有助于降低错配几率。
在CRISPR/Cas9基因组编辑技术中,sgRNA的设计和选择非常重要。但在定点修改中,同源修复模板(ssODNS)的设计也是一个关键问题。两种不同的DNA修复模式(HR和NHEJ)在双链断裂后被诱导。两种DNA修复方法的区别在于HR需要1个同源修复模板,即短链DNA,即单链寡核苷酸(ssODNs)。使用该模板的HR可以进行精确的点突变、插入和敲除DNA。当在靶位点引入1个大片段DNA序列时,修复模板也可以是含有同源臂的1个双链质粒。近年来,ssODNs替代了双链质粒在基因组位点修饰中的应用。为了提高修复效率,ssODN包括和靶向序列互补的,分别沿着至少3′方向和5′方向延伸至少40nt的序列(Ranetal.,2013)。在此基础上最好引入酶切位点,这样就可以用限制性酶切多态性实验(RFLP),很方便的检测基因编辑系统是否有效。
5.3Cas9蛋白的改造
由于CRISPR/Cas9技術的发展,研究人员除了开发野生型SpCas9系统之外,还开发了新成员。在2015年,张峰团队发现了一种更简单的核酸酶(-Cpf1),它比Cas9更简单它可以单独完成crRNA的加工和成熟,而不依赖于核糖核酸酶(RNase)和tracrRNA。-Cpf1不仅可以切割DNA序列,而且可以作用于RNA,并且没有脱靶现象(Kimetal.,2016)。同年,张峰研究团队还发现了1个新的蛋白质C2C2,仅针对细菌中特定的RNA序列进行切割。这些新发现可以为研究者解决未来的问题提供便利。
6展望
CRISPR/Cas9技术自2013年问世以来,已经迅速的普及到多种植物的遗传育种当中,相对于前两代TALENs\\ZFNs的基因编辑技术而言,敲除效率高、操作更加简便、耗材少,适用于很多实验室。在植物领域CRISPR/Cas9系统基因组编辑主要应用于模式植物和部分大作物,但要实现技术的应用普及至更多的非模式植物和小作物中,在未来需要进一步改进和完善。(1)特异性基因的发掘。虽然目前许多模式植物,尤其是大作物的全基因组测序工作已经完成。但是,很多非模式植物的基因组序列还尚未明确,许多关于重要性状的基因还有待鉴定,一定程度上阻碍了CRSPR/Cas9技术在植物基因工程育种中的应用。(2)载体构建效率有待提高。目前在非模式植物中,sgRNA的设计主要通过研究人员对植物基因外显子手动设计,且对sgRNA的特异性还要经过试验进一步验证确定,严重影响后期的敲除效率,在没有相关辅助软件的情况下,费时费力。(3)非模式遗传转化体系的建立。目前,还有许多非模式植物由于没有有效遗传转化体系难以应用CRISPR/Cas9基因编辑技术,加之该系统载体相对较大,对实现转化效率也有一定的影响。(4)HDR发生频率的提高。目前CRISPR/Cas9技术的研究主要是通过敲除靶位点,获得转基因植株。所以如何通过改造CRISPR/Cas9系统,将HDR在基因组靶位点引入相关功能基因,将成为CRISPR/Cas9技术在植物基因工程改良工作中的重点突破方向。相信随着CRISPR/Cas9技术的进一步发展这些问题终将被克服,它的出现必将给植物基因工程带来更好的发展。
参考文献:
BIBIKOVAM,GOLICM,GOLICKG,etal.,2002.TargetedchromosomalcleavageandmutagenesisinDrosophilausingzinc-fingernucleases[J].Genetics,161(3):1169-1175.
BOCHJ,BONASU,2010.XanthomonasAvrBs3family-typeIIIeffectors:Discoveryandfunction[J].AnnRevPhytopathol,48:419-436.
CENCICR,MIURAH,MALINAA,etal.,2014.Protospaceradjacentmotif(PAM)-distalsequencesengageCRISPRCas9DNAtargetcleavage[J].PLoSONE,9(10):e109213.
CRADICKTJ,ANTICOCJ,BAOG,2014.High-throughputcellularscreeningofengineerednucleaseactivityusingthesingle-strandannealingassayandluciferasereporter[J].MethodsMolBiol,(1114):339-352.
DEMIRCIY,ZHANGB,UNVERT,2018.CRISPR/Cas9:AnRNA-guidedhighlyprecisesynthetictoolforplantgenomeediting[J].CellPhysiol,233(3):32-36.
D′HALLUINK,RUITERR,2013.Directedgenomeengineeringforgenomeoptimization[J].IntJDevBiol,57(6-8):621-627.
DOUDNAJA,CHARPENTIERE,2014.Genomeediting:ThenewfrontierofgenomeengineeringwithCRISPR-Cas9[J].Science,346(6213):1258096.
ELORRIAGAE,2016.Asexualgenedriveinpopulus?ResultsfromCRISPR/Cas9mutagenesisoffloralgenesforgeneticcontainment[C].PAGXXVI,SanDiego,CA.
EYQUEMJ,MANSILLA-SOTOJ,GIAVRIDIST,etal.,2017.TargetingaCARtotheTRAClocuswithCRISPR/Cas9enhancestumourrejection[J].Nature,543(7643):113.
FAUSERF,SCHIMLS,PUCHTAH,2014.BothCRISPR/Cas-basednucleasesandnickasescanbeusedefficientlyforgenomeengineeringinArabidopsisthaliana[J].PlantJ,79(2):348-359.
HENDELA,KILDEBECKEJ,FineEJ,etal.,2014.Quantifyinggenome-editingoutcomesatendogenouslociwithSMRTsequencing[J].CellRep,7(1):293.
HUCH,DENGGM,SUNXX,etal.,2017.EstablishmentofanefficientCRISPR/Cas9-mediatedgeneeditingsysteminbanana[J].SciAgricSin,50(7):1294-1301.[胡春華,邓贵明,孙晓玄,等,2017.香蕉CRISPR/Cas9基因编辑技术体系的建立[J].中国农业科学,50(7):1294-1301.]
JIAH,WANGN,2014.TargetedgenomeeditingofsweetorangeusingCas9/sgRNA[J].PLoSONE,9(4):e93806.
KHATODIAS,BHATOTIAK,TUTEJAN,2017.DevelopmentofCRISPR/Cas9mediatedvirusresistanceinagriculturallyimportantcrops[J].Bioengineered,8(3):274-279.
KIMD,KIMJ,HURJK,etal.,2016.Genome-wideanalysisrevealsspecificitiesofCpf1endonucleasesinhumancells[J].NatBiotechnol,34(8):863-868.
KLEINSTIVERBP,PATTANAYAKV,PREWMS,etal.,2016.High-fidelityCRISPR-Cas9nucleaseswithnodetectablegenome-wideoff-targeteffects[J].Nature,529(7587):490-495.
LANDERES,2016.TheheroesofCRISPR[J].Cell,164(2):18-28.
LEIY,LUL,LIUHY,etal.,2014.CRISPR-P:Awebtoolforsyntheticsingle-guideRNAdesignofCRISPR-systeminplants[J].MolPlant,7(9):1494-1496.
LOWDERLG,ZHANGD,BALTESNJ,etal.,2015.ACRISPR/Cas9toolboxformultiplexedplantgenomeeditingandtranscriptionalregulation[J].PlantPhysiol,169(2):971-985.
MENGY,HOUY,WANGH,etal.,2017.Targetedmutage-nesisbyCRISPR/Cas9systeminthemodellegumeMedicagotruncatula[J].PlantCellRep,36(2):371-374.
MIKAMIM,TOKIS,ENDOM,2015.ComparisonofCRISPR/Cas9expressionconstructsforefficienttargetedmutagenesisinrice[J].PlantMolBiol,88(6):561-572.
OSAKABEY,OSAKABEK,2015.Genomeeditingwithengineerednucleasesinplants[J].PlantCellPhysiol,56(3):389-400.
PANC,YEL,QINL,etal.,2016.CRISPR/Cas9-mediatedefficientandheritabletargetedmutagenesisintomatoplantsinthefirstandlatergenerations[J].SciRep,78(6):46916.
PETOLINOJF,2015.Genomeeditinginplantsviadesignedzincfingernucleases[J].InVitroCellDevBiolPlant,51(1):1-8.
PUCHTAH,FAUSERF,2014.Syntheticnucleasesforgenomeengineeringinplants:Prospectsforabrightfuture[J].PlantJ,78(5):727-741.
PUCHTAH,2017.ApplyingCRISPR/Casforgenomeengineeringinplants:thebestisyettocome[J].CurrOpinPlantBiol,25(36):1-8.
RANFA,HSUPD,WRIGHTJ,etal.,2013.GenomeengineeringusingtheCRISPR-Cas9system[J].NatProtoc,8(11):2281-2308.
RENC,LIUXJ,ZHANGZ,etal.,2016.CRISPR/Cas9-mediatedefficienttargetedmutagenesisinChardonnay(VitisviniferaL.)[J].SciRep,(6):32289.
SAMANTAMK,DEYA,GAYENS,2016.CRISPR/Cas9:Anadvancedtoolforeditingplantgenomes[J].TransgRes,25(5):561-573.
SANJANANE,CONGL,ZHOUY,etal.,2012.Atranscriptionactivator-likeeffectortoolboxforgenomeengineering[J].NatProtoc,7(1):171-192.
SHENB,ZHANGJ,WUHY,etal.,2013.Generationofgene-modifiedmiceviaCas9/RNA-mediatedgenetargeting[J].CellRes,23(5):720-723.
SHENB,ZHANGW,ZHANGJ,etal.,2014.EfficientgenomemodificationbyCRISPR-Cas9nickasewithminimaloff-targeteffects[J].NatMethods,11(4):399-402.
SUGANOSS,SHIRAKAWAM,TAKAGIJT,etal.,2014.CRISPR/Cas9-mediatedtargetedmutagenesisintheliverwortMarchantiapolymorphaL.[J].PlantCellPhysiol,55(3):475-481.
SUNX,HUZ,CHENR,etal.,2015.TargetedmutagenesisinsoybeanusingtheCRISPR-Cas9system[J].SciRep,(5):10342.
SUNYG,ZHANGX,WUCY,etal.,2016.Engineeringherbicide-resistantriceplantsthroughCRISPR/Cas9-mediatedhomologousrecombinationofacetolactatesynthase[J].MolPlant,9(4):628-631.
SVITASHEVS,YOUNGJK,SCHWARTZC,etal.,2015.Targetedmutagenesis,precisegeneediting,andsite-specificgeneinsertioninmaizeusingCas9andguideRNA[J].PlantPhysiol,169(2):931-945.
UNCKLESSRL,CLARKAG,MESSERPW,2017.EvolutionofresistanceagainstCRISPR/Cas9genedrive[J].Genetics,205(2):827.
VANZEIJLA,WARDHANITAK,SEIFIKALHORM,etal.,2018.CRISPR/Cas9-mediatedmutagenesisoffourputativesymbiosisgenesofthetropicaltreeParasponiaandersoniirevealsnovelphenotypes[J].FrontPlantSci,(9):284.
WANGLX,WANGLL,TANQ,etal.,2016.EfficientinactivationofsymbioticnitrogenfixationrelatedgenesinlotusjaponicasusingCRISPR-Cas9[J].FrontPlantSci,7(703).
WANGMG,LUYM,BOTELLAJR,etal.,2017.Genetargetingbyhomology-directedrepairinriceusingageminivirus-basedCRISPR/Cas9system[J].MolPlant,10(7):1007-1010.
WANGS,ZHANGS,WANGW,etal.,2015.EfficienttargetedmutagenesisinpotatobytheCRISPR/Cas9system[J].PlantCellRep,1473-1476(9):10342.
WEIY,ZHANGXH,LIDL,2017.The“newfavorite”ofgeneeditingtechnology-singlebaseeditors[J].Hereditas,39(12):1115-1121.[魏瑜,張晓辉,李大力,2017.基因编辑之“新宠”—单碱基基因组编辑系统[J].遗传,39(12):1115-1121.]
XIEK,MINKENBERGB,YANGY,2015.BoostingCRISPR/Cas9multiplexeditingcapabilitywiththeendogenoustRNA-processingsystem[J].ProcNatlAcadSciUSA,112(11):3570-3575.
YANGH,WANGHY,SHIVALILACS,etal.,2013.One-stepgenerationofmicecarryingreporterandconditionalallelesbyCRISPR/Cas-mediatedgenomeengineering[J].Cell,154(6):1370-1379.
YINKQ,HANT,LIUG,etal.,2015.Ageminivirus-basedguideRNAdeliverysystemforCRISPR/Cas9mediatedplantgenomeediting[J].SciRep,7(5):14926.
ZHANGB,YANGX,YANGC,etal.,2016a.ExploitingtheCRISPR/Cas9systemfortargetedgenomemutagenesisinPetunia[J].SciRep,(6):20315.
ZHANGD,LIZ,LIJF,2016b.TargetedgenemanipulationinplantsusingtheCRISPR/Castechnology[J].JGenetGenom,43(5):251-262.
ZHANGL,ZHOUQ,2014.CRISPR/Castechnology:Arevolutionaryapproachforgenomeengineering[J].SciChinLifeSci,57(6):639-640.

