江苏省多基因聚合对水稻稻瘟病抗性的效应分析及Pb1基因功能标记开发
孙立亭 林添资 景德道 余波 钱华飞 曾生元 李闯 姚维成 杜灿灿 胡庆峰 周义文 龚红兵







摘要:【目的】研究多基因聚合對水稻稻瘟病抗性的效应,并开发Pb1基因功能标记,为江苏省培育持久抗稻瘟病品种及提高其育种效率提供理论依据。【方法】开发Pb1基因的功能标记,并结合Pita、Pib、Pi54、Pikm和Pizt功能标记检测2015─2017年参加江苏省预试的703份常规粳稻材料的抗病基因,分析其聚合方式与稻瘟病抗性的相关性。【结果】在Pb1基因编码区上游926~1085 bp设计1个存在/缺失标记M1,其引物可扩增获得160 bp的片段。703份供试材料中,Pita、Pib和Pi54基因频率高于Pikm、Pb1和Pizt,虽然抗病频率(稻瘟病综合指数≤5.0的材料所占比例)在2015─2017年呈逐年快速升高趋势,但3年间达中抗及以上等级的材料所占比例均较低。不含抗病基因的材料11份,综合指数≤5.0的材料4份,抗病频率为36.36%;当所含抗病基因数≤3个时,随着含抗病基因数的增加,对应的材料数量、出现频率和综合指数≤5.0的材料数量均明显增加,当所含抗病基因数≥4个时,随着含抗病基因数的增加,对应的材料数量、出现频率和综合指数≤5.0的材料数量均明显降低。抗病基因数越多,抗病频率越高,当抗病基因数达6个时,抗病频率达100.00%,说明聚合的抗病基因数与抗病频率呈正相关。3个抗病基因聚合的最佳方式为:Pi54+Pib+Pb1或Pita+Pib+Pikm。4个抗病基因聚合的最佳方式为Pita+Pib+Pikm+Pizt或Pita+Pi54+Pib+Pb1。【结论】开发的Pb1基因功能标记可应用于水稻稻瘟病抗性育种,以提高选择效率。多基因聚合能提高水稻稻瘟病抗性,但抗性强弱取决于抗性基因间的互作效应,并不是简单的累加效应。
关键词: 稻瘟病;多基因聚合;分子标记;抗性育种
中图分类号: S511.220.32 文献标志码: A 文章编号:2095-1191(2019)05-0913-11
Abstract:【Objective】Studying the effect of multiple genes polymerization on rice blast resistance could guide bree-ding rice blast resistant varieties and improve breeding efficiency in Jiangsu Province. 【Method】Functional markers of Pb1 was developed, and resistance genes of 703 japonica rices for preliminary experiment in the year of 2015,2016 and 2017 in Jiangsu were detected using functional markers of Pita,Pib,Pi54,Pikm and Pizt,then the correlation between polymerization mode and rice blast resistance was analyzed. 【Result】A presence/deletion marker M1 was designed near 1016 bp upstream of the coding region of Pb1. Its primer could amplify 160 bp fragment. Among the 703 materials, the gene frequency of Pita,Pib and Pi54 was higher than that of Pikm,Pb1 and Pizt. Although the frequency of rice blast resistance (the proportion of materials with composite index of blast ≤5.0) increased rapidly year by year from 2015 to 2017,the moderate resistance and above level ratios were low during the three years. Among the 703 materials tested,11 did not contain resistance genes,of which 4 materials had a composite index ≤5.0 and the resistance frequency of 36.36%. When the number of disease-resistant genes was ≤3,the number of materials,frequency of occurrence and the number of materials whose composite index was ≤5.0 increased greatly with the increase of the number of disease resistant genes. When the number of disease resistant genes was ≥4,the number of materials,the frequency of gene occurrence and the number of materials whose composite index was ≤5.0 decreased greatly with the increase of the number of disease resistant genes.The higher the number of resistance genes,the higher the resistance frequency. When the number of resistance genes reached 6,the resistance frequency reached 100.00%. This indicated that the number of multiple genes polymerization was positively correlated with the resistance frequency. The optimal polymerization mode for three genes was Pi54+Pib+Pb1 or Pita+Pib+Pikm. The optimal polymerization mode for four genes was Pita+Pib+Pikm+Pizt or Pita+Pi54+Pib+Pb1. 【Conclusion】The developed functional markers of Pb1 can be used in rice blast resistance breeding to improve selection efficiency. Polygenic polymerization can improve the resistance level of rice blast,but the resistance depends on the interaction between resistant genes,not simply the cumulative effect.
Key words: rice blast; multiple genes polymerization; molecular marker; resistance breeding
0 引言
【研究意义】稻瘟病是由子囊菌(Magnaporthe oryzae)引发的一种严重的水稻病害,对水稻产量产生严重影响(Khush and Jena,2009;Skamnioti and Gurr,2009;刘万才等,2016)。长期实践证明,培育抗病品种是最环保、最经济有效的方法(Koide et al.,2009;Miah et al.,2013)。水稻对稻瘟病的抗性可分为水平抗性和垂直抗性,其中垂直抗性具有很强的小种专化性,易随小种的变化而丧失,尤其是含有单个主效抗病基因的品种(Mackill and Bonman,1992;孙大元等,2017)。水稻聚合多个抗病基因后其植株抗谱和抗性均得到提高,是获得持久抗性的有效方法之一(Hittalmani et al.,2000)。因此,分析我国江苏省多基因聚合对稻瘟病抗性的效应,对改良现有品种的稻瘟病抗性具有重要意义。【前人研究进展】随着分子生物学和基因定位技术的发展,水稻稻瘟病抗性基因的研究取得长足进展。为开展高效的分子育种,对分子标记进行筛选即能实现性状筛选,其原因是一个分子标记位点代表一个特定的等位基因,并与该基因控制的性状相联系,水稻抗稻瘟病基因的克隆为开发基因功能标记提供了基础和前提(Andersen and Lǖbberstedt,2003)。目前,成功克隆的稻瘟病抗性基因有Pib(Wang et al.,1999)、Pita(Bryan et al.,2000)、Pi5(Jeon et al.,2003)、Pid2(Chen et al.,2006)、Piz-t(Zhou et al.,2006)、Pi2(Zhou et al.,2006)、Pi9(Qu et al.,2006)、Pi37(Lin et al.,2007)、Pi21(Fukuoka et al.,2009)、Pid3(Shang et al.,2009)、Pb1(Hayashi et al.,2010b)、Pit(Hayashi et al.,2010a)、Pish(Takahashi et al.,2010)、Pi2(Chen et al.,2011)、Pia(Okuyama et al.,2011)、Pik-p(Yuan et al.,2011)、Pikm(Zhai et al.,2011)、Pik(Ashikawa et al.,2012)、Pik-h/Pi54(Das et al.,2012)、Pi1(Hua et al.,2012)、Pi56(Liu et al.,2013)、Pi63(Xu et al.,2014)和Pigm(Deng et al.,2017)等。其中Pita基因位于第12条染色体上,编码细胞质膜受体蛋白,该蛋白与稻瘟病菌的无毒基因AVR-Pita表达产物相互作用引发抗病反应(Bryan et al.,2000);Pib基因定位于第2条染色体上,对日本大多数稻瘟病菌小种有抗性,对我国的菌株ZB13和ZC15也有抗病反应(Wang et al.,1999);pik座位上有5个等位基因,分别为Pi-l、Pik-h、Pi54、Pikm和Pikp基因,其中,Pi54基因属于抗病基因的CC-NBS-LRR家族,对稻瘟病菌具有广谱抗性(Das et al.,2012),Pikm是由两个紧密连锁的具有独立功能NBS-LRR类基因(Pikm1-TS和Pikm2-TS)组成(Ashikawa et al.,2008);Pizt基因位于第6条染色体上的Piz位点,属于NBS-LRR类基因,其编码1033个氨基酸,含1个核苷酸结合位点(NBS)和3个富亮氨酸重复区(LRR)(Zhou et al.,2006)。大量研究表明,聚合多个抗性基因是培育广谱、持久抗性品种的一条有效途径。范方军等(2014)对2012年审定的江苏水稻品种进行抗病基因检测,结果发现绝大多数水稻品种含有稻瘟病抗性基因Pita、Pib、Pi54和Pikm。刘斌等(2018)研究发现,云南省56个主栽水稻品种中Pita2、pi5、Pi9和Pizt基因出现频率较高,且对稻瘟病抗性良好。张善磊等(2018)研究发现,國内外238个粳稻品种(系)中同时含有Pita、Pi5和Pikm基因的品种(系)表现出较强的穗颈瘟抗性。黄乾龙等(2019)研究发现,重庆自育骨干亲本携带抗病基因及聚合方式不同,稻瘟病抗性均存在差异。【本研究切入点】目前,稻瘟病抗性基因Pita、Pib、Pi54、Pikm、Pb1和Pizt等在江苏省的聚合效应仍不清楚。因此,有必要对江苏省常规粳稻的抗病基因与抗性效应进行分析,总结基因聚合模式与稻瘟病抗性间的关系,并根据基因型和抗病性制定育种策略。【拟解决的关键问题】通过开发Pb1基因功能标记,结合前人已开发的Pita、Pib、Pi54、Pikm和Pizt基因功能标记,对2015—2017年江苏省703个常规粳稻预试品种(系)进行抗病基因检测,并测定其稻瘟病抗性,分析抗病基因聚合方式与稻瘟病抗性的相关性,为江苏省抗病品种(系)的合理布局、培育持久抗病品种(系)及提高育种效率提供理论依据。
1 材料与方法
1. 1 试验材料
供试材料为2015─2017年参加江苏省预试的703常规粳稻材料。其中,2015年参加江苏省预试的材料222份,包括中熟中粳79份、迟熟中粳93份、早熟晚粳50份;2016年参加江苏省预试的材料221份,包括中熟中粳83份、迟熟中粳89份、早熟晚粳49份;2017年参加江苏省预试的材料260份,包括中熟中粳92份、迟熟中粳104份、早熟晚粳64份,均由江苏省农业科学院提供。引物由南京金斯瑞生物科技有限公司合成;Taq DNA聚合酶和dNTP购自上海捷瑞生物工程有限公司。主要仪器设备:PCR扩增仪(Biometra TProfessional PCR)、电泳槽(DYCZ-30C)、电泳仪(BIO-RAD)和高通量组织研磨仪(DHS TL2020)。
1. 2 Pb1基因功能标记开发与验证
Hayashi等(2010b)对日本品种St. No1中的Pb1和P5基因进行克隆及分析,结果发现Pb1位于一个以60 kb为单位的串联重复序列中,且Pb1位于第二个重复基序中,与第一个重复基序的P5相对应,Pb1与P5均具有穗瘟抗性,但Pb1的穗瘟抗性高于P5,两个基因的抗穗瘟差异取决于Pb1基因编码区上游1~2056 bp的特有序列。基于上述研究结果,通过NCBI搜索获得Pb1基因(登录号AB570371.1)和P5基因(登录号LOC_Os11g38480)及感病品种日本晴等位基因Os11g0598500(登录号LOC_Os11g38580)。将P5基因编码区上游序列和日本晴等位基因Os11g0598500Pb1与Pb1基因编码区上游序列进行序列比对,寻找差异位点,利用Primer 5.0设计特异引物,并利用镇稻88、镇稻18号和日本晴进行验证。
1. 3 PCR扩增
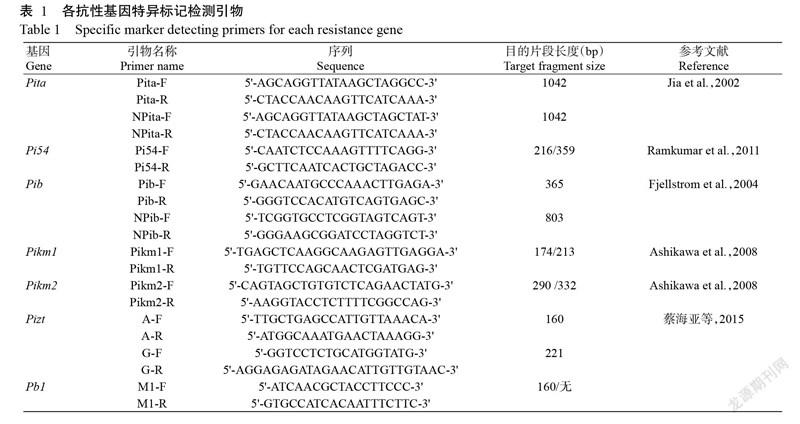
水稻基因组DNA提取采用SDS法(孙立亭等,2017)。根据等位基因PCR扩增原理,针对Pita、Pi54、Pib、Pikm、Pb1和Pizt抗感等位基因序列的差异设计特异引物(表1)。以基因组DNA为模板,PCR克隆稻瘟病抗性基因标记。反应体系10.0 μL:1.2 μL 10×Taq Buffer,50 ng/μL基因组DNA 1.5 μL,2 mmol/L上、下游引物各0.5 μL,1 mmol/L dNTP 0.3 μL,1000 U Taq DNA聚合酶0.1 μL,ddH2O补足至10.0 μL。扩增程序:95 ℃预变性5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 40 s,进行33个循环;72 ℃延伸10 min。Pita和Pib基因标记的扩增产物用4%聚丙烯酰胺凝胶电泳进行检测,其他基因标记的扩增产物用8%聚丙烯酰胺凝胶电泳进行检测。取PCR产物2.0 μL,以100 bp-ⅠDNA Ladder作为DNA Marker,在240 V恒压下电泳1 h。最后,采用银染法显示DNA条带。
1. 4 抗病基因检测
根据片段大小判断抗病基因功能标记。利用Pita-F/Pita-R引物可扩增出1042 bp条带,同时NPita-F/NPita-R引物扩增不出条带,则判定为该材料含有Pita基因。利用Pib-F/Pib-R引物可扩增出365 bp条带,同时NPib-F/NPib-R扩增不出条带,则判定为该材料含Pib基因。利用A-F/A-R引物可扩增出160 bp条带,同时G-F/G-R引物扩增不出条带,则该材料含Pizt基因。利用Pi54-F/Pi54-R引物扩增,若扩增片段为216 bp,则判定为该材料含Pi54基因,若扩增片段为359 bp,则判定为该材料不含Pi54基因。利用Pikm由Pikm1-F/Pikm1-R和Pikm2-F/Pikm2-R两对引物进行扩增,若Pikm1-F/Pikm1-R扩增片段为174 bp,则判定该材料含Pikm1基因,若扩增片段为213 bp则判定不含Pikm1基因;若Pikm2-F/Pikm2-R扩增片段为290 bp,则判定该材料含Pikm2基因,若扩增片段为332 bp,则判定该材料不含Pikm2基因。利用M1-F/M1-R引物扩增,若扩增片段为160 bp,则判定该材料含Pb1基因,若无条带,则判定该材料不含Pb1基因。
1. 5 稻瘟病抗性鉴定与抗病频率评价
供试材料的抗性鉴定综合指数评价分级标准如表2所示。鉴定结果参考《江苏省水稻新品种中间试验总结汇编(2015─2017)》江苏省种子管理站,2015,2016,2017)。综合指数=(叶瘟病级×25%+穗瘟发病率病级×25%+穗瘟损失指数病级×50%。
2 结果与分析
2. 1 稻瘟病抗性基因Pb1功能标记的开发及验证结果
通过比对Pb1和P5基因的编码区上游1~1016 bp序列,结果发现,Pb1基因在356 bp处比P5基因多17 bp,在1 bp处比P5基因少17 bp,但日本晴等位基因Os11g0598500不含此段序列(圖1)。通过比对Pb1基因编码区上游1017~2056 bp与日本晴等位基因Os11g0598500序列,结果发现Pb1基因与日本晴基因组序列存在31处单碱基替换,其他序列完全匹配(图2)。
本研究在Pb1基因编码区上游1016 bp附近设计1对存在/缺失标记M1(表1),该标记具体位于Pb1基因编码区上游926~1085 bp,其引物可扩增160 bp片段,若材料不含Pb1基因则扩增不出该片段,如图3所示镇稻88和镇稻18号(抗稻瘟病)均能扩增出160 bp的片段,而日本晴扩增不出对应的片段。Pb1基因的供体为Modan,而镇稻88含有Modan血缘且抗稻瘟病,测序结果表明,镇稻88中含有Pb1基因(图4和图5)。说明该功能标记可特异性检测Pb1基因。
2. 2 抗性基因检测及其分布情况
利用Pita、Pib、Pi54、Pikm、Pizt和Pb1等6个抗性基因的功能标记对703份供试材料进行抗性基因检测,结果发现含Pita、Pi54、Pib、Pikm、Pb1和Pizt基因的材料比例在2015─2017年发生明显变化,2015年分别为51.8%、40.1%、75.2%、20.7%、23.0%和22.1%,2016年分别为62.4%、53.4%、83.7%、24.9%、31.7%和27.1%,2017年分别为69.6%、54.6%、83.5%、18.8%、26.9%和22.3%(图6)。可见,2015—2017年江苏省预试材料中Pita、Pib和Pi54基因频率高于Pikm、Pb1和Pizt。
2. 3 稻瘟病的抗性鉴定结果
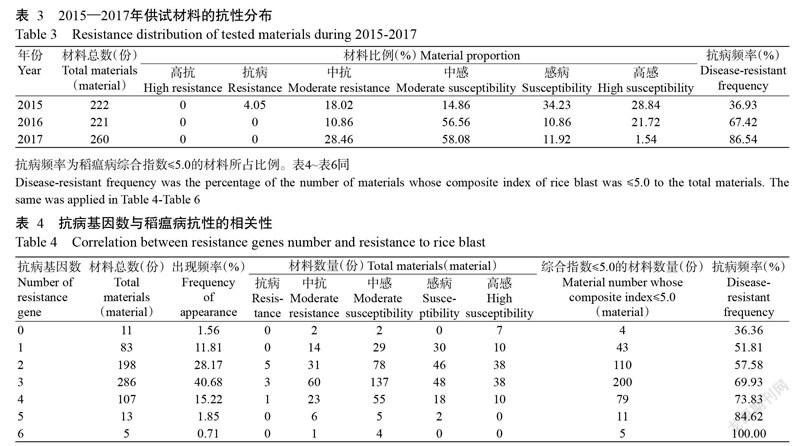
由表3可知,2015─2017年未鉴定出高抗材料,2015年抗病材料比例为4.05%,2016和2017年抗病材料均为0,2017年中抗材料比例(28.46%)明显高于2015年(18.02%)和2016年(10.86%),2015─2017年中感材料比例逐年升高,但感病和高感材料比例有所降低。整体来看,虽然抗病频率在2015─2017年呈逐年快速升高趋势,但3年間达中抗及以上等级的材料所占比例均偏低。
2. 4 抗病基因与综合病指的相关性分析结果
江苏省品种审定对稻瘟病的抗性要求综合指数≤5.0,因此,以综合指数5.0为界限研究抗病基因与稻瘟病抗性的关系。由表4可知,不含抗病基因的材料11份,综合指数≤5.0的材料4份,抗病频率为36.36%。当所含抗病基因数≤3个时,随着含抗病基因数的增加,对应的材料数量、出现频率和综合指数≤5.0的材料数量均明显增加;当所含抗病基因数≥4个时,随着抗病基因数的增加,对应的材料数量、出现频率和综合指数≤5.0的材料数量均明显降低。抗病基因数越多,抗病频率越高,当抗病基因数达3个时,抗病频率达69.93%,当抗病基因数达6个时,抗病频率达100.00%,表明抗病基因数与抗病频率呈正相关。703份供试材料中,含有5和6个抗病基因的出现频率较低,仅为1.85%和0.71%,因此本研究进一步分析3和4个抗病基因聚合方式与稻瘟病抗性的相关性。
2. 5 3个抗病基因的聚合效应分析结果
3个抗病基因的聚合效应如表5所示。703份供试材料中含有3个抗病基因的材料286份,其中稻瘟病综合指数≤5.0的材料有200份,抗病频率为69.93%。不同抗病基因聚合方式的抗病频率存在明显差异,其中抗病频率大于69.93%的聚合方式有7种,从高到低依次为:Pita+Pikm+Pizt=Pita+Pb1+Pizt=Pi54+Pib+Pb1>Pita+Pib+Pikm>Pita+Pib+Pizt>Pi54+Pib+Pikm>Pita+Pi54+Pikm,这7种聚合方式的出现频率从高到低依次为:Pita+Pib+Pikm>Pita+Pib+Pizt>Pi54+Pib+Pikm>Pi54+Pib+Pb1>Pita+Pi54+Pikm> Pita+Pikm+Pizt>Pita+Pb1+Pizt。综上所述,抗病频率最高且出现频率相对较高的基因聚合方式为Pi54+Pib+Pb1,出现频率最高且抗病频率相对较高的基因聚合方式为Pita+Pib+Pikm,即3个抗病基因聚合的最佳方式为Pi54+Pib+Pb1或Pita+Pib+Pikm。
2. 6 4个抗病基因的聚合效应分析结果
4个抗病基因的聚合效应见表6。703份供试材料中含4个抗病基因的材料107份,其中稻瘟病综合指数≤5.0的材料79份,抗病频率为73.83%。抗病频率大于73.83%的聚合方式有6种,从高到低依次为:Pita+Pib+Pikm+Pizt=Pita+Pib+Pikm+Pb1=Pita+Pi54+Pikm+Pb1>Pita+Pi54+Pib+Pizt>Pita+Pi54+Pib+Pikm>Pita+Pi54+Pib+Pb1,这6种聚合方式的出现频率由高到低依次为:Pita+Pi54+Pib+Pb1>Pita+Pi54+Pib+Pikm>Pita+Pib+Pikm+Pizt>Pita+Pi54+Pib+Pizt>Pita+Pib+Pikm+Pb1>Pita+Pi54+Pikm+Pb1。综上所述,抗病频率最高且出现频率相对较高的基因聚合方式为Pita+Pib+Pikm+Pizt,出现频率最高且抗病频率相对较高的基因聚合方式为Pita+Pi54+Pib+Pb1,即4个抗病基因聚合的最佳方式为Pita+Pib+Pikm+Pizt或Pita+Pi54+Pib+Pb1。
3 讨论
传统的水稻抗病育种是采用杂交、回交或复交等手段,费时费力,且通常达不到预期目的,但分子标记辅助选择育种可快速鉴定材料中是否含有抗性基因,且不受水稻生育期和环境的影响(宋兆强等,2017;肖宇龙等,2018)。分子标记分为连锁标记和功能标记,应用连锁标记常达不到育种目的,且连锁标记可能受遗传背景的限制,无法应用于非多态群体(Fujii et al.,2000)。Pb1基因是一种来自籼稻品种Modan的抗性基因,可介导水稻成株期持久的穗瘟病抗性(Hayashi et al.,2010)。有关Pb1基因分子标记的前人研究多集中在连锁标记(Fujii et al.,2000),但其选择效率不高且应用具有局限性。本研究开发了Pb1基因的功能标记并进行验证,可将其应用于水稻稻瘟病抗性育种,提高选择效率。
目前,大量水稻稻瘟病基因被定位及克隆,并开发出一批稻瘟病基因功能标记,其中Pita、Pib、Pikm和Pi54稻瘟病抗性基因及其功能标记已广泛存在于江苏省水稻品种(系)中(范方军等,2014)。本研究对2015─2017年703份江苏省预试材料抗病基因进行检测,结果发现Pita、Pib和Pi54基因分布最广,为江苏省近年主要的稻瘟病抗性基因,但稻瘟病的抗性鉴定结果显示,2015─2017年达中抗及以上等级的比例偏低,表明Pita、Pib和Pi54基因不能满足水稻对稻瘟病的抗性需求,需聚合更多抗病基因才能提高水稻持久抗稻瘟病能力。
多基因聚合并不是单个抗病基因抗谱的简单累加,而是抗性基因间极显著的互作效应(刘士平等,2003)。因此,多基因聚合育种的前提是了解抗性资源的基因遗传背景,明确基因聚合类型。本研究对2015─2017年江苏省预试材料所含抗病基因数与稻瘟病抗性进行相关性分析,结果发现抗病基因数与抗病频率呈正相关,但含5和6个抗病基因的材料较少,出现频率较低,因此本研究进一步分析3和4个抗病基因的聚合方式与稻瘟病抗性的相关性。由于3和4个基因聚合方式存在多样化,导致每种聚合方式的材料数量偏低,出现频率较低,因此,选择抗病基因聚合的最佳方式时既要考虑各聚合方式的抗病频率又要考虑其出现频率,最终确定3个抗病基因聚合的最佳方式为Pi54+Pib+Pb1或Pita+Pib+Pikm,4个抗病基因聚合的最佳方式为Pita+Pib+Pikm+Pizt或Pita+Pi54+Pib+Pb1。此外,由于抗性基因聚合方式Pita+Pi54+Pib+Pizt和Pita+Pi54+Pib+Pizt/Pikm的抗病频率均达80.00%以上,因此这两种基因聚合方式也可供育种工作者参考。
本研究中6个抗病基因均不含的材料有11份,其中有2份表现出中抗,可能是由于该品种(系)含有大量其他广谱抗性基因。在今后的研究中,应对更多的抗性基因进行定位及克隆,并开发出其特异性功能标记,建立完整的抗稻瘟病水稻资源抗性基因数据库,应用于水稻分子标记辅助选择育种,以提高水稻稻瘟病抗性。
4 结论
开发的Pb1基因功能標记可应用于水稻稻瘟病抗性育种,以提高选择效率。多基因聚合能提高水稻稻瘟病抗性,聚合的抗病基因数与抗病频率呈正相关,但稻瘟病抗性取决于抗性基因间的互作效应。
参考文献:
蔡海亚,周雷,闸雯俊,李三和,刘凯,焦春海,游艾青,徐得泽. 2015. 水稻抗稻瘟病基因Piz-t特异性分子标记开发及应用[J]. 分子植物育种,13(7):1457-1461. [Cai H Y,Zhou L,Zha W J,Li S H,Liu K,Jiao C H,You A Q,Xu D Z. 2015. Development and application of rice blast resistant gene Piz-t gene-specific molecular marker[J]. Molecular Plant Breeding,13(7):1457-1461.]
范方军,王芳权,刘永峰,王军,朱金燕,李文奇,仲维功,杨杰. 2014. Pi-b,Pi-ta,Pikm和Pi54对水稻穗颈瘟的抗性评价[J]. 华北农学报,29(3):221-226. [Fan F J,Wang F Q,Liu Y F,Wang J,Zhu J Y,Li W Q,Zhong W G,Yang J. 2014. Evaluation of resistance to rice panicle blast with resistant genes Pi-b,Pi-ta,Pikm and Pi54[J]. Acta Agriculturae Boreali-Sinica,29(3):221-226.]
黄乾龙,管玉圣,欧阳杰,郭爽,王楚桃,李贤勇. 2019. 重庆自育水稻骨干亲本稻瘟病抗性评价[J]. 南方农业学报,50(1):8-15. [Huang Q L,Guan Y S,Ouyang J,Guo S,Wang C T,Li X Y. 2019. Resistance evaluation of self-bred rice backbone parents germplasms against rice blast in Chongqing and resistance gene distribution analysis[J]. Journal of Southern Agriculture,50(1):8-15.]
江苏省种子管理站. 2015. 江苏省农作物品种中间试验总结汇编(2015)[G]. 南京:江苏省种子管理站. [Jiangsu Seed Station. 2015. Summary and compilation of intermediate trials of crop varieties in Jiangsu Province(2015)[G]. Nanjing:Jiangsu Seed Station.]
江苏省种子管理站. 2016. 江苏省农作物品种中间试验总结汇编(2016)[G]. 南京:江苏省种子管理站. [Jiangsu Seed Station. 2016. Summary and compilation of intermediate trials of crop varieties in Jiangsu Province(2016)[G]. Nanjing:Jiangsu Seed Station.]
江苏省种子管理站. 2017. 江苏省农作物品种中间试验总结汇编(2017)[G]. 南京:江苏省种子管理站. [Jiangsu Seed Station. 2017. Summary and compilation of intermediate trials of crop varieties in Jiangsu Province(2017)[G]. Nanjing:Jiangsu Seed Station.]
刘斌,毕振佳,何平,韩光煜,董丽英,杨勤忠,王云月. 2018. 云南省主栽水稻品种抗稻瘟病基因型鉴定[J]. 分子植物育种,16(22):7362-7371. [Liu B,Bi Z J,He P,Han G Y,Dong L Y,Yang Q Z,Wang Y Y. 2018. Genotype identification of blast resistance in major rice varieties in Yunnan Province[J]. Molecular Plant Breeding,16(22):7362-7371.]
刘士平,李信,汪朝阳,李香花,何予卿. 2003. 基因聚合对水稻稻瘟病的抗性影响[J]. 分子植物育种,1(1):22-26. [Liu S P,Li X,Wang Z Y,Li X H,He Y Q. 2003. Gene pyramiding to increase the blast resistance in rice[J]. Molecular Plant Breeding,1(1):22-26.]
刘万才,刘振东,黄冲,陆明红,刘杰,杨清坡. 2016. 近10 年农作物主要病虫害发生危害情况的统计和分析[J]. 植物保护,42(5):1-9. [Liu W C,Liu Z D,Huang C,Lu M H,Liu J,Yang Q P. 2016. Statistics and analysis of crop yield losses caused by main diseases and insect pests in recent 10 years[J]. Plant Protection,42(5):1-9.]
宋兆强,刘艳,王宝祥,王芳权,迟铭,刘金波,陈庭木,方兆伟,邢运高,徐波,杨波,杨杰,徐大勇. 2017. 稻瘟病抗性基因Pi-ta、Pi-b、Pi54和Pi-km的育种利用价值评价[J]. 江苏农业学报,33(5):968-974. [Song Z Q,Liu Y,Wang B X,Wang F Q,Chi M,Liu J B,Chen T M,Fang Z W,Xing Y G,Xu B,Yang B,Yang J,Xu D Y. 2017. Application value of blast resistant genes Pi-ta,Pi-b,Pi54 and Pi-km inrice breeding[J]. Jiangsu Journal of Agricultural Sciences,33(5):968-974.]
孙大元,周丹华,张景欣,王慧,朱小源,杨祁云,陈志强. 2017. 广谱抗源H4中2个主效抗病基因的单基因系构建及评价[J]. 江苏农业学报,33(1):1-5. [Sun D Y,Zhou D H,Zhang J X,Wang H,Zhu X Y,Yang Q Y,Chen Z Q. 2017. Development of near-isogenic lines for two blast resistance genes in an indicarice H4[J]. Jiangsu Journal of Agricultural Sciences,33(1):1-5.]
孙立亭,林添资,王云龙,牛梅,胡婷婷,刘世家,王益华,万建民. 2017. 水稻白条纹突变体st13的表型分析及基因定位[J]. 中国水稻科学,31(4):355-363. [Sun L T,Lin T Z,Wang Y L,Niu M,Hu T T,Liu S J,Wang Y H,Wan J M. 2017. Phenotypic analysis and gene mapping of a white stripe mutant st13 in rice[J]. Chinese Journal of Rice Science,31(4):355-363.]
肖宇龙,李湘民,余传元,雷建国,王智权,王晓玲,何虎,邱在辉,肖宇华. 2018. 江西省稻瘟病生理小种致病力测定及1组水稻优质新种质的稻瘟病抗性表现及应用评估[J]. 江西农业学报,30(4):9-13. [Xiao Y L,Li X M,Yu C Y,Lei J G,Wang Z Q,Wang X L,He H,Qiu Z H,Xiao Y H. 2018. Pathogenicity of physiological races of rice blast disease in Jiangxi Province,blast-resistant performance and application evaluation of a set of new high-quality rice germplasms[J]. Acta Agriculturae Jiangxi,30(4):9-13.]
張善磊,孙旭超,陈涛,赵春芳,朱镇,赵庆勇,周丽慧,赵凌,姚姝,王才林. 2018. Pi-ta、Pi-5、Pi-km和Pi-b基因在粳稻品种(系)中的分布及对穗颈瘟的抗性[J]. 江苏农业学报,34(5):961-971. [Zhang S L,Sun X C,Chen T,Zhao C F,Zhu Z,Zhao Q Y,Zhou L H,Zhao L,Yao S,Wang C L. 2018. Distribution of Pi-ta,Pi-5,Pi-km and Pi-b genes in japonica rice varieties(lines) and their relationship with neck blast resistance[J]. Jiangsu Journal of Agriculture Sciences,34 (5):961-971.]
Andersen J R,Lǖbberstedt T. 2003. Functional markers in plants[J]. Trends in Plant Science,8(11):554-560.
Ashikawa I,Hayashi N,Abe F,Wu J,Matsumoto T. 2012. Characterization of the rice blast resistance gene Pik cloned from Kanto 51[J]. Molecular Breeding,30(1):485-494.
Ashikawa I,Hayashi N,Yamane H,Kanamori H,Wu J,Matsumoto T,Ono K,Yano M. 2008. Two adjacent nucleotide-binding site-leucine-rich repeat class genes are required to confer Pikm-specific rice blast resistance[J]. Genetics,180(4):2267-2276.
Bryan G T,Wu K S,Farrall L,Jia Y L,Hershey H P,Mc Adams S A,Faulk K N,Donaldson G K,Tarchini R,Valent B. 2000. A single amino acid difference distinguishes resistant and susceptible alleles of the rice blast resistance gene Pi-ta[J]. The Plant Cell Online,12(11):2033-2046.
Chen J,Shi Y F,Liu W Z,Chai R Y,Fu Y P,Zhuang J Y,Wu J L. 2011. A Pid3 allele from rice cultivar Gumei2 confers resistance to Magnaporthe oryzae[J]. Journal of Genetics and Genomics,38(5):209-216.
Chen X,Shang J,Chen D,Lei C,Zou Y,Zhai W,Liu G,Xu J,Ling Z,Cao G,Ma B,Wang Y,Zhao X,Li S,Zhu L. 2006. A B-lectin receptor kinase gene conferring rice blast resistance[J]. The Plant Journal,46(5):794-804.
Das A,Soubam D,Singh P K,Thakur S,Singh N K,Sharma T R. 2012. A novel blast resistance gene,Pi54rh cloned from wild species of rice,Oryza rhizomatis confers broad spectrum resistance to Magnaporthe oryzae[J]. Functio-nal & Integrative Genomics,12(2):215-228.
Deng Y,Zhai K,Xie Z,Yang D,Zhu X,Liu J,Wang X,Qin P,Yang Y,Zhang G,Li Q,Zhang J,Wu S,Milazzo J,Mao B,Wang E,Xie H,Tharreau D,He Z. 2017. Epigene-tic regulation of antagonistic receptors confers rice blast resistance with yield balance[J]. Science,355(6328):962-965.
Fjellstrom R,Conaway-Bormans C A,McClung A M,Marchetti M A,Shank A R,Park W D. 2004. Development of DNA markers suitable for marker assisted selection of three Pi genes conferring resistance to multiple Pyricula-ria grisea pathotypes[J]. Crop Science,44(5):1790-1798.
Fujii K,Saito Y H,Saito K,Sugiura N,Hayashi N,Tsuji T,Izawa T,Iwasaki M. 2000. Identification of a RFLP marker tightly linked to the panicle blast resistance gene,Pb1,in rice[J]. Breeding science,50(3):183-188.
Fukuoka S,Saka N,Koga H,Ono K,Shimizu T,Ebana K,Hayashi N,Takahashi A,Hirochika H,Okuno K,Yano M. 2009. Loss of function of a proline-containing protein confers durable disease resistance in rice[J]. Science,325(5943):998-1001.
Hayashi K,Yasuda N,Fujita Y,Koizumi S,Yoshida H. 2010a. Identification of the blast resistance gene Pit in rice cultivars using functional markers[J]. Theoretical and Applied Genetics,121(7):1357-1367.
Hayashi N,Inoue H,Kato T,Funao T,Shirota M,Shimizu T,Kanamori H,Yamane H,Saito Y H,Matsumoto T,Yano M,Takatsuji H. 2010b. Durable panicle blast-resistance gene Pb1 encodes an atypical CC-NBS-LRR protein and was generated by acquiring a promoter through local genome duplication[J]. The Plant Journal,64(3):498-510.
Hittalmani S,Parco A,Mew T V,Zeigler R S,Huang N. 2000. Fine mapping and DNA marker-assisted pyrami-ding of the three major genes for blast resistance in rice[J]. Theoretical and Applied Genetics,100(7),1121-1128.
Hua L X,Wu J Z,Chen C X,Wu W H,He X Y,Lin F,Wang L,Ashikawa I,Matsumoto T,Wang L,Pan Q H. 2012. The isolation of Pi1,an allele at the Pik locus which confers broad spectrum resistance to rice blast[J]. Theoretical and Applied Genetics,125(5):1047-1055.
Jeon J S,Chen D,Yi G H,Wang G L,Ronald P C. 2003. Genetic and physical mapping of Pi5(t),a locus associated with broad-spectrum resistance to rice blast[J]. Molecular Genetics and Genomics,269(2):280-289.
Jia Y,Wang Z,Singh P. 2002. Development of dominant rice blast Pi-ta resistance gene markers[J]. Crop Science,42(6):2145-2149.
Khush G S,Jena K K. 2009. Current status and future prospects for research on blast resistance in rice(Oryza sative L.)[J]. Advances in Genetics,Genomics and Control of Rice Blast Disease. doi:10.1007/978-1-4020-9500-9_1.
Koide Y,Kobayashi N,Xu D,Fukuta Y. 2009. Resistance genes and selection DNA markers for blast disease in rice(Oryza sativa L.)[J]. Japan Agricultural Research Quarterly,43(4):255-280.
Lin F,Chen S,Que Z,Wang L,Liu X,Pan Q. 2007. The blast resistance gene Pi37 encodes a nucleotide binding site-leucine-rich repeat protein and is a member of a resistance gene cluster on rice chromosome 1[J]. Genetics,177(3):1871-1880.
Liu Y,Liu B,Zhu X,Yang J,Bordeos A,Wang G,Leach J E,Leung H,Liu Y. 2013. Fine-mapping and molecular marker development for Pi56(t),a NBS-LRR gene conferring broad-spectrum resistance to Magnaporthe oryzae in rice[J]. Theoretical and Applied Genetics,126(4):985-998.
Mackill D J,Bonman J M. 1992. Inheritance of blast resistance in near-isogenic lines of rice[J]. Phytopathology,82(7):746-749.
Miah G,Rafii M Y,Ismail M R,Peteh A B,Rahim H A,Aafa-liza R,Latif M A. 2013. Blast resistance in rice:a review of conventional breeding to molecular approaches[J]. Molecular Biology Reports,40(3):2369-2388.
Okuyama Y,Kanzaki H,Abe A,Yoshida K,Tamiru M,Saitoh H,Fujibe T,Matsumura H,Shenton M,Galam D C,Undan J,Ito A,Sone T,Terauchi R. 2011. A multifaceted genomics approach allows the isolation of the rice Pia-blast resistance gene consisting of two adjacent NBS-LRR protein genes[J]. The Plant Journal,66(3):467-479.
Qu S,Liu G,Zhou B,Bellizzi M,Zeng L,Dai L,Han B,Wang G L. 2006. The broad-spectrum blast resistance gene Pi9 encodes a nucleotide-binding site-leucine-rich repeat protein and is a member of a multi-gene family in rice [J]. Genetics,172(3):1901-1914.
Ramkumar G,Srinivasarao K,Mohan K M,Sudarshan I,Si-varanjani A K P,Gopalakrishna K,Neeraja C N,Balachandran S M,Sundaram R M,Prasad M S,Rani N S,Rama Prasad A M,Viraktamath B C,Madhav M S,Ramkumar G. 2011. Development and validation of functional marker targeting an InDel in the major rice blast disease resistance gene Pi54(Pikh)[J]. Molecular Bree-ding,27(1):129-135.
Shang J,Tao Y,Chen X,Zou Y,Lei C,Wang J,Li X,Zhao X,Zhang M,Lu Z,Xu J,Cheng Z,Wan J,Zhu L. 2009. Identification of a new rice blast resistance gene,Pid3,by genomewide comparison of paired nucleotide-binding site–leucine-rich repeat genes and their pseudogene alleles between the two sequenced rice genomes[J]. Gene-tics,182(4):1303-1311.
Skamnioti P,Gurr S J,2009,Against the grain:safeguarding rice from rice blast disease[J]. Trends in Biotechnology,27(3):141-150.
Takahashi A,Hayashi N,Miyao A,Hirochika H. 2010. Unique features of the rice blast resistance Pish locus revealed by large scale retrotransposon-tagging[J]. BMC Plant Bio-logy,10:175.
Wang Z X,Yano M,Yamanouchi U,Iwamoto M,Monna L,Hayasaka H,Katayose Y,Sasaki T. 1999. The Pib gene for rice blast resistance belongs to the nucleotide binding and leucine-rich repeat class of plant disease resistance genes[J]. The Plant Journal,19(1):55-64.
Xu X,Hayashi N,Wang C T,Fukuoka S,Kawasaki S,Takatsuji H,Jiang C J. 2014. Rice blast resistance gene Pikahei-1(t),a member of a resistance gene cluster on chromosome 4,encodes a nucleotide-binding site and leucine-rich repeat protein[J]. Molecular Breeding,34(2):691-700.
Yuan B,Zhai C,Wang W J,Zeng X S,Xu X K,Hu H Q,Lin F,Wang L,Pan Q H. 2011. The Pik-p resistance to Magnaporthe oryzae in rice is mediated by a pair of closely linked CC-NBS-LRR genes[J]. Theoretical and Applied Genetics,122(5):1017-1028.
Zhai C,Lin F,Dong Z Q,He X Y,Yuan B,Zeng X S,Wang L,Pan Q H. 2011. The isolation and characterization of Pik,a rice blast resistance gene which emerged after rice domestication[J]. New Phytologist,189(1):321-334.
Zhou B,Qu S,Liu G F,Dolan M,Sakai H,Lu G D,Bellizzi M,Wang G L. 2006. The eight amino-acid differences within three leucine-rich repeats between Pi2 and Piz-t resistance proteins determine the resistance specificity to Magnaporthe grisea[J]. Molecular Plant Microbe Interactions,19(11):1216-1228.
(責任编辑 陈 燕)

