银杏bHLH家族转录因子生物信息学及表达分析
冯磊 石元豹 汪贵斌 曹福亮

摘要:以银杏基因组数据为基础,对银杏bHLH转录因子进行筛选和分析,从银杏基因组中鉴定出72条bHLH转录因子。进一步分析发现,不同bHLH转录因子序列长度和分子量差异较大,而理论等电点及亲水性等比较接近;各家族成员均含有N端碱性氨基酸区和C端的螺旋环螺旋区;该家族可分为17个亚家族,相同亚家族成员保守基序的类型十分相似。通过启动子分析发现,多数银杏bHLH基因启动子均含有光响应元件、激素响应元件和逆境胁迫响应元件等。表达分析结果显示,有7条银杏bHLH基因的表达具有组织特异性,有6条基因在各组织中表达水平都比较高,预测其在银杏生物学过程中具有十分重要的作用。
关键词:银杏;bHLH转录因子;进化分析;表达分析
中图分类号:S792.95
文献标识码:A
文章编号:1000-4440(2019)02-0400-12
bHLH(Basic helix-loop-helix)转录因子是广泛存在于动、植物中的转录因子超家族,因其“碱性螺旋-环-螺旋”保守结构域而命名[1]。鸟类成髓细胞癌病毒中的c-MYC原癌基因是第1个被发现的bHLH家族成员基因[2],之后该类型的基因又陆续在多种动、植物中被发现[3]。该家族蛋白质的bHLH保守结构域大约含有60个氨基酸,其碱性区域分布在多肽链的N端,并负责与特定DNA的顺时作用元件结合;HLH区域位于C末端,参与二聚体或者异二聚体的形成,从而行使其功能。
动物中bHLH家族数量较多,被分为A~F等6个组,共计45个家族[4]。与动物相比,植物中bHLH家族转录因子相对较少,而且大多数植物的bHLH蛋白在动物的该家族分类中属于B组[5],根据植物中bHLH蛋白的序列同源性,又可将其分为不同的亚家族[5-6]。到目前为止,拟南芥中已鉴定的bHLH家族转录因子有162种,并且与水稻中的bHLH转录因子一起被分成了25个亚家族[7-9]。有学者对拟南芥、毛果杨、水稻、小立碗藓以及5种藻类的bHLH转录因子进行综合分析,并将其分成32个亚家族[10]。随着二代和三代测序技术的发展,苹果梨、樱桃、西瓜、甘薯、大白菜等越来越多植物的bHLH家族转录因子被鉴定出来[11-17]。
植物中bHLH家族不但数量较多,还参与植物生长发育、形态建成、逆境胁迫和次生代谢调控等多种生物学过程[18-19]。近年来,植物bHLH家族蛋白质的功能研究已经成为热点并且得到了快速发展。拟南芥作为模式植物,其bHLH转录因子功能研究相对较多。有研究者发现AtbHLH112对抗盐、抗旱和抗渗透胁迫有正调控作用,但是对拟南芥根系的发育有抑制作用[2021]。FIT转录因子可与At-bHLH38和AtbHLH39 共同调控拟南芥的铁代谢[2]。光敏色素作用因子PIF4,也是一个bHLH家族成员,对光敏色素B的信号转导有抑制作用[23]。另外,研究者在西瓜基因组中共计鉴定出96个bHLH转录因子,并预测其中多个bHLH转录因子可能在低温、ABA和盐胁迫中发挥重要作用[13]。还有研究者发现水稻中OsbHLHl基因可能与其抗低温胁迫相关[24]。
银杏是原产中国的特色经济树种,栽培历史悠久,用途广泛,经济价值高,拥有中华树木“国宝”之称[25]。植物中的bHLH家族研究在近年来得到了快速发展,但是到目前为止,在银杏中被筛选出的bHLH转录因子并不多,仅有1条被命名为bHLH91的转录因子被克隆并进行了表达分析[26-27]。本研究通过生物信息学方法,在全基因组范围内对银杏bHLH转录因子家族成员进行分离鉴定,并对鉴定出的银杏bHLH转录因子进行理化性质、系统进化、启动子结构和基因表达等方面进行分析,为bHLH转录因子功能的解析提供理论基础。
1 材料与方法
1.1 银杏bHLH家族的筛选
以Pfam数据库中bHLH家族的保守域PF00010为参考序列,用HMMER 3.0软件在测试银杏基因组库(GigaDB,http://gigadb.org/dataset/100209)中进行检索。检索得到的序列用NCBI-CDD(https://www.ncbi.nlm.nih.gov/cdd/)和SMART(http://smart.embl-heidelberg.de/)在线程序进行保守域预测,去除假阳性序列[28]。将预测的具有bHLH保守域的序列作为银杏bHLH家族候选转录因子,用于后续分析。
1.2 银杏bHLH家族的理化性质及保守域和保守基序分析
bHLH家族候選蛋白质的氨基酸数、分子量、理论等电点等理化性质用在线软件ExPASy中的Prot-Param模块(https://web.expasy.org/protparam/)进行分析。用DNAMAN(Lynnon Corporation,USA)对银杏bHLH家族候选基因编码的保守氨基酸序列进行多序列比对,并用MEME在线程序(http://meme-suite.org/tols/meme)进行氨基酸的保守基序分析[29]。
1.3 银杏bHLH转录因子家族的进化分析
从PlantTFDB数据库(http://planttfdb.cbi.pku.edu.cn/)中下载拟南芥bHLH转录因子序列作为参考,每个亚家族分别下载1~4条,共下载了49条At-bHLH家族转录因子[30]。将银杏候选bHLH蛋白序列与24个AtbHLH参考序列用MUSCLE法进行多序列比对,截取bHLH保守域序列并用软件MEGA5.0的Neighbo-Joining邻接法构建进化树,校验参数Bootstrap重复1000次[31]。
1.4 银杏bHLH转录因子家族基因结构分析
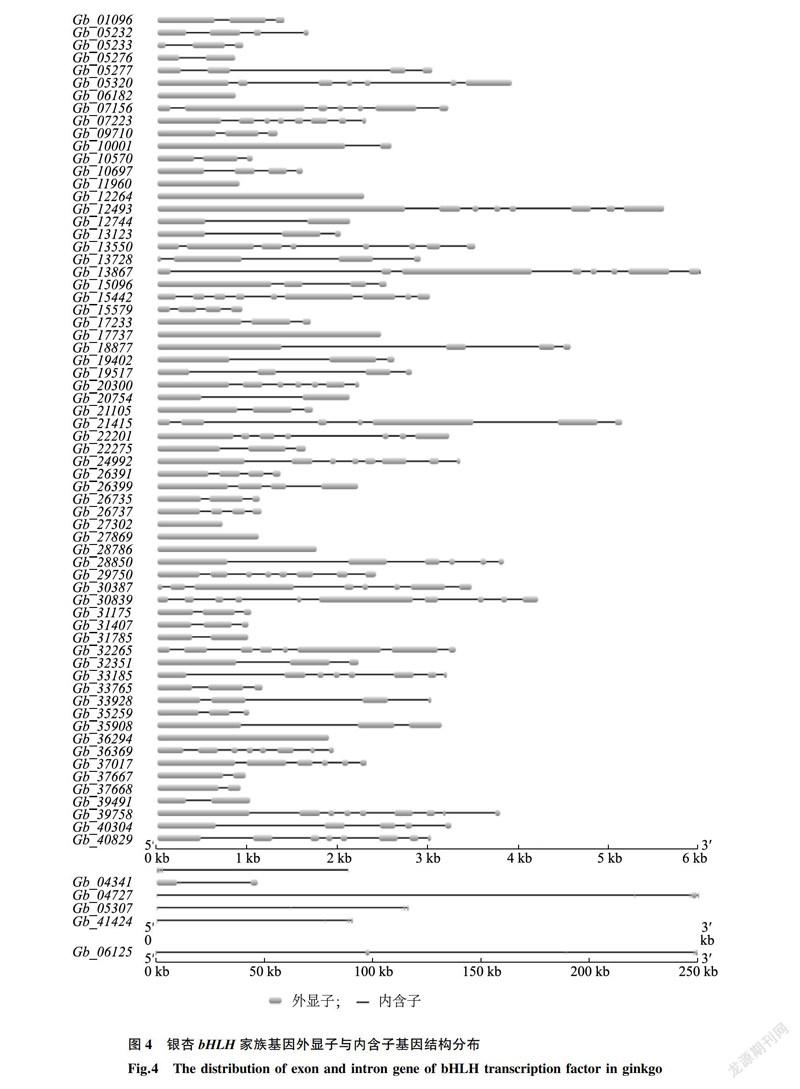
从银杏基因组数据库中提取筛选出bHLH基因CDS序列对应的基因组序列,用GSDS 2.0在线程序绘制基因外显子-内含子模式图。
1.5 银杏bHLH家族基因的启动子分析
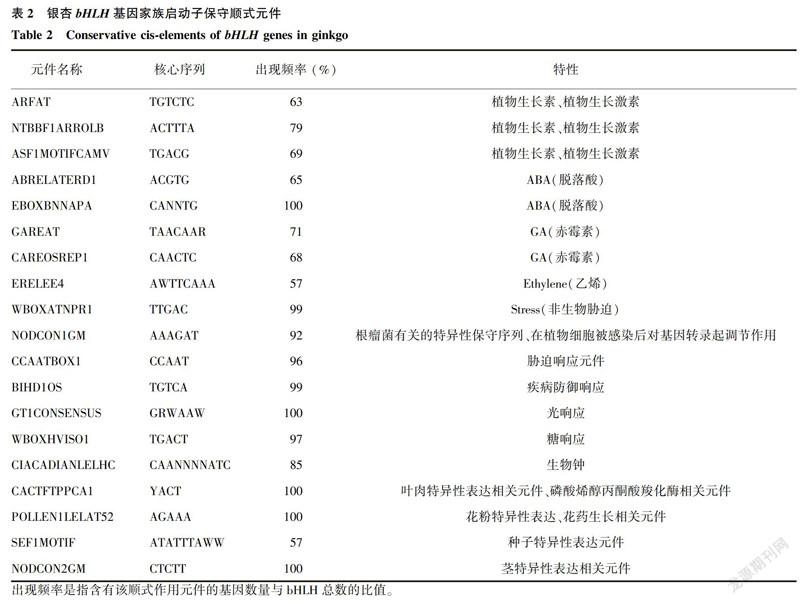
从银杏基因组数据库中截取银杏bHLH家族基因成员起始密码子上游2000 bp序列作为启动子区,利用植物顺式作用元件数据库SOGO(https://sogo.dna.affre.go.jp/cgi-bin/sogo.cgi?sid=&lang=en&pj=640&action=page&page=newplace#opennewwindow)分析基因的顺式作用元件。
1.6 银杏bHLH家族基因表达分析
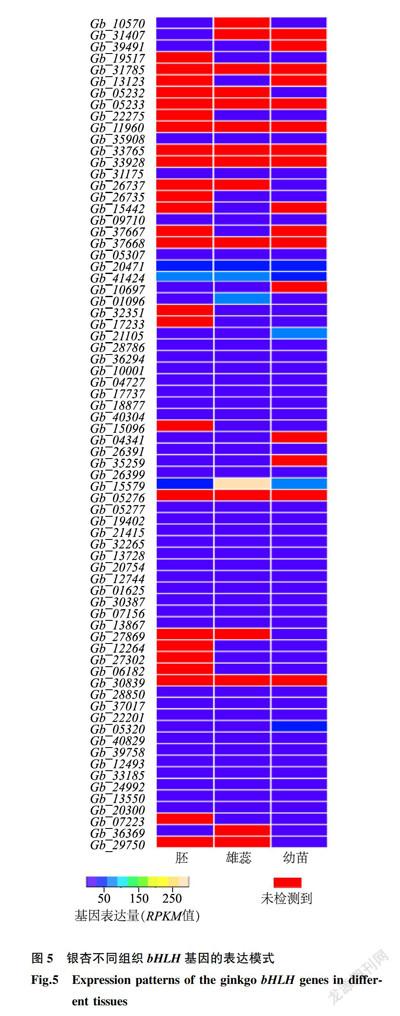
银杏基因表达数据下载于GigaDB,通过Heat-map 2.0作图,对采集于浙江省天目山的银杏胚(雌树)、雄蕊(雄树)和幼苗(茎和叶混合样)的bHLH基因表达进行分析。
2 结果与分析
2.1 银杏bHLH转录因子筛选与理化性质分析i
根据Pfam数据库bHLH家族的保守域PF00010特征文件,在银杏基因组数据库中共检索到133条可能的bHLH转录因子序列。候选序列用NCBI-CDD和SMART在线程序对保守结构域进行检测,剔除结构域不完整的序列,最后得到72个银杏bHLH转录因子家族成员。这些成员在GigaDB数据库中的登录号见表1。
利用ExPASy在线程序对银杏bHLH蛋白质的理化特性进行分析。由表1可见,银杏bHLH转录因子家族成员蛋白质氨基酸序列含有氨基酸数目为181~1471,平均值为498;预测其分子量为2.0220x104~1.6141x105,平均值为5.5240x104;理论等电点在4.75至9.38范围内。从整体看,有64%的银杏bHLH家族蛋白质等电点小于7,等电点在酸性范围内,蛋白质分子中富含酸性氨基酸。蛋白质疏水性分析结果表明,72条序列的疏水性均小于0,说明银杏bHLH蛋白质均为亲水蛋白质,但是不同蛋白质之间亲水性存在差异。另外,银杏bHLH蛋白质含有大量脂肪族氨基酸,其中脂肪族氨基酸指数最大的是Gb_15579。
2.2 银杏bHLH转录因子保守域分析
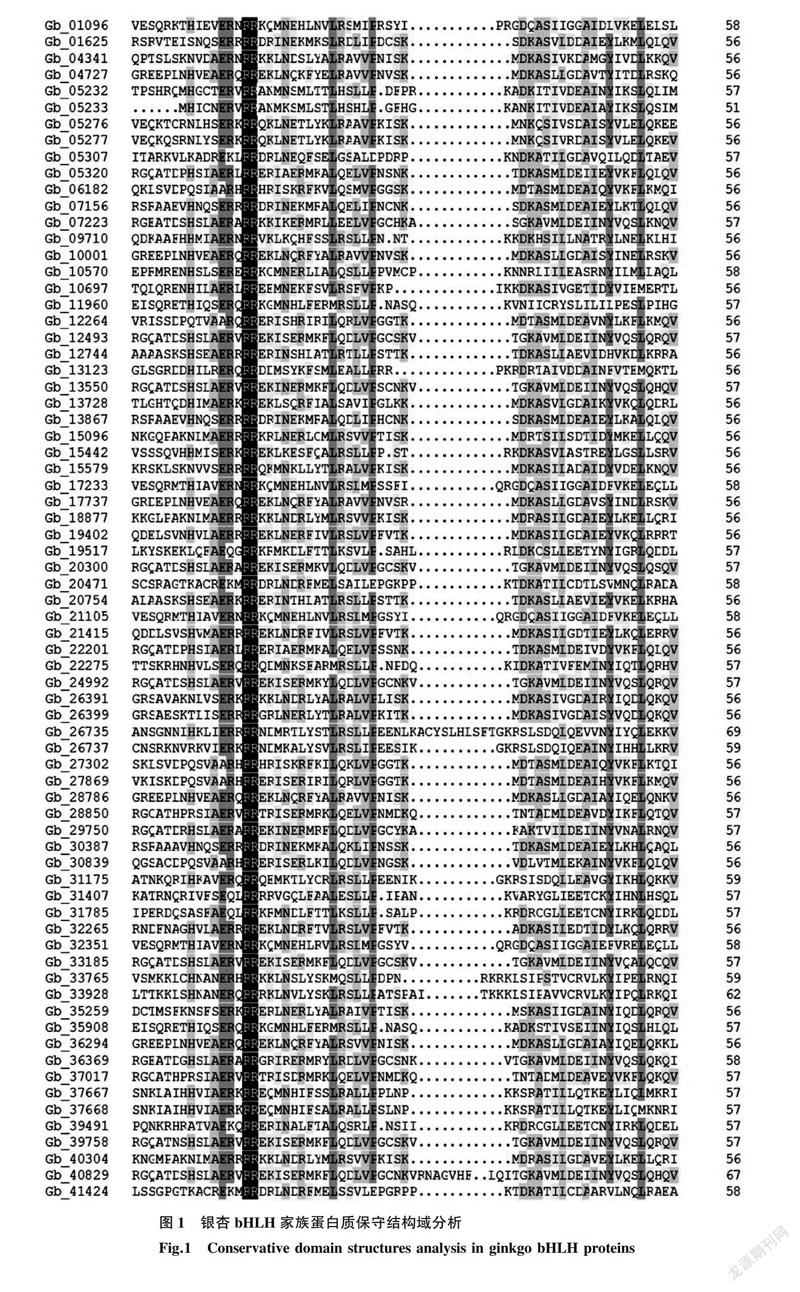
利用DNAMAN9.0对银杏bHLH家族蛋白质保守域进行多序列比对,发现银杏bHLH家族保守域长度多数在60个氨基酸左右,其中最长的有69个氨基酸,最短的有51个氨基酸(图1)。在结构域序列保守性方面,除了Loop区没有保守位点,其他区域都有不同数量的保守位点。在银杏bHLH蛋白质的保守域中共有24个位点上保守氨基酸的出现频率高于50%,与其他植物类似,其中谷氨酸Glu-12、精氨酸Arg-13、精氨酸Arg-15、精氨酸Arg-16亮氨酸Leu-26、脯氨酸Pro-32、酪氨酸Tyr-65、亮氨酸Leu-69保守性均高于86%,其中精氨酸Arg-15、精氨酸Arg-16保守性为100%。
2.3 银杏bHLH转录因子系统发生树分析
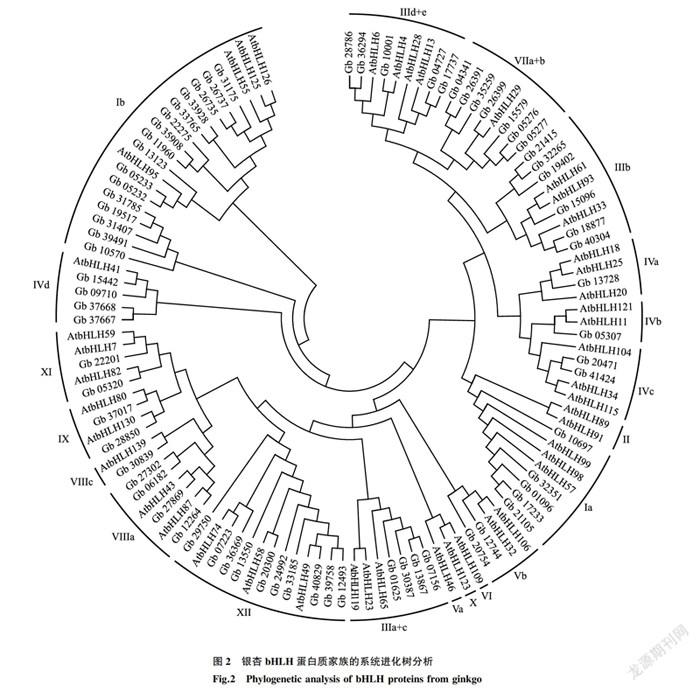
根据预测到的银杏bHLH蛋白质和随机选择的拟南芥bHLH各亚家族蛋白质保守结构域氨基酸序列的相似性,利用MEGA5.1构建系统发育树。参考拟南芥和水稻的bHLH家族转录因子的亚家族分类结果[9],预测得到的银杏72个bHLH转录因子和随机选择的拟南芥bHLH转录因子被分成了21个亚家族,其中第II、Va、VI和第X亚家族只含有拟南芥bHLH转录因子。也就是说,银杏的72个bHLH转录因子被分成了17个亚家族(图2)。在所有含有银杏bHLH蛋白质的17个亚家族中,第Ib亚家族中的银杏bHLH成员数量最多,共16个;第IVb、VIlc、IVc、Vb、XI和IX亚家族中的成员数量相对较少,数量仅为1~2个。根据进化树分类结果以及各亚家族某些成员在模式植物拟南芥和水稻中的作用,初步判断银杏bHLH蛋白质的功能。比如la亚家族蛋白质可能与细胞分裂和器官分化相关,Ib亚家族蛋白质可能与铁代谢调控相关,Ib亚家族可能在植物的抗低温胁迫中起重要作用等[32]。银杏bHLH转录因子在其各种生理过程中的具体功能,还需要进一步研究。
2.4 银杏bHLH转录因子保守基序分析
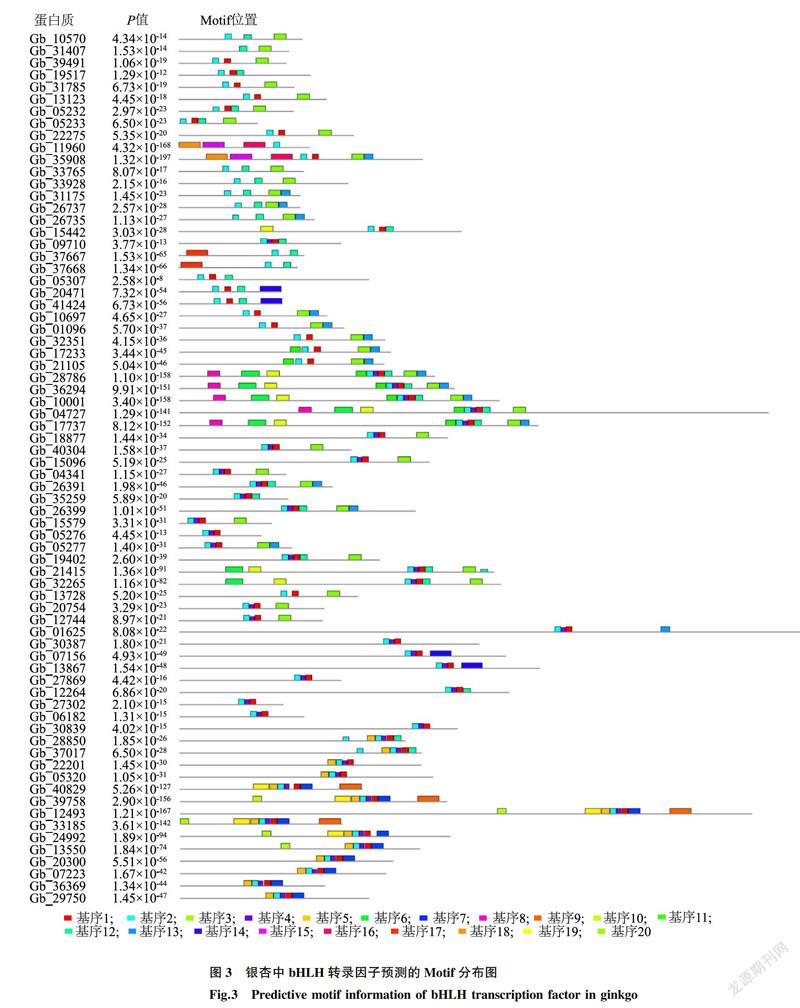
为了进一步研究银杏中bHLH蛋白质结构的多样性,通过MEME在线程序对银杏72个bHLH蛋白质进行保守基序(Motif)分析,识别获得20个保守基序(图3)。由图3可见,属于同一个亚家族的bHLH基因家族成员有着十分相似的Mo-tif类型和数量,但同时同一个亚家族成员的Motif模式也存在差异。在所有保守基序中,Motif 1、Motif2和Motif12是bHLH蛋白质保守结构域的特征保守基序。在所有银杏bHLH蛋白质中,序列Gb_31407的Motif 类型最为简单,仅有2个成员,最复杂的是Illd+e亚家族成员,其Motif数量为9~10个,且该亚家族成员除Gb_31407 没有Motif13以外,各成员含有的Motif类型完全相同。有一些Motif具有亚家族特异性,比如Motif15、Motif16和Motif18仅在亚家族lb中含有;Motif 17是IVd亚家族特有的保守基序;而Motif8和Motif 11只在亚家族IId+e被发现;XII亚家族中特有的Motif有4个,分别是Motif 7、Motif 9、Motif 19和Motif 20。
2.5 銀杏bHLH家族基因的基因结构分析
运用GSDS 2.0软件绘制银杏基因内含子-外显子结构图并对其结构进行分析和整理。由图4可知,银杏bHLH基因家族分为内含子富集和内含子缺失2类,其中Gb_06182、Gb_11960、Gb_12264、Gb_17737、Gb_27302、Gb_27869、Gb_28786、Gb_36294无内含子,Gb_39491、Gb_37668、Gb_37667、Gb_31785、Gb_20754、Gb_12744、Gb_10001、Gb_05276仅1个内含子,以上成员均属于内含子缺失组;Gb_30839含9个内含子(占比1%),Gb_15442、Gb_39758含8个内含子(占比3%),Gb_40829、Gb_36369、Gb_33185、Gb_32265、Gb_30387、Gb_29750、Gb_24992、Gb_13867、Gb_13550、Gb_12493、Gb_07223含7个内含子(占比15%),Gb_65320、Gb_07156、Gb_20300、Gb_21415、Gb_22201含6个内含子(占比7%),Gb_37017、Gb_28850含5个内含子(占比3%),以上成员均属内含子富集组。其中含有2个内含子的bHLH基因17个,所占成员总数比例最多,达24%。此外,基因结构分析结果表明,银杏bHLH基因家族各成员基因长度具有明显的差异。其中最短的Gb_05276 只有859 bp;而Gb_01625序列最长,达到了257612 bp。
2.6 银杏bHLH家族基因启动子分析
银杏bHLH基因启动子序列分析结果(表2)显示,72个bHLH基因的启动子调控区域均含有与光调控、脱落酸、叶肉特异性表达、花粉特异性表达、茎特异性表达有关的转录因子作用元件,例如GT1CONSENSUS、EBOX BNNAPA、CACTFTPPCA1、POLLEN1LELAT52和NODCON2GM。统计结果表明,有63%和79%的银杏bHLH基因成员含有植物生长激素响应元件(ARFAT和NTBBF1ARROLB),65%的bHLH基因含脱落酸响应元件(ABRELAT-ERD1),71%和68%的bHLH基因含有赤霉素响应元件(GAREAT和CAREOSREP1),57%的bHLH基因含有乙烯响應元件(ERELEE4),92%的bHLH基因含有与根瘤菌有关的特异性保守序列响应元件(NODCON1GM),96%的bHLH基因含有胁迫响应元件(CCAATBOX1),99%的bHLH基因含有疾病防御响应元件(BIHD1OS),97%的bHLH基因含有糖响应元件(WBOXHVISO1),57%的bHLH基因含有种子特异性表达元件(SEF1MOTIF)。说明各bHLH基因的表达与银杏抗逆境以及生长发育有重要联系。
2.7 银杏bHLH家族基因表达分析
从银杏基因组数据库中下载基因表达数据,将其中bHLH基因的表达数据用heatmap2.0作图(图5)。由图5可知,在银杏所有bHLH家族基因中有8个在胚、雄蕊和幼苗3个器官中都没有检测到表达,另外有7个基因的表达具有组织特异性。由图5可以看出,在胚中表达水平较高的有Gb_20471(36.86)、Gb_41424(81.93)和Gb_15579(40.27),另外Gb_05307、Gb_28786和Gb_01625表达量(RP_KM值)也达到10以上;雄蕊中表达量超过10的基因有19个,需要特别指出的是Gb_41424和Gb_15579基因的表达量分别高达79.48和297.03,而且Gb_15579在3个样品中表达量都比较高,在雄蕊中表达量是所有测试基因中表达最高的;在银杏幼苗中表达量超过10的基因有17个,其中Gb_20471、Gb_41424、Gb_21105和Gb_15579的表达量分别高达36.61、57.86、64.26和70.70。总体来看Gb_20471、Gb_41424、Gb_28786和Gb_15579在3个样品中表达水平都比较高。其中Gb_28786和Gb_15579分别属于ld+e和Ia+c亚家族,可能参与银杏中茉莉酸代谢的调控;Gb_20471和Gb_41424属于IVe亚家族,可能参与植物金属稳态的调节。
3 讨论
本研究通过对银杏全基因组进行搜索,最终筛选出了72个bHLH基因,并且对它们的理化性质、保守结构域、基因结构、系统进化和基因表达等情况进行了详细的分析。银杏bHLH转录因子的保守区域中有His、Glu、Arg和Leu等高度保守的氨基酸残基[33]。通过系统发育分析可将银杏和拟南芥的bHLH成员分为21个亚家族11.34-35]。本研究对筛选的银杏bHLH的表达模式进行了分析,发现有些基因在胚(雌树)、雄蕊(雄树)和幼苗(茎和叶混合样)3种样品中都未被检测到表达,可能这些基因在特定时间或某种胁迫条件下才会表达。还有些基因的表达具有组织特异性,也有几个基因在3种样品中的表达水平都比较高,说明有些bHLH基因在某些特定组织中才会发挥作用,也有些基因在所有组织中都发挥作用。
近年来,bHLH转录因子在植物次生代谢调控,中的作用受到许多研究者的关注,相关研究取得很大进展。Gonzalez等[36]和Xie等[3]发现了多个bHLH转录因子与拟南芥花青苷代谢相关。Dom-brecht等[38]和Schweizer等[39还发现多个bHLH转录因子在黄酮代谢中发挥重要调控作用。此外,bHLH家族转录因子还在吡啶类生物碱代谢、糖苷生物碱代谢、紫杉烷类代谢、异喹啉类生物碱代谢和萜类代谢等多种植物代谢途径中被发现[4041]。黄酮和萜类是银杏中主要的药用成分,其代谢通路中多数关键基因已经被研究,但是其转录调控研究还在摸索阶段。bHLH转录因子在银杏黄酮和萜类代谢中的调控作用是将来研究的一个重要方面。
总之,转录因子是一类重要的基因调控蛋白质,生物体的一系列生理生化活动都离不开转录因子的调节,它不仅可以调节生物体的新陈代谢和生长发育,而且在响应外界胁迫方面也起着重要的作用[42]。本研究利用生物信息学方法,对银杏bHLH基因家族进行了系统深入的分析,为未来的基因克隆提供了丰富的资源,可为将来深入研究该家族基因的表达调控、结构和功能等提供参考。
参考文献:
[1]BUCK M J,ATCHLEY W R.Phylogenetic analysis of plant basic helix-loop-helix proteins[J].J Mol Evol,2003,56(6):742-750.
[2]MURRE C,MCCAW P,BALTIMORE D.A new DNA binding and dimerizing motif in Immunoglobulin enhancer binding,Daugtherless,MyoD,and Myc proteins[J].Cell,1989,56(5):777-783.
[3]王勇江,陈克平,姚勤.bHLH转录因子家族研究进展[J].遗传,2008,30(7):821-830.
[4]王勇,姚勤,陈克平.动物bHLH转录因子家族成员及其功能[J].遗传,2010,34(4):307-330.
[5]刘晓月,王文生,傅彬英.植物BHLH转录因子家族的功能研究进展[J].生物技术进展,2011,1(6):391-397.
[6]ATCHLEY W A,FTTCH W M.A natural classification of the basic helix-loop-helix class of transcription factors[J].Proceedings ofthe National Academy of Sciences of the United States of America,1997,94(10):5172-5176.
[7]TOLEDOOG,HUQ E,QUAIL P H.The Arabidopsis basic/helix-loop-helix transcription factor family(w)[J].Plant Cell,2003,15(8):1749-1770.
[8]BAILEY P C,MARTIN C,T0LEDO OG,et al.Update on the basic helix-loop-helix transcription factor gene family in Arabidop-sis thaliana[J].The Plant Cell,2003,15(11):2497-2501.
[9LI X,DUAN X,JIANG H,et al.Genome-wide analysis of basic helix-loop-helix transcription factor family in rice and arabidopsis1[J].Physiologia Plantarum,2006,141(4):1167-1184.
[10]CARRETERO P L,GALSTYAN A,ROIG V I,et al.Genome-wide classification and evolutionary analysis of the bHLH family oftranscription factors in arabidopsis,poplar,rice,moss,and algael[J].Physiologia Plantarum,2010,153(3):1398-1412.[11]楊金华.苹果bHLH转录因子家族的鉴定及表达分析[D].杨凌:西北农林科技大学,2017.
[12]李晓刚,李慧,杨青松,等.杜梨bHLH转录因子家族两成员的序列特征及对非生物胁迫的转录响应[J].江苏农业科学,2017,45(22):40-45.
[13]李永强,应朱,郭卫东,等.樱桃bHLH转录因子家族基因鉴定及表达分析[J].分子植物育种,2018(14):1-13.
[14]黄宁,刘朋,霍俊伟,等.蓝果忍冬果实花青素含量及合成相关基因表达分析[J].南方农业学报,2017,48(7):1139-1147.
[15]何洁,顾秀容,魏春华,等.西瓜bHLH转录因子家族基因的鉴定及其在非生物胁迫下的表达分析[J].园艺学报,2016,43(2):281-294.
[16]裴苓荃.甘薯bHLH基因家族的鉴定与初步分析[D].徐州:江苏师范大学,2017.
[17]SONG X M,HUANG Z N,DUAN W K,et al.Genome-wide analysis of the bHLH transcription factor family in Chinese cabbage(Brassica rapa ssp. pekinensis)[J].Molecular Genetics and Genomies,2014,289(1):77-91.
[18]王翠,兰海燕.植物bHLH转录因子在非生物胁迫中的功能研究进展[J].生命科学研究,2016,20(4):358-364.
[19]刘文文,李文学.植物bHLH转录因子研究进展[J].生物技术进展,2013,3(1):7-11.
[20]WANG WS,ZHUJ,LU Y T.Overexpression of AtbHLH112 suppresses lateral root emergence in Arabidopsis[J].FunctionalPlant Biology,2014,41(4):342-352.
[21]LIU Y,JI X,NIE X,et al.Arabidopsis AtbHLH112 regulates the expression of genes involved in abiotic stress tolerance by bindingto their E-box and GCG-box motifs[J].New Phytologist,2015,207(3):692-709.
[22]YUAN Y,WUH,WANG N,et al.FTT interacts with AtbHLH38 and AtbHLH39 in regulating iron uptake gene expression for ironhomeostasis in Arabidopsis[J].Cell Res,2008,18(3):385-397.
[23]HUQ E,QUAIL P H.PIF4,a phytochrome-interacting bHLH fac-
tor,functions as a negative regulator of phytochrome B signaling in Arabidopsis[J].The EMBO Journal,2002,21(10):2441-2450.
[24]WANG Y J,ZHANGZG,HEXJ,et al.A rice transcription factor OsbHLH1 is involved in cold stress response[J].Theoreticaland Applied Genetics,2003,107(8):1402-1409.
[25]曹福亮.中國银杏[M].南京:江苏科学技术出版社,2002:406.
[26]何昌文,朱丽,沈珊,等.银杏bHLH91转录因子基因的克隆及表达分析[J].广西植物,2018,38(2):202-209.
[27]LIN X,ZHANG J,LI Y,et al.Functional genomics of a living fossil tree,Ginkgo,based on next-generation sequencing technology[J].Physiol Plant,2011,143(3):207-218.
[28]LETUNIC I,DOERKS T,BORK P.SMART:recent updates,new developments and status in 2015[J].Nucleic Acids Res,2015,43(D1):257-260.
[29]BAILEY T L,WILIAMS N,MISLEH C,et al.MEME:discovering and analyzing DNA and protein sequence motifs[J].NucleicAcids Res,2006,34(suppl_2):369-373.
[30]JINJ,TIANF,YANG D C,et al.PlantTFDB 4.0:toward a central hub for transcription factors and regulatory interactions inplants[J].Nucleie Acids Res,2017,45(D1):1040-1045.
[31]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].MolBiol Evol,2011,28(10):2731-2739.
[32]NIU X,GUAN Y,CHEN S,et al.Genome-wide analysis of basic helix-loop-helix(bHLH )transcription factors in Brachypodium dis-tachyon[J].BMC Genomics,2017,18(1):619.
[33]陈红霖,胡亮亮,王丽侠,等.绿豆bHLH转录因子家族的鉴定与生物信息学分析[J].植物遗传资源学报,2017,18(6):1159-1167.
[34]陈堤颖,刘娟,袁媛,等.黄苓bHLH转录因子基因家族生物信息学及表达分析[J].中草药,2018,49(3):671-677.
[35]张子佳,王迪,傅彬英.水稻转录因子bHLH家族基因响应环境胁迫表达谱分析[J].分子植物育种,2008,6(3):425-431.
[36]GONZALEZ A,ZHAO M,LEAVITT J M,et al.Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex in Arabidopsis seedlings[J].Plant J,2008,53(5):814-827.
[37]XIE Y,TAN H,MA Z,et al.DELLA proteins promote anthocyanin biosynthesis via sequestering MYBL2 and JAZ suppressors ofthe MYB/bHLH/WD40 complex in Arabidopsis thaliana[J].MolPlant,2016,9(5):711-721.
[38]DOMBRECHT B,XUE G P,SPRAGUESJ,et al.MYC2 differentially modulates diverse jasmonate-dependent functions in Arabi-dopsis[J].Plant Cell,2007,19(7):2225-2245.
[39]SCHWEIZER F,FERNANDEZ-CALVO P,ZANDER M,et al. Arabidopsis basic helix-loop-helix transcription factors MYC2,MYC3,and MYC4 regulate glucosinolate biosynthesis,insect performance,and feeding behavior[J].Plant Cell,2013,25(8):3117-3132.
[40]GOOSSENS J,MERTENS J,GOOSSENS A.Role and functioning
of bHLH transcription factors in jasmonate signalling[J].J ExpBot,2017,68(6):1333-1347.
[41]PATRA B,PATTANAIK S,SCHLUTTENHOFER C,et al.A network of jasmonate-responsivebHLH factors modulate monoter-penoid indole alkaloid biosynthesis in Catharanthus roseus[J].New Phytol,2018,217(4):1566-1581.
[42]Arce A L,CabelloJ V,Chan R L.Patents on plant transcription factors[J].Recent Pat Biotechnol,2008,2(3):209-217.

