茶碱激活型RNA分子开关的构建及在枯草芽胞杆菌中调节外源基因表达的性能
缪胜男,杨婷尧,崔文璟,周哲敏
茶碱激活型RNA分子开关的构建及在枯草芽胞杆菌中调节外源基因表达的性能
缪胜男,杨婷尧,崔文璟,周哲敏
江南大学 生物工程学院 工业生物技术教育部重点实验室,江苏 无锡 214122
枯草芽胞杆菌在工业生物技术以及合成生物学领域作为一种重要的微生物可广泛用作代谢工程、重组蛋白表达以及新型基因电路的底盘。在中构建基于非编码RNA的高精准调节元件,能够实现不依赖蛋白质因子的基因表达调控,丰富基因表达通用工具。通过基因工程手段,设计了基于茶碱适体域的核糖开关E和适体核酶AZ调节元件,并与不同的内源组成型启动子适配,构建出茶碱激活型基因表达控制元件。测定这两种调节元件与6种组成型启动子组合匹配下报告基因GFP的荧光强度,鉴定并分析各调控元件的工作性能。并进一步以红色荧光蛋白mCherry和普鲁兰酶两种不同的异源蛋白验证核糖开关或适体核酶与启动子的最优组合。结果表明,同一种RNA调节元件与不同启动子组合呈现不同水平的调控效率。在核糖开关与启动子的组合中,启动子PsigW和核糖开关E组合 (sigWE) 对GFP表达的诱导率最高,达到16.8。在适体核酶与启动子的组合中,AZ与启动子P43、PrpoB组合 (P43AZ和rpoBAZ) 的诱导率最高,分别达到了6.1和6.2。进一步验证结果显示,sigWE调控mCherry的诱导率最高 (9.2),而P43E调控普鲁兰酶的诱导率最高 (32.8),产酶水平达到了81 U/mL。核糖开关和适体核酶对GFP、mCherry、普鲁兰酶均能实现调控,但是不同元件组合的调控性能有所差异,对不同基因的调控效果也不尽相同。
核糖开关,适体核酶,枯草芽胞杆菌,基因调控
枯草芽胞杆菌作为一种重要的革兰氏阳性模式菌,是外源基因高效表达、实现工业酶高效生产和代谢工程的理想宿主。枯草芽胞杆菌也可作为合成微生物底盘,用于承载人工设计的基因电路,实现天然系统不能够完成的新功能[1-3]。近年随着遗传工具的不断扩展,诸如各种不同调控类型的启动子、标准化的核糖体结合位点以及部分ρ因子非依赖型终止子的构建、开发和鉴定[4],这一宿主已经被广泛认为是一种能与大肠杆菌相媲美的合成生物学底盘。
细菌通过基因调控来应对自身代谢和周围环境的变化,因此基因调控对于合理应用体内物质和能量具有重要意义[5],同时基因表达调控在蛋白质表达、代谢工程、合成生物学领域也多有应用。已有的基因表达工具多依赖诱导型启动子实现调控,这种系统需要一个或多个辅助蛋白来激活目标基因表达[6]。其中最经典的就是受IPTG诱导的启动子,IPTG通过与转录阻遏蛋白LacI结合,使LacI从操纵位点上脱离下来从而诱导下游基因,但是IPTG诱导对菌体造成生长压力,会阻碍细菌的生长;另一种常用的受木糖诱导的启动子,通过与XylR阻遏蛋白相互作用进行调控,但是木糖诱导调控会参与代谢途径,产生相互干扰。RNA元件因为系统元件小,构成简单,无需蛋白参与逐渐成为研究热点,通过在mRNA上融合适体域,使其对相应配体的存在作出响应,从而调控基因表达[7]。
用于调控原核生物基因表达的非编码RNA元件有多种,其中应用最广泛的有两类:核糖开关(Riboswitch) 和适体核酶(Aptazyme)[8-11]。核糖开关由一个可以响应高亲和力配体的适体域(Aptamer) 和表达平台域组成;通过利用适体域区域固有的三级结构来识别特异性的配体并结合配体[12],能够感知配体的浓度。核糖开关在转录水平和翻译水平均可实现调控[13],转录水平的调控是通过在表达平台域形成终止子或者抗终止子影响RNA聚合酶的功能,从而引起转录的终止或激活[14-15];翻译水平的调控通过配体和适体域的结合改变茎环结构,从而暴露或者隐藏SD序列,激活或终止翻译的进行。核糖开关现已在大肠杆菌、枯草芽胞杆菌等其他菌株中作为调控元件调控外源基因的表达。Mandal等[16]发现并验证了受甘氨酸调控的天然核糖开关,通过配体结合两个适体域后的协同作用来确保多余的甘氨酸能一方面作为柠檬酸循环的碳源,另一方面用来维持足够的氨基酸保证蛋白质的合成。Amy等[17]在蓝藻中通过筛选和合理设计开发了茶碱核糖开关,实现了翻译水平上的调控;Cui等[18]进一步证明茶碱诱导型核糖开关能在枯草芽胞杆菌中被成功激活,同时核糖开关具有配体依赖性。
适体核酶是人工设计出来的由天然或改造后的核酶(Ribozyme) 以及不同适体域组合而成的新型元件。其中,锤头状核酶是小型的自分裂RNA,首次在类病毒和植物病毒的RNA中发现[19],可催化特定磷酸二酯键在转环复制过程中的异构化反应。适体核酶具有作用方式多样、调控严谨、结构高度模块化等特点[20],既能和配体高特异性结合,又有核酶的催化活性。在无配体存在时,不具有或者具有较低的活性,而在配体存在时,配体会与适体核酶上的适体域结合从而诱发核酶的自我裂解作用,由无活性转变为有活性状态,从而发挥对下游基因的调控作用。Wieland等[21]通过在锤头状核酶的茎环Ⅲ上融合茶碱适体域,在大肠杆菌中实现对GFP表达的调控。RNA元件的发现和研究进一步拓展了人们对基因表达调控的认知,而且RNA元件作为一种基因功能研究的工具,对于特异表达启动子的研究和应用具有重要意义。
上游启动子对于核糖开关和适体核酶调控蛋白的性能会产生很大影响。因此本研究通过在枯草芽胞杆菌中将核糖开关和适体核酶与不同的组成型启动子进行匹配,分析启动子兼容性对调节元件的影响,对RNA元件的性质进行分析;同时分析了适体核酶和核糖开关调控不同蛋白的适用性,为进一步研究和应用提供实验依据。研究结果发现茶碱核糖开关和茶碱适体核酶能够与多种来源的内源启动子匹配,但不同启动子与核糖开关和适体核酶匹配后呈现的调控性能有差异;茶碱核糖开关和茶碱适体核酶对GFP、mCherry、普鲁兰酶均能实现调控。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
实验所用质粒详见表1。
1.1.2 主要试剂和仪器
PrimeSTAR®Max DNA 聚合酶购自TaKaRa公司;2*GenRec重组试剂盒购自通用生物系统(安徽) 有限公司;质粒提取试剂盒购自天根生化科技(北京) 有限公司;胶回收试剂盒购自AXYGEN公司;DMSO、茶碱购自生工生物工程(上海) 股份有限公司。PCR仪、蛋白电泳仪购自BIO-RAD公司;分光光度计购自上海美谱达仪器有限公司;多功能酶标仪购自BioTeck公司。
1.2 方法
1.2.1 重组质粒的构建
质粒pBSG03A (由P43启动子控制GFP表达的大肠杆菌-枯草芽胞杆菌穿梭表达载体) 由本实验室保存。以该质粒为模板采用定点突变和Infusion重组的方式构建突变体,所构建的质粒如表1所示,表2为构建相应质粒时的引物。在PCR得到的基因产物中加入Ⅰ酶反应2 h以消除模板,对PCR产物进行纯化后对其进行基因组装,然后转入大肠杆菌感受态细胞进行转化并提取质粒测序,待序列验证正确后,再将其转入枯草芽胞杆菌,进行验证。

表1 本研究所用质粒
1.2.2 茶碱核糖开关的构建
基于茶碱调控的核糖开关(图1) 在未加入茶碱时,SD被隐藏在茎环内,核糖体无法结合SD,基因无法进行表达;加入茶碱配体后,核糖开关构象发生改变,SD被暴露出来,翻译开始进行。根据启动子序列设计引物(表2),以质粒pBSG11/pP43EGFP[18]为模板进行PCR,获得含有不同启动子与茶碱核糖开关E组合匹配的重组表达质粒,将重组表达质粒转入168中,获得含有茶碱核糖开关与不同启动子组合匹配的基因工程菌。

表2 本研究所用引物序列
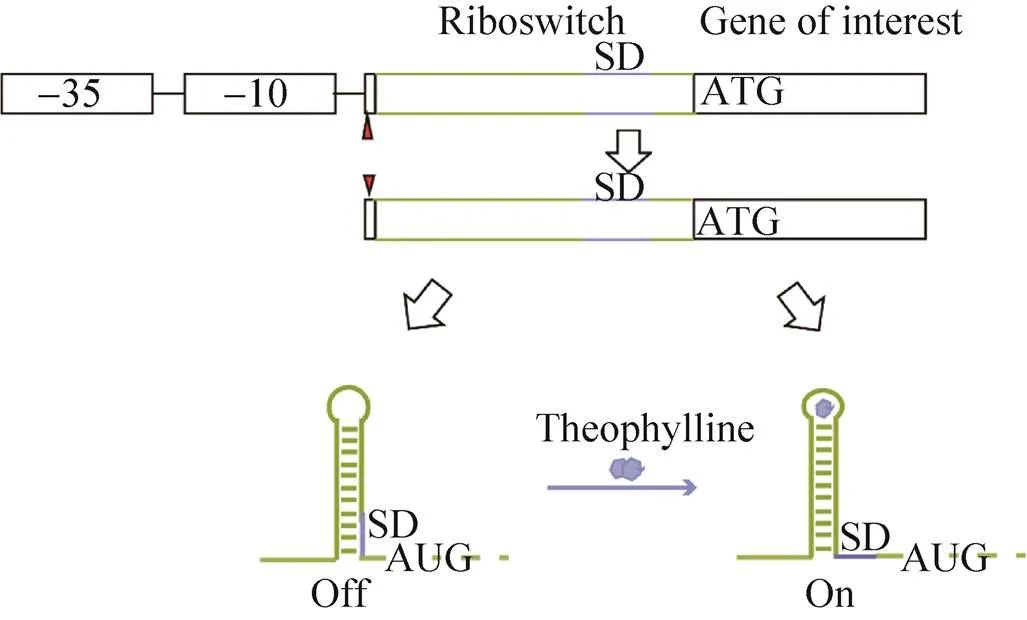
图1 茶碱核糖开关调节基因表达原理示意图
1.2.3 茶碱适体核酶的构建
基于茶碱调控的适体核酶(图2) 在未加入配体时,SD被隐藏在茎环内,核糖体无法结合SD,基因无法进行表达;加入茶碱配体后,构象发生改变,核酶发生自我剪切,SD暴露出来,翻译开始进行。根据Aptamer Database所提供的茶碱适体域序列和来源于曼氏血吸虫的锤头状核酶序列设计适体核酶序列并化学合成,获得含适体核酶序列的质粒pUCSmTheo-AZ。然后利用引物sm-aptazyme-F/sm-aptazyme-R (表2),以pUCSmTheo- AZ为模板进行PCR,获得含有适体核酶序列的片段PAZSDE1,回收并纯化,再以该片段作为大引物,pBSG03A质粒为模板进行PCR,得到重组表达质粒pP43AZGFP-A;以重组表达质粒pP43AZGFP-A为模板,用引物SDE1 CM 6N对通信模块(Communication module,CM)部分进行PCR建库筛选(方法1.2.4) 获得具有调控功能的质粒pP43AZGFP/pP43AZGFP-CM3nt-BG8。再将重组表达质粒转入.168中,即得基因工程菌PBP43AZGFP。再根据启动子序列设计引物(表2),以质粒pP43AZGFP为模板,进行PCR,获得含有不同启动子与茶碱适体核酶AZ组合匹配的重组表达质粒,将重组表达质粒转入168中,获得含有茶碱适体核酶与不同启动子组合匹配的基因工程菌。

图2 茶碱适体核酶调控基因表达原理示意图
1.2.4 茶碱适体核酶通信模块文库的构建和筛选
茶碱适体核酶的适体域和核酶结构依靠两者之间的通信模块连接[22]。适体域结合茶碱后,将RNA二级结构的变化通过CM传递给核酶,从而使核酶从无活性的状态转变为活化的自裂解状态,释放被封闭的SD序列,从而启动蛋白质的翻译。本研究中为了获得高效的CM,设计了3–5 nt的随机序列作为CM,构建CM文库。以重组表达质粒pP43AZGFP-A为模板,用引物SDE1 CM 6N (表2) 进行PCR。PCR产物用Ⅰ消化2 h,转入JM109,收集平板上所有菌落,提取总质粒,转入168。37 ℃过夜培养,挑取不同单菌落于96深孔板中培养12 h,取样,检测600和GFP表达;然后在96深孔板中添加终浓度4 mmol/L的茶碱,再培养12 h,取样,检测600和GFP表达。
1.2.5 绿色荧光蛋白GFP和红色荧光蛋白mCherry报告基因表达水平的测定
将测序正确的枯草芽胞杆菌在平板划线,37 ℃过夜培养。挑取单菌落接种到含有卡那霉素的5 mL LB培养基中,37 ℃、200 r/min过夜培养,再以1%的接种量将种子液分别接至含有2% DMSO、2% DMSO和终浓度4 mmol/L茶碱的50 mL LB培养基中扩大培养,24 h后取发酵液,12 000 r/min离心5 min收集菌体,PBS缓冲液(0.01 mol/L,pH 8.0) 洗涤3次,再用PBS将其悬浮并稀释一定浓度,取200 μL至96孔板,检测600和GFP荧光强度。GFP荧光强度在激发光495 nm、发射光525 nm处进行检测。mCherry的荧光强度在激发光587 nm、发射光610 nm处进行检测。荧光强度以a.u.表示。
1.2.6 普鲁兰酶的表达及酶活的测定
将测序正确的枯草芽胞杆菌进行平板划线,挑取单菌落PBP43EPUL、PBsigWEPUL、PBP43AZPUL、PBrpoBAZPUL于含有卡那霉素的5 mL LB培养基中,37 ℃、200 r/min过夜培养,再以1%的接种量将种子液分别接至含有2% DMSO、2% DMSO和终浓度4 mmol/L茶碱的50 mL的LB培养基中扩大培养,24 h后取样检测600和基因表达。样品于12 000 r/min离心5 min分别收集 菌体和上清,将菌体PBS缓冲液洗涤3次,再用含溶菌酶的TE缓冲液(10 mmol/L Tris-HCl, 1 mmol/L EDTA,pH 8.0) 将其悬浮,在37 ℃下反应30 min,并进行超声破碎,离心取上清,获得酶液。
取50 μL 底物、40 μL缓冲液和10 μL适当稀释酶液混合,65 ℃下反应15 min,加入200 μL 3,5-二硝基水杨酸溶液(DNS),煮沸5 min显色,待冷却后用蒸馏水补足体积至500 μL,测定550,每个反应做3个平行,每组测定做适当空白。将每分钟产生与1 μmol葡萄糖相同的还原力所需酶量定义为1 U。
1.2.7 SDS-PAGE
将所取的培养液12 000 r/min离心5 min收集菌体,PBS缓冲液洗涤3次,再用含有1 mg/mL溶菌酶的TE 缓冲液悬浮,37 ℃温育30 min,超声破碎适当时间,12 000 r/min离心5 min;取100 μL离心上清,加入25 μL 5×上样缓冲液,沸水浴10 min。使用5%的浓缩胶和12%的分离胶进行蛋白分离,考马斯亮蓝R250染色显示条带。
In-gel fluorescence[23]:样品12 000 r/min离心5 min收集菌体,PBS缓冲液洗涤3次,再用含有1 mg/mL溶菌酶的TE缓冲液悬浮,37 ℃温育30 min,超声破碎适当时间,12 000 r/min离心 5 min;取100 μL离心上清,加入25 μL 5×上样缓冲液。使用不含SDS的蛋白胶进行分离,在紫外光下成像,可特异性地显示目的蛋白,过滤掉其他不发光的背景蛋白。
2 结果与分析
2.1 不同组成型启动子匹配茶碱核糖开关后调控报告基因GFP表达的性能
为了深入探讨茶碱核糖开关在中对不同内源启动子的兼容程度,本研究从DBTBS库中选择了6种序列明确的内源启动子PrpoB[24]、Pylbp[25]、PspoVG[26]、PminC[27]、PsigW[28]、PyqeZ[28]与茶碱核糖开关E融合,转化后构建出重组菌PBrpoBEGFP、PBylbpEGFP、PBspoVGEGFP、PBminCEGFP、PBsigWEGFP、PByqeZEGFP。经过4 mmol/L的茶碱处理24 h后,检测标准化的GFP荧光强度(F.I/600),通过与只经过溶剂DMSO处理的对照组对比,检测诱导率(诱导后基因表达/未诱导基因表达),作为茶碱核糖开关功能的主要评价参数。结果如图3所示,茶碱核糖开关与6种组成型启动子融合均能够改变组成型表达特点,与仅用DMSO处理的对照组相比,经过4 mmol/L的茶碱处理后GFP的表达强度均有所提高,6株重组菌呈现出不同水平的诱导率。其中核糖开关E与启动子PrpoB匹配构成的调控元件rpoBE介导的GFP表达水平最高,处理24 h后达到(2 047 620±9 570) a.u.,与核糖开关E和启动子P43构成的调控元件P43E[18]相比,基因表达水平相当,但诱导率略低;而sigWE介导的表达调控呈现的诱导率最高,达到16.8。
随后,为了确证GFP荧光强度检测结果,利用SDS-PAGE和In-gel fluorescence法检测茶碱处理前后GFP的蛋白表达水平,结果如图4所示,经过4 mmol/L茶碱处理24 h后,GFP相对于未处理的对照组表达水平均明显提升,并且P43E和rpoBE介导的GFP表达水平显著高于其他含有相同核糖开关所介导的GFP表达水平,与图3得到的结果一致。这一结果充分表明茶碱核糖开关E能够与多种来源的内源启动子匹配,匹配后可实现基因的诱导调控,但不同启动子与核糖开关E匹配后呈现的调控性能有差异。
3.1.3患者因素 在X线摄片过程中,由于患者有外伤,疼痛较重,不能正确配合技术人员摆位,只能摄取一些被动体位。或者是一些患者身上有不能取下的金属异物或不能去除干净的膏药,导致伪影的发生。还有一些患者不能控制自己的运动,产生运动伪影。特别是一些脑外伤合并胸部外伤的患者,他们摄片时往往处于一种浅而短的呼吸状态,这样胸廓运动造成伪影使X线片模糊不清[2]。这些都会使图像质量下降,从而使诊断的正确性降低。
2.2 适体核酶与不同内源组成型启动子的适配及调控功能鉴定
为了构建受适体核酶调控的基因表达元件,需设计茶碱适体域与核酶的融合元件。在此,需要设计并筛选适宜于连接茶碱适体域和锤头状核酶的通信模块(CM)。通过构建由3–5 nt随机序列构成的CM文库,系统筛选与此两个部分兼容性强的模块。以质粒pP43AZGFP-A为模板,用含随机序列的引物SDE1 CM 6N构建文库。高通量筛选结果显示,获取了含有不同调控功能的适体核酶(图5A),其中选择含有元件BG8 (5′-GGT···32 nt···TCT-3′)的菌株PBP43AZGFP作为后续的研究菌株。将元件BG8在后续的研究中命名为适体核酶AZ。同时引入Zhong等[29]使用的WHBS (加权氢键得分) 参数进行分析。在此,利用这一参数探讨枯草芽胞杆菌中通信模块的结构特征与本底表达水平之间的相关性。结果如图5B所示,WHBS与本底表达在0.05水平上显著相关,因此可通过WHBS预测含有适体核酶菌株的本底表达来进行理性设计。

图3 启动子-核糖开关组合调节GFP表达

图4 SDS-PAGE和In-gel fluorescence检测启动子-核糖开关组合调控GFP表达

图5 通信模块对适体核酶功能的影响
为了进一步探讨茶碱适体核酶在中对不同内源启动子的兼容性,本研究选择与上述相同的6种s内源启动子PrpoB、Pylbp、PspoVG、PminC、PsigW、PyqeZ与适体核酶AZ融合,转化后构建出重组菌PBrpoBAZGFP、PBylbpAZGFP、PBspoVGAZGFP、PBminCAZGFP、PBsigWAZGFP、PByqeZAZGFP。经过4 mmol/L的茶碱处理24 h后,检测GFP荧光强度(F.I/600),通过与只经过溶剂DMSO处理的对照组相比(本底渗漏表达水平),计算诱导率,作为茶碱适体核酶功能的主要评价参数。结果如图6所示,与适体核酶适配后,6种组成型启动子均呈现受茶碱激活的调控特征,与仅用DMSO处理的对照组相比,经过4 mmol/L的茶碱处理后GFP的表达强度均有不同程度提高,并呈现出不同水平的诱导率。其中适体核酶AZ与启动子PrpoB匹配构成的调控元件rpoBAZ介导的GFP表达水平最高,处理24 h后为 (172 773±2 625) a.u.,且rpoBAZ介导的表达调控呈现的诱导率也最高,达到6.2。适体核酶AZ与启动子Pylbp匹配构成的调控元 件ylbpAZ介导的GFP的本底表达水平最低,为 (4 637±1 119) a.u.。
随后,为了确证GFP荧光强度检测结果,利用SDS-PAGE和In-gel fluorescence法检测经茶碱处理后GFP的表达水平,结果如图7所示,与未处理的对照组相比,经过4 mmol/L茶碱处理24 h后GFP的表达水平均显著提高,且P43AZ和rpoBAZ介导的GFP表达水平显著高于其他启动子-适体核酶元件所介导的GFP表达水平,与图6得到的结果一致。这一结果表明茶碱适体核酶AZ能够与多种来源的内源启动子匹配,匹配后可实现基因的诱导调控,但不同启动子与适体核酶AZ匹配后呈现的调控性能有差异。
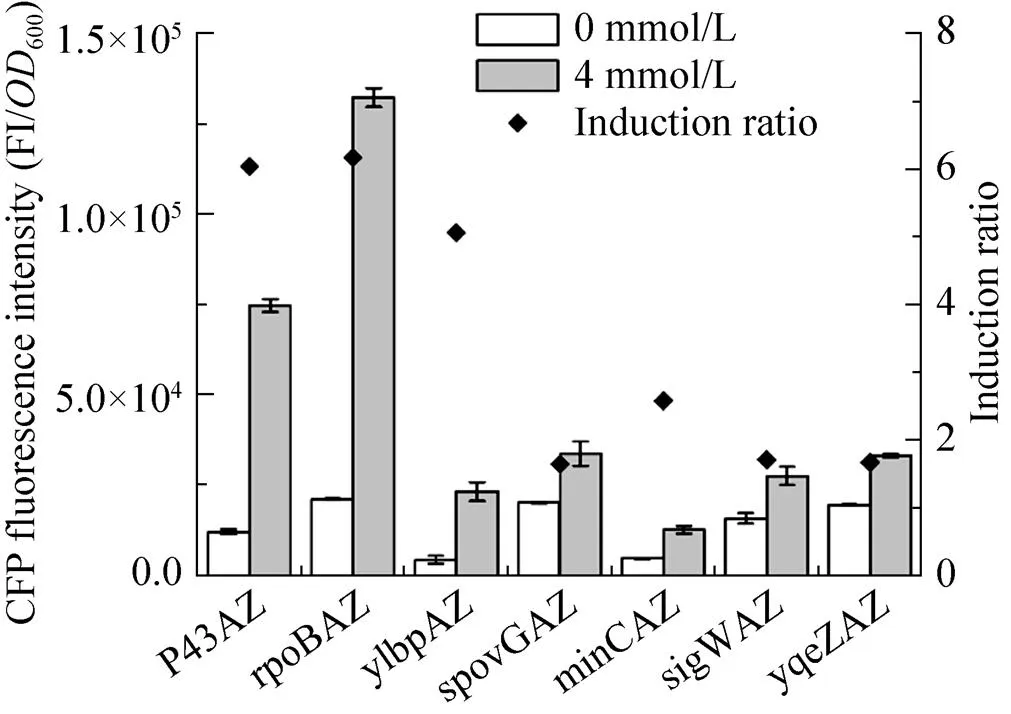
图6 启动子-适体核酶组合调控GFP表达

图7 SDS-PAGE和In-gel fluorescence检测启动子-适体核酶组合调控GFP表达
2.3 核糖开关和适体核酶调控mCherry的表达性能
为了研究核糖开关和适体核酶对不同基因的兼容性,分别选取上述研究中表达调控元件P43E、sigWE、P43AZ、 rpoBAZ用来进一步分析这些元件的适用性和兼容性,评价核糖开关以及人工适体核酶基因表达元件工作性能。首先,使用mCherry作为检验蛋白,与上述元件进行融合,构建菌株PBP43EmCherry、PBsigWEmCherry、PBP43AZmCherry、PBrpoBAZmCherry。结果如图8A所示,P43E介导的mCherry的荧光强度最高,达到 (128 607±984) a.u.,而sigWE元件介导的mCherry表达的诱导率最高,达到9.2。且核糖开关和适体核酶对mCherry的调控在蛋白表达水平和诱导率水平上与GFP相比趋势相似。SDS-PAGE和In-gel fluorescence (图8B、8C) 结果显示,与只经过DMSO处理的对照组相比,加入4 mmol/L茶碱后,均能启动mCherry的表达。这一结果表明茶碱激活型核糖开关与适体核酶能够与多种来源的内源启动子匹配组合,实现基因的诱导调控,并且不同启动子和元件匹配后呈现的调控效果不同。核糖开关调控的外源基因激活后表达水平较适体核酶更高,而适体核酶调控的基因表达严谨性较核糖开关更强。
2.4 茶碱核糖开关和适体核酶调控元件对普鲁兰酶的调控性能
为了扩展茶碱调节的RNA元件应用范围,检验元件的可靠性。将普鲁兰酶融合到P43E、sigWE和P43AZ、rpoBAZ的下游,构建出菌株PBP43EPUL、PBsigWEPUL、PBP43AZPUL、PBrpoBAZPUL。通过测定细胞破碎上清中的酶量和酶活性变化,检测这4种元件调控普鲁兰酶的性能。结果如图9所示,核糖开关E与P43启动子匹配构成的调控元件P43E介导的普鲁兰酶的表达量最高,产酶水平也最高,为 (80.99±2.84) U/mL,诱导率也最高,达到32.8。SDS-PAGE结果显示,在97.2 kDa下方有一条表达条带(图9A) 为普鲁兰酶目的条带,其中表达量最高的是诱导后的PBP43EPUL,进一步分析发现,尽管不同组合的RNA元件调控的性能有差异,但是核糖开关和适体核酶对于酶的表达均能实现调控,启动子P43介导的RNA元件结果进一步表明,融合同一组成型启动子的条件下,核糖开关诱导后的激活水平较适体核酶更高,适体核酶调控的严谨性较核糖开关更好。

图9 启动子-核糖开关组合和启动子-适体核酶组合调控PUL的表达
3 结论
本文成功构建了核糖开关和适体核酶调控表达的枯草芽胞杆菌,并对它们的性质进行了分析。核糖开关和适体核酶对于不同的启动子均具有调控功能,且从本底基因表达、诱导后基因表达、诱导率的水平综合评价,核糖开关的调控范围大,诱导后GFP荧光强度可高至(2 047 620±9 570) a.u.,诱导率最高可至16.8,而适体核酶调控的严谨性好,本底荧光强度可低至(4 637±1 119) a.u.。
核糖开关和适体核酶均能够在匹配组成型启动子后被茶碱激活,启动多种外源基因的表达。两者介导的基因表达调控性能有所差异。在同一组成型启动子条件下,匹配核糖开关的调控元件激活后能够输出较高的蛋白表达水平,而自身同样具有较高渗漏表达的特点;匹配适体核酶的元件受茶碱激活前后呈现较低的本底表达,同时外源基因表达水平也相对较低。核糖开关高表达、高诱导率的特点可以有效地驱动枯草芽胞杆菌中的基因调控,而适体核酶低渗漏表达的特点对于设计枯草芽胞杆菌中更复杂的遗传线路具有重要意义。
[1] Liu YF, Liu L, Li JH, et al. Synthetic biology toolbox and chassis development in. Trends Biotechnol, 2019, 37(5): 548–562.
[2] Liu L, Liu YF, Shin HD, et al. Developingspp. as a cell factory for production of microbial enzymes and industrially important biochemicals in the context of systems and synthetic biology. Appl Microbiol Biotechnol, 2013, 97(14): 6113–6127.
[3] Guiziou S, Sauveplane V, Chang HJ, et al. A part toolbox to tune genetic expression in. Nucleic Acids Res, 2016, 44(15): 7495–7508.
[4] Nguyen QA, Schumann W. Use of IPTG-inducible promoters for anchoring recombinant proteins on thespore surface. Prot Expr Purif, 2014, 95: 67–76.
[5] Han XD, Liu W, Wu CX, et al. Riboswitch in gene expression regulation. Chin J Biochem Mol Biol, 2011, 27(12): 1094–1100 (in Chinese).韩祥东, 刘薇, 吴存祥, 等. 核糖开关与基因表达调控. 中国生物化学与分子生物学报, 2011, 27(12): 1094–1100.
[6] Seeliger JC, Topp S, Sogi KM, et al. A riboswitch-based inducible gene expression system for Mycobacteria. PLoS ONE, 2012, 7(1): e29266.
[7] Wang SW, Li XX. The structures and mechanisms of riboswitches.Biotechnol Bull, 2010, (5): 16–22, 33 (in Chinese).王少伟, 李锡香. 核糖开关的结构和调控机理. 生物技术通报, 2010, (5): 16–22, 33.
[8] Nudler E, Mironov AS. The riboswitch control of bacterial metabolism. Trends Biochem Sci, 2004, 29(1): 11–17.
[9] Wieland M, Berschneider B, Erlacher MD, et al. Aptazyme-mediated regulation of 16S ribosomal RNA. Chem Biol, 2010, 17(3): 236–242.
[10] Nomura Y, Kumar D, Yokobayashi Y. Synthetic mammalian riboswitches based on guanine aptazyme. Chem Commun, 2012, 48(57): 7215–7217.
[11] Wickiser JK, Winkler WC, Breaker RR, et al. The speed of RNA transcription and metabolite binding kinetics operate an FMN riboswitch. Mol Cell, 2005, 18(1): 49–60.
[12] Serganov A, Patel DJ. Molecular recognition and function of riboswitches. Curr Opin Struct Biol, 2012, 22(3): 279–286.
[13] Romby P, Charpentier E. An overview of RNAs with regulatory functions in gram-positive bacteria. Cell Mol Life Sci, 2010, 67(2): 217–237.
[14] Quarta G, Kim N, Izzo JA, et al. Analysis of riboswitch structure and function by an energy landscape framework. J Mol Biol, 2009, 393(4): 993–1003.
[15] Fauzi H, Agyeman A, Hines JV. T box transcription antitermination riboswitch: Influence of nucleotide sequence and orientation on tRNA binding by the antiterminator element. Biochim Biophys Acta, 2009, 1789(3): 185–191.
[16] Mandal M, Lee M, Barrick JE, et al. A glycine-dependent riboswitch that uses cooperative binding to control gene expression. Science, 2004, 306(5694): 275–279.
[17] Ma AT, Schmidt CM, Golden JW. Regulation of gene expression in diverse cyanobacterial species by using theophylline-responsive riboswitches. Appl Environ Microbiol, 2014, 80(21): 6704–6713.
[18] Cui WJ, Han LC, Cheng JT, et al. Engineering an inducible gene expression system forfrom a strong constitutive promoter and a theophylline-activated synthetic riboswitch. Microb Cell Fact, 2016, 15(1): 199.
[19] Forster AC, Symons RH. Self-cleavage of plus and minus RNAs of a virusoid and a structural model for the active sites. Cell, 1987, 49(2): 211–220.
[20] Win MN, Smolke CD. A modular and extensible RNA-based gene-regulatory platform for engineering cellular function. Proc Natl Acad Sci USA, 2007, 104(36): 14283–14288.
[21] Wieland M, Hartig JS. Improved aptazyme design andscreening enable riboswitching in bacteria. Angew Chem Int Ed Engl, 2008, 47(14): 2604–2607.
[22] Win MN, Smolke CD. Higher-order cellular information processing with synthetic RNA devices. Science, 2008, 322(5900): 456–460.
[23] Wittig I, Karas M, Schägger H. High resolution clear native electrophoresis for in-gel functional assays and fluorescence studies of membrane protein complexes. Mol Cell Proteom, 2007, 6(7): 1215–1225.
[24] Boor KJ, Duncan ML, Price CW. Genetic and transcriptional organization of the region encoding the βsubunit ofRNA polymerase. J Biol Chem, 1995, 270(35): 20329–20336.
[25] Yu XX, Xu JT, Liu XQ, et al. Identification of a highly efficient stationary phase promoter in. Sci Rep, 2015, 5: 18405.
[26] Eymann C, Mittenhuber G, Hecker M. The stringent response, σH-dependent gene expression and sporulation in. Mol Gen Genet, 2001, 264(6): 913–923.
[27] Lee S, Price CW. The minCD locus oflacks thedeterminant that provides topological specificity to cell division. Mol Microbiol, 1993, 7(4): 601–610.
[28] Cao M, Wang T, Ye R, et al. Antibiotics that inhibit cell wall biosynthesis induce expression of theσWand sσMregulons. Mol Microbiol, 2002, 45(5): 1267–1276.
[29] Zhong GC, Wang HM, Bailey CC, et al. Rational design of aptazyme riboswitches for efficient control of gene expression in mammalian cells. eLife, 2016, 5: e18858.
Construction and application of theophylline-activated RNA switches in the regulation of expression of recombinant proteins in
Shengnan Miao, Tingyao Yang, Wenjing Cui, and Zhemin Zhou
Key Laboratory of Industrial Biotechnology (MOE), School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu, China
can be widely used as an important microorganism for metabolic engineering and recombinant proteins expression in industrial biotechnology and synthetic biology. However, it is difficult to make accurate regulation of exogenous gene by biological tools in, which limits the application ofin synthetic biology. The purpose of this study is to develop regulatory tools for precise control of gene expression by using non-coding RNAs, by which the activation of heterologous gene could be achieved without the auxiliary protein factors. We constructed the synthetic riboswitch E and aptazyme AZ using the theophylline aptamer. Six different native promoters fromwere functionally adapted with the E and AZ to fabricate an array of novel regulatory elements activated by theophylline. Then, we determined the performance of these elements using green fluorescence protein as reporter, and then further verified using red fluorescence protein and pullulanase as cargo proteins.Results showed that the same kind of RNA elements with different promoters showed different levels of efficiency. Promoter PsigWand E combination (sigWE) had the highest induction rate in. Compared with the control group, it can produce the induction rate of 16.8. Promoter PrpoBand AZ combination (rpoBAZ) showed the highest induction rate of 6.2. SigWE mediated mCherry induction rate was 9.2, and P43E mediated pullulanase induction rate was 32.8, in which enzyme activity reached 81 U/mL. This study confirmed that GFP, mCherry and pullulan can all be regulated by riboswitch and aptazyme, but there were differences between different combinations of promoters with RNA regulators.
riboswitch, aptazyme,, gene regulation
January 21, 2019;
March 19, 2019
Supported by: National Natural Science Foundation of China (No. 21878125).
Zhemin Zhou. Tel: +86-510-85325210; Fax: +86-510-85197551; E-mail: zhmzhou@jiangnan.edu.cn
国家自然科学基金(No. 21878125) 资助。

缪胜男, 杨婷尧, 崔文璟, 等. 茶碱激活型RNA分子开关的构建及在枯草芽胞杆菌中调节外源基因表达的性能. 生物工程学报, 2019, 35(8): 1478–1490.Miao SN, Yang TY, Cui WJ, et al. Construction and application of theophylline-activated RNA switches in the regulation of expression of recombinant proteins in Bacillus subtilis. Chin J Biotech, 2019, 35(8): 1478–1490.
(本文责编 陈宏宇)

