基于卷积神经网络的细胞图像分割与类型判别
胡威 汪春梅 张见
摘 要: 组织细胞图像形态各异、大小不一、纹理变化多样等特点,导致难以精准地分割细胞区域的问题,对此提出了一种基于卷积神经网络(CNN)和边缘聚类方法的新算法.对原始切片采用染色校正预处理,提高色彩对比度,利用CNN得到初步分割结果,结合边缘聚类方法提升初步分割结果的连续性和完整性.在此基础上,结合计算机视觉技术,获得分割图像中细胞颗粒的基本属性特征,并使用Softmax分类器判别细胞类型.实验结果表明:相较于经典的卷积神经网络、阈值分割、模糊聚类等细胞图像分割算法,该算法在分割结果的完整度方面提升了6.15个百分点.
关键词: 卷积神经网络(CNN); 边缘聚类; 图像分割; 分类判别
中图分类号: TP 399文献标志码: A文章编号: 1000-5137(2019)01-0106-07
Abstract: The characteristics of tissue cells in different shapes,sizes and varied textures make it difficult to segment cell areas precisely.We propose a new algorithm for the segmentation of cell images combined with convolutional neural networks(CNN) and edge clustering method.The original slices were stained with pretreatment to improve the color contrast.The convolutional neural network was used to obtain the initial segmentation results.Edge clustering method was used to improve the continuity and integrity of the initial segmentation results.On this basis,we combine computer vision techniques to obtain the basic attribute features of cell particles in the segmented images.And Softmax classifier was used to determine the cell type.The results of experiments show that compared with classic convolutional neural networks,threshold segmentation,fuzzy clustering and other cell image segmentation algorithms,the cell segmentation method proposed in this paper improves the integrity of the segmentation result by 6.15%.
Key words: convolutional neural networks(CNN); edge clustering; image segmentation; classification discrimination
0 引 言
細胞图像分割是诊断环节中的基础和关键,细胞图像分割的质量直接影响诊断结果.然而常规组织细胞染色切片图往往会受颜色、噪声、光照变化等多种外界因素干扰,并且细胞颗粒往往存在形态各异、大小不一、纹理变化多样、边界模糊、重叠交织、细胞区域与背景区域对比不明显等情况,导致难以精准地分割细胞区域[1].
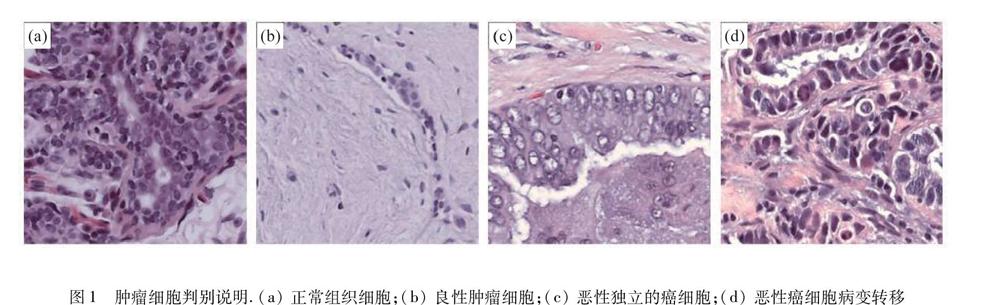
在目前较流行的医学影像诊断技术中,根据医学细胞图像判别组织生理状况,有着不可替代的参考价值,并被应用于各个方面.例如:病理学家可根据细胞面积大小判别其生长周期,观察细胞分裂、凋亡等相关生理周期;依据细胞核相对整个细胞面积占比,判别其是否发生病变;观察组织细胞的聚集交织程度,评判其是否形成肿瘤;观察组织细胞间疏密程度,判断其是否发生病变转移;依据各细胞间平均面积,预测细胞凋亡时间等.目前病理学家对癌症病变的判别主要依据为细胞颗粒间的大小差异,一般状况下,相较于正常组织细胞,癌细胞颗粒较大,核质比较高,如图1所示[2].
1 卷积神经网络简介
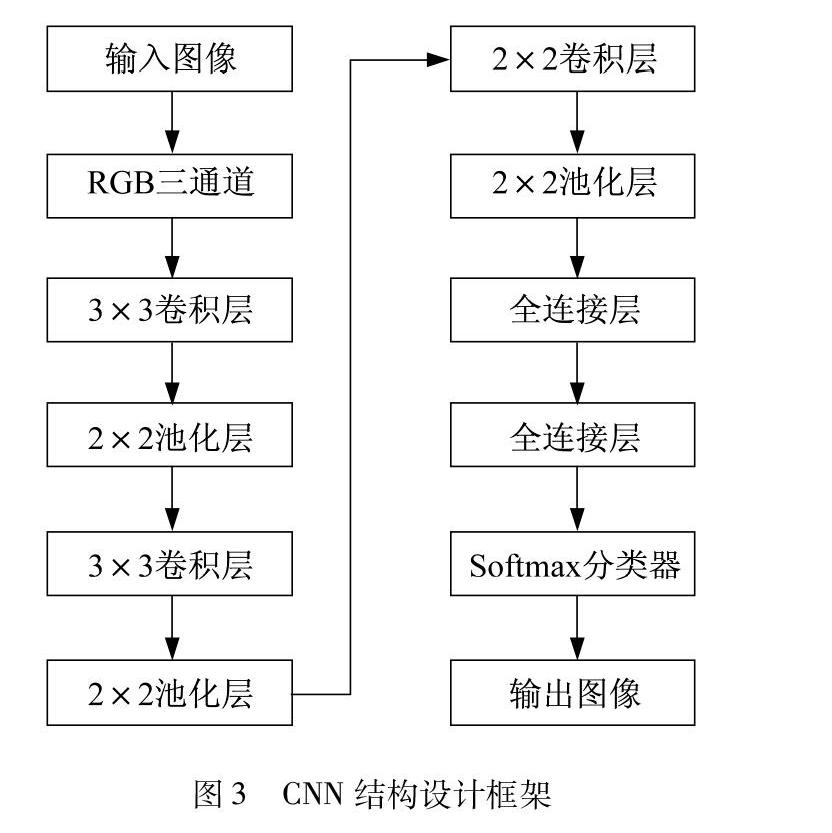
CNN是一种多层的神经网络结构,其中每层由多个二维平面网络结构组成,包含了若干独立神经元.CNN一般由卷积层、池化层以及全连接层构成.基于CNN在特征的提取分析方面具备如下优势:1) 能有效地将特征提取和分类判别结合到同一网络结构中进行同步训练;2) 以图片样本可直接作为网络输入,且以卷积核对图片样本进行遍历卷积计算,最大限度保留原始图像的数据信息,提高特征提取的有效性;3) 网络结构对输入图像中产生的平移旋转、局部扭曲等特殊变化具备很强的适应性[7].
卷积层的每个节点连接的是上层输入的邻域子图,用于表达图像的局部特征.
池化层的处理是对卷积之后得到的特征进行聚类整合,包括平均池化和最大池化两种方式,其作用是统计各个区域相似特征的维数,通过降低特征维数,避免实验数据结果出现过拟合的情况,在局部感受域范围内,计算其最大值或平均值,作为网络节点的输出值.
全连接层的作用是表示输出特征,通常由特征映射构成.特征输出向量的维数一般与最后一个特征映射输出的网络节点数量相同,利用输出的特征向量训练分类器模型实现对目标类别的辨识.
2 边缘聚类方法
邊缘聚类方法的核心思想是依据CNN分割的初步结果,确定边缘像素的位置和颜色特征,然后将边缘集合聚类为位置邻近和颜色相似的各个边缘子集,最后进行边界扩充,以获得完整连续的细胞分割效果,提升分割结果的完整性和连续性[8].具体操作大致分为3个步骤[9]:
1) 计算相邻边缘节点的色差;
2) 计算任意两个子集之间的最小位置差和最大颜色差;
3) 计算任意两个子集之间的平均位置差和平均颜色差.
本研究采用分步边缘聚类方法,使聚类结果逐渐逼近目标结果,通过大量实验确定相关阈值.该方法虽然使阈值个数有所增加,但可以简化聚类结果,提升算法的整体效果.
3 图像分割实验
3.1 实验过程
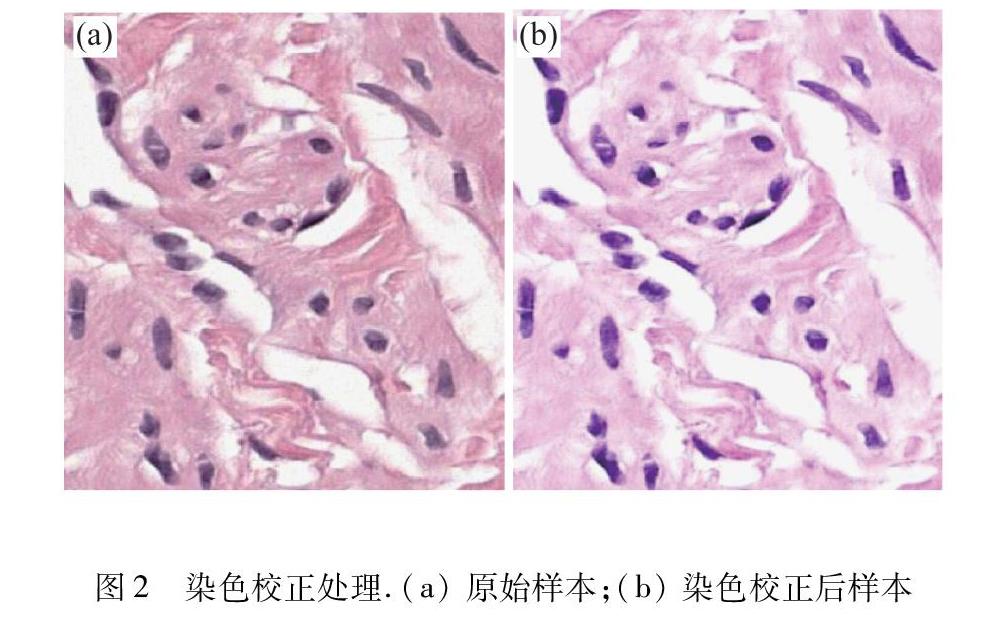
本实验采用荷兰Radboud大学医学中心公开数据集中的CAMELYON17数据,全幻灯片图像(WSI)以TIFF格式展示,病变级别注释以XML文件形式表示.选择有标注的50张由苏木精-伊红染色的WSI,每张图片颜色由RGB三通道合成.从原始样本中统一切割出256 pixels×256 pixels大小的样本图,从其中40张样本图中获得2621440个样本作为训练数据集,剩下的10张中获得655360个样本用作评估算法的测试集.实验验证邻域边长取8 pixels效果较为理想.对获得的样本进行染色校正预处理,结果如图2所示.
3.2 实验结果
为评价本算法的分割效果,选取一些较为常用的图像分割算法与本算法结果进行比较,各算法分割结果对比如图4所示.其中,图4(a)为原始细胞图像真实值;图4(b)是基于模糊均值聚类算法的分割效果[10],即用隶属度确定每个像素点所属类型的一种聚类算法,可用于细胞的分割研究,过程原理与分类器相似;图4(c)是基于阈值分割法(OTSU)的区域图像分割结果[11],该方法广泛应用于对自然目标图像的分割;图4(d)是基于统计直方图全局最小均方误差的图像分割效果,该方法也可应用于细胞分割领域;图4(e)是利用CNN对原始图像样本进行特征提取,从而实现细胞图像分割的方法,该方法考虑到网络深度对实验分割的影响;图4(f),(g)分别是利用vgg16和vgg19网络结构对原始图像样本进行图像分割的效果;图4(h)是本算法的图像分割效果.
4 类型判别
在实际临床医学诊断过程中,组织细胞形态、大小、色泽纹理等变化往往是医生判别其生理结构的重要依据.例如较正常组织细胞,恶性肿瘤细胞往往在形态尺寸上较大,细胞核面积占比也较大,颜色较深,各细胞之间重叠交杂严重,并多以抱团聚集状态呈现,在医学成像中区分度较高,判别较明显.基于上述细胞图像实现的分割效果,选取了细胞颗粒的面积和周长两种特征作为判别的客观依据,综合评估细胞尺寸大小,以实现对细胞生理状态二分类.实验过程中结合了目前较为流行的计算机视觉技术,以评判预测细胞生理状态种类.具体做法大致分为3步:
1) 为便于获取细胞颗粒基本属性,将原始切片样本以1∶20比例进行缩放,图5(a)为原始细胞图像按比例放大后的效果图;对分割后的结果进行灰度二值化反转,如图5(b)所示;再利用分割后的细胞图像结合OpenCV平台中findcontours函数实现特征轮廓描绘,为便于区分细胞颗粒,在显示输出中随机生成不同颜色的轮廓描绘曲线,中心点为颗粒计数,如图5(c)所示.以此获取细胞颗粒面积、周长特征属性,用于类型判别.
2) 将获得的细胞颗粒的周长和面积数据构成二维特征矩阵,送入支持向量机中训练,该实现过程结合了Matlab软件中libsvm函数求解一个线性约束的凸二次规划问题,实验中选取常用的径向基函数(RBF)作为核函数,惩罚因子c和g分别取1.0和0.5.
3) 将输出类别标签设为阴性A(-)、阳性B(+),以实现细胞大小二分类,为判别细胞生理形态的好坏提供客观数据结果.其中:A(-)类为正常细胞形态,大小适中;B(+)类为病变细胞,尺寸较大.分类数据结果以混淆矩阵形式给出,如表2所示.
5 结 论
提出了基于CNN和边缘聚类的细胞图像分割方法,利用CNN在目标识别、图像分类等特征提取方面具备较高准确度,以及边缘聚类方法在图像边界信息获取和相融相似区域上的处理优势,并利用与染色校正相结合的方法,增强颜色对比度,进一步提升了分割算法的准确度.实验结果表明:本算法对细胞图像具有较好的分割效果.此外,还结合计算机视觉技术,对细胞属性进行二分类,为病理学家后续的临床诊断提供定量的分析结果,降低主观因素造成的诊断偏差,提升诊断效率.
参考文献:
[1] STEGMAIER J,SPINA T V,FALCAO A X,et al.Cell segmentation in 3D confocal images using supervoxel merge-forests with CNN-based hypothesis selection [C]//2018 IEEE 15th International Symposium on Biomedical Imaging.Washington D.C.:IEEE,2018:1-5.
[2] ARAJO T,ARESTA G,CASTRO E,et al.Classification of breast cancer histology images using convolutional neural networks [J].Public Library of Science One,2017,12(6):e0177544.
[3] XU J,LUO X,WANG G,et al.A deep convolutional neural network for segmenting and classifying epithelial and stromal regions in histopathological images [J].Neurocomputing,2016,191:214-223.
[4] SONG Y,LING Z,CHEN S,et al.Accurate segmentation of cervical cytoplasm and nuclei based on multiscale convolutional network and graph partitioning [J].IEEE Transaction on Bio-medical Engineering,2015,62(10):2421-2433.
[5] HATIPOGLU N,BILGIN G.Classification of histopathological images using convolutional neural network [C]//Proceeding of International Conference on Image Processing Theory Tools and Applications.Paris:IEEE,2014:1-5.
[6] RUIFROK A C,JOHNSTON D A.Quantification of histochemical staining by color deconvolution [J].Analytical and Quantitative Cytology and Histology,2001,23(4):291-299.
[7] 杨金鑫,杨辉华,李灵巧.结合卷积神经网络和超像素聚类的细胞图像分割方法 [J].计算机应用研究,2018,35(5):1569-1572,1577.YANG J X,YANG H H,LI L Q.Cell image segmentation method combining convolutional neural network and superpixel clustering [J].Application Research of Computers,2018,35(5):1569-1572,1577.
[8] ZHANG R,LI L,BAO C M,et al.The community detection algorithm based on the node clustering coefficient and the edge clustering coefficient [C]//Proceeding of the 11th World Congress on Intelligent Control and Automation.Shenyang:IEEE,2014:1-5.
[9] 付慧,刘峡壁,贾云得.用于文本区域提取的边缘像素聚类方法 [J].计算机辅助设计与图形学学报,2006,18(5):729-734.FU H,LIU X B,JIA Y D.Edge pixel clustering method for text region extraction [J].Journal of Computer-Aided Design and Computer Graphics,2006,18(5):729-734.
[10] TANG J R,ISA N A M,CH′NGE S.A Fuzzy-C-means-clustering approach:quantifying chromatin pattern of non-neoplastic cervical squamous cells [J].Public Library of Science One,2015,10(11):e0142830.
[11] CHEN Q,ZHAO L,LU J,et al.Modified two-dimensional OTSU image segmentation algorithm and fast realisation [J].IET Journals and Magazines,2012,6(4):426-433.
(責任编辑:包震宇)

