利用基因组数据对长爪沙鼠微卫星位点的筛选与优化
冯丹丹,陈 沫,杜小燕,陈振文*
(1.首都医科大学基础医学院,北京 100069; 2.中国医学科学院药物研究所,北京 100050)
长爪沙鼠是源自我国的实验动物资源,因其独特的解剖结构以及对微生物易感性等原因,已广泛地应用于脑神经、微生物、营养、代谢及肿瘤等诸多领域研究,被誉为“多功能”实验动物。但有关长爪沙鼠的遗传学研究相对较少,尤其是遗传标记物的开发和应用远滞后于对其生物学特性和实验应用研究。目前国内外仅有Neumann等[1]通过用克隆方法找到的9个长爪沙鼠微卫星位点并对野生长爪沙鼠和人工饲养群体进行了多态性分析。赵太云,谭元卿等[2-3]利用种间扩增转移法,在536对小鼠微卫星引物中筛选出了长爪沙鼠微卫星位点 130 个。依据这些微卫星位点,浙江省和北京市分别用28个微卫星位点建立了《长爪沙鼠遗传质量控制》地方标准[4-5],中国实验动物学会也据此发布了实验动物行业标准[6]。这有效解决了长爪沙鼠遗传质量无据可依的状态,也为长爪沙鼠从实验用动物提升为实验动物奠定了基础。但在实际应用中发现标准中遗传质量检测所用的微卫星位点存在一定不足,表现为个别位点多态性不够高、个别位点扩增效果不够好、缺乏近交系遗传检测方法和位点等,因此有必要进一步寻找更多和更有效的长爪沙鼠微卫星位点。我们前期工作中对长爪沙鼠全基因组进行了测序分析,结果发现长爪沙鼠基因组中存在大量重复序列,但是这些微卫星能否成功扩增和是否适于遗传检测尚不确定。本研究中我们根据微卫星序列特征的标准选取了重复序列357个[7],并根据这些序列的侧翼序列设计并合成相应的引物进行PCR扩增,最终成功筛选出135个能有效扩增的微卫星位点,通过近交系和封闭群长爪沙鼠基因组对位点进行测试,优化出适用于长爪沙鼠近交系区分和封闭群遗传分析的微卫星位点。这些微卫星位点的获取进一步丰富了长爪沙鼠的遗传信息,为建立长爪沙鼠遗传数据库积累了数据[8],同时为制定长爪沙鼠近交系遗传质量标准和进一步完善和修改目前标准提供了依据,为下一步建立长爪沙鼠遗传质量国家标准奠定了基础。
1 材料和方法
1.1 实验动物
选取普通级长爪沙鼠脑缺血和糖尿病模型近交系F20代动物各6只[9-10],性别、年龄、体重不限,自行培育;普通级封闭群长爪沙鼠12只,性别、年龄、体重不限,由首都医科大学实验动物部提供。本实验通过首都医科大学实验动物和动物实验伦理委员会批准(No.:AEEI-2017-032),在实验过程中按照实验动物使用的3R原则给予动物人道的关怀。
1.2 主要试剂与仪器
试剂:Taq聚合酶、dNTP、10× PCR buffer和50 bp DNA marker(大连宝生物工程有限公司);低熔点琼脂糖(西班牙Biowest Agarose公司)。仪器:PCR仪(美国Bio-Rad公司);PAC-30型电泳仪(美国Bio-Rad公司);凝胶图像仪(美国Bio-Rad公司);漩涡混合器(北京科尔德科贸有限公司)等。
1.3 实验方法
1.3.1 长爪沙鼠基因组DNA的提取
将0.1 g肝组织切成片并置于2 mL组织消化缓冲液中,然后加入20 mg/mL蛋白酶K 10 μL,充分混匀后在55℃下消化12 h。然后用苯酚∶氯仿(比例=1∶1)萃取消化混合物,用乙醇沉淀DNA并溶解在TE缓冲液中。用Nanodrop 2000 C 微量分光光度计检测所得DNA质量,用TE溶液将已知浓度的DNA原液稀释为50 ng/μL,-20℃保存备用,将DNA原液保存于-80℃。
1.3.2 微卫星位点的选择与引物合成
选择位于长爪沙鼠基因组序列中符合微卫星标准的重复序列357个[4],根据每个微卫星位点的侧翼特异序列应用Primer 5软件进行引物的设计,每个位点2~4条引物,所有引物均由北京天一辉远有限公司合成。通过PCR实验对以上位点的PCR反应条件进行优化,确定能成功扩增的引物、最佳退火温度和Mg2+离子浓度。
1.3.3 PCR扩增
PCR反应体系的建立:总反应体系20 μL,其中:10× buffer 2 μL,上下游引物(100 μmol/μL)各0.1 μL,dNTP Mg2+plus(100 μmol/L)1.2 μL,Taq酶(5 U/μL)0.2 μL,基因组DNA(50~100 ng/μL) 1 μL,双蒸水(ddH2O)15.4 μL。
PCR扩增条件:95℃预变性5 min,94℃变性30 s,退火温度30 s,72℃延伸30 s,35个循环,接72℃继续延伸7 min。PCR产物于4℃保存。
1.3.4 PCR产物凝胶电泳
称取琼脂糖2 g,加入到100 mL 0.5× TBE中,用微波炉加热约5~10 min,自然冷却至50℃左右,加入EB混匀,使EB终浓度为0.5 μg/mL,灌入插好梳子的凝胶槽中,约30 min后拔出梳子。将凝胶放入电泳槽中。吸取5 μL样品加1 μL 6× loading buffer混匀,加入凝胶孔中。在凝胶样品中央或两侧加入3~5 μL的50 bp DNA marker。电泳条件为120 V,35 min,将电泳后的琼脂糖凝胶放入凝胶成像系统采集图像并分析结果。
2 结果
2.1 微卫星位点的筛选
针对预选出的357个串联重复序列,根据其侧翼序列合成了357对微卫星位点引物,经PCR扩增,135个微卫星位点获得成功扩增的PCR产物(135/357,37.82%),并筛选出引物的最适退火温度,部分典型位点扩增见图1。 135个微卫星位点的名称、引物序列、目的片段大小、核心序列类型和重复次数及PCR反应条件见表1。在筛选出的135个微卫星位点中,二核苷酸和三核苷酸重复的位点分别为112和23个,微卫星核苷酸重复次数范围为3~28次,目的片段大小范围为51 ~ 271 bp。

注:A:ccmu152位点;B:ccmu183位点。M:50 bp marker;图示数字为PCR反应时所用退火温度(℃)。图1 典型的微卫星位点退火温度梯度优化电泳结果(ccmu152和ccmu183两个位点)Note. A, Locus ccmu152. B, Locus ccmu183. M, 50 bp marker. Numbers represent annealing temperatures (℃).Figure 1 Typical electrophoretic map of annealing temperature gradient optimization for loci ccmu152 and ccmu183
表1135个微卫星位点的名称、引物序列、目的片段大小、核苷酸类型和重复次数及PCR反应条件
Table1Sequencing and polymerase chain reaction (PCR) conditions of 135 microsatellite loci

位点Loci引物序列(上游)(5’→3’)Primer sequences (upstream)引物序列(下游)(5’→3’)Primer sequences (downstream)目的片段大小(bp)Target sequence size核苷酸类型和重复次数Nucleotide type and number of repetitions退火温度(℃)Annealing temperatureMg2+浓度(mmol/L)Mg2+concentrationccmu131TGGTAGATGCTGGTTTTGCTGATAGTACAGCCTCCCCATTGGAT250(TG)23601.5ccmu132GGCGCTTAACCTACTCAGTCCAAACCACAGATCACGTCAGTCC300(AG)28601.5ccmu133CCTGAGCTTGTCCGTGAATCCCTAGTAGGATGCTTTCCCTCTTGT219(AAT)16601.5ccmu134CGGTACAACTGGTACAAAGCGGAGCCGGGGCTTTACACTAT263(AAT)16601.5ccmu135CTCTGAGCTTCAAGGAGCCCAGCCAGTTCTCCACCTCTG119(AAT)16601.5ccmu136CACAGGTTAAAGTGTAGGGGCATAAAAGTTGGAGGCCGAGTG285(AG)28601.5ccmu137TGCTGCTTGTCTTCCACCTTCTGACCTCCACACTCATGCC139(TG)18601.5ccmu138GTCTGGGAGGATGGAGAGGTTGTCGTTCCCGTTGTTCTGT250(TG)18601.5ccmu139GGTGCCCTGGAGTGAGCATCCCGCAGCTGGCGAGTTATC281(TG)18601.5ccmu140TGCTCTGTATCGCTTTGGTTGAACTTAGCATACAAAGCCCAGCCT223(TG)18601.5ccmu141CAGAAAACTCAAGTTCTCAGGTTTATGGTCACCACCATCTCTATGC284(TG)18601.5ccmu142GCTTTTAGTTGTACAGGCTGTGGGCAGTGATAGACCTTGCTCCA207(TG)18601.5ccmu143GCAGACGCCAGTCGCTAAAAAAACACAAGTGTTCTTGAGGAGAAA230(AC)19601.5ccmu144ACTATCCCATGAAATAGAGTCTTGAGTTTCAGCATTCCACCACAG307(AC)19601.5ccmu145ACTTCAAGATTCTTCTGCCAGGTACCAGATGCCCCAGTCTGTA224(AC)19601.5ccmu146TTTTAGCGTGGCCATTCCTACCCACTGGGAGACAGTGAGATTC279(AGG)13601.5ccmu147CCTGGATAAGGGAGGCAATCTCTGAAAACCTGAGTGAGCGTC230(TAA)12601.5ccmu148GACTCATGCAACCAACAACACAGCACACCCTTTGATTCTGTTTC208(TAA)13601.5ccmu149TAGGGTCTGAGGTTAAGTGCCGGACAAAAAGACTAGAACTGGGTC218(AAT)16601.5ccmu150TGTGGACTGAGTGCGTGATCTCTTCGAGGATAGGCAGTTT291(CT)28601.5ccmu151CAAATAAACGGCAATGCCTCAAGAGTGATAATGCAGGTGCCC172(TG)18601.5
(续表1)
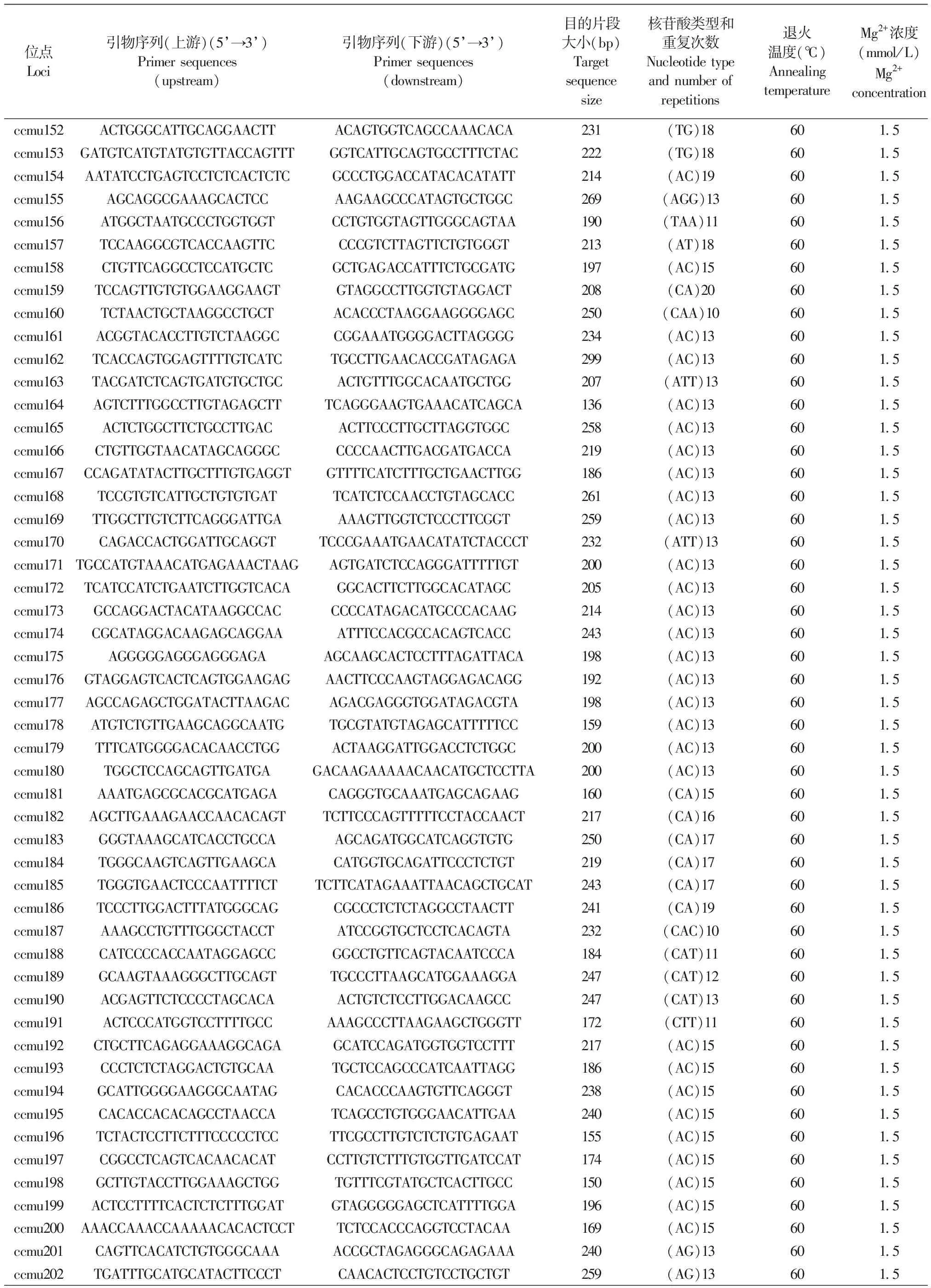
位点Loci引物序列(上游)(5’→3’)Primer sequences (upstream)引物序列(下游)(5’→3’)Primer sequences (downstream)目的片段大小(bp)Target sequence size核苷酸类型和重复次数Nucleotide type and number of repetitions退火温度(℃)Annealing temperatureMg2+浓度(mmol/L)Mg2+concentrationccmu152ACTGGGCATTGCAGGAACTTACAGTGGTCAGCCAAACACA231(TG)18601.5ccmu153GATGTCATGTATGTGTTACCAGTTTGGTCATTGCAGTGCCTTTCTAC222(TG)18601.5ccmu154AATATCCTGAGTCCTCTCACTCTCGCCCTGGACCATACACATATT214(AC)19601.5ccmu155AGCAGGCGAAAGCACTCCAAGAAGCCCATAGTGCTGGC269(AGG)13601.5ccmu156ATGGCTAATGCCCTGGTGGTCCTGTGGTAGTTGGGCAGTAA190(TAA)11601.5ccmu157TCCAAGGCGTCACCAAGTTCCCCGTCTTAGTTCTGTGGGT213(AT)18601.5ccmu158CTGTTCAGGCCTCCATGCTCGCTGAGACCATTTCTGCGATG197(AC)15601.5ccmu159TCCAGTTGTGTGGAAGGAAGTGTAGGCCTTGGTGTAGGACT208(CA)20601.5ccmu160TCTAACTGCTAAGGCCTGCTACACCCTAAGGAAGGGGAGC250(CAA)10601.5ccmu161ACGGTACACCTTGTCTAAGGCCGGAAATGGGGACTTAGGGG234(AC)13601.5ccmu162TCACCAGTGGAGTTTTGTCATCTGCCTTGAACACCGATAGAGA299(AC)13601.5ccmu163TACGATCTCAGTGATGTGCTGCACTGTTTGGCACAATGCTGG207(ATT)13601.5ccmu164AGTCTTTGGCCTTGTAGAGCTTTCAGGGAAGTGAAACATCAGCA136(AC)13601.5ccmu165ACTCTGGCTTCTGCCTTGACACTTCCCTTGCTTAGGTGGC258(AC)13601.5ccmu166CTGTTGGTAACATAGCAGGGCCCCCAACTTGACGATGACCA219(AC)13601.5ccmu167CCAGATATACTTGCTTTGTGAGGTGTTTTCATCTTTGCTGAACTTGG186(AC)13601.5ccmu168TCCGTGTCATTGCTGTGTGATTCATCTCCAACCTGTAGCACC261(AC)13601.5ccmu169TTGGCTTGTCTTCAGGGATTGAAAAGTTGGTCTCCCTTCGGT259(AC)13601.5ccmu170CAGACCACTGGATTGCAGGTTCCCGAAATGAACATATCTACCCT232(ATT)13601.5ccmu171TGCCATGTAAACATGAGAAACTAAGAGTGATCTCCAGGGATTTTTGT200(AC)13601.5ccmu172TCATCCATCTGAATCTTGGTCACAGGCACTTCTTGGCACATAGC205(AC)13601.5ccmu173GCCAGGACTACATAAGGCCACCCCCATAGACATGCCCACAAG214(AC)13601.5ccmu174CGCATAGGACAAGAGCAGGAAATTTCCACGCCACAGTCACC243(AC)13601.5ccmu175AGGGGGAGGGAGGGAGAAGCAAGCACTCCTTTAGATTACA198(AC)13601.5ccmu176GTAGGAGTCACTCAGTGGAAGAGAACTTCCCAAGTAGGAGACAGG192(AC)13601.5ccmu177AGCCAGAGCTGGATACTTAAGACAGACGAGGGTGGATAGACGTA198(AC)13601.5ccmu178ATGTCTGTTGAAGCAGGCAATGTGCGTATGTAGAGCATTTTTCC159(AC)13601.5ccmu179TTTCATGGGGACACAACCTGGACTAAGGATTGGACCTCTGGC200(AC)13601.5ccmu180TGGCTCCAGCAGTTGATGAGACAAGAAAAACAACATGCTCCTTA200(AC)13601.5ccmu181AAATGAGCGCACGCATGAGACAGGGTGCAAATGAGCAGAAG160(CA)15601.5ccmu182AGCTTGAAAGAACCAACACAGTTCTTCCCAGTTTTTCCTACCAACT217(CA)16601.5ccmu183GGGTAAAGCATCACCTGCCAAGCAGATGGCATCAGGTGTG250(CA)17601.5ccmu184TGGGCAAGTCAGTTGAAGCACATGGTGCAGATTCCCTCTGT219(CA)17601.5ccmu185TGGGTGAACTCCCAATTTTCTTCTTCATAGAAATTAACAGCTGCAT243(CA)17601.5ccmu186TCCCTTGGACTTTATGGGCAGCGCCCTCTCTAGGCCTAACTT241(CA)19601.5ccmu187AAAGCCTGTTTGGGCTACCTATCCGGTGCTCCTCACAGTA232(CAC)10601.5ccmu188CATCCCCACCAATAGGAGCCGGCCTGTTCAGTACAATCCCA184(CAT)11601.5ccmu189GCAAGTAAAGGGCTTGCAGTTGCCCTTAAGCATGGAAAGGA247(CAT)12601.5ccmu190ACGAGTTCTCCCCTAGCACAACTGTCTCCTTGGACAAGCC247(CAT)13601.5ccmu191ACTCCCATGGTCCTTTTGCCAAAGCCCTTAAGAAGCTGGGTT172(CTT)11601.5ccmu192CTGCTTCAGAGGAAAGGCAGAGCATCCAGATGGTGGTCCTTT217(AC)15601.5ccmu193CCCTCTCTAGGACTGTGCAATGCTCCAGCCCATCAATTAGG186(AC)15601.5ccmu194GCATTGGGGAAGGGCAATAGCACACCCAAGTGTTCAGGGT238(AC)15601.5ccmu195CACACCACACAGCCTAACCATCAGCCTGTGGGAACATTGAA240(AC)15601.5ccmu196TCTACTCCTTCTTTCCCCCTCCTTCGCCTTGTCTCTGTGAGAAT155(AC)15601.5ccmu197CGGCCTCAGTCACAACACATCCTTGTCTTTGTGGTTGATCCAT174(AC)15601.5ccmu198GCTTGTACCTTGGAAAGCTGGTGTTTCGTATGCTCACTTGCC150(AC)15601.5ccmu199ACTCCTTTTCACTCTCTTTGGATGTAGGGGGAGCTCATTTTGGA196(AC)15601.5ccmu200AAACCAAACCAAAAACACACTCCTTCTCCACCCAGGTCCTACAA169(AC)15601.5ccmu201CAGTTCACATCTGTGGGCAAAACCGCTAGAGGGCAGAGAAA240(AG)13601.5ccmu202TGATTTGCATGCATACTTCCCTCAACACTCCTGTCCTGCTGT259(AG)13601.5
(续表1)

位点Loci引物序列(上游)(5’→3’)Primer sequences (upstream)引物序列(下游)(5’→3’)Primer sequences (downstream)目的片段大小(bp)Target sequence size核苷酸类型和重复次数Nucleotide type and number of repetitions退火温度(℃)Annealing temperatureMg2+浓度(mmol/L)Mg2+concentrationccmu203AGTGGCTGGAAACAACCCAACCTCTCACTGAACCAACGTCA318(AG)13601.5ccmu204ACTCCTACATTGAAGCCGTCTCGGTCACTATGGCCCCAGAAG222(AG)13601.5ccmu205GGATTGTGTCTGCAGGTCAGTATGATGCACCAGCCATAGGTT241(AG)13601.5ccmu206TGAGTGACTTGAGTGACAGTACCTGTGACTGGATGAGGCTTGC224(AG)13601.5ccmu207TCAGGAAAACCAAAACAAATTGGAACTTTCCTTTCCCCACCGGAT302(AG)13601.5ccmu208GAGCTGTGTGAGGACCAGTTTGTCTGAGTGCTTTGACAACGTAT172(AC)5601.5ccmu209CAGCCCCAAGCCAAAGGTTTTTGCCAACTGGGTGGGATAAGA200(AC)5601.5ccmu210GACAGGCTCTAGCAGCAGAAATACAGTCACAGGAGAGTGCT186(AG)13601.5ccmu211CTTCTGATCATTCCCTGCGTGTCTCTGGTGCCTGCCTCTAA152(AC)13601.5ccmu212CTGTTCACCATTCACTTAACACCAAGGCTGATGAAAGTTGCCCT156(AC)13601.5ccmu213TACAGAACACAGATATTGGGCACATACACCCACCTCAAATGCG199(AC)5601.5ccmu214TCCCTATGCCTCTTCCCTAGTCTAGTAAGAGGGGCAGGGGATG170(AC)5601.5ccmu215GAGGGACCATAGGAAGCAGAAGACTCCCACATGTCACCCCA52(AC)5601.5ccmu216TTCACAGGTACAGTCATGGGCCAACAAGTGGTCTTGTCAGAAA63(AC)5601.5ccmu217TCTTGCTCTCTATCCCTCTCCATCCAGACTCAGAAAGGCAACC126(AC)5601.5ccmu218AGTAGCCATTTGCATGAGGACATTCCCTAGTGATCTATTGGGTTGG137(AC)5601.5ccmu219TGACTCTTGTGAGCTATCCTCTGTGCTACCTTCTGTCCTCCTCA180(AC)5601.5ccmu220CACGCAGGTGCTTATCTCCACAGGGTGAACAGAACGAGGT136(AC)5601.5ccmu221ACCCTCATTTCATACCCTGCCATTGGCCATTGGGCATAGCA271(CA)20601.5ccmu222AAAACTCCTCGGGAAGGTGGACGAATTTGGGGCGCATAAG289(CA)20601.5ccmu223ATGCCTGTGTTGTATCAGGCTCCAGGTTTTCCCAGGTTCAC208(CA)20601.5ccmu224TGCTGACCCCTACTCACAGTGCCTTGGTTCTTAGAAAGCCC246(CA)20601.5ccmu225TATGGTTCTTGCAGAGGGCGTGCATGGAAATTGATCCGCT283(CA)20601.5ccmu226CCCGGGGTCTGTGTACAATCAGCTGTCGTCTTGACTGTGTT240(CA)20601.5ccmu227ATGTTAACCACCACGGGTCCCGTGTCCAGTAGGAGGTTGT249(CA)20601.5ccmu228TAAACAAGAGCTGGGTGCCTCCCCTCATGGGCAGAACGAA300(CA)20601.5ccmu229TTGCGATGCACATGAAGTGATTTTATCCTCCGTGCTCCCG232(CAC)12601.5ccmu230ACATGTTTCCAATTGGGCTGCACTGCTTTCAGAATGGATTTTTGT239(CA)20601.5ccmu231GTCTGGGCTTGTCTAGGTGCTCCCAGGACTGTGGCCTATT242(TC)22601.5ccmu232TGACAACAGCCAGCACAGAGCCAGATTGGTTCCAAACAGCAG259(TC)22601.5ccmu233AGTCACGCGGGAGTAGAACTGCTCTCCTTGTGGAGACCCT191(TC)22601.5ccmu234AATGGGGGCCATCTTCTCCACGCTGTCTTGTGTGTCATCC177(TC)22601.5ccmu235CCTGGTGTACTCCATTTGCTCTGTGCGTTGTCCAGTTGTCTT243(TC)22601.5ccmu236GAAGAGGCTACACGATGGGGAGCCTTGGTGTTTCAGTTTCA231(TC)22601.5ccmu237CTCCTGTGCAGGCACACTTAGATCCCAGCACCCACAGAAG307(TC)22601.5ccmu238TGTGTCAGCCATAAGTCCCCAACTGGGGCACTGTCAACAT279(TC)22601.5ccmu239ACACCATGGATAAAGACCCTGTAAACTTTCTTCAGCTTCCACT114(TC)22601.5ccmu240TGTGGGATACTGGTGCACTGCTGCTTTCCAGTTGTGGTGC234(TC)22601.5ccmu241CTGTTAGCACGGGACTGACTAGACATTATGGCTTGTTCCCCA226(TC)22601.5ccmu242GATGCCTGGTCCCCAATCTTCTGTTCGAGAAATGGCCACC287(TC)22601.5ccmu243GCCTCGTTGGAGAAAGTGTGAAGACACCATGACCACAGCAA164(TC)22601.5ccmu244GGGCCAGCTGATGCTTAGATTTGGTGCTAACATTCCTGGCA144(TC)22601.5ccmu245CCACTGCCACTGTTTACCCAGGTCTGTCTGTTAGATCTGCCAAT277(TC)22601.5ccmu246GGACTTGTGGCCTTGTAGGAGGGTCATCGTGGAGAGCATGG272(TC)22601.5ccmu247AGGTATGGCCTCGTTAGAGGAGTGTCAGAGGGTTAGAGTCCAT232(TC)22601.5ccmu248TACCTGGAGGGGCAGCTAAAGATCATGCCTAAGGGGGAGG343(TC)22601.5ccmu249GAGTTGGGAAAACTCAGCGTTCCCAAGTTTTCTTTTTCATCACTG245(TC)22601.5ccmu250TCCCTAGACCTGCAAAACCACACCCCCATCCCAAGATTCCTA214(TC)22601.5ccmu251TTGTGTCTAATTCCCTCCTCCCGTGCTTCTCTACCGGGTACT152(CA)22601.5ccmu252CCTAGACAGAAACAAGGGCCACCCCACAAAGCACTGATTAGC224(CA)22601.5ccmu253ACTGGGGACCATGGTAAGTCTATTACCTTTGTGGAGTACTTGGT277(ATT)3601.5
(续表1)

位点Loci引物序列(上游)(5’→3’)Primer sequences (upstream)引物序列(下游)(5’→3’)Primer sequences (downstream)目的片段大小(bp)Target sequence size核苷酸类型和重复次数Nucleotide type and number of repetitions退火温度(℃)Annealing temperatureMg2+浓度(mmol/L)Mg2+concentrationccmu254TCTGAGTGAAACGTAAGTACACCTAGTTTCTGCTCTCTGACATGGA210(ATT)3601.5ccmu255ACCAACTATTCCCACTGAGACAGCTTACCACAGACATTCCCCA240(ATT)3601.5ccmu261TTTGGGCAGTTCACCACGATGTCCCGCCTGTTTGAAGGTA316(GA)11601.5ccmu262AGTGTGTCCGTGTCTTCCTTCAGGAGAAATCCAATGCTGTGC146(GA)9601.5ccmu263AGGCTACACCATACCTGACCTCTGAGTCTGGAGTCTGCTGT249(GA)18601.5ccmu264AGGCTAGCTTCTGGATGCACGTAGGTCTCCAGATCAAGGGC249(GCT)10601.5ccmu265TGGGTAGGATGGCAGGGATATATGACTCCTGGGCAGTTGTTT161(GCT)10601.5ccmu266ACCACCACCTCCAAAGAACTGACAATGCCACCTCCCTGAAT151(AC)15601.5ccmu267ACTTGAGATTCTTCAGCAGCAAATCTCAGTCAGTGACACTCAAA200(AC)15601.5ccmu268TCCCAAGTGCGCAGTCATAGACCCTGTGTGTTTCTCCAAAGT225(AC)15601.5ccmu269GCTCCAAACACAGGGCGATAAGGGTCCCTGGTATCTGAGT247(AC)15601.5ccmu270CCCATACATTCCTCATTGCACTGCCACTGAGGCTTCATTCAT221(AC)15601.5
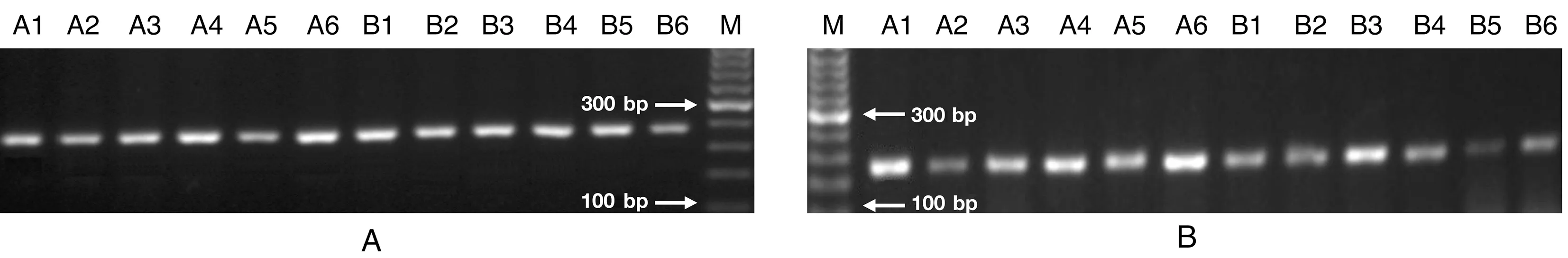
注:A:引物ccmu138的PCR产物;B:引物ccmu218的PCR产物。M:50 bp marker;A1 ~ A6:长爪沙鼠脑缺血模型;B1 ~ B6:长爪沙鼠糖尿病模型。图2 两个微卫星位点 ccmu138和ccmu218的PCR产物琼脂糖凝胶电泳图Note. A, PCR product of ccmu138 primers. B, PCR product of ccmu218 primers. M, 50 bp marker. A1-A6, Cerebral ischemia Mongolian gerbils. B1-B6, Diabetic Mongolian gerbils.Figure 2 Agarose gel electrophoresis of PCR products for loci ccmu138 and ccmu218
2.2 适于长爪沙鼠近交系区分的微卫星位点优化
选取本课题组自行培育的长爪沙鼠脑缺血和糖尿病模型近交系F20代动物各6只,通过135个微卫星位点扩增结果分析,结果发现各位点在同一品系内均呈单态性,而有10个微卫星位点在2种模型品系间存在差异,包括ccmu138、ccmu146、ccmu156、ccmu161、ccmu175、ccmu189、ccmu218、 ccmu235、ccmu250和ccmu261。这些位点可将长爪沙鼠2种模型近交系品系区分开来,图2为2个位点对不同近交系的扩增结果。
2.3 微卫星位点多态性优化
应用135个筛选出的长爪沙鼠微卫星位点引物对12只封闭群长爪沙鼠样本进行扩增,结果有23个微卫星位点出现多态性,包括ccmu148、ccmu151、ccmu163、ccmu164、ccmu165、ccmu176、ccmu183、ccmu187、ccmu190、ccmu192、ccmu198、ccmu201、ccmu214、ccmu231、ccmu232、ccmu233、ccmu241、ccmu244、ccmu248、ccmu249、ccmu264、ccmu267和ccmu269微卫星位点,图3显示ccmu148和ccmu176位点对12只封闭群动物的扩增结果,两个位点在封闭群多个样品中显示多态性。
3 讨论
微卫星DNA也称为简单重复的DNA片段,其重复单位一般为1 ~ 6 bp,重复数为10 ~ 20次左右,在哺乳动物基因组中随机分布。由于其具有在基因组中分布广泛、多态性高、易于检测、共显性遗传等优点,成为第二代遗传标记物。微卫星在评价遗传多样性、构建遗传连锁图谱、绘制系统发生树、连锁分析、疾病诊断和亲子鉴定等方面显示出巨大优势,并在动植物的遗传研究中得到了广泛应用[11-12],并成为一种重要的、成熟的遗传学检测工具广泛应用于各类实验动物遗传质量检测研究中[13-14]。

注:A:ccmu148引物的PCR产物;B:ccmu176引物的PCR产物。M:50 bp marker;1 ~ 12:封闭群长爪沙鼠。图3 两个微卫星位点ccmu148和ccmu176的PCR产物琼脂糖凝胶电泳图Note. A, PCR product of ccmu148 primers. B, PCR product of ccmu176 primers. M, 50 bp marker. 1-12, outbred group of Mongolian gerbils.Figure 3 Agarose gel electrophoresis of PCR products for the loci ccmu148 and ccmu176
微卫星DNA标记在大、小鼠遗传多态性的研究已经取得较好成果,但长爪沙鼠开发出的微卫星位点数量较少。除Neumann等[1]通过用克隆方法找到的9个长爪沙鼠微卫星位点外,本实验室利用种间扩增转移法,在536对小鼠微卫星引物中筛选出了长爪沙鼠微卫星位点 130 个,并已登录到GenBank中[2-3]。我们利用国内外筛选的139个微卫星位点通过封闭群长爪沙鼠进行多态性分析,优化出了28个具有多态性的位点,以此为依据建立了封闭群长爪沙鼠遗传质量检测方法,并被浙江省和北京市长爪沙鼠遗传质量控制地方标准和中国实验动物行业标准所采纳[4-6]。在建立长爪沙鼠遗传检测方法时,由于可供选择的位点数量少,为了能真实地反映群体遗传概貌,不得采用更多的位点组合,使一些仅有2个等位基因的位点也纳入了其中。这大大增加了工作量,也使得检测周期延长和耗费过多。同时,以往尚未见有对近交系长爪沙鼠遗传检测方法的研究报道,因此有必要筛选更多的长爪沙鼠微卫星位点为下一步补充和完善目前的长爪沙鼠遗传检测方法及建立近交系长爪沙鼠遗传检测方法提供候选位点。
本研究中我们通过分析长爪沙鼠全基因组的测序结果,选择符合微卫星标准的位点357个,并根据微卫星位点的侧翼序列合成相应的引物,能够针对性地筛选适用于长爪沙鼠的微卫星位点,优于实验室前期利用种间扩增转移的方法。我们首先用长爪沙鼠混合基因组对位点进行PCR扩增,在357个微卫星位点中成功扩增出135个条带清晰且无杂带的微卫星位点,包括112个二核苷酸位点和23个三核苷酸位点。其余222个微卫星位点没有扩增出理想条带,扩增失败的原因可能为以下方面:所设计的引物特异性不佳,形成引物二聚体从而产生非特异性扩增条带;模板DNA的质量也能影响个别位点的扩增,尤其是对相对较长的微卫星片段的影响更加明显。本实验室前期研究发现长爪沙鼠近交系是脑缺血研究良好的模型动物,也是理想的II型糖尿病模型,两种模型的建立不但丰富了实验动物资源,也为研究人类疾病的机制与药物开发提供良好的模型材料。所以本研究利用筛选出的135个微卫星位点分析已知的长爪沙鼠脑缺血和糖尿病模型近交系动物进行验证,结果多数位点为单态性,尤其在同一品系内均为一致的电泳图带。而2个品系间有10个微卫星位点存在差异,能区分长爪沙鼠脑缺血和糖尿病模型近交系群体,对于模型动物的推广应用具有重要的意义。由于这2个近交系均来源于20代前的共同父母,是在F7代时分离培育成2个品系,因此遗传背景很大程度上是一致的,以往的遗传检测方法很难将其区分,说明这10个位点可用于近交系的遗传监测。遗传多样性是生物多样性的核心和重要组成部分,对遗传多样性的研究有利于了解物种或种群的进化历史和分类地位,为资源的保存与利用提供理论依据。封闭群动物群体中存在遗传多样性,选择用于群体遗传分析的微卫星位点更加注重其多态性,即等位基因数量越多,其所蕴含的信息量越大,遗传分析的结果更加真实可靠。为了建立有效的长爪沙鼠群体遗传检测方法,开展长爪沙鼠群体遗传多样性分析,本研究随机选取了长爪沙鼠封闭群动物12只,应用筛选出的135个微卫星位点进行扩增,结果有23个多态性位点,一共发现了63个等位基因,其中等位基因数最多的达6个。这为补充和完善封闭群长爪沙鼠遗传质量检测方法提供了新的可供选择的微卫星位点。

